Особенности использования флуоресцентных репортерных белков для изучения развития корневых систем на примере тыквенных (Cucurbitaceae)
Автор: Ильина Е.Л., Кирюшкин А.С., Демченко К.Н.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Методы исследований
Статья в выпуске: 5 т.55, 2020 года.
Бесплатный доступ
Современные исследования тонких процессов развития растений невозможно представить без использования широкого спектра флуоресцентных белков (R. Day и M. Davidson, 2009; D. Chudakov с соавт., 2010), применение которых, однако, ограничено из-за несовершенства приемов их визуализации в растительных тканях. Растения представляют собой сложные объекты для микроскопических исследований: даже применение наиболее совершенных методов имеет значительное ограничение по глубине проникновения света по причине его рассеивания и поглощения клеточными стенками. Соответственно, для изучения распределения репортерных флуоресцентных белков в крупных органах, типичных для большинства растений, необходима фиксация растительного материала и приготовление толстых гистологических срезов с помощью микротома с вибрирующим лезвием. Химические вещества, традиционно используемые для фиксации, обезвоживания и заключения растительных образцов в заливочную среду приводят к изменению структуры флуоресцентных белков и, как следствие, к потере флуоресценции. Поэтому сохраняется актуальность оптимизации протоколов фиксации тканей растений, приготовления срезов, а также изучения распределения флуоресцентных белков при помощи лазерной сканирующей конфокальной микроскопии. В представленной работе нами впервые предложен комплексный подход к фиксации тканей трансгенных растений и приготовлению срезов при изучении паттернов клеточного ответа на ауксин и экспрессии транскрипционных факторов с применением лазерной сканирующей конфокальной микроскопии. Нашей целью было обобщение современных подходов в использовании эффективной универсальной методики визуализации тканевого и клеточного паттернов распределения флуоресцентных репортерных белков на срезах крупных органов немодельных растений. Первый шаг для использования флуоресцентных белков в растениях - создание генетических конструкций, несущих гены интереса и гены репортерных флуоресцентных белков. При этом необходимо наличие оптимальной методики трансформации для выбранного вида растений. В работе описано применение технологии клонирования Gateway® для получения векторов для трансформации растений, отвечающих современным экспериментальным задачам. Для изучения локализации ауксина in vivo мы получили серию векторов с генами, кодирующими флуоресцентные белки (eGFP, tdTomato, mRuby3), под контролем ауксин-чувствительного промотора DR5 (E. Ilina с соавт., 2012). Продемонстрировано преимущество использования ярких флуоресцентных белков с ядерной локализацией (mNeonGreen-H2B, tdTomato-H2B, mRuby3-H2B), а также возможность их применения для дополнительной визуализации ядер клеток в сочетании с высокоспецифичным окрашиванием клеточных стенок красителем SCRI Renaissance2200. Представлена методика конструирования векторов для изучения тканевого паттерна экспрессии генов регуляторов развития на примере ранее идентифицированных нами генов транскрипционных факторов GATA24 (А. Kiryushkin с соавт., 2019) и LBD16 у представителей семейства Cucurbitaceae (Тыквенные). На примере кассеты pAtUBQ10::DsRED1 (E. Limpens с соавт., 2004), несущей ген красного флуоресцентного белка под управлением конститутивного промотора, продемонстрирована высокая эффективность использования флуоресцентных белков для скрининга трансгенных органов растений. Представлена новая методика фиксации и просветления растительных тканей, содержащих репортерные флуоресцентные белки, и приготовления срезов на примере трансгенных корней у тыквенных. Показано преимущество использования расплавленной агарозы для ориентации растительных объектов при пробоподготовке по сравнению с заливочными средами. Также показано значение модифицированной нами заключающей просветляющей среды ClearSee (D. Kurihara с соавт., 2015) для повышения фотостабильности флуоресцентного белка на срезах. Таким образом, продемонстрированы преимущества, которые предоставляет применение комплекса современных методических подходов пробоподготовки и лазерной сканирующей конфокальной микроскопии для изучения биологии развития крупных немодельных растений.
Агробактериальная трансформация, флуоресцентные белки, конфокальная микроскопия, развитие растений, транскрипционные факторы
Короткий адрес: https://sciup.org/142229429
IDR: 142229429 | УДК: 582.98:581.43:57.086:577.21 | DOI: 10.15389/agrobiology.2020.5.1040rus
Текст научной статьи Особенности использования флуоресцентных репортерных белков для изучения развития корневых систем на примере тыквенных (Cucurbitaceae)
Исследования молекулярных и физиологических механизмов развития растений сегодня невозможно представить без использования широкого спектра репортерных флуоресцентных белков (5). Области их применения многообразны. Это изучение тканевого паттерна активности экспрессии генов; визуализация распределения, транспорта и взаимодействия белков; мечение органелл, клеток и тканей; поиск новых лекарств; современный селекционный процесс и т.д. (6-8). Однако в тканях многих растений применение репортерных флуоресцентных белков имеет существенные ограничения.
Arabidopsis признан удобным объектом для микроскопических исследований. Большинство работ с применением флуоресцентных белков проводятся на этом модельном растении. Arabidopsis имеет тонкие корни толщиной около 100 мкм, что делает возможным изучение распределения флуоресцентных белков в тканях корня при помощи конфокальной микроскопии прижизненно без приготовления срезов. Листья у Arabidopsis также тонкие, но оптически непрозрачные из-за хлорофилла, что позволяет изучать распределение флуоресцентных белков только в поверхностных слоях. Для Arabidopsis разработана методика просветления тканей ClearSee, которая сохраняет флуоресценцию некоторых репортерных белков и позволяет изучать их распределение в оптически непрозрачных органах (9), а также совмещать визуализацию флуоресцентных белков и окрашенных неспецифическими красителями Direct Red 23 или Direct Yellow 96 клеточных стенок (10), что дает возможность соотнести локализацию экспрессии гена с конкретной клеткой или тканью. Известна также методика иммуногистохимических исследований без приготовления срезов органов ePro-ClearSee, которая была адаптирована для исследований в области эпигенетики и позволяет изучать модификации гистонов и ДНК (11). Кроме того, модифицирован способ окрашивания по Шиффу с последующим просветлением органов, позволяющий изучать распределение индиго (продукта GUS-окрашивания) по всей толщине любого органа Arabidopsis (12). Однако такое окрашивание несовместимо с применением флуоресцентных белков.
Абсолютное большинство растений, в том числе важные сельскохозяйственные культуры, обладают более сложным анатомическим строением по сравнению с Arabidopsis , их органы состоят из большего числа слоев клеток в тканях и, соответственно, обладают большими размерами. Максимальная глубина проникновения лазера конфокального микроскопа в ткани растений относительно невелика по причине рассеивания и поглощения света клеточными стенками и достигает максимального значения 100 мкм.
Мультифотонная лазерная микроскопия достаточно сложна в применении, хотя и позволяет осуществить более глубокое проникновение в толщу тканей растений при использовании длинноволнового (900-1000 нм) фемтосекундного лазера (13, 14). Новейшая технология плоскостного освещения (light-sheet fluorescence microscopy), которая базируется на широкопольной флуоресцентной микроскопии, позволяет проникать в образец на глубину до 500 мкм (15-17). Однако это применимо только к прозрачным или просветленным объектам животного и растительного происхождения. Даже для Arabidopsis эта технология, успешно совмещенная с деконволюцией и реконструкцией изображения (Fiji плагин Multiview-Reconstruction, , имеет значительные ограничения по глубине проникновения света и разрешающей способности, как и традиционные методы микроскопии (18).
Таким образом, несмотря на несомненный прогресс последних лет в области микроскопических технологий, для изучения распределения репортерных флуоресцентных белков или слияния их генов с генами изучаемых белков в толстых органах растений необходима оптимизация протокола фиксации растительного материала и приготовления толстых гистологических срезов. Химические вещества, традиционно используемые для фиксации, обезвоживания и заключения образцов растительных тканей в заливочную среду приводят к изменению структуры флуоресцентных белков и, как следствие, к потере флуоресценции. Для работы с флуоресцентными белками растений некоторые авторы применяют криомикротомию, с помощью которой можно получить толстые (25-50 мкм) срезы растительных тканей, где сохраняется флуоресценция таких белков (19). Однако подобные подходы не получили широкого распространения.
При работе с растительными объектами целесообразно использовать наиболее яркие и фотостабильные флуоресцентные белки, например mNeonGreen, mRuby3 или mScarlet (20, 21), что дает возможность проводить сканирование образцов на большую глубину. Также необходимо учитывать возможность автофлуоресценции клеток в различных частях спектра. Следовательно, векторы для трансформации растений должны соответствовать задачам эксперимента и объекту. Существующие наборы векторов для маркирования органелл растительной клетки (22) или же коммерческие генетические конструкции не всегда подходят наилучшим образом к конкретной научной задаче, поэтому существует необходимость применения гибкой и производительной технологии, позволяющей сконструировать оптимальный для такой задачи вектор. Технологии клонирования Gateway® (23) и Golden Gate (24) эффективны, обеспечивают направленное клонирование и позволяют осуществить сборку практически любых сочетаний фрагментов ДНК (например, промотора, кодирующей части и терминатора) в векторе, пригодном для трансформации растений.
Настоящая работа посвящена методическим приемам изучения молекулярно-генетических механизмов развития у крупных немодельных растений. Нами впервые предложен комплексный подход к фиксации тканей трансгенных растений и приготовлению срезов при изучении паттернов клеточного ответа на ауксин и экспрессии транскрипционных факторов с применением лазерной сканирующей конфокальной микроскопии. Описано применение технологии клонирования Gateway® для получения новых генетических конструкций (векторов) для трансформации растений, отвечающих современным экспериментальным задачам.
Нашей целью было обобщение современных подходов в использовании эффективной универсальной методики визуализации тканевого и клеточного паттернов распределения флуоресцентных репортерных белков на срезах крупных органов немодельных растений на примере тыквенных.
Описание методики . Создание молекулярно -генетических конструкций с использованием технологии клонирования Gateway®. Общий принцип технологии клонирования Gateway® . Для использования репортерных флуоресцентных белков в растениях необходимы генетические конструкции (векторы), в составе которых гены, кодирующие эти белки, будут доставлены в клетки. При конструировании векторов, пригодных для трансформации растений, была использована технология клонирования Gateway® («Thermo Fisher Scientific», США) (25). Для осуществления LR plus клоназной реакции в одной пробирке соединяли три различных вектора ввода (entry vector), содержащих соответственно промотор изучаемого гена, кодирующую часть и терминатор транскрипции, и вектор назначения (destination vector), после чего вносили ферментативную смесь LR ClonaseTM II Plus («Thermo Fisher Scientific», США; далее LR plus кло-наза), которая осуществляет реакцию рекомбинации (рис., А).
Также для конструирования векторов для трансформации растений использовали ферментативную смесь LR ClonaseTM II, осуществляющую перенос одного фрагмента ДНК из вектора ввода в вектор назначения посредством рекомбинации между attL и attR сайтами. В основе технологии лежит система рекомбинации бактериофага лямбда (26). Векторы ввода и назначения содержат разноименные гомологичные участки ДНК бактериофага лямбда attL и attR, способные рекомбинировать друг с другом под действием LR plus клоназы, которая представляет собой смесь ферментов интегразы и эксцизионазы бактериофага лямбда и белок Integration Host Factor, кодируемый бактерией Escherichia coli (26). Разноименные attL-участки находятся по краям фрагментов ДНК, клонированных в вектор ввода, что позволяет осуществлять их направленный перенос в определенном порядке в вектор назначения. Для получения векторов ввода использовали ферментативную смесь BP Clonase™, которая осуществляет рекомбинацию между attB сайтами, фланкирующими целевой фрагмент ДНК, и attP сайтами вектора-донора (donor vector). Клонирование целевого фрагмента ДНК в вектор ввода проводили также традиционно, используя последовательные рестрикции и лигирования целевого фрагмента ДНК и вектора ввода.
В представляемой работе были использованы следующие векторы ввода: 369_pENTRattL4attR1_BSAI («Thermo Fisher Scientific», США) для клонирования промоторной части, pUC18-entry 8 (27) для клонирования репортерной части и донорный вектор pDONR P2-P3 («Thermo Fisher Scientific», США) для клонирования терминатора. В качестве вектора назначения для LR клоназной реакции использовали вектоp pKGW-GG-RR (28), содержащий прилегающий к att сайтам слитый репортерный ген eGFP-GUS (25), кодирующий зеленый флуоресцентный белок eGFP и β -глюкуронидазу (GUS), а также кассету pAtUBQ10::DsRED1 (29) для скрининга трансгенных корней по флуоресценции белка DsRED1. В качестве вектора назначения для LR plus клоназной реакции использовали вектоp 242 pKGW-RR-MGW, содержащий ту же кассету pAtUBQ10::DsRED1 (см. рис., А), и вектор 236 pKGW-RR-MGW, не содержащий такой кассеты (для отбора трансгенного материала).
Все виды клоназных реакций (LR plus-, LR- и BP) проводили согласно рекомендациям производителя («Thermo Fisher Scientific», США). Наличие и правильность сборки кассеты в векторе назначения проверяли методом ПЦР и секвенированием. Вектор назначения трансформировали в клетки штамма R1000 Rhizobium rhizogenes и затем использовали для трансформации проростков кабачка и огурца согласно разработанной ранее методике (3), приводящей к созданию композитных растений, состоящих из побега дикого типа и трансгенной корневой системы.
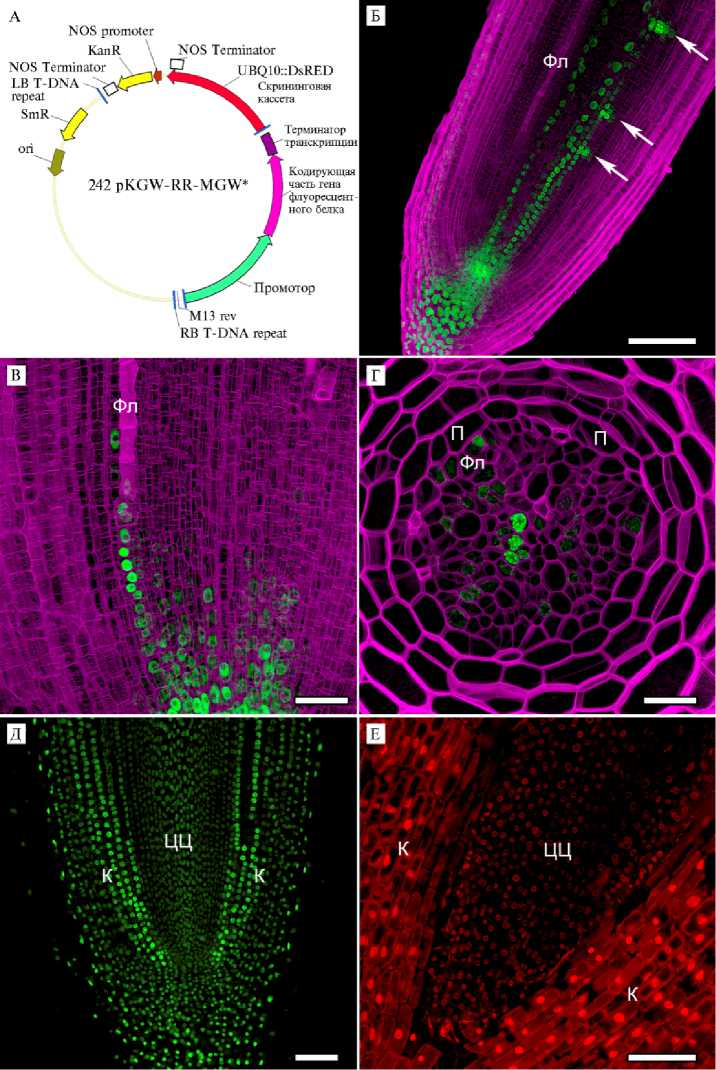
Примеры использования репортерных флуоресцентных белков при изучении паттернов клеточного ответа на ауксин и экспрессии транскрипционных факторов с применением конфокальной лазерной сканирующей микроскопии (микроскоп LSM 780, «Zeiss», Германия). Продольные срезы кончиков фиксированных корней, толщина среза 65 мкм. Проекции максимальной интенсивности оптических срезов.
А. Общая схема вектора 242 pKGW-RR-MGW с клонированным в него промотором, кодирующей частью гена флуоресцентного белка и терминатором транскрипции. Обозначена кассета pUBQ10::DsRED для скрининга трансгенного материала.
Б. Визуализация максимума клеточного ответа на ауксин в трансгенных корнях у проростков огурца, несущих вставку DR5::mRuby3-H2B . Зеленый канал — клеточный ответ на ауксин (флуоресценция mRuby3 с ядерной локализацией), пурпурный канал — клеточные стенки, окрашенные SCRI Renaissance2200. Белые стрелки указывают на примордии бокового корня. Фл — протофлоэма. Масштабная линейка 100 мкм.
-
В, Г. Визуализация паттерна экспрессии гена СpLBD16b в кончике корня у проростков кабачка, несущего вставку СpLBD16b::mNeonGreen-H2B , на продольном (В) и поперечном срезе (Г). Зеленый канал — распределение СpLBD16b (флуоресценция mNeonGreen с ядерной локализацией), пурпурный канал — клеточные стенки, окрашенные SCRI Renaissance2200. Фл — протофлоэма, П — перицикл. Масштабная линейка 50 мкм.
Д. Визуализация ядер клеток в трансгенных корнях у проростков кабачка, несущих вставку UBQ10::H2B-Venus . Зеленый канал, ядра клеток (флуоресценция Venus). К — кора, ЦЦ — центральный цилиндр. Масштабная линейка 100 мкм.
-
Е. Визуализация ядер клеток в трансгенных корнях у проростков кабачка, несущих вставку pAct::tdTomato-H2B . Красный канал, ядра клеток (флуоресценция tdTomato). К — кора, ЦЦ — центральный цилиндр. Масштабная линейка 100 мкм.
Конструирование векторов для изучения локализации in vivo клеточного ответа на ауксин . Ауксин известен как один из наиболее важных факторов, влияющих на развитие корневой системы у высших растений (30). Для изучения роли ауксина в инициации и развитии бокового корня у тыквенных была сконструирована серия векторов, содержащих синтетический ауксин-чувствительный промотор DR5 (31) и репортерную часть для детекции клеточного ответа на ауксин. Вектор pKGW-RR-MGW- DR5::eGFP-GUS был получен посредством LR клоназной реакции между вектором ввода pUC18-entry8- DR5 , содержащим промотор DR5 , и вектором назначения pKGW243-GG-RR (3). Распределение клеточного ответа на ауксин изучали на целых трансгенных корнях кабачка ( DR5::eGFP-GUS ), окрашенных на активность β -глюкуронидазы (GUS-окрашивание), а также на продольных и поперечных срезах трансгенных корней у кабачка. Недостаток GUS-окрашивания заключается в миграции индиго (продукт β -глюкуронидазной реакции), что может приводить к искажению данных по тканевой локализации ответа на фитогормон, поэтому для более детального изучения роли ауксина в инициации бокового корня кабачка целесообразно использовать флуоресцентные белки с ядерной локализацией.
Также были синтезированы генетические конструкции 242 pKGW-RR-MGW- DR5::mRuby3-H2B и 242 pKGW-RR-MGW- DR5::tdTomato-H2B , обеспечивающие локализацию репортерного белка в ядре. Распределение клеточного ответа на ауксин анализировали на продольных и поперечных срезах (60 мкм) кончиков трансгенных корней кабачка и огурца, несущих конструкции DR5::NLS-eGFP-GUS , DR5::mRuby3-H2B и DR5::tdTomato-H2B . Применение репортерных белков tdTomato и mRuby3, слитых с гистоном человека H2B (32), позволило получить наиболее точные данные по распределению клеточного ответа на ауксин в делящихся клетках в процессе инициации бокового корня в меристеме родительского корня у огурца (см. рис., Б) по сравнению с репортерной конструкцией DR5::NLS-eGFP-GUS (3), содержащей сигнал ядерной локализации NLS. Также важно, что яркость белков tdTomato и mRuby3 в три раза превышает яркость белка eGFP, кроме того, mRuby3 обладает повышенной фотостабильностью (33, 34). Было показано, что формирование максимума ответа на ауксин, связанного с инициацией примордия бокового корня, у огурца происходит на удалении 200300 мкм от инициалей рядов перед первым антиклинальным делением клеток перицикла, а также эндодермы. Кроме того, показано вовлечение ауксина в дальнейшее развитие примордия бокового корня.
Таким образом, применение промоторов, чувствительных к изучаемому фитогормону, в соединении с репортерными конструкциями дает возможность проанализировать вовлечение гормона в тот или иной морфоге- нетический процесс. Предлагаемый подход подразумевает использование наиболее ярких фотостабильных флуоресцентных белков с ядерной локализацией, которые позволяют в десятки раз повысить яркость метки по сравнению с белками с цитоплазматической локализацией, поскольку при изучении ранних стадий инициации бокового корня или схожих процессов необходимо детектировать слабый сигнал от нескольких клеток перицикла, расположенных в толще корня. При этом маркеры ядерной локализации также необходимо подбирать под экспериментальную задачу. Так, применение сигнала ядерной локализации NLS не позволяло достоверно установить наличие или отсутствие маркера в делящейся клетке, поскольку в процессе деления клетки ядерная оболочка разбирается и флуоресцентный белок диффундирует в цитоплазму. Вместе с тем NLS визуализировал ядра в форме, близкой к форме нативных ядер. Соединение репортерных белков с гистоном H2B позволяет визуализировать фигуры митоза, так как гистон в процессе митоза остается связанным с ядерной ДНК. Однако у тыквенных некоторым недостатком слияния репортера с гистоновым белком может быть неравномерность распределения метки в фиксированных альдегидами хроматических ядрах (см. рис., Г).
Конструирование векторов для изучения тканевого паттерна экспрессии генов регуляторов развития . Для исследования тонких процессов развития органа растений, например инициации бокового корня, возникает необходимость выяснить, в каких тканях и клетках экспрессируется изучаемый ген. Ассиметричные клеточные деления часто опосредуют дифференцировку клеток в многоклеточных организмах и играют важную роль в развитии новых органов. У большинства цветковых растений боковые корни образуются из клеток перицикла, расположенных на ксилемном полюсе. Эти клетки ассиметрично делятся на мелкие центральные и фланкирующие клетки большего размера с разной судьбой. У Arabidopsis LATERAL ORGAN BOUNDARIES DOMAIN 16 (LBD16) и другие члены семейства LBD играют важную роль в установлении асимметрии клеток-основательниц бокового корня. LBD16 специфично экспрессируется в паре клеток перицикла на ксилемном полюсе до первого антиклинального деления в процессе инициации бокового корня. Клетки перицикла на ксилемном полюсе, в которых происходит экспрессия LBD16 , предположительно являются клетками-основательницами бокового корня (35), спецификация которых происходит в базальной части меристемы корня и зависит от осциллирующей экспрессии генов, включая ген транскрипционного фактора GATA23 (36).
Использовав базы данных NCBI , Phytozome , Cucurbit Genomics Database , PlantTFDB , мы идентифицировали в геноме кабачка (Cucurbita pepo) гены CpGATA24 (37) и CpLBD16b — ортологи ауксин-регулируемых генов Arabidopsis GATA23 и LBD16, вовлеченных в определение компетенции клеток перицикла к инициации бокового корня.
Для изучения тканевой локализации активности генов-ортологов кабачка СpLBD16b и CpGATA24 с помощью LR plus клоназной реакции были сконструированы векторы 242 pKGW-RR-MGW- pCpLBD16b::mNeonGreen-H2B и 242 pKGW-RR-MGW- pCpGATA24::mNeonGreen-H2B . Нуклеотидные последовательности промоторов генов СpLBD16b и CpGATA24 амплифици-ровали на матрице геномной ДНК кабачка и клонировали в вектор 369_pENTRattL4attR1_BSAI. В качестве репортера для локализации активности промоторов этих генов выбрали один из самых ярких и фотостабильных 1046
белков с коротким временем созревания mNeonGreen (38, 39), слитый с гистоном H2B. Нуклеотидная последовательность mNeonGreen-H2B была амплифицирована на матрице плазмиды («Allele Biotechnology and Pharmaceuticals, Inc.», США, плазмида #H2B-213) (20) и клонирована в вектор pUC18-entry8. В качестве терминатора транскрипции использовали вектор 373_pENTRattR2attL3-TermAct, содержащий терминатор гена Actin2 Arabi-dopsis (24). Результирующие бинарные векторы использовались для трансформации растений кабачка и изучения тканевой локализации активности промоторов генов CpGATA24 (37) и CpLBD16b (см. рис., В, Г). Впервые было выявлено участие CpGATA24 в инициации примордия на стадии первых антиклинальных делений в перицикле, а также отсутствие экспрессии CpLBD16b в клетках перицикла на ксилемном полюсе корня. Кроме того, было показано, что экспрессия CpLBD16b локализована в инициальных клетках рядов корня и формирующейся протофлоэме (см. рис., В, Г).
Таким образом, экспрессия репортерных генов, кодирующих флуоресцентные белки, особенно с ядерной локализацией, под контролем видоспецифичных промоторов изучаемых генов позволяет получить надежные данные о тканевом распределении экспрессии этих генов и об их вовлечении в тот или иной процесс морфогенеза даже в крупных органах растений.
Конститутивные промоторы как инструмент для визуализации. Отбор трансгенного материала — необходимая стадия экспериментов по трансформации растений. Например, при агробактериальной трансформации растений кабачка штаммами R. rhizogenes в месте инокуляции образуется каллус, состоящий из клеток, несущих вставку Т-ДНК бинарного вектора, и из нетрансформированных клеток, а также, возможно, из клеток, в которых вставка подверглась сайленсингу. Впоследствии из каллуса начинают развиваться трансгенные корни, состоящие только из трансформированных клеток, корни дикого типа и химерные корни, состоящие из клеток разных типов; последнее создает трудности при анализе трансгенных корней. Эффективный скрининг трансгенных корней позволяет осуществить кассета, состоящая из гена, кодирующего флуоресцентный белок под управлением конститутивного промотора, находящаяся в пределах Т-ДНК в бинарном векторе. Спектр эмиссии скринингового и ре-портерного белков должен различаться (традиционно подбирают пары с флуоресценцией в красной и зеленой областях спектра). В случае вектора 242 pKGW-RR-MGW для отбора трансгенных корней использовали кассету pAtUBQ10::DsRED1 , состоящую из гена, кодирующего красный флуоресцентный белок DsRED1, под контролем промотора конститутивного гена убиквитина Arabidopsis AtUBQ10 (см. рис., А), при этом репортерные гены кодировали флуоресцентные белки из зеленой части спектра (см. рис., Б-Г).
В большинстве микроскопических исследований возникает необходимость визуализации ядер клеток. Обычно ядра клеток подкрашивают красителями на ДНК (например, DAPI или йодид пропидия), однако такие красители неравномерно проникают в глубину препарата и окрашивают все нуклеиновые кислоты (ДНК митохондрий и хлоропластов, а иногда и РНК). Современный подход к визуализации ядер — применение конститутивных промоторов, под контролем которых находится ген, кодирующий флуоресцентный белок с ядерной локализацией. Наличие такой кассеты в Т-ДНК вектора позволяет одновременно осуществлять скрининг трансгенного материала корней и визуализировать ядра в клетках на срезах изучаемого органа.
Для визуализации ядер в тканях корня тыквенных мы получили новые генетические конструкции, в которых гены, кодирующие скрининговые белки, контролируются конститутивными промоторами. В векторе
242 pKGW-RR-MGW- pUBQ10::H2B-Venus репортерный ген желтого флуоресцентного белка Venus слит с геном человеческого гистона H2B и находится под управлением промотора гена убиквитина Arabidopsis ( pUBQ10 ). Для репортерного белка Venus характерно короткое время созревания, высокая фотостабильность и повышенная яркость по сравнению с eGFP (33, 40). Вектор 236 pKGW-RR-MGW- pACT2::tdTomato-H2B содержит репортер-ный ген tdTomato , слитый с геном гистона H2B под контролем конститутивного промотора гена актина Arabidopsis ( pACT2 ).
Было показано, что оба промотора (гена убиквитина и гена актина), хотя и обеспечивают достаточно высокий уровень экспрессии генов, кодирующих флуоресцентные белки, но все же проявляют различную активность в тканях корня (см. рис., Д, Е). В клетках коры и ризодермы оба промотора были активнее, чем в тканях центрального цилиндра корня, что создает трудности при изучении инициации бокового корня и других процессов, протекающих в перицикле и остальных тканях центрального цилиндра.
Таким образом, конститутивные промоторы в сочетании с геном, кодирующим флуоресцентный белок, служат ценным инструментом, который может быть использован как для скрининга трансгенного материала, так и для визуализации ядер клеток. Однако выбор конститутивного промотора требует внимания исследователя. Так, во многих векторах для трансформации растений в скрининговой кассете используется сильный конститутивный промотор вируса мозаики цветной капусты (CaMV) p35S . Ранее нами уже было показано, что при трансформации растений кабачка «пустым» вектором pMDC162-GFP (23) p35S промотор, находящийся в Т-ДНК этого вектора в кассете p35S::GFP , вызывает эктопическую экспрессию гена GUS , перед которым нет промотора и который, следовательно, не должен экспрессироваться в тканях корня (3). В литературе также описаны случаи влияния промотора 35S на экспрессию целевой кассеты, что вызывает получение артефактов в исследованиях (41, 42). Наличие нативного промотора pAtUBQ10 в кассете pAtUBQ10::DsRED1 вектора 242 pKGW-RR-MGW не приводит к искажениям в экспрессии целевой кассеты (3). Также известен факт неравномерной активности промоторов конститутивных генов в разных тканях органов растений. Так, p35S более активен в клетках центрального цилиндра, что проявляется, как яркое окрашивание ядер клеток центрального цилиндра и тусклое — клеток коры (43). В исследованиях наиболее часто используются промоторы конститутивно экспрессирующихся генов Arabidopsis — гена убиквитина и гена актина (18, 29, 44, 45), хотя известны и другие промоторы конститутивных генов растений, например клонированный из тополя Populus tomentosa PtMCP (46). Также описан промотор гена RIBOSOMAL PROTEIN S5A (RPS5A) , который селективно экспрессируется в делящихся клетках (47) и может быть использован в исследованиях как маркер пролиферативной активности.
Методика пробоподготовки для локализации флуоресцентных белков на срезах фиксированных органов. Важнейшая задача в исследованиях с использованием репортерных флуоресцентных белков — сохранение их способности к флуоресценции в течение как можно более длительного времени. Для этого мы модифицировали методику фиксации растительных тканей и приготовления срезов (на примере трансгенных корней кабачка и огурца).
В связи с тем, что при фиксации в 100 мМ фосфатном буфере клетки корня претерпевают плазмолиз, мы изменили состав фиксатора (48), предложенного Brian Lin (Tufts University, Boston, MA, США). Снижение молярности фосфатного буфера в 5 раз (до 20 мМ) позволило решить проблему плазмолиза. Аналогичный подход также был использован при иммунолокализации цитоскелета на срезах фиксированных симбиотических клубеньков (49, 50). Для большинства флуоресцентных белков (GFP, Venus, tdTomato, mRuby3) оптимально значение pH 7,2. Однако для предотвращения быстрого затухания флуоресцентного белка mNeonGreen в корнях трансгенных растений мы использовали буфер с pH 8,0. Финальный состав фиксатора был следующим: 1 % параформальдегид, 5 % диметилсульфоксид (DMSO), 75 мM L-лизин, 10 мM м-периодат натрия в 20 мМ фосфатном буфере, pH 7,2-8,0. Фиксировали кончики главного корня длиной 6-8 мм. Для удаления воздуха из тканей растительный материал в емкостях с фиксатором помещали в эксикатор под вакуум (-1 атм) на 3-5 мин, затем вакуум снимали, процедуру повторяли 3-4 раза. Корни выдерживали в фиксаторе в течение 1 ч при комнатной температуре. После фиксации материал трижды промывали 20 мМ фосфатным буфером, содержащим 75 мM L-лизин, в течение 15-20 мин.
Фиксированный материал помещали в резиновые формы и заливали расплавленной 2 % LE агарозой («Lonza Group», Швейцария) при температуре 40-50 ° С. Готовые агарозные блоки наклеивали на плоские металлические держатели секундным цианакрилатным клеем Супер Момент («Хенкель», Россия). Для приготовления толстых срезов (65 мкм) корней использовали автоматический прецизионный микротом с вибрирующим лезвием HM650V («Thermo Fisher Scientific Microm International GmbH», Германия). Для повышения качества срезов применяли сапфировый нож («Delaware Diamond Knives, Inc.», США). Оптимальной средой для резки была дистиллированная вода, поскольку в различных буферах — фосфатном, трис-буферном солевом растворе (TBS), буфере, стабилизирующем микротрубочки (MTSB), — срезы корней кабачка значительно деформировались. Важно, что клеточные стенки были окрашены специфическим красителем — 0,1 % SCRI Renaissance 2200 (SR2200, «Renaissance Chemicals, Ltd», Великобритания) (51) в dH 2 O (pH 8,0). Срезы окрашивали в течение 1 ч без последующей отмывки. Краситель готовили непосредственно перед применением. Если это было необходимо, ядра клеток альтернативно подкрашивали 0,3 мкг/мл DAPI в течение 30-50 мин.
Срезы помещали в заключающую среду под покровные стекла. Мы показали, что большинство флуоресцентных белков (GFP, Venus, tdTomato) после предложенной фиксации сохраняют флуоресценцию в жидкой заключающей среде CFMR2 («Citifluor, Division of Electron Microscopy Sciences», США). Однако оптимальным, в том числе и для трансгенных корней, несущих mNeonGreen, оказалось заключение срезов в модифицированный нами просветляющий состав ClearSee (9), содержащий монохлорид L-лизина (L-lysine monochloride, 13,7 г/л), ксилитол (100 г/л), дезоксихолат натрия (150 г/л) и мочевину (240 г/л в 20 мМ фосфатном буфере, pH 8,0).
Таким образом, при работе с флуоресцентными белками существует множество методических сложностей, которые необходимо учитывать. Так, для сохранения свечения флуоресцентных белков необходимо сделать выбор в пользу заключения материала в 2-3 % агарозу, в которой при резке удобно удерживать небольшие фрагменты растительного материала. Как уже было показано ранее, для иммунолокализации цитоскелета в азотфик-сирующих корневых клубеньках бобовых (49, 50) материал не следует дегидратировать в серии спиртов и заключать в заливочные среды. При заключении трансгенного материала, несущего репортерные флуоресцентные белки, в парафин или в воск Стидмана (среду с более щадящей пробопод-готовкой, подходящей для иммунолокализации чувствительных белков) (52-
-
54) требуется проводка по спиртам для обезвоживания, что приводит к быстрому затуханию флуоресценции таких белков. При монтаже в расплавленную агарозу воздействие температуры (около 40 ° С) кратковременно, что не приводит к денатурации флуоресцентного белка. Концентрация агарозы также требует оптимизации: чем мягче объект, тем ниже должна быть концентрация агарозы. Слишком плотная агароза может приводить к сильному сжатию и обезвоживанию мягкого объекта, что в случае кончика корня выглядит как сжатие зоны растяжения. Вместо традиционно используемых йодида пропидия (PI) или FM4-64 (окрашивание плазмалеммы) мы предлагаем упрощенный протокол окрашивания специфическим для клеточных стенок красителем SCRI Renaissance 2200, который позволяет четко визуализировать анатомическое строение на срезах (см. рис., В, Г). Наше исследование также показало важность выбора оптимальной заключающей среды для срезов, поскольку от этого параметра зависит стабильность и длительность флуоресценции.
Лазерная сканирующая конфокальная микроскопия для визуализации флуоресцентных белков в фиксированных тканях. Анализ препаратов и получение иллюстраций проводили с применением конфокального лазерного сканирующего микроскопа LSM 780 («Zeiss», Германия) с использованием объективов 10½/0,45 и 20½/0,8 серии Plan-Apochromat с высокой числовой апертурой, а для 40½/1,3 — с масляной иммерсией. Для детекции репортерных флуоресцентных белков eGFP и mNeonGreen использовали лазер с длиной волны 488 нм, для желтого белка Venus — 514 нм, для красных белков mRuby3 и tdTomato — 561 нм. Клеточные стенки (SCRI Renaissance 2200) или ядра (DAPI) детектировали при возбуждении лазером с длиной волны 405 нм. Для визуализации неокрашенных анатомических структур применяли дифференциальный интерференционный контраст в дополнительном канале проходящего света. Для получения и обработки изображений использовали программу ZEN версии 2.3 («Zeiss», Германия).
На современном этапе исследований визуализацию флуоресцентных белков в толстых срезах тканей растений нельзя представить без применения конфокальной микроскопии. К возможностям, которые предоставляет лазерный сканирующий микроскоп, относится значительное повышение пространственного разрешения сигнала, что позволяет разделить структуры, сливающиеся при применении широкопольной микроскопии из-за внефокусного света. Приготовление относительно тонких срезов (7-16 мкм) исследуемых объектов приводит к трудностям при восстановлении пространственной структуры участка ткани или органа, однако только тонкие срезы могут быть исследованы при помощи традиционных методов световой микроскопии. Применение толстых срезов (50-70 мкм) в комбинации с конфокальной микроскопией дает возможность получить серии оптических срезов (z-стек). Далее в зависимости от целей исследования можно представить z-стек в виде двумерного изображения с использованием алгоритма проекции максимальной интенсивности или получить 3D-рекон-струкцию структуры. Такой подход успешно используется для изучения процессов развития различных органов растений, в частности апикальных меристем побегов и корней, а также развития бокового корня (55, 56).
Существенную проблему, с которой сталкиваются при исследовании развития органов растений, составляет перекрывание спектров нескольких флуоресцентных белков, а также автофлуоресценция элементов клеток и тканей (57-59). Главные источники автофлуоресценции при изучении растительных тканей — хлорофилл (флуоресценция в красной области спектра)
и лигнин (флуоресценция в зеленой и желтой областях спектра). Автофлуоресценция маскирует и затрудняет регистрацию флуоресценции исследуемых флуорохромов. Она бывает достаточно сильной, и ее можно ошибочно принять за флуоресценцию репортеров, что приводит к искажению данных. Применение конфокальной микроскопии позволяет разделить автофлуоресценцию и сигнал от репортеров. Для элиминации автофлуоресценции, а также для разделения перекрывающихся спектров нескольких флуоресцентных репортерных белков применяют алгоритм линейного спектрального разделения (linear spectral unmixing) (58, 60). Оценку характера автофлуоресценции удобно проводить, снимая спектры эмиссии на неокрашенном препарате (61). Обычно в изучаемых препаратах присутствует несколько красителей/флуоресцентных белков, каждый из которых маркирует структуры клетки или отдельные клетки. При получении изображения флуорофоры детектируются либо по отдельности, либо в виде смеси в зависимости от их пространственного распределения внутри объекта. Цель алгоритма линейного спектрального разделения заключается в определении относительного вклада интенсивности каждого флуорофора для каждого пикселя (60). Если спектры всех флуорофоров, находящихся в образце, известны, то их содержание в пределах пикселя может быть вычислено по спектральной кривой каждого пикселя, которая определяется и записывается в процессе лямбда-сканирования. Как только происходит определение спектрального вклада каждого флуорофора, лямбда-стек может быть разделен на отдельные каналы для каждого флуорофора. В частности, спектр автофлуоресценции можно представить как отдельный канал и вывести из финального изображения образца. Таким образом, применение комплекса современных подходов в конфокальной лазерной микроскопии позволяет преодолеть проблемы крупных и плотных образцов, получая объемную картину распределения флуоресцентных белков с высоким разрешением.
Итак, нами разработана и применена на практике новая комплексная методика, позволяющая изучить распределение репортерных флуоресцентных белков на фиксированных срезах крупных органов немодельных растений. Показана важность тщательного подбора генетических конструкций со скрининговой и репортерной кассетой с учетом особенностей растительного объекта, в первую очередь — автофлуоресценции. Оптимальным решением является конструирование таких векторов в соответствии с задачами конкретного эксперимента, поэтому необходимое условие применения предлагаемого подхода — наличие методики трансформации для изучаемого вида, которая позволяет доставить Т-ДНК вектора в геном растения. Критический фактор — состав и pH фиксатора, которые необходимо адаптировать с учетом как растительного объекта, так и используемого флуоресцентного репортерного белка. Для сохранения свечения флуоресцентных белков также важен способ приготовления срезов. Оптимальна заливка материала в агарозу. Для визуализации клеточных стенок и морфологии тканей нами предложен упрощенный протокол окрашивания с SCRI Renaissance 2200. Также важен правильный подбор среды для заключения полученных срезов, так как ее свойства влияют на фотостабильность белка во время изучения препаратов. Наконец, точность конечного результата будет зависеть от разрешающей способности выбранного метода микроскопирования. Применение флуоресцентных репортерных белков для изучения процессов развития растений многообразно и не ограничивается изучением пространственного распределения клеточного ответа на изучаемый фитогормон, тканевой локализации активности промотора гена, кодирующего один из регуляторов развития, или непосредственно самого белка. Экспрес- сия генов флуоресцентных белков под контролем промоторов генов интереса представляет собой крайне специфичную флуоресцентную метку, вырабатываемую непосредственно растением и маркирующую не только структуры клетки, но и физиологические процессы, чего невозможно достигнуть при окрашивании образца флуоресцентными красителями. Однако для любого исследования с применением флуоресцентных белков необходимо тщательное планирование эксперимента и пробоподготовки для обеспечения сохранности образца.
Список литературы Особенности использования флуоресцентных репортерных белков для изучения развития корневых систем на примере тыквенных (Cucurbitaceae)
- Cesarino I., Ioio R.D., Kirschner G.K., Ogden M.S., Picard K.L., Rast-Somssich M.I., Somssich M. Plant science's next top models. Annals of Botany, 2020, 126(1): 1-23 (doi: 10.1093/aob/mcaa063).
- Che G., Gu R., Zhao J., Liu X., Song X., Zi H., Cheng Z., Shen J., Wang Z., Liu R., Yan L., Weng Y., Zhang X. Gene regulatory network controlling carpel number variation in cucumber. Development, 2020, 147(7): dev184788 (doi: 10.1242/dev.184788).
- Ilina E.L., Logachov A.A., Laplaze L., Demchenko N.P., Pawlowski K., Demchenko K.N. Composite Cucurbita pepo plants with transgenic roots as a tool to study root development. Annals of Botany, 2012, 110(2): 479-489 (doi: 10.1093/aob/mcs086).
- Osipowski P., Pawelkowicz M., Wojcieszek M., Skarzynska A., Przybecki Z., Pl^der W. A high-quality cucumber genome assembly enhances computational comparative genomics. Mol. Genet. Genomics, 2020, 295(1): 177-193 (doi: 10.1007/s00438-019-01614-3).
- Day R.N., Davidson M.W. The fluorescent protein palette: tools for cellular imaging. Chemical Society Reviews, 2009, 38(10): 2887-2921 (doi: 10.1039/B901966A).
- Chudakov D.M., Matz M.V., Lukyanov S., Lukyanov K.A. Fluorescent proteins and their applications in imaging living cells and tissues. Physiological Review, 2010, 90(3): 1103-1163 (doi: 10.1152/physrev.00038.2009).
- Mathur J. The illuminated plant cell. Trends in Plant Science, 2007, 12(11): 506-513 (doi: 10.1016/j.tplants.2007.08.017).
- Berg R.H., Beachy N.R. Fluorescent protein applications in plants. Methods in Cell Biology, 2008, 85: 153-177 (doi: 10.1016/S0091-679X(08)85008-X).
- Kurihara D., Mizuta Y., Sato Y., Higashiyama T. ClearSee: a rapid optical clearing reagent for whole-plant fluorescence imaging. Development, 2015, 142(23): 4168-4179 (doi: 10.1242/dev.127613).
- Ursache R., Andersen T.G., Marhavy P., Geldner N. A protocol for combining fluorescent proteins with histological stains for diverse cell wall components. Plant J., 2018, 93(2): 399-412 (doi: 10.1111/tpj.13784).
- Nagaki K., Yamaji N., Murata M. ePro-ClearSee: a simple immunohistochemical method that does not require sectioning of plant samples. Scientific Reports, 2017, 7: 42203 (doi: 10.1038/srep42203).
- Truernit E., Bauby H., Dubreucq B., Grandjean O., Runions J., Barthélémy J., Palauqui J.-C. High-resolution whole-mount imaging of three-dimensional tissue organization and gene expression enables the study of phloem development and structure in Arabidopsis. Plant Cell, 2008, 20(6): 1494 (doi: 10.1105/tpc.107.056069).
- Mizuta Y., Kurihara D., Higashiyama T. Two-photon imaging with longer wavelength excitation in intact Arabidopsis tissues. Protoplasma, 2015, 252(5): 1231-1240 (doi: 10.1007/s00709-014-0754-5).
- FeyY J.A., Moreno N. Imaging plant cells by two-photon excitation. Protoplasma, 2004, 223(1): 1-32 (doi: 10.1007/s00709-003-0026-2).
- Girkin J.M., Carvalho M.T. The light-sheet microscopy revolution. Journal of Optics, 2018, 20(5): 053002 (doi: 10.1088/2040-8986/aab58a).
- Ovecka M., Vaskebova L., Komis G., Luptovciak I., Smertenko A., Samaj J. Preparation of plants for developmental and cellular imaging by light-sheet microscopy. Nature Protocols, 2015, 10: 1234 (doi: 10.1038/nprot.2015.081).
- Valuchova S., Mikulkova P., Pecinkova J., Klimova J., Krumnikl M., Bainar P., Heckmann S., Tomancak P., Riha K. Imaging plant germline differentiation within Arabidopsis flowers by light sheet microscopy. eLife, 2020, 9: e52546 (doi: 10.7554/eLife.52546).
- Ovecka M., von Wangenheim D., Tomancak P., Samajova O., Komis G., Samaj J. Multiscale imaging of plant development by light-sheet fluorescence microscopy. Nature Plants, 2018, 4(9): 639-650 (doi: 10.1038/s41477-018-0238-2).
- Knapp E., Flores R., Scheiblin D., Modla S., Czymmek K., Yusibov V. A cryohistological protocol for preparation of large plant tissue sections for screening intracellular fluorescent protein expression. BioTechniques, 2012, 52(1): 31-37 (doi: 10.2144/000113778).
- Shaner N.C., Lambert G.G., Chammas A., Ni Y., Cranfill P.J., Baird M.A., Sell B.R., Allen J.R., Day R.N., Israelsson M., Davidson M.W., Wang J. A bright monomeric green fluorescent protein derived from Branchiostoma lanceolatum. Nat. Methods, 2013, 10(5): 407409 (doi: 10.1038/nmeth.2413).
- Bindels D.S., Haarbosch L., van Weeren L., Postma M., Wiese K.E., Mastop M., Aumonier S., Gotthard G., Royant A., Hink M.A., Gadella Jr T.W.J. mScarlet: a bright monomeric red fluorescent protein for cellular imaging. Nat. Methods, 2016, 14: 53 (doi: 10.1038/nmeth.4074).
- Ivanov S., Harrison M.J. A set of fluorescent protein-based markers expressed from constitutive and arbuscular mycorrhiza-inducible promoters to label organelles, membranes and cytoskeletal elements in Medicago truncatula. Plant J, 2014, 80(6): 1151-1163 (doi: 10.1111/tpj.12706).
- Curtis M.D., Grossniklaus U. A Gateway cloning vector set for high-throughput functional analysis of genes in planta. Plant Physiology, 2003, 133(2): 462-469 (doi: 10.1104/pp.103.027979).
- Engler C., Youles M., Gruetzner R., Ehnert T.-M., Werner S., Jones J.D.G., Patron N.J., Marillonnet S. A Golden Gate modular cloning toolbox for plants. ACS Synthetic Biology, 2014, 3(11): 839-843 (doi: 10.1021/sb4001504).
- Karimi M., Inze D., Depicker A. GATEWAY(TM) vectors for Agrobacterium-mediated plant transformation. Trends in Plant Science, 2002, 7(5): 193-195 (doi: 10.1016/S1360-1385(02)02251-3).
- Landy A. Dynamic, structural, and regulatory aspects of lambda site-specific recombination. Annu. Rev. Biochem., 1989, 58(1): 913-941 (doi: 10.1146/annurev.bi.58.070189.004405).
- Hornung E., Krueger C., Pernstich C., Gipmans M., Porzel A., Feussner I. Production of (10E,12Z)-conjugated linoleic acid in yeast and tobacco seeds. Biochimica et Biophysica Acta (BBA) — Molecular and Cell Biology of Lipids, 2005, 1738(1-3): 105-114 (doi: 10.1016/j.bbalip.2005.11.004).
- Op den Camp R.H.M., De Mita S., Lillo A., Cao Q., Limpens E., Bisseling T., Geurts R. A phylogenetic strategy based on a legume-specific whole genome duplication yields symbiotic cytokinin type-A response regulators. Plant Physiology, 2011, 157(4): 2013-2022 (doi: 10.1104/pp.111.187526).
- Limpens E., Ramos J., Franken C., Raz V., Compaan B., Franssen H., Bisseling T., Geurts R. RNA interference in Agrobacterium rhizogenes-transformed roots of Arabidopsis and Medicago truncatula. Journal of Experimental Botany, 2004, 55(399): 983-992 (doi: 10.1093/jxb/erh122).
- Tsai F.Y., Coruzzi G. Light represses transcription of asparagine synthetase genes in photosynthetic and nonphotosynthetic organs of plants. Mol. Cell. Biol., 1991, 11(10): 4966-4972 (doi: 10.1128/mcb.11.10.4966).
- Ulmasov T., Murfett J., Hagen G., Guilfoyle T.J. Aux/IAA proteins repress expression of reporter genes containing natural and highly active synthetic auxin response elements. Plant Cell, 1997, 9(11): 1963-1971 (doi: 10.1105/tpc.9.11.1963).
- Nam H.-S., Benezra R. High levels of Id1 expression define B1 type adult neural stem cells. Cell Stem Cell, 2009, 5(5): 515-526 (doi: 10.1016/j.stem.2009.08.017).
- Shaner N.C., Steinbach P.A., Tsien R.Y. A guide to choosing fluorescent proteins. Nat. Methods, 2005, 2(12): 905-909 (doi: 10.1038/nmeth819).
- Bajar B.T., Wang E.S., Lam A.J., Kim B.B., Jacobs C.L., Howe E.S., Davidson M.W., Lin M.Z., Chu J. Improving brightness and photostability of green and red fluorescent proteins for live cell imaging and FRET reporting. Scientific Reports, 2016, 6(20889): 1-12 (doi: 10.1038/srep20889).
- Goh T., Joi S., Mimura T., Fukaki H. The establishment of asymmetry in Arabidopsis lateral root founder cells is regulated by LBD16/ASL18 and related LBD/ASL proteins. Development, 2012, 139(5): 883-893 (doi: 10.1242/dev.071928).
- De Rybel B., Vassileva V., Parizot B., Demeulenaere M., Grunewald W., Audenaert D., Van Campenhout J., Overvoorde P., Jansen L., Vanneste S., Möller B., Wilson M., Holman T., Van Isterdael G., Brunoud G., Vuylsteke M., Vernoux T., De Veylder L., Inze D., Weijers D., Bennett M.J., Beeckman T. A novel Aux/IAA28 signaling cascade activates GATA23-dependent specification of lateral root founder cell identity. Current Biology, 2010, 20(19): 1697-1706 (doi: 10.1016/j.cub.2010.09.007).
- Kiryushkin A.S., Ilina E.L., Puchkova V.A., Guseva E.D., Pawlowski K., Demchenko K.N. Lateral root initiation in the parental root meristem of cucurbits: Old players in a new position. Front. Plant Sci., 2019, 10: 365 (doi: 10.3389/fpls.2019.00365).
- Hostettler L., Grundy L., Käser-Pebernard S., Wicky C., Schafer W.R., Glauser D.A. The bright fluorescent protein mNeonGreen facilitates protein expression analysis in vivo. G3: Genes, Genomes, Genetics, 2017, 7(2): 607-615 (doi: 10.1534/g3.116.038133).
- Balleza E., Kim J.M., Cluzel P. Systematic characterization of maturation time of fluorescent proteins in living cells. Nat. Methods, 2017, 15: 47 (doi: 10.1038/nmeth.4509).
- Nagai T., Ibata K., Park E.S., Kubota M., Mikoshiba K., Miyawaki A. A variant of yellow fluorescent protein with fast and efficient maturation for cell-biological applications. Nat. Biotechnol., 2002, 20(1): 87-90 (doi: 10.1038/nbt0102-87).
- Yoo S.Y., Bomblies K., Yoo S.K., Yang J.W., Choi M.S., Lee J.S., Weigel D., Ahn J.H. The 35S promoter used in a selectable marker gene of a plant transformation vector affects the expression of the transgene. Planta, 2005, 221(4): 523-530 (doi: 10.1007/s00425-004-1466-4).
- Zheng X.L., Deng W., Luo K.M., Duan H., Chen Y.Q., McAvoy R., Song S.Q., Pei Y., Li Y. The cauliflower mosaic virus (CaMV) 35S promoter sequence alters the level and patterns of
- activity of adjacent tissue- and organ-specific gene promoters. Plant Cell Rep., 2007, 26(8): 11951203 (doi: 10.1007/s00299-007-0307-x).
- Samac D.A., Tesfaye M., Dornbusch M., Saruul P., Temple S.J. A comparison of constitutive promoters for expression of transgenes in alfalfa (Medicago sativa). Transgenic Res., 2004, 13(4): 349-361 (doi: 10.1023/B:TRAG.0000040022.84253.12).
- An Y.-Q., McDowell J.M., Huang S., McKinney E.C., Chambliss S., Meagher R.B. Strong, constitutive expression of the Arabidopsis ACT2/ACT8 actin subclass in vegetative tissues. The Plant Journal, 1996, 10(1): 107-121 (doi: 10.1046/j.1365-313X.1996.10010107.x).
- Vermeer J.E.M., von Wangenheim D., Barberon M., Lee Y., Stelzer E.H.K., Maizzel A., Geldner N. A spatial accommodation by neighboring cells is required for organ initiation in Arabidopsis. Science, 2014, 343(6167): 178-183 (doi: 10.1126/science.1245871).
- Chen Z., Wang J., Ye M.-X., Li H., Ji L.-X., Li Y., Cui D.-Q., Liu J.-M., An X.-M. A novel moderate constitutive promoter derived from poplar (Populus tomentosa Carrière). Int. J. Mol. Sci., 2013, 14(3): 6187-6204 (doi: 10.3390/ijms14036187).
- Benjamins R., Quint A., Weijers D., Hooykaas P., Offringa R. The PINOID protein kinase regulates organ development in Arabidopsis by enhancing polar auxin transport. Development, 2001, 128(20): 4057-4067.
- McLean I.W., Nakane P.K. Periodate-lysine-paraformaldehyde fixative a new fixative for immunoelectron microscopy. Journal of Histochemistry & Cytochemistry, 1974, 22(12): 1077-1083 (doi: 10.1177/22.12.1077).
- Kitaeva A.B., Kusakin P.G., Demchenko K.N., Tsyganov V.E. Key methodological features of tubulin cytoskeleton studies in nodules of legume plants. Agricultural Biology [Sel'skokhozyaistvennaya Biologiya], 2018, 53(3): 634-644 (doi: 10.15389/agrobiology.2018.3.634eng).
- Kitaeva A.B., Demchenko K.N., Tikhonovich I.A., Timmers A.C.J., Tsyganov V.E. Comparative analysis of the tubulin cytoskeleton organization in nodules of Medicago truncatula and Pisum sativum: Bacterial release and bacteroid positioning correlate with characteristic microtubule rearrangements. New Phytol., 2016, 210(1): 168-183 (doi: 10.1111/nph.13792).
- Musielak T.J., Schenkel L., Kolb M., Henschen A., Bayer M. A simple and versatile cell wall staining protocol to study plant reproduction. Plant Reprod., 2015, 28(3): 161-169 (doi: 10.1007/s00497-015-0267-1).
- Zdyb A., Demchenko K., Heumann J., Mrosk C., Grzeganek P., Göbel C., Feussner I., Pawlowski K., Hause B. Jasmonate biosynthesis in legume and actinorhizal nodules. New Phytol., 2011, 189(2): 568-579 (doi: 10.1111/j.1469-8137.2010.03504.x).
- Stumpe M., Göbel C., Demchenko K., Hoffmann M., Klösgen R.B., Pawlowski K., Feussner I. Identification of an allene oxide synthase (CYP74C) that leads to formation of a-ketols from 9-hydroperoxides of linoleic and linolenic acid in below-ground organs of potato. The Plant Journal, 2006, 47(6): 883-896 (doi: 10.1111/j.1365-313X.2006.02843.x).
- Demchenko K., Zdyb A., Feussner I., Pawlowski K. Analysis of the subcellular localisation of lipoxygenase in legume and actinorhizal nodules. Plant Biology, 2012, 14(1): 56-63 (doi: 10.1111/j.1438-8677.2011.00480.x).
- Ilina E.L., Kiryushkin A.S., Semenova V.A., Demchenko N.P., Pawlowski K., Demchenko K.N. Lateral root initiation and formation within the parental root meristem of Cucurbita pepo: is auxin a key player? Annals of Botany, 2018, 122(5): 873-888 (doi: 10.1093/aob/mcy052).
- Prunet N., Jack T.P., Meyerowitz E.M. Live confocal imaging of Arabidopsis flower buds. Developmental Biology, 2016, 419(1): 114-120 (doi: 10.1016/j.ydbio.2016.03.018).
- Dickinson M.E., Bearman G., Tille S., Lansford R., Fraser S.E. Multi-spectral imaging and linear unmixing add a whole new dimension to laser scanning fluorescence microscopy. BioTechniques, 2001, 31(6): 1272-1278 (doi: 10.2144/01316bt01).
- Kraus B., Ziegler M., Wolff H. Linear fluorescence unmixing in cell biological research. In: Modern research and educational topics in microscopy /A. Méndez-Vilas, J. Diaz (eds.). Formatex, Badajoz, Spain, 2007: 863-872.
- Mylle E., Codreanu M.-C., Boruc J., Russinova E. Emission spectra profiling of fluorescent proteins in living plant cells. Plant Meth., 2013, 9(1): 10 (doi: 10.1186/1746-4811-9-10).
- Zimmermann T., Rietdorf J., Pepperkok R. Spectral imaging and its applications in live cell microscopy. FEBS Letters, 2003, 546(1): 87-92 (doi: doi:10.1016/S0014-5793(03)00521-0).
- Коржевский Д.Э., Кирик О.В., Сухорукова Е.Г., Колос Е.А., Карпенко М.Н., Суфиева Д.А., Назаренкова А.В. Молекулярная морфология. Методы флуоресцентной и конфокальной лазерной микроскопии. СПб, 2014.


