Особенности экспрессионного профиля микроРНК в клетках меланомы и опухолевом микроокружении
Автор: Комина Анна Владимировна, Аксененко Мария Борисовна, Палкина Надежда Владимировна, Артемьев Сергей Александрович, Рукша Татьяна Геннадьевна
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 5 (71), 2015 года.
Бесплатный доступ
Меланома кожи относится к многокомплексным неопластическим системам, характеризующимся высокой степенью гетерогенности опухолевой ткани. МикроРНК являются разновидностью эпигенетических регуляторов экспрессии генов. На сегодняшний день известно более 1 500 разновидностей микроРНК, а также установлено, что около 90 % генома человека регулируется посредством микроРНК. В данном исследовании проведен сравнительный анализ экспрессии микроРНК на основе микрочипирования в образцах клеток меланомы, опухолевого окружения, а также меланоцитарных невусов. Выявленные различия в уровне экспрессии микроРНК между тремя исследуемыми группами требуют дальнейшего разъяснения с целью уточнения функциональной роли отдельных микроРНК в развитии меланомы, а также для потенциального использования в качестве прогностических и диагностических маркеров
Микрорнк, меланома, микроэррей
Короткий адрес: https://sciup.org/14056582
IDR: 14056582 | УДК: 577.218:616-006.81
Текст научной статьи Особенности экспрессионного профиля микроРНК в клетках меланомы и опухолевом микроокружении
МикроРНК представляют собой последовательности, состоящие из 18–22 нуклеотидов, являются посттранскрипционными регуляторами экспрессии генов. На сегодняшний день выявлено несколько тысяч разновидностей микроРНК. Известно, что профили микроРНК являются тканеспецифичными, а также могут изменяться при развитии патологических процессов. Единичная микроРНК может регулировать активность сотен генов. С помощью микроРНК регулируется 90 % человеческого генома. Одним из основных механизмов действия микроРНК является снижение экспрессии целевой мРНК посредством связывания с комплементарными фрагментами в 3` не-транслируемом регионе [9]. Основанное на данных о регуляции микроРНК механизмов развития различных заболеваний, одним из направлений экспериментальной терапии является ингибирование микроРНК анти-смысловыми «анти-микроРНК» или, напротив, повышение уровня/активности микроРНК [5]. Для идентификации микроРНК, ассоциированных с конкретным патологическим состоянием, используется профилирование микроРНК, которое позволяет определять экспрессию широкого спектра микроРНК. В дальнейшем выявленные изменения могут быть использованы для определения диагностической и прогностической значимости отдельных микроРНК [8]. Изменения профиля микроРНК выявлены при воспалении, развитии сердечно-сосудистых заболеваний, злокачественных новообразований и пр. [6, 11].
В Российской Федерации в 2013 г. вклад меланомы кожи (МК) в структуру заболеваемости злокачественными новообразованиями кожи составил 12 %, при этом МК явилась причиной смерти от злокачественных новообразований кожи в 31 % случаев [1]. Меланома – это гетерогенное новообразование с включением в молекулярный патогенез множества сигнальных путей, которые, в том числе, обеспечивают высокую пластичность опухолевых клеток и, как следствие, резистентность к противоопухолевой терапии. Известно, что микроокружение опухоли при меланоме играет существенную роль в развитии и прогрессировании заболевания. В частности, повышение продукции эндотелиальными клетками ИЛ-6, -8 вызывает активацию пролиферации клеток меланомы [17], костно-мозговые супрессоры блокируют опухоль-реактивные Т-лимфоциты [10]. МикроРНК также выступают как паракринные и аутокринные регуляторы биологического поведения опухолевого микроокружения. Так, показано, что miR-155, miR-31, miR-214 in vitro и in vivo способствуют трансформации фибробластов в опухоль-ассоциированные фибробласты при раке яичников [3].
Целью исследования являлось изучение особенностей профиля микроРНК в доброкачественных меланоцитарных новообразованиях кожи: меланоцитарных невусах, в клетках меланомы и ткани опухолевого окружения.
Материал и методы
Образцы меланомы кожи (n=10) и доброкачественных меланоцитарных новообразований (n=5) были получены в КГБУЗ «Красноярское краевое патологоанатомическое бюро». Исследование утверждено локальным этическим комитетом (протокол № 59/2014 от 02.12.2014). Образцы окрашивались гематоксилином и эозином и просматривались на процент содержания опухолевых клеток. Для отделения опухолевого микроокружения осуществлялась макродиссекция ткани, после чего повторно изготавливались срезы, окрашенные гематоксилином и эозином, с целью проверки на отсутствие опухолевых клеток в ткани микроокружения.
Выделение и очистка микроРНК из образцов осуществлялись в два этапа: на первом этапе производилась экстракция РНК смесью фенол/ хлороформ с использованием комплекта реагентов «РИБО-золь-В» (Amplisens, Россия), после чего, на втором этапе, осуществлялась очистка микроРНК из экстракта на колонках при помощи набора mirVana™ miRNA Isolation Kit (Ambion, Canada) в соответствии с протоколом производителя. Оценка концентрации микроРНК выполнялась с помощью флуориметра Qubit® 2.0 (Life technologies Holdings Pte Ltd., USA) c применением набора Qubit® HS RNA Assay kit и набора Qubit® microR-NA Assay kit (Eugene, Oregon, USA). Наличие примесей белка и фенолов определялось с помощью оценки коэффициентов поглощения при длинах волн 260/280 и 260/230 на спектрофотометре NanoVue Plus (GE Healthcare, USA). Дальнейшая оценка профиля экспрессии микроРНК выполнялась с помощью системы микрочипирования GeneAtlas® Personal Microarray System (Affymetrix, Santa
Clara, CA, USA). С этой целью не менее 130 нг микроРНК подвергалось полиаденилированию и последующему биотинилированию с использованием набора FlashTag™ Biotin HSR RNA labeling Kit (Affymetrix, Santa Clara, CA, USA) в соответствии с протоколом производителя. Полученные комплексы подвергались гибридизации на микрочипы GeneChip miRNA 4.1 in Array Strip (Affyme-trix, Santa Clara, CA), созданные на основе базы микроРНК mirBase v.20. Для постановки реакции гибридизации использовался набор GeneChip® Hybridization, Wash, and Stain Kit (Affymetrix, Santa Clara, CA) в течение 20 ч при температуре 48°С. По окончании гибридизации производилась отмывка микрочипов от несвязавшихся молекул и окраска реагентами набора GeneChip® Hybridization, Wash, and Stain Kit (Affymetrix, Santa Clara, CA). Далее осуществлялась детекция флуоресценции в ячейках микрочипа в станции детекции системы (Imaging station). Контроль качества гибридизации производился на всех этапах работы от постановки реакции полиаденилирования до детекции посредством использования контрольных образцов. Обработка данных флуоресценции производилась при помощи программы Expression Console software (Build 1.4.0.38) (Affymetrix, USA).
Статистическая обработка осуществлялась при помощи программы Transcriptome analysis Console 2.0. Различия в экспрессии микроРНК между меланоцитарными невусами, клетками меланомы и микроокружением меланомы признавались значимыми при р<0,05. При этом для последующего анализа были представлены только различия в уровнях экспрессии в 3 и более раз. Упорядочивание данных, имеющих значимые различия, производилось при помощи программы Transcrip-
Таблица 1
МикроРНК, имеющие различия в уровнях экспрессии в меланоме и меланоцитарных невусах
|
№ п/п |
микроРНК |
Кратность различий |
p |
Гены-мишени (по базам данных TargetScan, MiRanda, DIANA-miR-Path, экспериментально подтвержденные) |
|
1 |
hsa-miR-155-5p |
11,86 |
0,033 |
BACH1, JARID2, ARID2 |
|
2 |
hsa-miR-126-3p |
7,80 |
0,006 |
SPRED1, PLK2, IRS1 |
|
3 |
hsa-miR-21-5p |
7,33 |
0,017 |
CCL-1, PDCD-4, GATAD2B, ZNF367, RECK, SOX5, STAG2, YOD1, PELI1, MALT1, NFIB, GPR64, MEIS1, PBRM1, PLEKHA1, KBTBD7, RMND5A |
|
4 |
hsa-miR-4286 |
6,21 |
0,005 |
– |
|
5 |
hsa-miR-20b-5p |
4,29 |
0,043 |
CDKN1A, STAT3, CRIM1, HIF1A, BAMBI |
|
6 |
hsa-miR-363-3p |
4,2 |
0,025 |
HIPK3 (экспериментально не подтверждено) |
|
7 |
hsa-miR-6769b-5p |
3,83 |
0,003 |
– |
|
8 |
hsa-miR-150-5p |
3,70 |
0,046 |
MYB |
|
9 |
hsa-miR-6893-5p |
3,09 |
0,014 |
– |
|
10 |
hsa-miR-211-5p |
–24,77 |
0,000074 |
MAPRE2, SHC1 (экспериментально не подтверждено) |
|
11 |
hsa-miR-200c-3p |
–19,06 |
0,027 |
ZEB2, ZEB1, ZFPM2 |
|
12 |
hsa-miR-508-5p |
–13,71 |
0,0004 |
– |
|
13 |
hsa-miR-203a |
–12,47 |
0,012 |
– |
|
14 |
hsa-miR-513a-5p |
–12,00 |
0,004 |
– |
|
15 |
hsa-miR-506-3p |
–10,77 |
0,002 |
AMOTL1, ATP7A, TMEM134, WIPF1, |
Примечание: * – отрицательными числами отмечено, во сколько раз уровень экспрессии микроРНК в меланоме ниже, чем в невусах.
Таблица 2
МикроРНК, имеющие различия в уровнях экспрессии в меланоме и опухолевом микроокружении
|
№ п/п |
микроРНК |
Кратность различий |
p |
Гены-мишени |
|
1 |
hsa-miR-106a-5p |
11,46 |
0,040 |
UBE2W, SERF1A, SERF1B, PLEKHA3, MAP3K2, FGD4, MYCN, PTPN4, GPR137C, RRAGD (экспериментально не подтверждено) |
|
2 |
hsa-miR-223-3p |
5,63 |
0,037 |
RHOB, NFIA |
|
3 |
hsa-miR-221-3p |
4,95 |
0,035 |
ZEB2, CDKN1B |
|
4 |
hsa-miR-4745-5p |
4,68 |
0,010 |
– |
|
5 |
hsa-miR-4739 |
4,21 |
0,010 |
ZSWIM6, AFF4 |
|
6 |
hsa-miR-363-3p |
4,04 |
0,032 |
HIPK3 (экспериментально не подтверждено) |
|
7 |
hsa-miR-107 |
4,02 |
0,041 |
– |
|
8 |
hsa-miR-514b-5p |
3,32 |
0,045 |
– |
|
9 |
hsa-miR-6769b-5p |
3,18 |
0,009 |
– |
|
10 |
hsa-miR-3175 |
3,16 |
0,027 |
– |
|
11 |
hsa-miR-6805-5p |
3,15 |
0,018 |
– |
Таблица 3
МикроРНК, имеющие различия в уровнях экспрессии в меланоцитарных невусах и микроокружении меланомы
№ п/п микроРНК Кратность различий p Гены-мишени 1 hsa-miR-509-3p 36,14 0,005 – 2 hsa-miR-514b-5p 27,73 0,0006 – 3 hsa-miR-211-5p 19,08 0,0006 – 4 hsa-miR-513a-5p 15,7 0,00079 – 5 hsa-miR-6747-3p 10,12 0,014 – 6 hsa-miR-506-3p 9,93 0,013 – 7 hsa-miR-4800-3p 9,84 0,016 – 8 hsa-miR-508-5p 9,34 0,00000087 – 9 hsa-miR-4695-3p 8,13 0,022 – 10 hsa-miR-197-3p 7,86 0,015 – 11 hsa-miR-6761-3p 7,3 0,044 – 12 hsa-miR-449-3p 6,71 0,0056 – 13 hsa-miR-4467 6,64 0,020 – 14 hsa-miR-6754-3p 6,51 0,033 – 15 hsa-miR-3591-3p 6,21 0,0078 – 16 hsa-miR-6794-3p 6,02 0,029 – 17 hsa-miR-4326 5,96 0,019 – 18 hsa-miR-3178 5,93 0,017 – 19 hsa-miR-6741-3p 5,86 0,015 – 20 hsa-miR-149-5p 5,79 0,019 ZNF726 tome analysis Console 2.0 методом иерархической кластеризации на тепловой карте. Базовые характеристики измененных микроРНК определялись с помощью базы данных Mirbase ( от 12.02.2015).
Результаты исследования и обсуждение
По результатам микрочипирования было выявлено 9 микроРНК, уровни которых были повышены в 3 и более раз в клетках меланомы по сравнению с меланоцитарными невусами; экспрессия 83 разновидностей микроРНК была снижена при меланоме по сравнению с невусами. 11 микроРНК отличались повышенной экспрессией в клетках меланомы по сравнению с опухолевым окружением, а также
94 микроРНК были выше по уровню экспрессии в 3 и более раз в ткани меланоцитарных невусов по сравнению с микроокружением меланомы; 5 микроРНК характеризовались сниженным уровнем экспрессии в меланоцитарных невусах по сравнению с микроокружением меланомы (табл. 1, 2, 3).
Таким образом, можно резюмировать, что изменения в экспрессии микроРНК превалируют в клетках меланомы по сравнению с доброкачественными меланоцитарными новообразованиями; минимальны изменения между опухолевым окружением меланомы и образцами меланоцитарных невусов. Подобные результаты также соотносятся с анализом экспрессионного профиля микроРНК
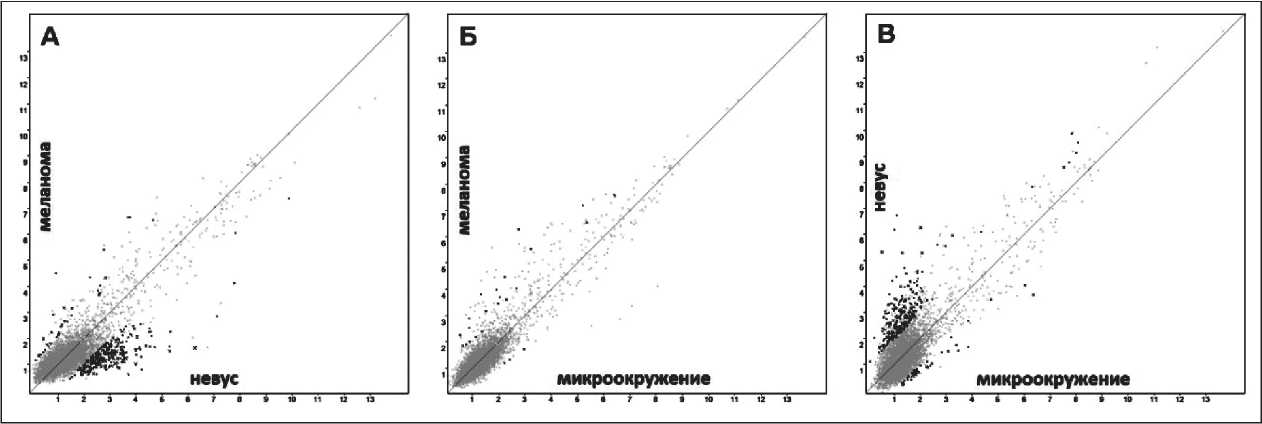
Рис. 1. Диаграмма разброса уровней экспрессии микроРНК:
А – в меланоме в сравнении с меланоцитарными невусами; Б – в меланоме в сравнении с микроокружением меланомы;
В – в невусах в сравнении с микроокружением меланомы. Серые точки, расположенные близко к центральной диагонали, указывают на уровни экспрессии микроРНК, не имеющие достоверных различий между сравниваемыми группами. Темные точки выше диагональной линии соответствуют микроРНК, уровень экспрессии которых в группах, отмеченных на оси ординат, в 2 и более раза выше такового в группах, отмеченных по оси абсцисс (p<0,05). Темные точки ниже диагональной линии соответствуют микроРНК, уровень экспрессии которых в группах, отмеченных на оси ординат, в 2 и более раза ниже такового в группах, отмеченных по оси абсцисс (p<0,05)
с помощью иерархической кластеризации, что отражено на тепловой карте (рис. 1).
Несомненно, стоит отметить, что опухолевое микроокружение имело значительно меньше различий по профилю экспрессии микроРНК с меланомой, чем с меланоцитарными невусами (рис. 2). Это может являться отражением феномена «перепрограммирования» опухолевого окружения, и синтеза опухоль-ассоциированными клетками (макрофагами, фибробластами, лимфоцитами) факторов, аналогичных продуцируемым опухолевыми клетками [16]. МикроРНК могут играть значимую регуляторную роль в этом процессе.
С помощью баз данных TargetScan, MiRanda, DIANA-miR-Path в дальнейшем был выполнен анализ генов-мишеней для микроРНК с максимально измененными уровнями. При анализе генов-мишеней микроРНК, уровни которых отличались в исследуемых группах, не все гены-мишени являлись идентифицированными (табл. 1, 2, 3).

Рис. 2. Тепловая карта, представляющая экспрессию микроРНК в меланоцитарных невусах (1), меланоме (2), микроокружении меланомы (3). Отчетливо прослеживается разделение в отдельные группы по профилю микроРНК меланоцитарных невусов и меланомы
Как показали исследования, генами-мишенями miR-21 являются, в том числе, опухолевые супрессоры (PDCD-4, RECK), регулятор пролиферации клеток STAG2, а также другие, ассоциированные с канцерогенезом, гены (MALT1, MEIS1, PELI1). В целом, можно отметить, что результаты нашего исследования соотносятся с литературными данными в отношении miR-21.
Ранее в исследованиях также указывалось на повышение уровней miR-155 при меланоме кожи. С помощью гибридизации выявлены особенности локализации микроРНК по отношению к опухоли – преимущественно на периферии опухолевых комплексов [7]. Повышение уровня miR-155 регистрируется при других видах злокачественных новообразований: немелкоклеточном раке легкого, глиобластоме [4, 15]. И если для микроРНК, уровни которых изменены в клетках меланомы, по сравнению с показателями в клетках доброкачественных меланоцитарных новообразований, характерными генами-мишенями являются последние, преимущественно кодирующие регуляторы клеточного цикла, апоптоза, подвижности и пр., то в отношении опухолевого микроокружения и клеток меланомы мишенями являются гены, кодирующие белки, обеспечивающие базовые функции клетки: транспорт аминокислот, связывание с ДНК, а также транскрипционные факторы.
Наименее изученными на сегодняшний день являются микроРНК, которые по результатам
Список литературы Особенности экспрессионного профиля микроРНК в клетках меланомы и опухолевом микроокружении
- Злокачественные новообразования в России в 2013 году (заболеваемость и смертность)/Под ред. А.Д. Каприна, В.В. Старинского, Г.В. Петровой. М., 2015. 250 с
- Chan S.H., Wu C.W., Li A.F., Chi C.W., Lin W.C. miR-21 microRNA expression in human gastric carcinomas and its clinical association//Anticancer Res. 2008. Vol. 28 (2A). P. 907-911
- Chou J., Werb Z. MicroRNAs play a big role in regulating ovarian cancer-associated fibroblasts and the tumor microenvironment//Cancer Discovery. 2012. Vol. 2 (12). P. 1078-1080. CD-12-0465 DOI: 10.1158/2159-8290
- Costa P.M., Cardoso A.L., Nobrega C., Pereira de Almeida L.F., Bruce J.N., Canoll P., Pedroso de Lima M.C. MicroRNA-21 silencing enhances the cytotoxic effect of the antiangiogenic drug sunitinib in glioblastoma//Hum. Mol. Genet. 2013. Vol. 22 (5). P. 904-918 DOI: 10.1093/hmg/dds496
- Dimmeler S., Nicotera P. MicroRNAs in age-related diseases//EMBO Mol. Med. 2013. Vol. 5 (2). P. 180-190 DOI: 10.1002/emmm.201201986
- Flowers E., Aouizerat B.E. MicroRNAassociated with dyslipidemia and coronary disease in humans//Physiol. Genomics. 2013. Vol. 45 (24). P. 1199-1205 DOI: 10.1152/physiolgenomics.00106.2013
- Grignol V., Fairchild E.T., Zimmerer J.M., Lesinski G.B., Walker M.J., Magro C.M., Kacher J.E., Karpa V.I., Clark J., Nuovo G., Lehman A., Volinia S., Agnese D.M., Croce C.M., Carson W.E 3rd. miR-21 and miR-155 are associated with mitotic activity and lesion depth of borderline melanocytic lesions//Br. J. Cancer. 2011. Vol. 105 (7). P. 1023-1029 DOI: 10.1038/bjc.2011.288
- Pimentel F., Bonilla P., Ravishankar Y.G., Contag A., Gopal N., LaCour S., Lee T., Niemz A. Technology in MicroRNA Profiling: Circulating MicroRNAs as Noninvasive Cancer Biomarkers in Breast Cancer//J. Lab. Autom. 2014. pii: 2211068214561788
- Su W., Hopkins S., Nesser N.K., Sopher B., Silvestroni A., Ammanuel S., Jayadev S., Möller T., Weinstein J., Garden G.A. The p53 transcription factor modulates microglia behavior through microRNA-dependent regulation of c-Maf//J. Immunol. 2014. Vol. 192 (1). P. 358-366 DOI: 10.4049/jimmunol.1301397
- Umansky V., Sevko A. Overcoming immunosuppression in the melanoma microenvironment induced by chronic inflammation//Cancer Immunol Immunother. 2012. Vol. 61 (2). P. 275-282 DOI: 10.1007/s00262-011-1164-6
- Van Rooij E., Kauppinen S. Development of microRNA therapeutics is coming of age//EMBO Mol. Med. 2014. Vol. 6 (7). P. 851-864 DOI: 10.15252/emmm.201100899
- Yamamichi N., Shimomura R., Inada K., Sakurai K., Haraguchi T., Ozaki Y., Fujita S., Mizutani T., Furukawa C., Fujishiro M., Ichinose M., Shiogama K., Tsutsumi Y., Omata M., Iba H. Locked nucleic acid in situ hybridization analysis of miR-21 expression during colorectal cancer development//Clin. Cancer Res. 2009. Vol. 15 (12). P. 4009-4016 DOI: 10.1158/1078-0432.CCR-08-3257
- Yan L.X., Huang X.F., Shao Q., Huang M.Y., Deng L., Wu Q.L., Zeng Y.X., Shao J.Y. MicroRNA miR-21 overexpression in human breast cancer is associated with advanced clinical stage, lymph node metastasis and patient poor prognosis//RNA. 2008. Vol. 14 (11). P. 2348-2360 DOI: 10.1261/rna.1034808
- Yang C.H., Yue J., Pfeffer S.R., Handorf C.R., Pfeffer L.M. MicroRNAmiR-21 regulates the metastatic behavior of B16 melanoma cells//J. Biol. Chem. 2011. Vol. 286 (45). P. 39172-39178. doi: 10.1074/jbc. M111.285098
- Yang M., Shen H., Qiu C., Ni Y., Wang L., Dong W., Liao Y., Du J.High expression of miR-21 and miR-155 predicts recurrence and unfavourable survival in non-small cell lung cancer//Eur. J. Cancer. 2013. Vol. 49 (3). P. 604-615 DOI: 10.1016/j.ejca.2012.09.031
- Yang P., Markowitz G.J., Wang X.F. The hepatitis B virus-associated tumor microenvironment in hepatocellular carcinoma//Natl. Sci. Rev. 2014. Vol. 1 (3). P. 396-412
- Zigler M., Kamiya T., Brantley E.C., Villares G.J., Bar-Eli M. PAR-1 and thrombin: the ties that bind the microenvironment to melanoma metastasis//Cancer Res. 2011. Vol. 71 (21). P. 6561-6566 DOI: 10.1158/0008-5472.CAN-11-1432


