Особенности выделения белков для анализа протеома растений гороха Pisum sativum L. при симбиозе с клубеньковыми бактериями
Автор: Кириенко А.Н., Леппянен И.В., Грибченко Э.С., Долгих Е.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Симбиогенетика
Статья в выпуске: 5 т.52, 2017 года.
Бесплатный доступ
Горох посевной Pisum sativum L. - удобная модель для изучения молекулярно-генети-ческих механизмов становления азотфиксирующего симбиоза с клубеньковыми бактериями, поскольку для этого вида создана представительная коллекция мутантов с нарушением процесса развития симбиоза на разных стадиях. Сравнительный анализ протеомов у исходных сортов, линий гороха и мутантов может быть удобным подходом при выявлении и анализе регуляторов, контролирующих процесс образования азотфиксирующих клубеньков. Однако, как показывает обзор данных литературы, дифференциальные изменения протеома корней гороха при симбиозе почти не изучались. Подготовка проб - ключевой этап в протеомных исследованиях. От эффективности выделения белков из исходного материала и очистки от небелковых примесей зависят качество получаемых после двумерного (2-D) электрофореза гелей и возможность их анализа. К методам выделения белков для протеомных исследований предъявляются особые требования по минимизации числа этапов работы, что необходимо для сохранности белков, а также по эффективному удалению примесей, которые могут негативно влиять на качество разделения и последующую оценку изменений в синтезе белков. Наша работа была посвящена поиску наиболее эффективного способа выделения общего пула белков из корней гороха посевного ( P. sativum L.), инокулированного ризобиями. Для этого мы сравнили три метода выделения белка: стандартный, предлагаемый компанией «Bio-Rad Laboratories» (США); метод, основанный на использовании фенола и ацетата аммония; метод с применением трихлоруксусной кислоты. В работе использовали растения гороха сорта Frisson, для инокуляции применяли штамм Rhizobium leguminosarum bv. viciae CIAM1026. После выделения белков из корней проростков гороха, зараженных ризобиями (1 сут после инокуляции), было показано, что лучший результат достигается при использовании фенола с последующим осаждением белков ацетатом аммония. Гели анализировали на наличие примесей, затрудняющих поиск различающихся белковых продуктов, эффективность выделения общего белка и видимые следы деградации белков. Для выделенных белков был проведен дифференциальный 2-D электрофорез с использованием флуоресцентных меток цианин-2 (Cy2) и цианин-5 (Cy5), основанный на изоэлектрическом фокусировании белков с помощью стрипов с диапазоном рН 3-10 и последующим разделением в полиакриламидном геле (ПААГ). Установлено, что при выделении белков с помощью фенола и ацетата аммония удалось получить достаточно представительные протеомы корней проростков гороха. Дифференциальный 2-D электрофорез позволил выявить различия между контрольным вариантом (неинокулированные корни) и вариантом, полученным после инокуляции ризобиями (инокулированные корни). Разработанный метод может быть использован в дальнейшем для протеомных исследований на растениях гороха.
Горох посевной, бобово-ризобиальный симбиоз, протеомный анализ, дифференциальный 2-d электрофорез
Короткий адрес: https://sciup.org/142214078
IDR: 142214078 | УДК: 633.358:631.461.5:57.052:577.112 | DOI: 10.15389/agrobiology.2017.5.1012rus
Текст научной статьи Особенности выделения белков для анализа протеома растений гороха Pisum sativum L. при симбиозе с клубеньковыми бактериями
При формировании симбиоза между бобовыми растениями и клубеньковыми бактериями (ризобиями) в растении происходят существенные изменения метаболических процессов, гормонального статуса, структурные перестройки, связанные с реорганизацией цитоскелета (1). Эти изменения направлены на формирование на корнях новых органов — симбиотических клубеньков, в которых осуществляется азотфиксация. В последние годы с распространением методов комплексного анализа, которые позволяют изучать изменения транскрипционной активности генов растений под влиянием разнообразных факторов (транскриптомика), появилась возможность выявлять регуляторы, вовлеченные в контроль развития симбиоза. Однако такие исследования проводятся главным образом на модельных видах Medicago truncatula Gaertn. и Lotus japonicus L., геномы которых расшифрованы благодаря относительно небольшим размерам
* Исследования выполнены при поддержке Российского научного фонда (грант ¹ 16-16-10043).
( ∼ 470-500 млн п.н.) (2, 3). Показано, что в процессе развития симбиоза в корнях бобовых растений изменяется транскрипционная активность нескольких тысяч генов (4-6). При этом функциональное значение таких изменений в экспрессии генов остается достаточно далеким от понимания.
Горох ( Pisum sativum L.) обладает очень большим геномом (около 4300 млн п.н.), который пока не расшифрован. Исследования по выявлению в нем генов, дифференциально экспрессирующихся в ответ на инокуляцию, представляют достаточно сложную задачу. При этом уникальные коллекции мутантов по sym генам, контролирующим развитие симбиоза со стороны растения, определяют интерес к использованию этого объекта. Протеомика служит комплексным походом для изучения изменений в составе белков под влиянием биотических и абиотических факторов. Она позволяет проводить сравнительные исследования на горохе и его мутантах и выявлять не известные ранее регуляторы, необходимые для формирования симбиоза. Однако протеомные исследования белков гороха, изменяющихся в ответ на обработку ризобиями, почти не проводились. Было изучено перибактероидное пространство симбиосом при развитии симбиоза гороха с азотфиксирующими бактериями (7). Проанализированы протеомы гороха при антагонистических взаимодействиях с заразихой городчатой ( Orobanche crenata ) и микосфереллой ( Mycosphaerella pinode ) (8, 9), при развитии вегетативных органов и семян (10-12), формировании устойчивости к температурному стрессу (13). Исследовано влияние салициловой и жасмоновой кислот на белковый состав корней и листьев гороха (14-16).
Подготовка проб — ключевой этап в протеомных исследованиях с использованием 2-D электрофореза (17). Она особенно затруднена в работе с растениями, ткани которых богаты протеазами и веществами, препятствующими протеомному анализу: полисахаридами, липидами и фенольными соединениями (18). Такие соединения мешают разделению белков и их анализу из-за появления на гелях горизонтальных и вертикальных полос, размазанности, снижения числа ярких пятен. Кроме того, ткани растений содержат меньше белка по сравнению с тканями животных и микроорганизмами, поэтому эффективное извлечение белков важна для получения успешных результатов 2-D электрофореза (17, 18). Протеомика позволяет качественно и количественно сравнивать белки в различных пробах, поэтому потери белка в процессе анализа имеют решающее значение. При экстракции белков из пробы необходимо сохранить их качество и количество, для чего выбранный метод должен содержать как можно меньше шагов, чтобы минимизировать потери (19, 20).
В настоящей работе мы впервые сравнили эффективность трех методов выделения общего белка из корней гороха посевного. Метод выделения белков с использованием фенола и ацетата аммония оказался наиболее предпочтительным для проведения дифференциального 2-D электрофореза.
Целью работы был подбор оптимального метода выделения общего пула белков из корней гороха посевного, инокулированных ризобиями, для получения качественного результата при 2-D электрофорезе.
Методика. Семена гороха (Pisum sativum L., сорт Frisson) стерилизовали 5 мин в серной кислоте, 3 раза промывали дистиллированной водой и проращивали на 1 % водном агаре в темноте в течение 4 сут. Проростки переносили в горшки со стерильным вермикулитом, пропитанным жидкой средой Jensen (21). Инокуляцию проводили штаммом Rhizobium legumi-nosarum bv. viciae CIAM1026 (OD600 ≈ 0,5) по 1 мл на проросток. В качестве контроля использовали неинокулированные проростки. Растения выращи- вали в фитотроне Sanyo MLR-351H (Япония) при 21 °C, 16-часовом светом дне, 60 % влажности. Для анализа отбирали фрагменты главных корней, соответствующие зоне восприимчивости к ризобиальной инфекции, через 24 ч после инокуляции ризобиями. Образцы замораживали в жидком азоте и хранили при -80 °С. Для выделения белков корни (по 100 мг для каждой методики) растирали в жидком азоте в охлажденных ступках.
Выделение белков по стандартной методике компании «Bio-Rad Laboratories» (США) предполагало гомогенизацию материала в буфере для изоэлектрического фокусирования (ИЭФ). Материал переносили в эппендорфы и добавляли буфер для ИЭФ, содержащий 25 мM Трис-HCl (pH 8,0), 9 М мочевину, 4 % 3-(3-холамидо-1-пропилдиметиламмонио)-1-пропансульфонат (ЧАПС), 50 мМ дитиотреитол (ДТТ), 0,2 % амфолиты («Bio-Rad Laboratories», США). Смесь инкубировали 20 мин на льду, затем центрифугировали 15 мин при 12000 g, +4 ° С. Надосадочную жидкость, содержащую белки (150 мкл), наносили на стрипы для регидратации.
Для выделения белков с помощью фенола и ацетата аммония к материалу добавляли охлажденный до +4 ° С буфер для экстракции, содержащий 0,1 M Трис-HCl (pH 8,0), 30 % сахарозу, 10 мM ДТТ, 2 % додецилсульфат натрия (ДСН), коктейль протеазных ингибиторов («Sigma», США). Смесь центрифугировали («Hettich 320R», Германия) 15 мин при 12000 g и +4 ° С. Надосадочную жидкость смешивали в соотношении 1:1 с фенолом (pH 8,0) («Invitrogen», США), перемешивали на шейкере («Vortex Genius», Германия) 30 с и центрифугировали при 10000 g в течение 5 мин при +4 ° С. Верхнюю фазу переносили в новый эппендорф, белки осаждали 5 объемами охлажденного 100 мМ ацетата аммония в метаноле 30 мин при - 20 ° С, затем центрифугировали в течение 5 мин при 10000 g. Осадок промывали 2 раза 100 мМ ацетатом аммония в метаноле и дважды — 80 % ацетоном. Осадки белков сушили на воздухе и растворяли в буфере для изофокусировки, содержащем 25 мM Трис-HCl (pH 8,0), 9 М мочевину, 4 % CHAPS, 50 мМ ДТТ, 0,2 % амфолиты («Bio-Rad Laboratories», США).
Чтобы выделить общий белок с помощью трихлоруксусной кислоты (ТХУ), к растертым корням добавляли ее 10 % раствор и 0,07 % β -меркаптоэтанол, приготовленный на ацетоне. Полученную смесь обрабатывали ультразвуком 3 раза по 20 с при амплитуде 10 мкм (Soniprep 150 Plus, «MSE», Великобритания). Суспензию инкубировали 1 ч при - 20 ° С, перемешивая каждые 15 мин на шейкере, а затем центрифугировали 20 мин при 9000 g и +4 ° С. Полученный осадок промывали дважды раствором 0,07 % β -меркаптоэтанола в ацетоне, затем сушили в вакуумном испарителе (Concentrator Plus, «Eppendorf», США), растворяли в буфере, содержащем 25 мM Трис-HCl (pH 8,0), 9 М мочевину, 4 % CHAPS, 50 мМ ДТТ, 0,2 % амфолиты («Bio-Rad Laboratories», США), инкубировали 20 мин на льду и центрифугировали 10 мин при 12000g и +4 ° С. Полученную надосадочную жидкость, содержащую белки (150 мкл), наносили на стрипы для регидратации.
Изоэлектрическое фокусирование (ИЭФ) проводили с помощью стрипов длиной 7 см с градиентом pH 3-10, используя прибор Protean IEF Cell («Bio-Rad Laboratories», США). Предварительно выполняли регидратацию стрипов и загрузку полученных проб белка (по 150 мкл на стрип) в течение 12-14 ч. ИЭФ осуществляли при температуре +20 ° С, обессоливали образцы при 250 В в течение 15 мин, после этого линейно повышали напряжение до 4000 В в течение 2 ч, далее проводили собственно ИЭФ (до 10000 В•ч при ограничении по силе тока 35 мA на гель). После процедуры ИЭФ стрипы замораживали и хранили при - 80 ° С либо сразу вы-1014
полняли разделение во втором направлении.
Перед электрофорезом белков в полиакриламидном геле (ПААГ) инкубировали стрипы в буфере, содержащем 0,375 М Трис-HCl (рН 8,8), 6 М мочевину, 2 % ДСН, 2 % ДТТ, 20 % глицерин, в течение 10 мин, затем алкилирование в буфере с 0,375 М Трис-HCl (рН 8,8), 6 М мочевиной, 2 % ДСН, 20 % глицерин, 2,5 % йодацетамид (10 мин). Стрипы укладывали на концентрирующий гель, сверху наслаивали расплавленную 0,5 % агарозу (25 мM Трис-HCl, 192 мM глицин, 0,1 % ДСН) с добавлением бромфенолового синего (БФС), чтобы контролировать разделение. Электрофорез проводили в 15 % ПААГ с использованием 4 % концентрирующего геля, устанавливали 35 мА на гель до полного выхода БФС из геля в трис-глициновом буфере (25 мМ Трис-HCl, pH 8,3, 192 мМ глицин, 0,1 % ДСН). После завершения фореза гели промывали деионизированной водой и окрашивали в растворе Simple Blue («Invitrogen», США) по протоколу производителя. Гели, отмытые от краски, фотографировали на приборе G:BOX-CHEMI-XX9 («Syngene», Великобритания).
Для проведения дифференциального двумерного электрофореза (2D fluorescence difference gel electrophoresis, DIGE) общий белок, полученный после выделения с помощью фенола и ацетата аммония, растворяли в 200 мкл буфера для ИЭФ без ДТТ и амфолитов. Затем пробы белка конъюгировали с флуорофорами Су2 и (или) Су5 («Lumiprobe», Россия) в разных комбинациях. По 50 мг белкового экстракта инкубировали с 400 пМ красителя, растворенного в диметилформамиде, в течение 30 мин на льду в темноте. Реакцию мечения останавливали 10 мМ L-лизином («Sigma-Aldrich», США) в течение 10 мин на льду. Контрольные и опытные пробы, меченные разными красителями, смешивали, добавляли ДТТ и амфолиты и использовали для регидратации стрипов. После разделения белков гели визуализировали с помощью лазерного сканера Typhoon FLA 9500 («GE Healthcare», Германия). Для анализа фотографий гелей использовали программу ImageJ .
Результаты. В последнее десятилетие протеомика зарекомендовала себя как метод, который позволяет достаточно эффективно оценить изменения, происходящие в растении под влиянием различных биотических, абиотических и антропогенных факторов. Успешность метода зависит от подготовки проб и получения исходного экстракта белков (22). Это особенно важно для дифференциальной протеомики, которая связана с изучением различий между контрольными пробами и пробами, полученными после обработок. Выбор оптимального метода для выделения общего пула белков — ключевой фактор для получения надежных экспериментальных результатов (23). Биохимические свойства белков, такие как растворимость, суммарный заряд, а также особенности локализации (например, мембранные белки) и низкое количество белка в исходном материале, могут негативно сказаться на изучении их полного спектра. Кроме того, растительные клетки содержат значительные количества небелковых веществ — полисахаридов, липидов и органических кислот (24), а клеточная стенка состоит из большого количества клетчатки и пектина. Эти вещества оказывают существенное влияние на качество белковых экстрактов и, следовательно, на результаты 2D-электрофореза (25, 26). Оптимальный метод подготовки проб необходим для эффективного удаления небелковых веществ из образца.
Нами были опробованы три метода выделения общего белка из корней гороха после 24 ч инкубации с ризобиями. Во всех случаях разрушение ткани производилось в жидком азоте, что позволяло максимально эффективно гомогенизировать материал и разрушить клеточную стенку.
После дополнительных отмывок (в случае методов с использованием фенола и ацетата аммония или с ТХУ) все пробы были растворены в буфере, содержащем мочевину, детергент CHAPS, восстанавливающий агент ДТТ и коктейль протеазных ингибиторов. Эти вещества позволяют избежать деградации, модификации, потери и преципитации белков (27).
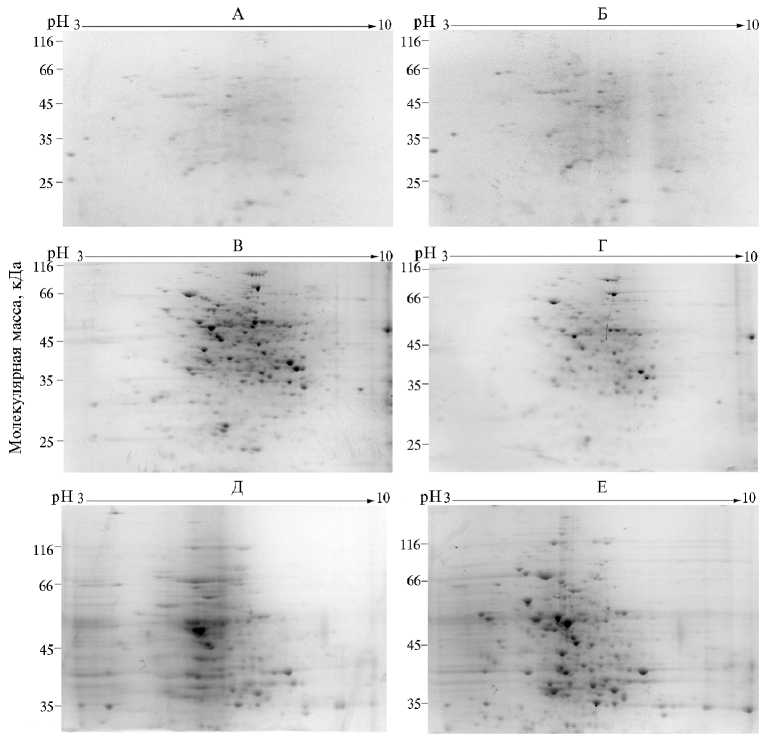
Рис. 1. 2-D электрофореграммы белков, выделенных из корней гороха ( Pisum sativum L.) сорта Frisson по стандартной методике компании «Bio-Rad Laboratories» (США) (А, Б), с использованием фенола и ацетата аммония (В, Г), с помощью трихлоруксусной кислоты (Д, Е): слева — контрольные пробы, справа — пробы из корней, инокулированных ризобиями (1 сут после инокуляции). Использованы стрипы с pH 3-10. Разделение во втором направлении проводили в 12 % ПААГ.
Получение экстракта белков гороха с использованием стандартной методики оказалось наименее подходящим для анализа. 2D-электрофорез проб обнаружил небольшое количество выделенного белка, в контрольной и обработанной пробах (рис. 1, А, Б). На геле также были видны следы загрязнений — полосы и фоновые шумы, что мешало поиску различий в спектре разделенных белков.
Выделение белков с помощью ТХУ оказалось эффективнее. Нам удалось экстрагировать большее количество белков по сравнению со стандартным методом, о чем судили по появлению более насыщенного спектра пятен на ПААГ. Однако 2-D электрофорез проб, полученных с помощью ТХУ (см. рис. 1, Д, Е), показал невозможность удаления фоновых шумов (горизонтальные полосы), появление которых связывают с остат-1016
ками нуклеиновых кислот, полисахаридами и фенольными соединениями (28, 29). Полисахариды также были причиной агрегации белков в образце. Такие белковые комплексы могут блокировать поры в ПААГ и делают невозможным прохождение пептидов в геле и их фокусировку в нужной области (30). По данным литературы, к недостаткам этого метода следует отнести в целом небольшое количество экстрагируемых белков, что было отмечено ранее в опытах по получению белкового экстракта из клеток сахарной свеклы Beta vulgaris L., кактуса Mammillaria gracilis Pfeiff. и молодило кровельного Sempervivum tectorum L. Такой недостаток можно исправить, используя большее количество исходного материала, но это, в свою очередь, увеличит количество загрязняющих агентов.
Метод с использованием фенола и ацетата аммония был впервые предложен W.J. Hurkman и C.K. Tanaka для протеомного анализа (19). Фенольную экстракцию применяли для выделения белков из картофеля, рапса, яблок, банановых и оливковых листьев, помидоров, люцерны, авокадо и бананов (17, 18, 24, 31-33). При изучении растительно-микробных взаимодействий он был использован для анализа протеома Lotus japonicus на поздних стадиях развития симбиоза с Mesorhizobium loti (34, 35). Метод занимает больше времени по сравнению с другими, однако в нашем случае он оказался наиболее результативным для получения качественных 2-D электрофореграмм. На геле отсутствовали ярко выраженные горизонтальные полосы, характерные для полисахаридного загрязнения (см. рис. 1, В, Г). Высокое качество геля достигалось за счет большого количества выделяемого белка, что делает возможным проведения более надежного сравнительного анализа контрольной пробы и пробы после обработки. К достоинствам метода можно отнести и эффективное извлечение из образца мембранных белков (19).
На основании полученных результатов этот метод был выбран для выделения белков при проведении дифференциального 2-D электрофореза (DIGE). Полученные белковые пробы инкубировали с флуоресцентными красителями, проводили ИЭФ и разделение в ПААГ (рис. 2).

Рис. 2. 2-D дифференциальные электрофореграммы белков, выделенных из корней гороха ( Pisum sativum L.) сорта Frisson с помощью фенола и ацетата аммония: А — контрольная проба (Cy2, синяя флуоресцентная метка) и проба после обработки ризобиями (Cy5, желтая флуоресцентная метка); Б — контрольная проба (Cy5, желтая флуоресцентная метка) и проба после обработки ризобиями (Cy2, синяя флуоресцентная метка). Использованы стрипы с pH 3-10. Разделение во втором направлении проводили в 12 % ПААГ. Белые пятна указывают на отсутствие, цветные — на наличие разницы в уровне синтеза белков. Стрелками отмечены белки, синтез которых усиливается при обработке.
Таким образом, метод выделения общего пула белков корней гороха, инокулированных ризобиями, с помощью фенола и ацетата аммония оказался наиболее эффективным и качественным при подготовке проб для проведения как стандартного электрофореза в ПААГ с последующей окраской Simple Blue, так и для дифференциального 2-D электрофореза. Он позволил выделить достаточное количество белка и избавиться от примесей, мешающих качественному электрофорезу, провести эффективное связывание с метками и получить картину высокой степени разрешения, которая необходима для определения разницы в спектре синтезируемых белков. В дальнейшем метод будет использован для изучения различий в спектре синтезируемых белков на последовательных этапах развития симбиоза между горохом посевным и ризобиями Rhizobium leguminosarum bv. viciae CIAM1026.
Список литературы Особенности выделения белков для анализа протеома растений гороха Pisum sativum L. при симбиозе с клубеньковыми бактериями
- Genre A., Russo G. Does a common pathway transduce symbiotic signals in plant-microbe interactions? Front Plant Sci., 2016, 7: 96 ( ) DOI: 10.3389/fpls.2016.00096
- Young J., Kambere M., Trask B., Lane R. Divergent V1R repertoires in five species: Amplification in rodents decimation in primates, and a surprisingly small repertoire in dogs. Genome Res., 2005, 15: 231-240 ( ) DOI: 10.1101/gr.3339905
- Cook D., Dreyer D., Bonnet D., Howell M., Nony E., VandenBosch K. Transient induction of a peroxidase gene in Medicago truncatula precedes infection by Rhizobium meliloti. Plant Cell, 1995, 7: 43-55 ( ) DOI: 10.1105/tpc.7.1.43
- Breakspear A., Liu C., Roy S., Stacey N., Rogers C., Trick M., Morieri G., Mysore K.S., Wen J., Oldroyd G.E., Downie J.A., Murray J.D. The root hair «infectome» of Medicago truncatula uncovers changes in cell cycle genes and reveals a requirement for Auxin signaling in rhizobial infection. Plant Cell, 2014, 26(12): 4680-4701 ( ) DOI: 10.1105/tpc.114.133496
- van Zeijl A., Op den Camp R.H., Deinum E.E., Charnikhova T., Franssen H., Op den Camp H.J., Bouwmeester H., Kohlen W., Bisseling T., Geurts R. Rhizobium lipo-chitooligosaccharide signaling triggers accumulation of cytokinins in Medicago truncatula roots. Mol. Plant., 2015, 8: 1213-1226 ( ) DOI: 10.1016/j.molp.2015.03.010
- Larrainzar E., Riely B.K., Kim S.C., Carrasquilla-Garcia N., Yu H.J., Hwang H.J., Oh M., Kim G.B., Surendrarao A.K., Chasman D., Siahpirani A.F., Penmetsa R.V., Lee G.S., Kim N., Roy S., Mun J.H., Cook D.R. Deep sequencing of the Medicago truncatula root transcriptome reveals a massive and early interaction between nodulation factor and ethylene signals. Plant Physiol., 2015, 169: 233-265 ( ) DOI: 10.1104/pp.15.00350
- Saalbach G., Erik P., Wienkoop S. Characterisation by Proteomics of peribacteroid space and peribacteroid membrane preparations from pea (Pisum sativum) symbiosomes. Proteomics, 2002, 2: 325-337 (doi: 10.1002/1615-9861(200203)2:33.0.CO;2-W).
- Castillejo M.Á., Curto M., Fondevilla S., Rubiales D., Jorrín J.V. Two-dimensional electrophoresis based proteomic analysis of the pea (Pisum sativum) in response to Mycosphaerella pinodes. J. Agric. Food Chem., 2010, 58(24): 12822-1283 ( ) DOI: 10.1021/jf1036917
- Castillejo M.Á., Fernández-Aparicio M., Rubiales D. Proteomic analysis by two-dimensional differential in gel electrophoresis (2D DIGE) of the early response of Pisum sativum to Orobanche crenata. J. Exp. Bot., 2011, 63(1): 107-119 ( ) DOI: 10.1093/jxb/err246
- Schiltz S., Gallardo K., Huart M., Negroni L., Sommerer N., Burstin J. Proteome reference maps of vegetative tissues in pea. An investigation of nitrogen mobilization from leaves during seed filling. Plant Physiol., 2004, 135: 2241-2260 ( ) DOI: 10.1104/pp.104.041947
- Bourgeois M., Jacquin F., Savois V., Sommerer N., Labas V., Henry C., Burstin J. Dissecting the proteome of pea mature seeds reveals the phenotypic plasticity of seed protein composition. Proteomics, 2009, 9: 254-271 ( ) DOI: 10.1002/pmic.200700903
- Bourgeois M., Jacquin F., Cassecuelle F., Savois V., Belghazi M., Aubert G., Quillien L., Huart M., Marget P., Burstin J. A PQL (protein quantity loci) analysis of mature pea seed proteins identifies identifies loci determining seed protein composition. Proteomics, 2011, 9: 1581-1594 ( ) DOI: 10.1002/pmic.201000687
- Dumont E., Bahrman N., Goulas E., Valot B., Sellier H., Hilbert J.L., Lejeune-Hénaut I., Delbreil B. A proteomic approach to decipher chilling response from cold acclimation in pea (Pisum sativum L.). Plant Sci., 2011, 180: 86-98 ( ) DOI: 10.1016/j.plantsci.2010.09.006
- Тарчевский И.А., Яковлева В.Г., Егорова А.М. Протеомный анализ салицилат-индуцированных белков листьев гороха (Pisum sativum L.). Биохимия, 2010, 75(5): 689-697.
- Тарчевский И.А., Егорова А.М. Протеомный анализ влияния циклогексимида на корни гороха. Физиология растений, 2015, 62(6): 883-895.
- Yakovleva V.G., Egorova A.M., Tarchevsky I.A. Proteomic analysis of the effect of methyl jasmonate on pea seedling roots. Doklady Biochemistry and Biophysics, 2013, 449: 90-93 ( ) DOI: 10.1134/S1607672913020105
- Saravanan R.S., Rose J.K.C. A critical evaluation of sample extraction techniques for enhanced proteomic analysis of recalcitrant plant tissues. Proteomics, 2004, 4: 2522-2532 ( ) DOI: 10.1002/pmic.200300789
- Carpentier S.C., Witters E., Laukens K., Deckers R.S., Panis B. Preparation of protein extracts from recalcitrant plant tissues: An evaluation of different methods for two-dimensional gel electrophoresis analysis. Proteomics, 2005, 5: 2497-2507 ( ) DOI: 10.1002/pmic.200401222
- Hurkman W.J., Tanaka C.K. Solubilization of plant membrane proteins for analysis by two-dimensional gel electrophoresis. Plant Physiol., 1986, 81: 802-806 ( ) DOI: 10.1104/pp.81.3.802
- Westermeier R., Naven T. Proteomics in practice: a laboratory manual of proteome analysis. Wiley-VCH Verlag-GmbH, Weinheim, 2002: 342 ( ) DOI: 10.1002/3527600175
- van Brussel A.A.N., Tak T., Wetselaar A., Pees E., Wijffelman C.A. Small Leguminosae as test plants for nodulation of Rhizobium leguminosarum and other rhizobia and agrobacteria harbouring a leguminosarum sym-plasmid. Plant Sci. Lett., 1982, 27: 317-325 ( ) DOI: 10.1016/0304-4211(82)90134-1
- Isaacson T., Damasceno C.M.B., Saravanan R.S., He Y., Catala C. Sample extraction techniques for enhanced proteomic analysis of plant tissues. Nature Protocols, 2006, 1: 769-774 ( ) DOI: 10.1038/nprot.2006.102
- Freeman W.M., Hemby S.E. Proteomics for protein expression profiling in neuroscience. Neurochem Res., 2004, 29: 1065-1081 ( ) DOI: 10.1023/B:NERE.0000023594.21352.17
- Wang W., Scali M., Vignani R., Spadafora A., Sensi E. Protein extraction for two-dimensional Electrophoresis from olive leaf, a plant tissue containing high levels of interfering compounds. Electrophoresis, 2003, 24: 2369-2375 ( ) DOI: 10.1002/elps.200305500
- Rose J.K.C., Bashir S., James J.G., Jahn M.M., Saravanan R.S. Tackling the plant proteome: practical approaches, hurdles and experimental tools. Plant J., 2004, 39: 715-733 ( ) DOI: 10.1111/j.1365-313X.2004.02182.x
- Vâlcu C.M., Schlink K. Efficient extraction of proteins from woody plant samples for two-dimensional electrophoresis. Proteomics, 2006, 6: 1599-1605 ( ) DOI: 10.1002/pmic.200500660
- Görg A., Weiss W., Dunn M.J. Current two-dimensional electrophoresis technology for Proteomics. Proteomics, 2004, 4(12): 3665-3685 ( ) DOI: 10.1002/pmic.200401031
- Leimgruber R.M., Malone J.P., Radabaugh M.R., LaPorte M.L., Violand B.N., Monahan J.B. Development of improved cell lysis, solubilization and imaging approaches for proteomic analyses. Proteomics, 2002, 2: 135-144 (doi: 10.1002/1615-9861(200202)2:2%3C135::AID-PROT135%3E3.0.CO;2-1).
- Morris A.C., Djordjevic M.A. Proteome analysis of cultivar-specific interactions between Rhizobium leguminosarum biovar trifolii and subterranean clover cultivar Woogenellup. Electrophoresis, 2001, 22(3): 586-598 (doi: 10.1002/1522-2683(200102)22:33.0.CO;2-L).
- Bodzon-Kulakowska A., Bierczynska-Krzysik A., Dylag T., Drabik A., Suder P. Methods for samples preparation in proteomic research. J. Chromatogr. B, 2007, 849: 1-31 ( ) DOI: 10.1016/j.jchromb.2006.10.040
- Schenkluhn L., Hohnjec N., Niehaus K., Schmitz U., Colditz F. Differential gel electrophoresis (DIGE) to quantitatively monitor early symbiosis-and pathogenesis-induced changes of the Medicago truncatula root proteome. J. Proteomics., 2010, 73(4): 753-768 ( ) DOI: 10.1016/j.jprot.2009.10.009
- Mijnsbrugge K.V., Meyermans H., Van Montagu M., Bauw G., Boerjan W. Wood formation in poplar: identification, characterization, and seasonal variation of xylem proteins. Planta, 2000, 210: 589-598 ( ) DOI: 10.1007/s004250050048
- Mihr C., Braun H.P. Proteomics in plant biology. In: Handbook of proteomics/P. Conn, (ed.). Humana Press, New Jersey, Totowa, 2003: 409-416 ( ) DOI: 10.1007/978-1-59259-414-6_28
- Nautrup-Pedersen G., Dam S., Laursen B.S., Siegumfeldt A.L., Nielsen K., Goffard N., Stærfeldt H.H., Friis C., Sato S., Tabata S., Lorentzen A., Roepstorff P., Stougaard J. Proteome analysis of pod and seed development in the model legume Lotus japonicus. J. Proteome Res., 2010, 9(11): 5715-5726 ( ) DOI: 10.1021/pr100511u
- Dam S., Dyrlund T.F., Ussatjuk A., Jochimsen B., Nielsen K., Goffard N., Ventosa M., Lorentzen A., Gupta V., Andersen S.U., Enghild J.J., Ronson C.W., Roepstorff P., Stougaard J. Proteome reference maps of the Lotus japonicus nodule and root. Proteomics, 2013, 14(2-3): 230-240 ( ) DOI: 10.1002/pmic.201300353


