Поиск ассоциации полиморфных локусов генов HIF1A, VEGFA с риском развития рака яичников у женщин Республики Башкортостан
Автор: Андреева Е.А., Аминова Э.Т., Фаисханова Р.Р., Загитов И.Р., Валова Я.В., Федорова Ю.Ю., Нургалиева А.Х., Дарвиш М., Сакаева Д.Д., Хуснутдинова Э.К., Прокофьева Д.С.
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 1 т.24, 2025 года.
Бесплатный доступ
Введение. Основные гены - кандидаты рака яичников (BRCA1/2) объясняют не более 15-20 % случаев заболевания, поэтому проблема поиска новых молекулярно-генетических маркеров остается актуальной. Цель исследования - анализ ассоциации полиморфных локусов rs11549465/ HIF1A , rs3025039/ VEGFA , rs2146323/ VEGFA с риском развития рака яичников у женщин, проживающих в Республике Башкортостан. Материал и методы. В исследование включены образцы ДНК пациенток с диагнозом РЯ (n=205) и женщин без онкопатологии на момент забора крови (n=259), проживающих в Республике Башкортостан. Генотипирование проводилось методом ПЦР Real Time на основе технологии TaqMan.
Рак яичников, молекулярно-генетическое исследование, ассоциативный анализ, генетические полиморфизмы
Короткий адрес: https://sciup.org/140309650
IDR: 140309650 | УДК: 618.11-006.6-092:575.174.015.3(470.57) | DOI: 10.21294/1814-4861-2025-24-1-79-91
Текст научной статьи Поиск ассоциации полиморфных локусов генов HIF1A, VEGFA с риском развития рака яичников у женщин Республики Башкортостан
studY oF tHe associatioN BetWeeN HIF1A aNd VEGFA geNe polYMoRpHisMs aNd oVaRiaN caNceR RisK iN WoMeN FRoM BasHKoRtostaN
e.a. andreeva1,2, e.t. aminova1, R.R. Faiskhanova3, i.R. Zagitov3,Y.V. Valova1,4, Yu.Yu. Fedorova1, a.Kh. Nurgalieva1, M. darvish5,d.d. sakaeva6, e.K. Khusnutdinova1,6,7, d.s. prokofyeva1
-
1 Ufa State University of Science and Technology
-
32, Zaki Validi St., Ufa, 450076, Russia
-
2Ural Medical Institute
-
9, Kurchatov St., Chelyabinsk, 454092, Russia
-
3Republican Clinical Oncology Dispensary
73/1, October Ave., Ufa, 450054, Russia
-
4Ufa Research Institute of Occupational Health and Human Ecology
-
94, Stepan Kuvykin St., Ufa, 450106, Russia
-
5Arak University of Medical Sciences, School of Medicine
-
3JPR+5FC, Basij Square, Arak, Markazi Province, Iran
-
6Bashkir State Medical University
-
3, Lenin St., Ufa, 450008, Russia
-
7Institute of Biochemistry and Genetics, Ufa Scientific Center, Russian Academy of Sciences
71, October Ave., Ufa, 450054, Russia
Background. The major candidate genes for ovarian cancer (BRCA1/2) explain no more than 15–20 % of cases; therefore it is important to focus on the search for new molecular genetic markers. the aim of the study was to analyze the association of rs11549465/HIF1A, rs3025039/VEGFA, and rs2146323/VEGFA polymorphic variants with the risk of developing ovarian cancer in women from the Republic of Bashkortostan. Material and Methods . Our research included DNA samples of women with ovarian cancer (n=205) and women without cancer at the time of blood sampling (n=259) from the Republic of Bashkortostan. Genotyping was carried out using the Real Time PCR method based on TaqMan technology. Results. Polymorphic variants, such as rs11549465/HIF1A, rs3025039/VEGFA, and rs2146323/VEGFA were not associated with the risk of developing ovarian cancer in women of the Republic of Bashkortostan. However, the rs11549465/HIF1A polymorphic locus was significantly correlated with the grade of tumor cell differentiation, and the rs3025039/VEGFA was associated with lymph node metastasis. conclusion. These polymorphic variants may be associated with ovarian cancer prognosis. To confirm this association, it is necessary to conduct research on a large sample size.
В структуре злокачественных новообразований (ЗНО) женского населения во всем мире рак яичников (РЯ) является восьмой по распространенности формой. В мире ежегодно регистрируется 324 402 случая РЯ, при этом число смертельных случаев составляет 206 839. В 2022 г. в России было зарегистрировано 14 606 новых случаев и 9 333 смерти по причине ЗНО указанной локализации [1]. Отсутствие патогномоничных симптомов заболевания существенно снижает эффективность ранней диагностики. В большинстве наблюдений диагностируют РЯ III–IV стадий, когда активно развивается метастатический процесс.
С этиологической точки зрения РЯ может быть наследственным и спорадическим. Наследственные формы РЯ составляют 5–10 % случаев ЗНО указанной локализации [2]. Основными генами – кандидатами РЯ являются гены BRCA1/2, однако нарушения в них объясняют не более 15–20 % случаев заболевания [3]. Исходя из вышеизложенного, проблема поиска новых молекулярно-генетических маркеров остается актуальной.
Большое влияние на процесс онкогенеза и метастазирование оказывает микросреда, особенно в условиях гипоксии. Гипоксия ассоциирована с нестабильностью генома, подавлением иммунитета. Кроме того, гипоксия является предиктором неблагоприятного клинического исхода, независимо от стадии опухолевого процесса, гистологических особенностей и состояния лимфатических узлов [4]. В нормальной ткани концентрация кислорода составляет около 7 %, в опухолевой ткани она колеблется от 1 до 7 %. Низкое количество кислорода способствует стабилизации белкового продукта гена HIF1a, который стимулирует эндотелиальные клетки мигрировать в развивающуюся опухоль и создавать новые кровеносные сосуды. Новые сосуды создаются хаотично, препятствуя правильному снабжению клеток кислородом и питательными веществами, таким образом, происходит усиление гипоксии. Белок HIF 1a стимулирует также экспрессию синтазы оксида азота (NOS), которая, участвуя в метаболизме аргинина, освобождает оксид азота, обладающий сосудорасширяющим свойством, а также способна увеличить выработку фактора роста эндотелия сосудов (VEGF) [5]. В свою очередь, VEGF повышает проницаемость сосудов, ведет к дезорганизации сосудистой стенки, что усугубляет гипоксию и способствует распространению клеток опухоли и росту метастазов (рис. 1) [6]. В настоящее время появляется все больше данных, указывающих на внутриклеточную роль белка VEGF, который может быть ассоциирован с регуляцией роста и дифференцировкой клеток при канцерогенезе [7].
Полиморфный локус rs3025039 гена VEGFA ассоциирован с различными заболеваниями, включая онкологические. Однако данные исследований противоречивы. Так, в одних работах сообщается, что аллель T выступает протективным фактором [8, 9], а в других – рисковым [10–12]. Полиморфный вариант rs2146323 также представляет собой распространенный локус в гене VEGFA, оказывающий влияние на транскрипционную активность гена. F. Lose et al. [13] проведена оценка влияния полиморфизмов в данном гене на общую выживаемость у больных РЯ, в том числе был исследован полиморфный локус rs2146323. Однако авторы не нашли достаточных подтверждений вклада данного полиморфного варианта в показатель общей выживаемости пациенток.
Несмотря на ряд проектов в дизайне «случай-контроль» [7, 14–16], фактическая связь между однонуклеотидным полиморфным локусом rs11549465 в гене HIF1A и риском возникновения и развития злокачественных опухолей остается до конца нераскрытой. Изучаемый полиморфный локус может значительно усиливать транскрипционную активность как в нормоксической, так и в гипоксической среде, как показано in vitro [17]. H.N. Li et al. [18] в результате метаанализа по данным 49 исследований «случай-контроль», включающих 12 920 больных онкологическими заболеваниями и 13 363 условно здоровых донора, установлено, что изучаемый локус ассоциирован с предрасположенностью к развитию злокачественных опухолей. Таким образом, изучение вклада полиморфных локусов генов HIF1a и VEGFA остается актуальным.
Цель исследования – поиск ассоциации полиморфных локусов rs11549465/ HIF1A , rs3025039/ VEGFA , rs2146323/ VEGFA с риском развития РЯ у женщин, проживающих в Республике Башкортостан.
Материал и методы
В исследовании использованы образцы ДНК, выделенные из периферической крови методом фенол-хлороформной экстракции, 205 больных РЯ и 259 женщин без онкопатологии на момент забора крови, проживающих в Республике Башкортостан (табл. 1, 2). Из 205 исследованных пациенток 7 (3,4 %) являются носительницами зародышевых мутаций в гене BRCA1 . В контрольной группе носительниц патогенных генетических вариантов
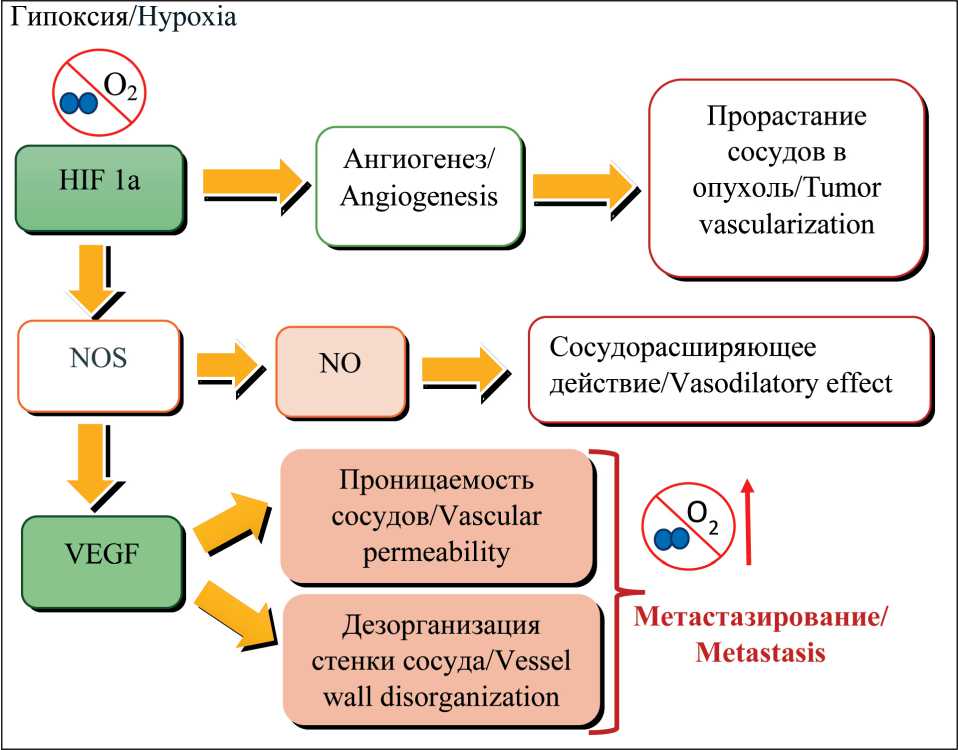
Рис. 1. Влияние белков HIF1a и VEGF на прогрессию и метастазирование опухоли. Примечание: рисунок выполнен авторами Fig.1. Effect of HIF1a and VEGF proteins on tumor progression and metastasis. Note: created by the authors
|
Характеристика выборки больных раком яичников characteristics of patients with ovarian cancer |
Таблица 1/table 1 |
||
|
Параметр/Parameter |
Больные РЯ/OC patients (n=205) |
Параметр/Parameter |
Больные РЯ/OC patients (n=205) |
|
Этническая принадлежность/Ethnicity |
Муцинозные опухоли/Mucinous tumors |
12 (5,9 %) |
|
|
Русские/Russians |
99 (48,2 %) |
Высокодифференцированные |
|
|
Татарки/Tatars |
66 (32,2 %) |
серозные опухоли/ |
5 (2,4 %) |
|
Башкирки/Bashkirs |
9 (4,4 %) |
Well-differentiated serous tumors |
|
|
Украинки/Ukrainians |
9 (4,4 %) |
Эндометриоидные опухоли/ |
2 (0,98 %) |
|
Чувашки/Chuvash |
7 (3,4 %) |
Endometrioid tumors |
|
|
Мордва/Mordovians |
4 (1,9 %) |
Светлоклеточные опухоли/Clear cell |
2 (0,98 %) |
|
Марийки/Mari |
2 (0,9 %) |
tumors |
|
|
Белоруски/Belarusians |
1 (0,5 %) |
Регионарные и отдаленные метастазы/ Regional and distant metastases |
|
|
Метисы/Mixed-race |
8 (3,9 %) |
||
|
Да/Yes |
70 (34,1 %) |
||
|
Репродуктивная функция/Reproductive function |
|||
|
Нет/No |
135 (65,9 %) |
||
|
Сохранена/Preserved |
62 (30,4 %) |
||
|
Менопауза/Menopause |
143 (69,6 %) |
Метастатическое поражение лимфатических узлов/ |
|
|
Стадия заболевания/Stage of disease |
Да/Yes |
61 (29,8 %) |
|
|
I–II стадия/Stage I–II |
70 (34,2 %) |
||
|
Нет/No |
144 (70,2 %) |
||
|
III–IV стадия/Stage III–IV |
135 (65,8 %) |
||
|
BRCA-статус/BRCA-status |
|||
|
Степень злокачественности/Tumor grade |
Наличие мутаций/Evidence of mutations |
7 (3,4 %) |
|
|
Низкая/Low-grade |
109 (53,2 %) |
||
|
Отсутствие мутаций/ No evidence of mutations |
|||
|
Высокая/High-grade |
96 (46,8 %) |
198 (96,6 %) |
|
|
Гистологический тип/Histological type |
|||
|
Низкодифференцированные серозные опухоли/ Low-differentiated serous tumors |
184 (89,8 %) |
||
|
Примечание: таблица составлена авторами. |
|||
|
Note: created by the authors. |
|||
Таблица 2/table 2
Характеристика контрольной группы characteristics of the control group
|
Параметр/Рarameter |
Контрольная группа/Control group (n=259) |
|
Этническая принадлежность/Ethnicity |
|
|
Русские/Russians |
124 (47,9 %) |
|
Татарки/Tatars |
84 (32,4 %) |
|
Башкирки/Bashkirs |
31 (12,0 %) |
|
Чувашки/Chuvash |
3 (1,2 %) |
|
Украинки/Ukrainians |
2 (0,7 %) |
|
Узбечки/Uzbeks |
1 (0,4 %) |
|
Калмычки/Kalmyks |
1 (0,4 %) |
|
Белоруски/Belarusians |
1 (0,4 %) |
|
Метисы/Mixed-race |
12 (4,6 %) |
|
Репродуктивная функция/Reproductive function |
|
|
Сохранен/Preserved |
137 (53,0 %) |
|
Менопауза/Menopause |
122 (47,0 %) |
|
BRCA-статус/BRCA-status |
|
|
Наличие мутаций/Evidence of mutations |
0 (0 %) |
|
Отсутствие мутаций/No evidence of mutations |
259 (100 %) |
|
Примечание: таблица составлена авторами. |
Note: created by the authors.
в главных генах – кандидатах рака яичников не обнаружено.
Для генотипирования использовались наборы реагентов «SNP-Скрин», производимые компанией «Синтол». Статистическая обработка результатов исследования проводилась с помощью MC Office Excel, а также с помощью программы Ген Эксперт. При попарном сравнении частот аллелей и генотипов в группах больных и контроля применялся критерий χ2 для таблиц сопряженности 2×2 с поправкой Йетса на непрерывность. Относительный риск развития заболевания оценивали с помощью показателя odds ratio (OR) с указанием 95 % доверительного интервала (95 %, CI). При статистическом критерии p<0,05 различия оценивались как значимые.
Результаты
Проведен анализ распределения частоты аллелей и генотипов полиморфных вариантов rs11549465/ HIF1A , rs3025039/ VEGFA , rs2146323/ VEGFA в группах больных РЯ и индивидов без онкопатологии на момент забора крови, проживающих в Республике Башкортостан. Распределение частот указанных полиморфных локусов соответствовало равновесию Харди–Вайнберга (p>0,05) (табл. 3). Распределение частот минорных аллелей согласуется с информацией, приведенной в популяционной базе данных RUSeq [19]. Так как исследуемая выборка не однородна по этническому происхождению, проведено сравнение частот аллелей и генотипов по отдельности среди русской (табл. 4) и татарской этнических групп (табл. 5). При этом показано отсутствие значимых различий в распределении частот аллелей и генотипов исследуемых полиморфных локусов (p>0,05). При изучении распределения частот аллелей и генотипов исследуемых полиморфных локусов в общей выборке (табл. 6) не обнаружено значимых различий (p>0,05).
Проведено изучение распределения частот аллелей и генотипов среди больных РЯ и женщин контрольной группы в зависимости от статуса менопаузы (табл. 7, 8). При разделении исследуемой группы в зависимости от статуса менопаузы значимых различий не обнаружено (p>0,05). Исследуемая выборка больных также была разделена на группы в зависимости от стадии заболевания на момент установки диагноза (табл. 9). При этом не выявлено значимых различий в распределении частот аллелей и генотипов (p>0,05).
При разделении группы больных РЯ в зависимости от степени дифференцировки опухоли обнаружены значимые различия. Установлено, что редкий аллель T полиморфного локуса rs11549465 гена HIF1A значимо чаще встречался у больных РЯ с низкодифференцированными опухолями, по сравнению с больными РЯ, у которых опухоли были высокодифференцированными (10,7 vs 2,5 %, p=0,01). Гомозиготный генотип по аллелю T также значимо чаще встречался у женщин с опухолями высокой степени злокачественности (p=0,04). При изучении частот аллелей и генотипов полиморфных локусов rs3025039/VEGFA, rs2146323/VEGFA в указанных выборках значимые различия не обнаружены (табл. 10).
При сравнении групп больных РЯ в зависимости от наличия отдаленных и регионарных метастазов (табл. 11) значимые различия в распределении частот аллелей и генотипов не обнаружены (p>0,05). Также проведен сравнительный анализ частот аллелей и генотипов в группах больных РЯ с метастатическим поражением лимфатических узлов и без него (табл. 12). Показано, что редкий аллель T полиморфного локуса rs3025039 гена VEGFA значимо чаще встречался в группе больных РЯ без метастатического поражения лимфоузлов (20,3 vs 5 %, p=0,02). Изучение частот аллелей и генотипов полиморфных локусов rs11549465/ HIF1A и rs2146323/ VEGFA значимых различий не демонстрирует (p>0,05).
Обсуждение
Одним из наиболее изученных полиморфизмов гена HIF1A является rs11549465 С>Т. Указанное изменение приводит к аминокислотной замене Pro582Ser (C1772T). Наличие указанного полиморфизма влияет на функционирование мРНК HIF1-α. I. Gladek et al. показали наличие ассоциации гетерозиготного генотипа CT c повышенной экспрессией фактора транскрипции HIF1-α [15].
Наше исследование демонстрирует отсутствие ассоциации данного полиморфного локуса с риском развития РЯ. Однако нами было установлено, что минорный аллель T данного полиморфного варианта достоверно чаще встречался у больных РЯ с опухолью высокой степени злокачественности (p=0,01). Гомозиготный генотип TT также достоверно чаще встречался в этой же группе пациенток (p=0,04). В литературе сообщается об отсутствии ассоциации полиморфизмов HIF1A с риском развития рака яичников в группе лиц европеоидного происхождения (исследования Ece Konac et al.) [20]. Кроме того, H. Suzuki et al. показано, что rs11549465/ HIF1A не связан со стадией опухоли и прогнозом [21]. Однако метаанализ, проведенный H.N. Li et al., демонстрирует взаимосвязь полиморфного локуса rs11549465/ HIF1A с риском развития злокачественных новообразований вне зависимости от локализации (TT vs CC: OR=2,06, 95 % CI=1,34–3,16). В данном метаанализе выявлена взаимосвязь указанного полиморфного варианта с риском развития рака предстательной железы (TT vs CC: OR=7,63, 95 % CI=1,83–31,8; TT vs CC/CT: OR=6,60, 95 % CI=2,07–21,0), злокачественных новообразований полости рта (TT vs CC: OR=2,61, 95 % CI=1,19–5,72; TT vs CC/CT: OR=13,2, 95 % CI=1,08–162), рака поджелудочной железы (TT vs
Таблица 3/table 3
Распределение частот аллелей и генотипов в соответствии с равновесием Харди–Вайнберга distribution of allele and genotype frequencies according to Hardy–Weinberg equilibrium
|
Полиморфный локус/ P olymorphic locus |
Выборка больных РЯ/ Cohort of OC patients (n=205) |
Контроль/ Сontrol group (n=259) |
|||
|
χ2 p |
χ |
p |
|||
|
rs11549465/ HIF1A |
0,03 1 |
3,67 |
0,06 |
||
|
rs3025039/ VEGFA |
0,13 0,72 |
0,13 |
0,72 |
||
|
rs2146323/ VEGFA |
2,69 2,69 |
3.48 |
0,06 |
||
|
Примечание: таблица составлена авторами. |
|||||
|
Note: created by the authors. |
|||||
|
Таблица 4/table 4 |
|||||
|
Сравнение частот аллелей и генотипов среди русской этнической группы |
|||||
|
comparison of allele and genotype frequencies among representatives of the Russian ethnic group |
|||||
|
Полиморфный локус, генотипы Polymorphic locus, genotypes, |
, аллели/ alleles |
Больные РЯ (русские)/ OC patients (Russians) (n=99) |
Контроль (русские)/ Сontrol group (Russians) (n=124) |
OR (95 %) CI) |
p |
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC |
88 (88,8 %) |
107 (85,9 %) |
1,30 (0,52–3,22) |
|
|
CT |
11 (11,2 %) |
13 (10,9 %) |
1,04 (0,40–2,70) |
0,27 |
|
|
TT |
0 (0 %) |
4 (3,3 %) |
0,16 (0,01–3,12) |
||
|
Аллели/Alleles |
C |
187 (94,4 %) |
227 (91,3 %) |
1,60 (0,79–3,72) |
0,27 |
|
T |
11 (5,6 %) |
21 (8,7 %) |
0,63 (0,27–1,46) |
||
|
rs3025039/ VEGFA Генотипы/ rs3025039/ VEGFA Genotypes |
CC |
74 (75,0 %) |
88 (71,2 %) |
1,22 (0,63–2,33) |
|
|
CT |
23 (22,5 %) |
33 (26,1 %) |
0,82 (0,42–1,61) |
0,84 |
|
|
TT |
2 (2,5 %) |
3 (2,7 %) |
0,92 (0,15–5,66) |
||
|
Аллели/Alleles |
C |
171 86,3 %) |
209 (84,2 %) |
1,17 (0,66–2,09) |
0,59 |
|
T |
27 (13,8 %) |
39 (15,8 %) |
0,85 (0,48–1,52) |
||
|
rs2146323/ VEGFA |
CC |
32 (32,7 %) |
54 (43,2 %) |
0,64 (0,32–1,26) |
|
|
Генотипы/rs2146323/ VEGFA |
CA |
47 (47,3 %) |
44 (35,1 %) |
1,66 (0,86–3,19) |
0,29 |
|
Genotypes |
AA |
20 (20,0 %) |
26 (21,6 %) |
0,91 (0,41–2,02) |
|
|
Аллели/Alleles |
C |
111 (56,4 %) |
152 (60,8 %) |
0,83 (0,52–1,32) |
0,44 |
|
A |
87 (43,6 %) |
96 (39,2 %) |
1,20 (0,76–1,91) |
||
|
Примечание: таблица составлена авторами. |
|||||
|
Note: created by the authors. |
|||||
|
Таблица 5/table 5 |
|||||
|
Сравнение частот аллелей |
и генотипов у представителей татарской этнической группы |
||||
|
comparison of allele frequencies and genotypes among representatives of the tatar ethnic group |
|||||
|
Полиморфный локус, генотипы |
, аллели/ |
Больные РЯ (татарки)/ |
Контроль (татарки)/ |
) OR (95 %) CI) |
p |
|
Polymorphic locus, genotypes, |
alleles |
OC patients (Tatars) (n=66) Control group (Tatars) (n=84 |
|||
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC |
57 (86,7 %) |
78 (92,5 %) |
0,53 (0,14–2,01) |
|
|
CT |
9 (13,3 %) |
5 (5,7 %) |
2,56 (0,60–10,91) |
0,29 |
|
|
TT |
0 (0 %) |
1 (1,9 %) |
0,38 (0,02–9,68) |
||
|
Аллели/Alleles |
C |
123 (93,3 %) |
161 (95,3 %) |
0,69 (0,20–2,35) |
0,55 |
|
T |
9 (6,7 %) |
7 (4,7 %) |
1,44 (0,43–4,90) |
||
|
rs3025039/ VEGFA Генотипы/ rs3025039/ VEGFA Genotypes |
CC |
48 (73,9 %) |
54 (64,8 %) |
1,54 (0,65–3,65) |
|
|
CT |
18 (26,1 %) |
28 (33,3 %) |
0,61 (0,30–1,68) |
0,45 |
|
|
TT |
0 (0 %) |
2 (1,9 %) |
0,38 (0,02–9,64) |
||
|
Аллели/Alleles |
C |
114 (87,0 %) |
136 (81,5 %) |
1,52 (0,60–3,30) |
0,29 |
|
T |
18 (13,0 %) |
32 (18,5 %) |
0,66 (0,30–1,44) |
||
|
rs2146323/ VEGFA Генотипы/ rs2146323/ VEGFA Genotypes |
CC |
20 (30,0 %) |
33 (39,3 %) |
0,66 (0,26–1,68) |
|
|
CA |
31 (46,7 %) |
34 (41,0 %) |
1,26 (0,52–3,04) |
0,68 |
|
|
AA |
15 (23,3 %) |
17 (19,7 %) |
1,24 (0,43–3,57) |
||
|
Аллели/Alleles |
C |
71 (53,3 %) |
100 (59,8 %) |
0,77 (0,41–1,43) |
0,4 |
|
A |
61 (46,7 %) |
68 (40,2 %) |
1,30 (0,70–2,4) |
||
Примечание: таблица составлена авторами.
Note: created by the authors.
|
Таблица 6/table 6 |
|||||
|
Распределение частот аллелей среди больных РЯ и контроля в общей выборке |
|||||
|
distribution of allele frequencies among patients with ovarian cancer and controls in the general sample |
|||||
|
Полиморфный локус, генотипы, аллели/ |
Больные РЯ/ |
Контроль/ |
OR (95 %) CI) |
p |
|
|
Polymorphic locus, genotypes, alleles |
OC patients (n=205) Control group (n=259) |
||||
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC |
179 (87,5 %) |
225 (86,8 %) |
1,07 (0,60–1,91) |
|
|
CT |
26 (12,5 %) |
28 (10,8 %) |
1,13 (0,61–2,09) |
0,26 |
|
|
TT |
0 (0 %) |
6 (2,5 %) |
0,20 (0,02–1,63) |
||
|
Аллели/Alleles |
C |
384 (92,2 %) |
478 (92,3 %) |
1,22 (0,72–2,09) |
0,46 |
|
T |
26 (7,8 %) |
40 (7,7 %) |
0,82 (0,48–1,40) |
||
|
CC |
146 (71,2 %) |
180 (69,5 %) |
1,09 (0,72–1,64) |
||
|
rs3025039/ VEGFA Генотипы/ |
CT |
0,84 |
|||
|
rs3025039/ VEGFA Genotypes |
53 (25,9 %) |
73 (28,3 %) |
0,89 (0,58–1,36) |
||
|
TT |
6 (2,9 %) |
6 (2,2 %) |
1,31 (0,40–4,37) |
||
|
Аллели/Alleles |
C |
345 (84,1 %) |
433 (83,6 %) |
1,04 (0,72–1,50) |
0,84 |
|
T |
65 (15,9 %) |
85 (16,4 %) |
0,96 (0,67–1,39) |
||
|
CC |
70 (34,2 %) |
102 (39,4 %) |
0,80 (0,53–1,22) |
||
|
rs2146323/ VEGFA Генотипы/ |
0,42 |
||||
|
rs2146323/ VEGFA Genotypes |
CA |
83 (40,3 %) |
104 (40,2 %) |
1,00 (0,67–1,52) |
|
|
AA |
52 (25,5 %) |
53 (20,5 %) |
1,33 (0,83–2,14) |
||
|
Аллели/Alleles |
C |
223 (54,4 %) |
308 (59,5 %) |
0,81 (0,61–1,08) |
0,16 |
|
A |
187 (45,6 %) |
210 (40,5 %) |
1,23 (0,92–1,64) |
||
|
Примечание: таблица составлена авторами. |
|||||
|
Note: created by the authors. |
|||||
|
Таблица 7/table 7 |
|||||
|
Распределение частот аллелей и генотипов в выборках больных РЯ и контроля в состоянии |
|||||
|
менопаузы |
|||||
|
distribution of allele and genotype frequencies in samples of patients with oc and controls in menopause |
|||||
|
Полиморфный локус, генотипы Polymorphic locus, genotypes, |
, аллели/ alleles |
Больные РЯ в менопаузе/ OC patients in menopause (n=143) |
Контроль, менопауза/ Control group in menopause OR (95 %) CI) (n=122) |
p |
|
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC |
126 (88,4 %) |
109 (89,7 %) |
0,87 (0,32–2,33) |
|
|
CT |
17 (11,7 %) %) |
9 (7,7 %) |
1,58 (0,55–4,57) |
0,24 |
|
|
TT |
0 (0 %) |
3 (2,6 %) |
0,18 (0,01–3,74) |
||
|
Аллели/Alleles |
C |
269 (94,2 %) |
227 (93,6 %) |
1,11 (0,45–2,74) |
0,82 |
|
T |
17 (5,8 %) |
15 (6,4 %) |
0,90 (0,36–2,23) |
||
|
CC |
106 (73,9 %) |
83 (68,4 %) |
1,31 (0,67–2,56) |
||
|
rs3025039/ VEGFA Генотипы/ rs3025039/ VEGFA Genotypes |
CT |
36 (25,0 %) |
34 (27,8 %) |
0,86 (0,43–1,72) |
0,46 |
|
TT |
1 (1,1 %) |
5 (3,8 %) |
0,29 (0,03–2,86) |
||
|
Аллели/Alleles |
C |
248 (86,4 %) |
200 (82,3 %) |
1,36 (0,75–2,47) |
0,3 |
|
T |
38 (13,6 %) |
44 (17,7 %) |
0,73 (0,40–1,33) |
||
|
CC |
43 (29,8 %) |
45 (36,8 %) |
0,73 (0,36–1,47) |
||
|
rs2146323/ VEGFA Генотипы/ |
0,5 |
||||
|
rs2146323/ VEGFA Genotypes |
CA |
65 (45,6 %) |
49 (40,0 %) |
1,26 (0,65–2,44) |
|
|
AA |
35 (24,6 %) |
28 (23,2 %) |
1,08 (0,50–2,33) |
||
|
Аллели/Alleles |
C |
151 (52,6 %) |
139 (56,8 %) |
0,84 (0,53–1,35) |
0,47 |
|
A |
135 (47,4 %) |
105 (43,2 %) |
1,19 (0,74–1,89) |
||
Примечание: таблица составлена авторами.
Note: created by the authors.
Таблица 8/table 8
Распределение частот аллелей и генотипов в выборках больных РЯ и контроля с сохранной репродуктивной функцией
distribution of allele and genotype frequencies in samples of patients with oc and controls with intact reproductive function
|
Полиморфный локус, генотипы, аллели/ Polymorphic locus, genotypes, alleles |
Больные РЯ с сохранной репродуктивной функцией/ OC patients with preserved reproductive function (n=62) |
Контроль с сохранной репродуктивной функцией/ Control group with preserved reproductive function (n=137) |
OR (95 %) CI) |
p |
|
CC |
54 (87,3 %) |
114 (83,0 %) |
1,40 (0,55–3,56) |
|
|
rs11549465/ HIF1A Генотипы/ |
||||
|
rs11549465/ HIF1A Genotypes CT |
7 (10,9 %) |
20 (14,4 %) |
0,73 (0,27–2,00) |
0,77 |
|
TT |
1 (1,8 %) |
3 (2,6 %) |
0,67 (0,07–6,62) |
|
|
C Аллели/Alleles |
115 (92,7 %) |
248 (90,2 %) |
1,39 (0,60–3,23) |
0,44 |
|
T |
9 (7,3 %) |
26 (9,8 %) |
0,72 (0,31–1,67) |
|
|
CC |
43 (69,5 %) |
97 (70,9 %) |
0,93 (0,47–1,86) |
|
|
rs3025039/ VEGFA Генотипы/ CT rs3025039/ VEGFA Genotypes |
18 (29,0 %) |
35 (25,5 %) |
1,20 (0,59–2,44) |
0,61 |
|
TT |
1 (1,5 %) |
5 (3,6 %) |
0,41 (0,06–2,99) |
|
|
C Аллели/Alleles |
104 (84,0 %) |
229 (83,6 %) |
1,02 (0,56–1,87) |
0,94 |
|
ллелиees T |
20 (16,0 %) |
45 (16,4 %) |
0,98 (0,53–1,78) |
|
|
CC |
24 (39,0 %) |
44 (31,8 %) |
1,37 (0,46–4,10) |
|
|
rs2146323/ VEGFA Генотипы/ |
||||
|
rs2146323/ VEGFA Genotypes CA |
21 (34,1 %) |
62 (45,2 %) |
0,62 (0,22–1,79) |
0,68 |
|
AA |
17 (26,8 %) |
31 (22,7 %) |
1,25 (0,37–4,19) |
|
|
C Аллели/Alleles |
69 (56,1 %) |
150 (54,5 %) |
1,06 (0,51–2,22) |
0,87 |
|
A |
55 (43,9 %) |
124 (45,5 %) |
0,94 (0,45–1,96) |
Примечание: таблица составлена авторами.
Note: created by the authors.
Таблица 9/table 9
Распределение частот аллелей и генотипов в группе больных РЯ в зависимости от стадии заболевания distribution of allele and genotype frequencies in group of patients with ОС depending on the stage of the disease
|
Полиморфный локус, генотипы, аллели/ Polymorphic locus, genotypes, alleles |
Больные РЯ I–II стадии/ Patients with OC Stage I–II (n=70) |
Больные РЯ III–IV стадии/ Patients with OC Stage III–IV (n=135) |
OR (95 %) CI) |
p |
|
CC |
63 (89,4 %) |
116 (85,7 %) |
1,40 (0,49–4,03) |
|
|
rs11549465/ HIF1A Генотипы/ |
||||
|
rs11549465/ HIF1A Genotypes CT |
6 (9,1 %) |
19 (14,3 %) |
0,60 (0,20–1,80) |
0,42 |
|
TT |
1 (1,5 %) |
0 (0 %) |
2,91 (0,12–2,74) |
|
|
C Аллели/Alleles |
132 (93,9 %) |
251 (92,9 %) |
1,19 (0,45–3,19) |
0,73 |
|
T |
8 (6,1 %) |
19 (7,1 %) |
0,84 (0,31–2,25) |
|
|
CC |
46 (65,7 %) |
110 (81,3 %) |
0,44 (0,20–0,99) |
|
|
rs3025039/ VEGFA Генотипы/ CT rs3025039/ VEGFA Genotypes |
23 (32,8 %) |
23 (17,2 %) |
2,36 (1,03–5,38) |
0,12 |
|
TT |
1 (1,5 %) |
2 (1,6 %) |
0,95 (0,06–5,59) |
|
|
C Аллели/Alleles |
115 (82,1 %) |
243 (89,8 %) |
0,52 (0,25–1,07) |
0,07 |
|
ллелиees T |
25 (17,9 %) |
27 (10,2 %) |
1,93 (0,94–3,98) |
|
|
CC |
20 (28,6 %) |
47 (35,0 %) |
0,74 (0,34–1,61) |
|
|
rs2146323/ VEGFA Генотипы/ |
||||
|
rs2146323/ VEGFA Genotypes CA |
33 (46,9 %) |
56 (41,2 %) |
1,26 (0,62–2,58) |
0,73 |
|
AA |
17 (24,5 %) |
32 (23,8 %) |
1,04 (0,45–2,39) |
|
|
C Аллели/Alleles |
73 (52,0 %) |
150 (55,6 %) |
0,87 (0,52–1,43) |
0,57 |
|
A |
67 (48,0 %) |
120 (44,4 %) |
1,16 (0,70–1,91) |
Примечание: таблица составлена авторами.
Note: created by the authors.
|
Таблица 10/table 10 Распределение частот аллелей и генотипов в группе больных РЯ в зависимости от степени злокачественности distribution of allele and genotype frequencies in the group of patients with ovarian cancer depending on |
|||||
|
the tumor grade |
|||||
|
Полиморфный локус, генотипы Polymorphic locus, genotypes, |
, аллели/ alleles |
Больные РЯ G1–G2/ Patients with OC Grade G1–G2 (n=109) |
Больные РЯ G3–G4/ Patients with OC Grade G3–G4 (n=96) |
OR (95 %) CI) |
p |
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC CT TT |
104 (95,1 %) 5 (4,9 %) 0 (0 %) |
77 (80,4 %) 17 (17,8 %) 2 (1,9 %) |
0,21 (0,06–0,80) 4,20 (1,09–16,16) 3,32 (0,13–83,30) |
0,04 |
|
Аллели/Alleles |
C T |
213 (97,5 %) 5 (2,5 %) |
171 (89,3 %) 21 (10,7 %) |
0,21 (0,06–0,77) 4,76 (1,31–17,34) |
0,01 |
|
rs3025039/ VEGFA Генотипы/ rs3025039/ VEGFA Genotypes |
CC CT TT |
74 (67,8 %) 31 (28,8 %) 4 (3,4 %) |
67 (69,8 %) 27 (28,3 %) 2 (1,9 %) |
1,10 (0,49–2,45) 0,98 (0,43–2,22) 0,55 (0,05–6,22) |
0,88 |
|
Аллели/Alleles |
C T |
179 (82,2 %) 39 (17,8 %) |
161 (84,0 %) 31 (16,0 %) |
1,13 (0,56–2,29) 0,88 (0,44–1,78) |
0,73 |
|
rs2146323/ VEGFA Генотипы/ rs2146323/ VEGFA Genotypes |
CC CA AA |
31 (28,3 %) 52 (47,8 %) 26 (23,9 %) |
41 (42,9 %) 41 (42,9 %) 14 (14,2 %) |
1,90 (0,65–5,59) 0,82 (0,29–2,31) 0,53 (0,13–2,15) |
0,44 |
|
Аллели/Alleles |
C A |
114 (52,2 %) 104 (47,8 %) |
123 (64,3 %) 69 (35,7 %) |
1,65 (0,78–3,50) 0,61 (0,29–1,29) |
0,19 |
|
Примечание: таблица составлена авторами. Note: created by the authors. |
|||||
|
Таблица 11/table 11 Распределение частот аллелей и генотипов в выборке больных РЯ в зависимости от наличия регионарных и отдаленных метастазов distribution of allele and genotype frequencies in a sample of patients with oc depending on the presence of metastases |
|||||
|
Больные РЯ Больные РЯ Полиморфный локус, с метастазами/ без метастазов/ генотипы, аллели/ OC patients with metastasis OC patients without metastasis Polymorphic locus, genotypes, alleles (n=70) (n=135) |
OR (95 %) CI) |
p |
|||
|
rs11549465/ HIF1A Генотипы/ rs11549465/ HIF1A Genotypes |
CC CT TT |
58 (83,3 %) 12 (16,7 %) 0 (0 %) |
121 (89,4 %) 12 (9,4 %) 2 (1,2 %) |
0,59 (0,21–1,65) 1,93 (0,67–5,51) 0,58 (0,02–4,54) |
0,36 |
|
Аллели/Alleles |
C T |
128 (91,7 %) 12 (8,3 %) |
254 (94,1 %) 16 (5,9 %) |
0,69 (0,26–1,81) 1,45 (0,55–3,82) |
0,44 |
|
rs3025039/ VEGFA Генотипы/ rs3025039/ VEGFA Genotypes |
CC CT TT |
54 (77,8 %) 14 (20,0 %) 2 (2,2 %) |
94 (69,3 %) 38 (28,4 %) 3 (2,3 %) |
1,55 (0,67–3,57) 0,63 (0,27–1,50) 0,98 (0,09–11,08) |
0,57 |
|
Аллели/Alleles |
C T |
122 (87,1 %) 18 (12,9 %) |
226 (83,5 %) 44 (16,5 %) |
1,42 (0,67–2,99) 0,71 (0,33–1,49) |
0,36 |
|
rs2146323/ VEGFA Генотипы/ rs2146323/ VEGFA Genotypes |
CC CA AA |
19 (26,5 %) 31 (44,1 %) 20 (29,4 %) |
50 (36,7 %) 54 (40,0 %) 31 (23,3 %) |
0,62 (0,25–1,57) 1,18 (0,51–2,77) 1,37 (0,53–3,54) |
0,58 |
|
Аллели/Alleles |
C A |
69 (49,2 %) 71 (50,8 %) |
154 (57,0 %) 116 (43,0 %) |
0,72 (0,40–1,31) 1,39 (0,76–2,52) |
0,28 |
Примечание: таблица составлена авторами.
Note: created by the authors.
Таблица 12/table 12
Распределение частот аллелей и генотипов в выборках больных РЯ в зависимости от метастатического поражения лимфатических узлов distribution of allele and genotype frequencies in samples of patients with oc depending on lymph node metastasis
|
Полиморфный локус, генотипы, аллели/ Polymorphic locus, genotypes, alleles |
Больные РЯ с метастатическим поражением лимфатических узлов/ OC patients with lymph node metastasis (n=61) |
Больные РЯ без метастатического поражения лимфатических узлов/ OC patients without lymph node metastasis (n=144) |
OR (95 %) CI) |
p |
|
CC |
48 (78,9 %) |
133 (92,5 %) |
0,31 (0,08–1,17) |
|
|
rs11549465/ HIF1A Генотипы/ CT rs11549465/ HIF1A Genotypes |
13 (21,1 %) |
9 (6,5 %) |
3,8 (0,97–15,35) |
0,12 |
|
TT |
0 (0 %) |
2 (1,1 %) |
1,58 (0,06–40,28) |
|
|
C Аллели/Alleles |
109 (89,5 %) |
275 (95,5 %) |
0,38 (0,11–1,34) |
0,12 |
|
T |
13 (10,5 %) |
13 (4,5 %) |
2,62 (0,75–9,18) |
|
|
CC |
55 (90,0 %) |
90 (62,5 %) |
5,40 (1,18–24,65) |
|
|
rs3025039/ VEGFA Генотипы/ CT rs3025039/ VEGFA Genotypes |
6 (10,0 %) |
50 (34,4 %) |
0,21 (0,05–0,97) |
0,06 |
|
TT |
0 (0 %) |
4 (3,1 %) |
0,65 (0,03–13,11) |
|
|
C Аллели/Alleles |
116 (95,0 %) |
230 (79,7 %) |
4,84 (1,12–20,95) |
0,02 |
|
ллелиees T |
6 (5,0 %) |
58 (20,3 %) |
0,21 (0,05–0,89) |
|
|
CC |
16 (26,7 %) |
52 (36,4 %) |
0,64 (0,18–2,22) |
|
|
rs2146323/ VEGFA Генотипы/ |
33 |
|||
|
rs2146323/ VEGFA Genotypes CA |
53,3 %) |
55 (37,9 %) |
1,87 (0,61–5,80) |
0,55 |
|
AA |
12 (20,0 %) |
37 (25,8 %) |
0,72 (0,18–2,86) |
|
|
C Аллели/Alleles |
65 (53,3 %) |
159 (55,3 %) |
0,92 (0,42–2,05) |
0,84 |
|
A |
57 (46,7 %) |
129 (44,7 %) |
1,08 (0,49–2,40) |
Примечание: таблица составлена авторами.
Note: created by the authors.
злокачественности, по сравнению с больными РЯ, у которых опухоли обладали низкой степенью злокачественности (p=0,01). Обнаружено, что гомозиготы по аллелю T полиморфного локуса rs11549465/ HIF1A значимо чаще встречались у пациенток с низкодифференцированными опухолями (p=0,04). Также мы обнаружили, что минорный


