Поиск и изучение генов, кодирующих металло-бета-лактамазы, у госпитальных штаммов Pseudomonas aeruginosa
Автор: Кузнецова Марина Валентиновна, Егорова Дарья Олеговна, Карпунина Тамара Исаковна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 3-4, 2011 года.
Бесплатный доступ
Изучены карбапенемоустойчивые нозокомиальные штаммы Pseudomonas аeruginosa, изолированные в хирургических стационарах г. Перми и пос. Лобаново. Установлено, что в геноме 34 культур присутствуют фрагменты ДНК, соответствующие по размеру генам blaVIM-2. Принадлежность амплифицированных генов к подгруппе blaVIM-2 была подтверждена результатами рестрикционного анализа с эндонуклеазой HindII. Сравнение нуклеотидных последовательностей blaVIM шести изолятов, принадлежащих к разным геномогруппам, показало высокий уровень сходства (99-100%) участков генов длиной 615-741 п.н. с геном blaVIM-2 референтного штамма P. aeruginosa COL-1 (GenBank GQ853417.1).
Pseudomonas аeruginosa, карбапенемы, металло-бета-лактамазы
Короткий адрес: https://sciup.org/147204561
IDR: 147204561 | УДК: 579.61:579.25:579.841.11
Текст научной статьи Поиск и изучение генов, кодирующих металло-бета-лактамазы, у госпитальных штаммов Pseudomonas aeruginosa
Карбапенемы – это один из важнейших классов антибиотиков, сочетающий исключительно широкий спектр антибактериальной активности, низкую токсичность и благоприятные фармакологические параметры (Галкин, 2007). Меропенем и имипенем, наряду с другими «антисинегнойными» средствами, являются препаратами выбора в терапии тяжелых нозокомиальных инфекций, вызванных P. aeruginosa (Руднов, 2005; Иванов и др., 2009).
Наиболее распространенным механизмом резистентности к карбапенемам у P. aeruginosa является их ферментная инактивация. Этот механизм может реализовываться с помощью бета-лактамаз трех классов: некоторыми ферментами классов А и D и металло-бета-лактамазами класса В (Queenan, Bush, 2007). Для P. aeruginosa описано пять основных групп карбапенемаз молекулярного класса В, подкласса В1: VIM, IMP, SPM, GIM и SIM. Все они содержат по два атома цинка и различаются аминокислотными последовательностями. Гены, кодирующие большую часть этих ферментов, в составе генных кассет включены в различные мобильные элементы, в основном интегроны 1-го класса.
VIM-1-карбапенемаза (for “Verona integron-encoded metallo-lactamase”) была впервые выделена из нозокомиального штамма P. aeruginosa в Вероне (Италия) в 1997 г. (Lauretti, Riccio, Mazzariol et al., 1999). Позднее описан VIM-2-фермент из штамма, изолированного еще в 1996 г. во Франции (Poirel, Naas, Nicholas et al., 2000). Госпитальные штаммы P. aeruginosa чаще всего продуцируют именно VIM-ферменты (Lee, Lim, Yum et al., 2002; Черкашин, Федорчук, Иванов и др., 2006; Шевченко, Эйдельштейн, Степанова, 2007). Внутри VIM-карбапенемаз выделяют две большие филогенетические линии: подгруппа VIM1 (VIM-1, 4, 5, 12, 14, 19) и подгруппа VIM2 (VIM-2, 3, 6, 8, 9, 10, 11, 13, 15, 16, 17, 18, 23), в отдельный кластер выделен единственный фермент VIM-7 (Toleman, Rols-ton, Jones, Walsh, 2004). Сегодня описано 25 вариантов этих ферментов, и их список постоянно пополняется #table1). Первый фермент IMP-группы (for “active on imipenem”) был описан у А. baumannii, изолированного в госпитале Италии (Riccio, Franceschini, Boschi et al., 2000). После этого IMP-карбапенемазы найдены во всем мире, включая США и Австралию. GIM-1-фермент (for “German imipenemase”) был изучен в Германии в 2002 г. (Castanheira, Toleman, Jones et al., 2004), SPM-1 (for “Sao Paulo metallo-lactamase”) впервые выделен у штаммов P. aeruginosa из Сан-Паулу в Бразилии (Poirel, Magalhaes, Lopes, Nordmann, 2004). Фермент SIM-1 (for “Seoul imipenemase”) – последний из металло-лактамаз описан в Корее (Lee, Yum, Yong et al., 2005). Если SPM-, GIM- и SIM-ферменты почти не распространись за пределы
страны их происхождения, то VIM и IMP-металло-бета-лактамазы описаны по всему миру и продолжают обнаруживаться не только у P. aeruginosa , но и у других видов бактерий (Queenan, Bush, 2007).
Цель настоящего исследования – поиск и изучение генов металло-бета-лактамаз у нозокомиальных штаммов P. aeruginosa .
Материалы и методы исследования
Исследовано 34 штамма P. aeruginosa , обладающих фенотипическими признаками продукции ме-талло-бета-лактамаз (положительных в ЭДТА-тесте) (Arakawa et al., 2000). Культуры выделены в 2008– 2009 гг. от больных крупных хирургических стационаров г. Перми (краевая клиническая больница – ККБ, городская клиническая больница – ГКБ) и пос. Лобаново Пермского края (центральная районная больница – ЛКБ).
ДНК получали, как описано G.G. Stone, R.D. Oberst, S. Hays et al. (1994). Петлю биомассы бактериальной культуры инокулировали в 100 мкл сверхчистой воды, прогревали при 95°С в твердотельном термостате «Термит» 15 мин, пробы охлаждали, центрифугировали 5 мин при 12 тыс. об./мин. Супернатант использовали в генетических исследованиях.
Генетическое типирование (фингерпринтинг) штаммов осуществляли с использованием методов
RAPD-ПЦР (с праймером М13) и BOX-ПЦР (Versa-lonic et al., 1994). Продукты реакции разделяли посредством электрофореза в 1.2%-ном агарозном геле.
Таблица 1
Праймеры для ПЦР-анализа генов металло-бета-лактамаз
Рестрикционный анализ амплифицированных фрагментов ДНК, с использованием праймеров к bla VIM-2 -гену, проводили с помощью эндонуклеазы Hind II. 5 мкл ПЦР-продукта обрабатывали 1 ед. ре-стриктазы (Fermentas, Литва) с использованием рекомендуемого буфера и при соответствующем температурном режиме.
«Healthcare» (США) использовался согласно рекомендациям производителя. Для секвенирующей ПЦР использовали те же праймеры, что и для наработки ампликонов. Предварительный анализ гена blaVIM-2 и «внутреннего» фрагмента генов blaVIM и поиск гомологичных последовательностей осуществляли, используя программы BLAST и базы данных GenBank/EMBL/DDBJ . Далее их анализировали с использованием программ CLUSTAL X 1.83 (Thompson et al., 1994), TREECON version 1.3b (Van de Peer et al., 1994). Анализ первичной структуры белков осуществляли с помощью программы Vector NTI 10 . Поиск гомологичных последовательностей выполнен с использованием программ BLAST. Выравнивание аминокислотных последовательностей проведено с помощью программы CLUSTAL X 1.83.
Результаты и их обсуждение
Скрининг штаммов на наличие генов, кодирующих металло-бета-лактамазы
Для определения родства выделенных изолятов и отбора штаммов для дальнейших исследований проведено их генетическое типирование. Штаммы P. aeruginosa , выделенные от больных ГКБ, распределились в две геномогруппы. Изоляты 2ПЛ, 31 и 60, полученные из ЛКБ, обладают индивидуальными RAPD-профилями. Все культуры P. aeruginosa ККБ образуют одну геномогруппу (табл. 2). На матрице ДНК исследуемых штаммов была проведена амплификация нуклеотидных последовательностей, гомологичных гену bla VIM-2 и консервативному участку генов группы VIM.
Таблица 2
Распределение исследуемых штаммов P. aeruginosa в геномогруппы
|
ЛПУ |
Изолят |
Геномо-группа |
|
ГКБ |
11р, 9р, 2р, 10к, 11к, 12к, 13к, 1-13, 2-1, 2-4, 2-9, 3-4, 3-6, 3-7, 3-9, 3-14, 3-16, 3-17, 3-22, 3-22а |
I |
|
125, 127, 128, 129 |
II |
|
|
ККБ |
195, 196, 197, 198, 201, 203 |
III |
|
ЛКБ |
2 ПЛ |
IV |
|
31 |
V |
|
|
60, 18 |
VI |
Специфичная амплификация была обнаружена у 34 штаммов, изолированных из 3 стационаров (ГКБ, ККБ, ЛКБ) (рис. 1). Амплифицированные участки ДНК длиной около 260 п.н. соответствуют по размеру консервативной части генов группы VIM, а ДНК-фрагменты размером 800 п.н. – полному bla VIM-2 -гену.
261 п.н.
---►
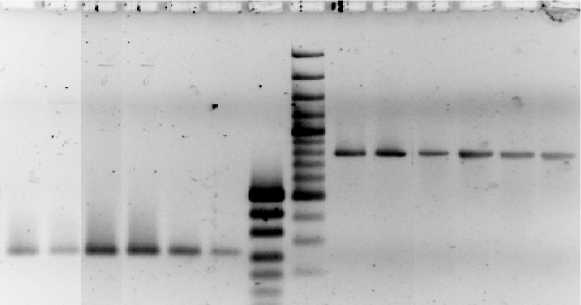
801 п.н. ◄---
1 2 3 4 5 6 7 8 9 10 11 12 13 14
А
В
Рис. 1 . Электрофореграмма продуктов амплификации консервативных последовательностей (А) и полных генов (В), кодирующих металло-бета-лактамазы, изолятов P. aeruginosa :
1, 9 – штамм 129 (ГКБ, геномогруппа II); 2,10 – штамм 3-16 (ГКБ, геномогруппа I); 3, 11 – штамм 198 (ККБ, геномогруппа III); 4, 12 – штамм 2ПЛ (ЛКБ, геномогруппа IV); 5, 13 – штамм 31 (ЛКБ, геномогруппа V); 6, 14 – штамм 60 (ЛКБ, геномогруппа VI); 7 – маркер молекулярных масс pUC19 ДНК/MspI (“Силекс”, Россия); 8 – маркер молекулярных масс O’GeneRulerTM 100bp Plus DNA Ladder (“Fermentas”, Литва)
Амплификация с праймерами к фланкирующим участкам генов других металло-бета-лактамаз не дала положительного результата.
ПДРФ-анализ амплифицированных участков
Для выявления сходства и различий между исследуемыми генами, кодирующими VIM-карба-пенемазы, был проведен анализ полиморфизма длин рестрикционных фрагментов амплифициро-ванных последовательностей. Набор рестрикционных фрагментов, полученный после обработки ампликонов blaVIM-2-гена исследуемых штаммов ре-стриктазой HindII, был представлен фрагментами ДНК длиной 414 и 387 п.н. (рис. 2). Длины полу- ченных рестрикционных фрагментов сопоставимы с длинами фрагментов на рестрикционных картах гена blaVIM-2 известных карбапенеморезистентных штаммов P. aeruginosa (рис. 3).
Анализ нуклеотидной последовательности амплифицированных фрагментов bla VIM-2
Определены нуклеотидные последовательности фрагментов и полных генов металло-бета-лактамаз группы VIM, амплифицированные с ДНК штаммов P. aeruginosa 3-16, 127, 2ПЛ, 31, 60, 198, являющихся представителями разных геномогрупп. Анализ нуклеотидной последовательности фрагментов длиной около 260 п.н. исследуемых штам- мов показал их 100%-ное сходство между собой, а также с известными последовательностями гена blaVIM-2 на участке с 114 по 353 нуклеотид. Исследуемые участки ДНК размером 615-741 п.н. были на 99–100% сходны с последовательностью blaVIM-2-гена известных штаммов (табл. 3, рис. 4). Степень сходства с геном blaVIM-2 референтного штамма P. aeruginosa COL-1 (GenBank AF191564.1) составила 99-100%. Обнаружены точечные нуклеотидные замены в последовательности исследуемых генов у 4 штаммов (табл. 3), в трех из них выявлена замена аминокислотного остатка.
У штаммов P. aeruginosa 2ПЛ, 31 и 3–16 обнаружена точечная мутация bla VIM-2 -гена в позиции 85, несущая замену серина на пролин, которая характерна для генов, кодирующих ферменты подгруппы VIM-1 и VIM-7 (Lauretti et al., 1999; Tole-man et al., 2007). Учитывая, что эта область находится на поверхности белка и удалена от активного центра, данные замены не влияют на связывание фермента с субстратом – антибиотиком. Несмотря на выявленные нуклеотидные замены результаты сравнительного анализа свидетельствуют о наличии bla VIM-2 -генов в штаммах P. aeruginosa , изолированных из крупных стационаров г. Перми и пос. Лобаново.
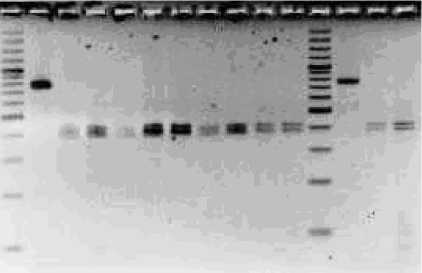
1 2 3 4 5 6 7 8 9 10 11 12 13 14 15
Рис. 2 . Электрофореграмма гидролиза ПЦР-продуктов амплификации bla VIM-2 -гена, обработанных Hind II:
1, 12 – маркер молекулярных масс O’GeneRulerTM 100bp Plus DNA Ladder; 2, 13 – ампликоны bla VIM-2 -гена; изоляты из ЛКБ: 3 – штамм 2ПЛ; 4 – штамм 31; 5 – штамм 60; изоляты из ККБ: 6 – штамм 198; 7
– штамм 201; 8 – штамм 203; изоляты из ГКБ: 9 – штамм 3-16; 10 – штамм 128; 11 – штамм 127; 14 – штамм 1-13; 15 – штамм 3-9
0.1
VIM2 (GQ853417.1)
VBI16 (ELT419746)
VBI15 (ELT419745)
VIM18 (АМ778091)
VIM3 (PS-26/00)
там (AY165025)
mill (AY605049)
2 ПЛ
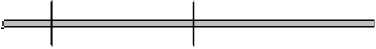
bla VIM-1
109 305 387
bla VIM-2
414 387
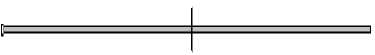
801 п.н.
Рис. 3 . Карты расположения сайтов реестрик-ции эндонуклеазы Hind II на генах bla VIM .
Карты построены с помощью программы Vector NTI для известных последовательностей, кодирующих металло-бета-лактамазы основных ферментов группы VIM (подгруппа VIM-1 – карта на примере bla VIM-1 идентична картам других ферментов данной подгруппы, подгруппа VIM-2 – карта на примере bla VIM-2 соответствует картам остальных ферментов данной подгруппы, за исключением bla VIM-13)
юо
3-16
— VIM8 (AY524987)
¥IM5(PSEK5)
VIM1 (Y180502)
таи 4 (FJ445404)
VIM4 (GU250441)
VIM13(EF577407)
Рис. 4 . Древо сходства нуклеотидных последовательностей полных генов металло-бета-лактамаз (группа VIM) бактерий рода Pseudomonas , построенное методом UPGMA :
“Bootstrap”-анализ проведен на 1000 повторностях. Жирным крупным шрифтом выделены штаммы, исследуемые в данной работе. В скобках указаны номера последовательностей, депонированных в GenBank, или обозначение штамма
Таблица 3
Анализ нуклеотидной последовательности фрагментов полных генов bla VIM-2
|
Штамм |
Размер секвенированного фрагмента, п.н. (расположение на гене bla VIM-2 P. aeruginosa COL-1 ) |
Сходство с bla VIM-2 P. aeruginosa COL-1 , % |
Нуклеотидные замены* |
Аминокислотные замены* |
|
|
2ПЛ |
665 |
с 24 п.н. по 689 п.н. |
99 |
Т85С |
S29P |
|
31 |
637 |
с 68 п.н. по 704 п.н. |
99 |
T85C |
S29P |
|
60 |
615 |
с 51 п.н. по 665 п.н. |
100 |
- |
- |
|
3-16 |
741 |
с 61 п.н. по 801 п.н. |
99 |
T85C |
S29P |
|
127 |
678 |
с 97 п.н. по 774 п.н. |
99 |
G744A |
нет замены |
|
198 |
637 |
с 63 п.н. по 698 п.н. |
100 |
- |
- |
*Номер замены соответствует месту нуклеотида и аминокислотного остатка от начала гена bla VIM-2
Заключение
Таким образом, устойчивость к карбапенемам 34 культур P. aeruginosa , изолированных из двух стационаров г. Перми и пос. Лобаново, обусловлена продукцией ими металло-бета-лактамаз группы VIM, и в частности – фермента VIM-2. Можно предположить, что в Пермском крае циркулируют преимущественно продуценты VIM-2-подобных ферментов. Полученные результаты согласуются с литературными данными о распространенности VIM-2-обусловленной карбапенеморезистентности внутрибольничных штаммов P. aeruginosa в России и странах Европы (Poirel, Naas, Nicholas et al., 2000; Черкашин, Федорчук, Иванов и др., 2006; Шевченко, Эйдельштейн, Степанова, 2007).
Список литературы Поиск и изучение генов, кодирующих металло-бета-лактамазы, у госпитальных штаммов Pseudomonas aeruginosa
- Галкин Д.В. Карбапенемы через 20 лет после открытия: современные микробиологические и клинические аспекты//Клин. микробиол. антимикроб. химиотер. 2007. Т. 9, № 2. С. 133-152.
- Иванов Д.В. и др. Особенности антибиотикочувствительности важнейших грамотрицательных возбудителей нозокомиальных инфекций//Вест. Росс. гос. мед. ун-та. 2009. № 2. С. 26-29.
- Руднов В.А. Антибиотикотерапия госпитальных инфекций, вызванных P. aeruginosae//Рус. мед. журн. 2005. Т. 13. № 7. С. 485-490.
- Черкашин Е.А., Федорчук В.В., Иванов Д.В. и др. Исследование распространенности метало-бе-талактамаз в Российской Федерации//Вестн. Моск. ун-та. Сер. Химия. 2006. №4 7(3). С. 83-86.
- Шевченко О.В., Эйдельштейн М.В., Степанова М.Н. Металло-лактамазы: значение и методы выявления у грамотрицательных неферментирующих бактерий//Клин. микробиол. антимикроб. химиотер. 2007. Т. 9. № 3. С. 211-218.
- Arakawa Y., Shibata N., Shibayama K. [et al] Convenient test for screening metallo-b-lactamaseproducing gram-negative bacteria by using thiol compounds//J. Clin. Microbiol. 2000. Vol. 38. P. 40-43.
- Castanheira M., Toleman M.A., Jones R.N. et al. Molecular characterization of -lactamase gene, bla-GIM-1, encoding a new subclass of metallo-blactamase//Antimicrob. Agents Chemother. 2004. Vol. 48. P.4654-4661.
- Lauretti L., Riccio M.L., Mazzariol A. et al. Cloning and characterization of [bla.sub.VIM], a new integron-borne metallo-[beta]-lactamase gene from a Pseudomonas aeruginosa clinical isolate//Antimicrob. Agents. Chemother. 1999. Vol. 43. P. 1584-1590.
- Lee K., Lim J.B., Yum J.H. et al. [bla.sub.VIM-2] cassette-containing novel integrons in metallo-[beta]-lactamase-producing Pseudomonas aeruginosa and Pseudomonas putida isolates disseminated in a Korean hospital//Antimicrob. Agents. Chemother. 2002. Vol. 46. P. 1053-1058.
- Lee K., Yum J.H., Yong D., et al. Novel acquired metallo-b-lactamase gene, blaSIM-1, in a class 1 integron from Acinetobacter baumannii clinical isolates from Korea. Antimicrob//Agents Chemother. 2005. Vol. 49. P. 4485-4491.
- Poirel L., Naas T., Nicholas D. et al. Characterization of VIM-2, a carbapenem-hydrolyzing metallobeta-lactamase and its plasmid-and integron-borne gene from a Pseudomonas aeruginosa clinical isolate in France//Antimicrob. Agents. Chemother. 2000. Vol. 44. P. 891-897.
- Poirel L., Magalhaes M., Lopes M., Nordmann P. Molecular analysis of metallo-beta-lactamase gene blaSPM-1-surrounding sequences from disseminated Pseudomonas aeruginosa isolates in Recife, Brazil//Antimicrob. Agents Chemother. 2004. Vol. 48. Р. 1406-1409.
- Queenan A.M., Bush K. Carbapenemases: the versatile b-lactamases//Clin. Microbiol. Rev. 2007. Vol. 20. P. 440-458.
- Riccio M.L., Franceschini N., Boschi L. et al. Characterization of the metallo-beta-lactamase determinant of Acinetobacter baumannii AC-54/97 reveals the existence of blaIMP allelic variants carried by gene cassettes of different phylogeny//Antimicrob. Agents Chemother. 2000. Vol. 44. P. 1229-1235.
- Shibata N., Doi Y., Yamane K. et al. PCR typing of genetic determinants for metallo-beta-lactamases and integrases carried by gram-negative bacteriaisolated in Japan, with focus on the class 3 integron//J. Clin. Microbiol. 2003. Vol. 41. P. 5407-5413.
- Stone G.G., Oberst R.D., Hays S. [et al.] Detection of Salmonella serovars from clinical samples by enrichment broth cultivation-PCR procedure//J. Clin. Microbiol. 1994. Vol. 32. P. 1742-1749.
- Thompson J.D., Higgins D.G., Gibson T.J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment son//Nucl. Acids. Res. 1994. Vol. 22. P. 4673-4680.
- Toleman M.A., Rolston K., Jones R., Walsh T.R. bla-VIM-7, an evolutionarily distinct metallo-betalactamase gene in a Pseudomonas aeruginosa isolate from the United States//Antimicrob. Agents Chemother. 2004. Vol. 48, N 1. P. 329-332.
- Tsakris A., Pournaras S., Woodford N. et al. Outbreak of infections caused by Pseudomonas aeruginosa producing VIM-1 carbapenemase in Greece//J. Clin. Microbiol. 2000. Vol. 38. P. 1290-1292.
- Van de Peer Y., DeWachter R. TREECON for Windows a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment//Comput. Appl. Biosci. 1994. Vol. 10. P. 569-570.


