Поиск протеомных маркеров рака молочной железы в составе суммарных экзосом крови
Автор: Тутанов Олег Сергеевич, Бакакина Юлия Сергеевна, Проскура Ксения Викторовна, Григорьева Алина Евгеньевна, Сяхович Виталий Эдуардович, Беляев Сергей Александрович, Рябчикова Елена Ивановна, Центалович Юрий Павлович, Лактионов Павел Петрович, Тамкович Светлана Николаевна
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 2 т.19, 2020 года.
Бесплатный доступ
Актуальность. Актуальной задачей повышения эффективности ранней диагностики онкологических заболеваний является поиск высокоспецифичных опухолевых маркеров в биологических жидкостях организма. Значительная часть экзосом ассоциирована с поверхностью форменных элементов крови, однако белковый спектр таких экзосом ранее не исследовался. Использование суммарных экзосом крови (экзосом плазмы и экзосом, ассоциированных с поверхностью клеток крови) может не только значительно повысить специфичность и чувствительность существующих методов, но и выявить новые онкомаркеры для жидкостной биопсии. Цель работы - поиск кандидатных белковых онкомаркеров рака молочной железы (РМЖ) путем сравнения 2D-протеомных карт суммарных экзосом крови здоровых женщин и больных РМЖ. Материал и методы. Экзосомы выделены из крови здоровых женщин и больных РМЖ методом ультрафильтрации с последующим ультрацентрифугированием и охарактеризованы при помощи трансмиссионной электронной микроскопии и иммуноцитохимии. Концентрацию белка в экзосомах определяли при помощи коммерческого набора NanoOrange Protein Quantitation kit (Invitrogen). Протеом экзосом исследован с помощью 2D-электрофореза с последующей идентификацией белков методом масс-спектрометрии. Результаты. Получены высокоочищенные препараты микровезикул суммарной крови размером не более 100 нм, на поверхности которых иммуноцитохимически были выявлены специфические для экзосом маркеры (CD63, CD9 и CD24). Сравнительный анализ протеомных карт экзосомальных белков здоровых женщин и больных РМЖ, полученных с помощью 2D-электрофореза, позволил установить значимые различия в уровне экспрессии и наборе белков в норме и патологии. Методом пептидного фингерпринта идентифицировано 11 перспективных протеомных маркеров РМЖ, из них LRG и у-цепь FGB выявлены в составе экзосом впервые (согласно базе Exocarta). Методом MALDI-TOF масс-спектрометрии идентифицировано 99 белков в препаратах экзосом крови здоровых женщин и больных РМЖ, из них 35 % выявлены в составе экзосом впервые (согласно базе Exocarta). В составе суммарных экзосом крови онкологических больных выявлено 17 (53 %) белков, ассоциированных с РМЖ. Заключение. Полученные результаты свидетельствуют о том, что суммарные экзосомы крови являются перспективным источником диагностического материала для поиска протеомных маркеров РМЖ. Идентифицированные протеомные онкомаркеры в их составе требуют дальнейшей валидации.
Экзосомы, трансмиссионная электронная микроскопия, масс-спектрометрия, протеомные маркеры, рак молочной железы
Короткий адрес: https://sciup.org/140254337
IDR: 140254337 | УДК: 616.19-006.6+577.112+57.088.2 | DOI: 10.21294/1814-4861-2020-19-2-49-61
Текст научной статьи Поиск протеомных маркеров рака молочной железы в составе суммарных экзосом крови
Рак молочной железы (РМЖ) занимает первое место среди онкологических заболеваний женщин. Заболеваемость и смертность от него продолжают неуклонно возрастать во всех странах мира, в связи с чем проблема рака молочной железы приобретает социальное значение. Применение современных методов лечения позволяет в 96 % добиться 5-летней выживаемости при РМЖ I стадии [1]. Однако у значительного числа больных диагностируется распространенный рак молочной железы. В ряде исследований продемонстрирована эффективность маммографии при раннем выявлении РМЖ, однако критики метода указывают на высокую частоту ложнонегативных и ложнопозитивных результатов [1]. Таким образом, актуальной проблемой молекулярной медицины является разработка раннего неинвазивного диагностического метода, обеспечивающего выявление РМЖ на асимптоматичной стадии.
В настоящее время ведутся разработки новых методов ранней диагностики рака, в том числе на основе анализа содержимого циркулирующих в крови экзосом [2]. К экзосомам относят внеклеточные везикулы диаметром 30–100 нм, секретируемые различными клетками во внеклеточное пространство [3]. В биологических жидкостях экзосомы являются стабильными структурами, при этом они несут уникальные белковые маркеры продуцирующих их клеток [2, 3], что определяет интерес к этим везикулам как к источнику диагностической информации [2, 4]. Действительно, анализ белкового состава циркулирующих в крови онкологических больных экзосом является перспективным направлением поиска опухолеспецифических маркеров злокачественных новообразований [5]. Несомненным преимуществом исследования протеома экзосом является возможность удалить мажорные белки плазмы крови и повысить концентрацию опухолеспецифических белков, в том числе и мембранных. Белки экзосом составляют менее 0,01 % общего протеома плазмы [6], и этого достаточно как для составления протеомных карт, так и для идентификации эк-зосомальных протеомных маркеров при помощи масс-спектрометрии.
Поскольку ранее было показано, что для осуществления межклеточной коммуникации экзо-сомы связываются с поверхностью клеток [7], мы предположили, что экзосомы могут быть связаны и с форменными элементами крови. Действительно, в пилотном исследовании нами получены данные о наличии экзосом на поверхности форменных элементов крови [8].
Целью исследования явился поиск канди-датных белковых онкомаркеров рака молочной железы путем сравнения 2D-протеомных карт суммарных экзосом крови здоровых женщин и больных РМЖ.
Материал и методы
Образцы крови 6 здоровых женщин, средний возраст 44 ± 4 года, были получены из Центральной клинической больницы СО РАН и 8 первичных больных РМЖ T1–2N0M0 – из Новосибирского областного онкологического диспансера (табл. 1). Исследование проводили с соблюдением принципов добровольности и конфиденциальности в соответствии с Основами законодательства РФ об охране здоровья граждан. Образцы крови после забора путем венепункции в вакутейнеры, содержащие К3EDTA, хранили при 4 °C и обрабатывали в течение ближайшего часа.
Суммарные экзосомы выделяли из крови здоровых женщин и больных РМЖ путем ультрафильтрации с последующим ультрацентрифугированием. В частности, к образцу крови объемом 4,5 мл добавляли равный объем буфера, элюирующий экзосомы, ассоциированные с форменными элементами [9], и инкубировали на ротационной мешалке 10 об/мин в течение 5 мин при комнатной температуре. Форменные элементы осаждали центрифугированием в течение 20 мин при 290 g и 4 °C, супернатант повторно центрифугировали в течение 20 мин при 1 200 g и 4 °C. Для удаления клеточного дебриса образцы плазмы центрифугировали при 17 000 g 4°C в течение 20 мин. Для удаления везикул размером более 100 нм супернатант разводили в 3 раза фосфатно-солевым буфером (10 мМ фосфатный буфер, 0,15 М NaCl, рН 7,5) (ФБ) и фильтровали через фильтр с диаметром пор 100 нм (Minisart high flow, 16553-K, Sartorius). Экзосомы осаждали ультрацентрифугированием (100 000 g, 90 мин, 4 °C), осадок ресуспендировали в 10 мл ФБ и дважды ультрацентрифугировали при тех же условиях. Экзосомы ресуспендировали в 200 мкл ФБ, замораживали в жидком азоте и хранили при –80 °С.
Для негативного контрастирования экзосомы сорбировали на медные сетки с формваровой подложкой, стабилизованной углеродом, в течение 1 мин и 10 сек контрастировали 2 % раствором фосфорно-вольфрамовой кислоты.
Для выявления специфических маркеров к 10 мкл суспензии экзосом добавляли 10 мкл 0,5 % бычьего сывороточного альбумина в ФБ, вносили по 3 мкл (100 мкг/мл) моноклональных антител к рецептору CD9 (Abcam) и инкубировали в течение 18 ч на шейкере Elpan 358S, затем сорбировали на сетки. Далее сетки промывали ФБ и инкубировали 2 ч с конъюгатом белка А и наночастиц золота во влажной камере при комнатной температуре, затем промывали ФБ в течение 2 мин и контрастировали фосфорно-вольфрамовой кислотой в течение 10 сек.
Подготовленные сетки изучали в просвечивающем электронном микроскопе JEM 1400 (Jeol, Japan) при ускоряющем напряжении 80 кВ. Изображения получали с помощью цифровой фотока- меры Veleta (ЕМ SIS, Germany). Размеры структур измеряли на мониторе цифровой фотокамеры с помощью программы iTEM (ЕМ SIS, Germany).
Для оценки концентрации белка экзосом использовали коммерческий набор NanoOrange Protein Quantitation kit (Invitrogen) в соответствии с рекомендациями производителя.
Для гель-электрофоретического разделения белки экзосом осаждали смесью хлороформа с метанолом (1:4). Для этого к 100 мкл экзосом добавляли 300 мкл деионизированной воды, 400 мкл метанола, 100 мкл хлороформа, тщательно перемешивали и центрифугировали при 14 000 g в течение 1 мин. Затем верхнюю водно-метанольную фазу отделяли, вносили 400 мкл метанола, тщательно перемешивали и центрифугировали при 20 000 g в течение 15 мин. Осадок высушивали на воздухе при комнатной температуре и растворяли в буфере для 2D-электрофореза [8].
Для получения протеомных карт экзосом использовали метод 2D-электрофореза [10]. Разделение белков методом 2D-электрофореза проводили с использованием стрипов с иммобилизованным градиентом (IPG-стрипы, 7 см), pH 4-7 (Bio-Rad Laboratories, США), как было описано ранее [8]. В качестве стандартной смеси белков для определения масс и pI использовали набор маркеров для 2D-электрофореза «Markers for Two Dimensional Electrophoresis» (1789 кДа, pI-спектр 7,6–3,8, Sigma). Визуализацию белковых пятен в гелях после завершения второго направления 2D-электрофореза осуществляли методом окрашивания нитратом серебра тиосульфатом натрия [11].
Для анализа окрашенные серебром гели сканировали с разрешением 300 точек на дюйм при помощи калибровочного денситометра GS-800 (Bio-Rad Laboratories, США) и статистически анализировали c использованием программного обеспечения PDQuest (version 8.0, Bio-Rad Laboratories, США). Анализ белковых пятен состоял из следующих этапов: вычисление относительного объема белковых пятен, рассчитанного как отношение выраженной в пикселях интенсивности пятна к суммарной интенсивности всех пятен на геле; выравнивание гелей; сопоставление белковых пятен. Достоверность полученных результатов оценивали по t-критерию Стьюдента при p<0,05.
Для масс-спектрометрической идентификации белков экзосом, по которым наблюдались отличия между исследуемыми группами, проводили их подготовку: вырезание из геля, деокрашивание от нитрата серебра, восстановление дисульфидных связей в молекуле белка и алкилирование остатков цистеина, расщепление белков на пептиды трипсином непосредственно в геле, экстракция пептидов из геля, очистка от солей и детергентов и концентрирование пептидов с помощью технологии ZipTip (фирмы «Milipore», США) согласно протоколу фирмы-производителя. Раз- деление полученных в результате триптического гидролиза пептидов проводили методом ВЭЖХ с использованием высокоэффективного жидкостного хроматографа Agilent 1290 Infinity LC System (фирмы «Agilent Technologies», Inc., США). Масс-спектрометрическую детекцию осуществляли на квадруполь-времяпролетном масс-спектрометре высокого разрешения Agilent 6550 iFunnel Q-TOF (фирмы «Agilent Technologies», Inc., США).
Для определения первичной последовательности экзосомальных белков, препаративные количества белков разделяли при помощи 10–20 % SDS-диск электрофореза и окрашивали при помощи Кумасси R-250 (Sigma, USA). Фрагменты ПААГ, содержащие исследуемые белки, отмывали от Кумасси R-250 и SDS и подвергали трипсинолизу, как было описано ранее [12]. Пептидные фрагменты белков экстрагировали из геля, концентрировали и обессоливали на микроколонках C18 ZipTips (фирмы «Milipore», США). Смесь пептидов элюировали с микроколонки на мишень приборной пластины насыщенным раствором матрикса.
Получение и регистрацию масс-спектров производили на тандемном времяпролетном масс-спектрометре MALDI-TOF autoflex speed series LIFT («Bruker Daltonics», ФРГ). Идентификацию белков проводили путём поиска соответствующих кандидатов в аннотированных базах данных NCBI и SwissProt с использованием программы Mascot (Matrix Science Ltd., London, www.matrixscience. com/, как было описано ранее [12]. Анализ идентифицированных белков проводили по методике Q. Yang et al. [13].
Результаты
Для поиска кандидатных белковых онкомаркеров РМЖ на первом этапе работы были сформированы группы клинически здоровых женщин и первичных больных РМЖ, выделены и охарактеризованы суммарные экзосомы крови.
Для подтверждения экзосомальной природы выделенных микровезикул «Обществом по изучению внеклеточных везикул» рекомендованы оценка размера и выявление экзосомальных мембранных или цитозольных белков [14]. Сочетание ультрафильтрации и двойного ультрацентрифугирования позволяет получить препараты везикул без примесей частиц более 100 нм, а форма и размер этих везикул соответствуют характеристикам экзосом, выделенных из других биологических жидкостей [15, 16]. В полученных нами препаратах основная часть везикул из крови здоровых женщин и онкологических больных имела размеры 40–100 нм и морфологические характеристики экзосом (рис. 1). Содержание везикул с поврежденной мембраной в препаратах не превышало 10 %, доля микровезикул (размером менее 30 нм) составила не более 15 %.
Для характеризации экзосом в текущей работе были использованы антитела к рецепторам семей- ства тетраспанинов CD9, которые опосредуют адгезию экзосом на поверхности клетки-реципиента и являются обязательным структурным компонентом мембраны экзосом [14]. Практически все суммарные экзосомы крови связывались с антителами к CD9, частицы золота выявлялись на их поверхности (рис. 1).
Для сравнительного анализа протеома суммарных экзосом крови здоровых женщин и больных РМЖ были получены 2D-протеомные карты (по три 2D-электрофореграммы/образец) (рис. 2). Было установлено, что в составе экзосом присутствуют белки с молекулярной массой от 10 до 250 кДа. Наиболее значимые различия между протеомными картами суммарных экзосом крови здоровых женщин и больных РМЖ были найдены в одиннадцати областях электрофоретической карты. Выявленные различия заключались в появлении/ исчезновении белков и изменении экспрессии присутствующих в норме белков. В частности, наблюдалось появление белковых пятен (области 2, 9) и исчезновение пятна (область 1 (II)) при РМЖ. Белковые пятна (области 1 (I), 3, 4, 5, 6, 7, 8, 10, 11) имели больший относительный объем на протеомных картах больных РМЖ по сравнению с контрольной группой (рис. 2).
Идентифицированные при помощи пептидного фингерпринта экзосомальные белки наиболее отличающихся 11 областей протеомных карт представлены в табл. 2. Ранее в составе экзосом методом масс-спектрометрии уже были обнаружены 11 из 13 (85 %) идентифицированных нами белков, они аннотированы в базе данных Exocarta (www. . Лейцин-богатый γ-2-гликопротеин (LRG) и α-цепь фибриногена (FGB) обнаружены нами в составе экзосом впервые.
Ранее методом MALDI-TOF масс-спектрометрии были идентифицированы 54 и 45 белков в препара-
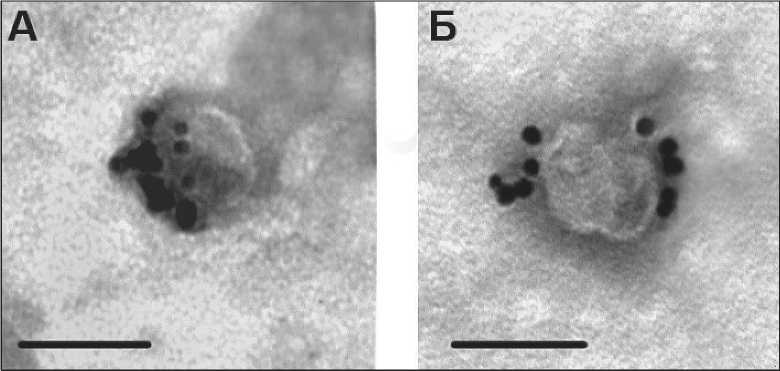
Рис. 1. Морфологическая характеристика суммарных экзосом, выделенных из крови здоровых женщин (а) и больных РМЖ (б). Экзосомы мечены антителами к рецептору CD9. Длина масштабной линии соответствует 100 нм. Трансмиссионная электронная микроскопия, негативное контрастирование фосфорновольфрамовой кислотой
Fig. 1. Morphological characteristics of total exosomes isolated from the blood of healthy women (a) and patients with breast cancer (b). Exosomes are labeled with antibodies to the CD9 receptor. The length of the scale line corresponds to 100 nm. Transmission electron microscopy, negative contrast with phosphoric tungsten acid
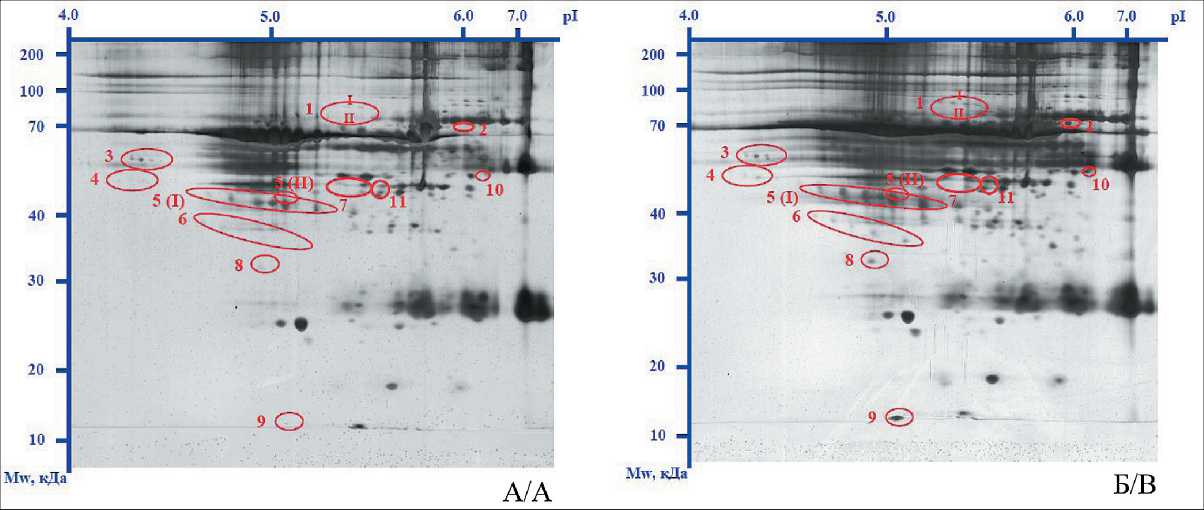
Рис. 2. Типичные протеомные карты суммарных экзосом крови здоровых женщин (а) и больных раком молочной железы (б). Двумерная электрофореграмма окрашена серебром. По оси абсцисс – изоэлектрическое фокусирование, по оси ординат – SDS-ПААГ-электрофорез. Молекулярные массы белков в кДа приведены слева. В окружности заключены белки, по относительному объему и наличию которых наблюдались отличия между исследуемыми группами
Fig. 2. Typical proteomic maps of the total blood exosomes of healthy women (a) and patients with breast cancer (b). Two-dimensional electrophoregram is painted silver. The abscissa axis is isoelectric focusing, the ordinate axis is SDS-PAGE electrophoresis. Molecular weights of proteins in kDa are given on the left. In the circle are proteins, according to the relative volume and presence of which differences were observed between the studied groups
Таблица 1/Table 1
Характеристика больных РМЖ Characteristics of patients with breast cancer
|
Параметр/Parameter |
Количество больных/ Number of patients |
|
|
Люминальный подтип/Lumina |
l subtype |
6 (75 %) |
|
Трижды-негативный подтип/ Triple negative subtype |
2 (25 %) |
|
|
Ki67 10 |
–15 % |
8 (100 %) |
|
T1 |
4 (50 %) |
|
|
Стадия/ |
T2 |
4 (50 %) |
|
Tumor stage |
N0 |
8 (100 %) |
|
M0 |
8 (100 %) |
|
|
Степень злокачественности/ Tumor grade |
II |
8 (100 %) |
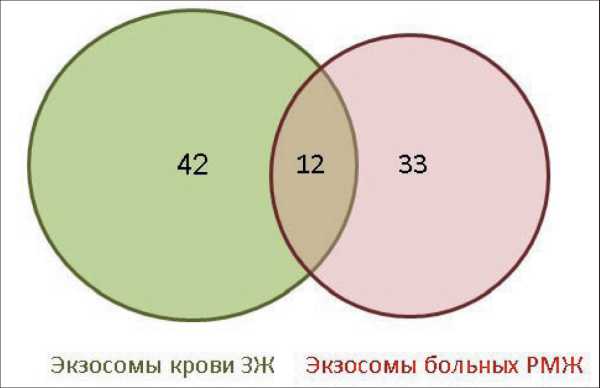
Рис 3. Диаграмма Венна – Эйлера идентифицированных белков суммарных экзосом крови
Fig. 3. Venn-Euler diagram of identified proteins of total blood exosomes
|
Р02787 |
6/6 |
|
Р02647 |
5/9 |
|
Р02671 |
3/1 |
|
Р04259 |
1/0 |
|
095613 |
1/0 |
|
043399 |
2/0 |
|
Р02765 |
3/2 |
|
Р02750 |
0/1 |
|
Р00738 |
10/6 |
|
Р00739 |
1/2 |
|
Р06727 |
2/1 |
|
Р10909 |
1/3 |
|
Р02679 |
3/2 |
|
Р02760 |
1/2 |
|
Р02790 |
5/4 |
|
Р02766 |
3/7 |
|
Р69905 |
2/2 |
TF
APOA1
FGA
KRT6B
PCNT
TPD52L2
AHSG
LRG1
HP
HPR
APOA4
CLU
FGG
AMBP
HPX
TTR
HBA2
Р02768 4/5 0/3 8/2 2/3 6/7 1/2 0/1 2/2 3/5 1/4
P02787 2/2 0/1 6/8 2/1 6/6
P02647 1/72/1 5/4 1/4 5/9
P02671 1/3
Q14005
P02538 4/1 1/0
3/4 2/1 3/1
1/2
Р04259 0/1 1/0 1/0 0/1 095613
Q9NZU7 Q15024 014543
Q14320 |/0
Q16775
Q13424
P16233
P02765
2/1
1/0
o/i ШМ6Я
2/3 0/2 5/4
1/0 0/1 3/1
1/0
1/0 1/0
2/0
щи
PO275O 1/2 1/0 4/0 1/1 0/1
P00738 3/0 0/2 4/5 3/3 10/6
P06727
2/1 1/2
0/1 Ш|2/1
Р10909 2/0 0/1 3/1 1/3 1/3
Р02679 2/0 1/0 3/1 1/0 3/2
Р02760 0/2 0/1 0/1 1/2
Р02766 1/1 0/1 3/3 0/2 3/7
Р00738 3/0 0/2 4/5 3/3 10/6
О ™ та Q Ф
1/0 0/1
шин
0/5
2/0 3/0 1/0 0/1 1/0 2/1
ШШ 1/о
1/0 2/6 1/1 2/3 5/0
4/0
ЙН —ii/o
1/0
0/1
0/1
1/0 1/0
1/0 2/0 5/0 1/0
0/2 1/0 2/0 10/0
0/1 1/0
1/0 1/3
0/1 2/2 1/1
2/0
0/2
1/0
1/0
1/0
1/0
1/0
2/2 3/0 0/1 1/0 1/2 0/1 0/1
Ф Ф <и Ф
о
П5
Ф
Ф
Ф
2/0
E о
Ф
E о
о
E
£ и га Ф " > и $ О
О
ru
Ф
Ф
1/3 0/1 3/1
ВТ 170 В® 1®Й 1/0 1/2
1/0
1/0
1/0
0/1
ГО E о
E ,E
1/0
1/2 2/5 3/0 2/0
0/1 0/1 0/2 1/1
1/0
0/1 4/1 1/0
0/1
0/1 ш 1/1 2/2 0/2
2/3 1/1 0/2
1/0 2/0
Ф
ЛЗ
Ф
1/0
1/0
0/1
1/0
0/1
1/0
0/1
вшши
1/1 4/40/1
1 /2 4/|зСДЦЙ
5: та га та v> 8 8 Е Е Е Е с с о о о Я га га с —
E
0/1 та
E о
ALB TF AP0A1
FGA IL16 KRT6A KRT6B PCNT TPD52L2 CABP1 EXOSC7
SOC53 FAM50A
HAGH SNTA1 PNLIP AHSG LRG1
HP HPR APOA4
CLU FGG AMBP HPX TTR
HP HBA2
Ф
Ф
та
ф
та о с ф
Ф
О Ф
о Ф
о
Ф
Ф
Ф
A/A
Б/В
Рис. 4. Тепловая карта идентифицированных белков в составе суммарных экзосом крови больных РМЖ, ассоциированных с развитием злокачественных новообразований (a) и РМЖ в частности (б)
Fig. 4. The heat map of the identified proteins as a part of the common blood exosomes of breast cancer associated with the development of malignant neoplasms (a) and breast cancer in particular (b)
Таблица 2/Table 2
Идентифицированные при помощи пептидного фингерпринта белки суммарных экзосом крови Total blood exosome proteins identified with peptide fingerprint
|
Номер пятна/ Spot number |
Номер белка в базе Uniprot/ Protein number in Uniprot base |
Название белка/ Protein name |
Теоретический мол вес белка, Da/ Theoretical protein weight (Da) |
Теоретическая изоэлектрическая точка белка/ Theoretical isoelectric point of protein |
|
1 (I) |
P02768 |
Serum albumin |
71362 |
5,92 |
|
1 (II) |
P06396 |
Gelsolin |
86095 |
5,90 |
|
2 |
P02768 |
Serum albumin |
71362 |
5,92 |
|
3 |
P02765 |
Alpha-2-HS-glycoprotein |
40123 |
5,43 |
|
4 |
P02750 |
Leucine-rich alpha-2-glycoprotein |
38405 |
6,46 |
|
5 (I) |
P00738 |
Haptoglobin beta chain |
27265 |
6,32 |
|
5 (I) |
P00739 |
Haptoglobin-related protein |
39029 |
6,63 |
|
5 (II) |
P06727 |
Apolipo-protein A-IV |
45398 |
5,28 |
|
6 |
P10909 |
Clusterin |
53064 |
5,89 |
|
7 (I) |
P02679 |
Fibrinogen gamma chain |
52138 |
5,37 |
|
7 (II) |
P02768 |
Serum albumin |
71362 |
5,92 |
|
8 |
P02760 |
Alpha-1-microglycoprotein |
20846 |
6,13 |
|
9 |
P00738 |
Haptoglobin alpha chain |
15945 |
5,57 |
|
9 |
P02766 |
Transthyretin |
13761 |
5,31 |
|
10 |
P02790 |
Hemopexin |
52417 |
6,55 |
Таблица 3/Table 3
Идентифицированные при помощи MALDI-TOF масс-спектрометрии белки суммарных экзосом крови здоровых женщин
Total blood exosome proteins identified by MALDI-TOF mass-spectrometry in healthy women
|
Номер белка в базе/ Gene Name |
Название белка/ Protein name |
Аббревиатура белка/ Protein Abbreviation |
Наличие в базе данных Exo-Carta/ ExoCarta Database Availability |
|
A0M8Q6 |
Immunoglobulin lambda constant 7 |
IGLC7 |
+ |
|
A6NCL7 |
Ankyrin repeat domain-containing protein 33B |
ANKRD33B |
– |
|
A8MU93 |
Uncharacterized protein C17orf100 |
C17orf100 |
– |
|
H0YGS3 |
Microfibrillar-associated protein 5 |
MFAP5 |
+ |
|
O14862 |
Interferon-inducible protein AIM2 |
AIM2 |
– |
|
O15054 |
Lysine-specific demethylase 6B |
KDM6B |
+ |
|
O15520 |
Fibroblast growth factor 10 |
FGF10 |
– |
|
O60832 |
H/ACA ribonucleoprotein complex subunit 4 |
DKC1 |
+ |
|
O95602 |
DNA-directed RNA polymerase I subunit RPA1 |
POLR1A |
– |
|
O95831 |
Apoptosis-inducing factor 1 |
AIFM1 |
- |
|
P00738 |
Haptoglobin beta chain |
HP |
+ |
|
P00739 |
Haptoglobin-related protein |
HPR |
– |
|
P01871 |
Ig mu chain C region |
IGHM |
+ |
|
P02545 |
Prelamin-A/C |
LMNA |
+ |
|
P02679 |
Fibrinogen gamma chain |
FGG |
+ |
|
P02750 |
Leucine-rich alpha-2-glycoprotein |
LRG1 |
– |
|
P02760 |
Alpha-1-microglycoprotein |
AMBP |
+ |
|
P02765 |
Alpha-2-HS-glycoprotein |
AHSG |
+ |
|
P02766 |
Transthyretin |
TTR |
+ |
|
P02768 |
Serum albumin |
ALB |
+ |
Окончание таблицы 3/End Table 3
|
P02787 |
Serotransferrin |
TF |
– |
|
P02790 |
Hemopexin |
HPX |
+ |
|
P05976 |
Myosin light chain 1/3 |
MYL1 |
+ |
|
P06396 |
Gelsolin |
GSN |
– |
|
P06727 |
Apolipo-protein A-IV |
APOA4 |
+ |
|
P0DOX6 |
Immunoglobulin mu heavy chain |
IGHM |
+ |
|
P10909 |
Clusterin |
CLU |
+ |
|
P11801 |
Serine/threonine-protein kinase H1 |
PSKH1 |
– |
|
P23109 |
AMP deaminase 1 |
AMPD1 |
– |
|
P26440 |
Isovaleryl-CoA dehydrogenase |
IVD |
– |
|
P31327 |
Carbamoyl-phosphate synthase [ammonia] |
CPS1 |
+ |
|
P31749 |
RAC-alpha serine/threonine-protein kinase |
AKT1 |
+ |
|
P48506 |
Glutamate-cysteine ligase catalytic subunit |
GCLC |
– |
|
P49748 |
Very long-chain specific acyl-CoA dehydrogenase |
ACADVL |
– |
|
P49763 |
Placenta growth factor |
PGF |
– |
|
P53674 |
Beta-crystallin B1 |
CRYBB1 |
– |
|
P61224 |
Ras-related GTP-binding protein B |
RAP1B |
+ |
|
P78356 |
Phosphatidylinositol 4-kinase type 2-beta |
PIP4K2B |
– |
|
Q08426 |
Peroxisomal bifunctional enzyme |
EHHADH |
+ |
|
Q11201 |
CMP-N-acetylneuraminate-beta-galactosamide-alpha-2,3-sialyltransferase 1 |
ST3GAL1 |
– |
|
Q49A33 |
Putative zinc finger protein 876 |
ZNF876P |
– |
|
Q49MG5 |
Microtubule-associated protein |
MAP9 |
– |
|
Q4LEZ3 |
Alanine and arginine-rich domain-containing protein |
AARD |
– |
|
Q504T8 |
Midnolin |
MIDN |
– |
|
Q69YQ0 |
Cytospin-A |
SPECC1L |
+ |
|
Q6P1J9 |
Parafibromin |
CDC73 |
– |
|
Q6ZS02 |
Putative GED domain-containing protein |
DNM1P46 |
– |
|
Q7Z553 |
MAM domain-containing glycosylphosphatidylinositol anchor protein 2 |
MDGA2 |
– |
|
Q86VE0 |
Myb-related transcription factor, partner of profilin |
MYPOP |
– |
|
Q8N8C0 |
Zinc finger protein 781 |
ZNF781 |
– |
|
Q9H6Z4 |
Ran-binding protein 3 |
RANBP3 |
+ |
|
Q9HBI5 |
Uncharacterized protein C3orf14 |
C3orf14 |
– |
|
Q9UK05 |
Growth/differentiation factor 2 |
GDF2 |
+ |
Примечание: курсивом отмечены универсальные белки.
Notes: italics indicate universal proteins.
Таблица 4/Table 4
Идентифицированные при помощи MALDI-TOF масс-спектрометрии белки суммарных экзосом крови больных РМЖ
Total blood exosome proteins identified by MALDI-TOF mass-spectrometry in patients with breast cancer
|
Номер белка в базе/ Gene Name |
Название белка/ Protein name |
Аббревиатура белка/ Protein Abbreviation |
Наличие в базе данных ExoCarta/ Availability in ExoCarta Database |
|
O14543 |
Suppressor of cytokine signaling 3 |
SOCS3 |
- |
|
O15050 |
TPR and ankyrin repeat-containing protein 1 |
TRANK1 |
+ |
Окончание таблицы 4/End Table 4
|
O43399 |
Tumor protein D54 |
TPD52L2 |
+ |
|
O95243 |
Methyl-CpG-binding domain protein 4 |
MBD4 |
- |
|
O95613 |
Pericentrin |
PCNT |
- |
|
P69905 |
Haptoglobin alpha chain |
HBA1 |
+ |
|
P00738 |
Haptoglobin beta chain |
HP |
+ |
|
P00739 |
Haptoglobin-related protein |
HPR |
- |
|
P02538 |
Cytokeratin-6A |
KRT6A |
+ |
|
P02647 |
Apolipoprotein A-I |
APOA1 |
+ |
|
P02671 |
Fibrinogen alpha chain |
FGA |
+ |
|
P02679 |
Fibrinogen gamma chain |
FGG |
+ |
|
P02750 |
Leucine-rich alpha-2-glycoprotein |
LRG1 |
- |
|
P02760 |
Alpha-1-microglycoprotein |
AMBP |
+ |
|
P02765 |
Alpha-2-HS-glycoprotein |
AHSG |
+ |
|
P02766 |
Transthyretin |
TTR |
+ |
|
P02768 |
Serum albumin |
ALB |
+ |
|
P02787 |
Serotransferrin |
TF |
- |
|
P02790 |
Hemopexin |
HPX |
+ |
|
P04259 |
Cytokeratin-6B |
KRT6B |
+ |
|
P06727 |
Apolipo-protein A-IV |
APOA4 |
+ |
|
P10909 |
Clusterin |
CLU |
+ |
|
P16233 |
Pancreatic triacylglycerol lipase |
PNLIP |
- |
|
P55199 |
RNA polymerase II elongation factor |
ELL |
- |
|
P62987 |
Ubiquitin-60S ribosomal protein L40 |
UBA52 |
- |
|
Q13424 |
Alpha-1-syntrophin |
SNTA1 |
+ |
|
Q13522 |
Protein phosphatase 1A |
PPP1R1A |
+ |
|
Q14005 |
Pro-interleukin-16 |
IL16 |
- |
|
Q14320 |
Protein FAM50A |
FAM50A |
- |
|
Q15024 |
Exosome complex component |
EXOSC7 |
+ |
|
Q15776 |
Zinc finger protein with KRAB and SCAN domains 8 |
ZKSCAN8 |
- |
|
Q16775 |
Hydroxyacylglutathione hydrolase |
HAGH |
+ |
|
Q4G0S7 |
Coiled-coil domain-containing protein 152 |
CCDC152 |
+ |
|
Q52M93 |
Zinc finger protein 585B |
ZNF585B |
- |
|
Q5M9N0 |
Coiled-coil domain-containing protein 158 |
CCDC158 |
+ |
|
Q68J44 |
Dual specificity phosphatase |
DUPD1 |
- |
|
Q8IUS5 |
Epoxide hydrolase 4 |
EPHX4 |
- |
|
Q8IYE0 |
Coiled-coil domain-containing protein 146 |
CCDC146 |
- |
|
Q8N9H8 |
Exonuclease mut-7 homolog |
EXD3 |
- |
|
Q8NDD1 |
Uncharacterized protein C1orf131 |
C1orf131 |
- |
|
Q8NEQ6 |
Steroid receptor-associated and regulated protein |
SRARP |
- |
|
Q8WXS5 |
Voltage-dependent calcium channel gamma-8 subunit |
CACNG8 |
- |
|
Q9NZU7 |
Calcium-binding protein 1 |
CABP1 |
+ |
|
Q9UKW4 |
Guanine nucleotide exchange factor VAV3 |
VAV3 |
- |
|
Q9Y4E5 |
E3 SUMO-protein ligase ZNF451 |
ZNF451 |
+ |
Примечание: курсивом отмечены универсальные белки.
Notes: italics indicate universal proteins.
тах суммарных экзосом крови здоровых женщин и больных РМЖ соответственно, из них 12 белков являются универсальными (рис. 3). Ранее в составе экзосом методом масс-спектрометрии уже были обнаружены 35 % идентифицированных нами белков, они аннотированы в базе данных Exocarta ( (табл. 3, 4). С использованием базы данных dbDEPC 3.0 (database of Differently Expressed Proteins in Human Cancer) [13] установлено, что в составе суммарных экзосом крови больных РМЖ 28 (88 %) белков ассоциировано со злокачественными новообразованиями (рис. 4а), из них 17 (53 %) – с развитием РМЖ (рис. 4б). Согласно литературным данным, из идентифицированных с помощью MALDI-TOF масс-спектрометрии белков, ассоциированных с развитием РМЖ, гипер-экспрессированными являются 9 (53 %) белков, гипоэкспрессированными – 6 (35 %), экспрессия не изменяется у 2 (12 %) белков.
Обсуждение
Малоинвазивная процедура взятия венозной крови представляет альтернативу традиционной биопсии. Именно поэтому на сегодняшний день анализ белкового состава циркулирующих в крови экзосом является перспективным направлением поиска опухолеспецифичных маркеров злокачественных новообразований.
Представленная работа является пилотной в поиске протеомных опухоле-ассоциированных маркеров в составе суммарных экзосом крови, пул которых формируется из экзосом плазмы и экзо-сом, ассоциированных с поверхностью форменных элементов. В литературе широко представлены данные по поиску протеомных опухолевых маркеров в составе экзосом плазмы [17, 18], обсуждаются достоинства использования белков экзосом плазмы по сравнению с белками плазмы [19], однако данные об ассоциированных с клеточной поверхностью экзосомах немногочисленны. На сегодняшний день известно, что экзосомы несут на своей поверхности специфические рецепторы, которые обеспечивают их взаимодействие с клетками-мишенями. В то же время клетки различных типов могут связывать и интернализовать циркулирующие экзосомы [20–23]. Пул циркулирующих в крови экзосом формируют экзосомы, секретируемые лейкоцитами, эритроцитами, тромбоцитами и эндотелиоцитами (т.е. либо форменные элементы крови или клетки, непосредственно
Список литературы Поиск протеомных маркеров рака молочной железы в составе суммарных экзосом крови
- Witten M., Parker C.C. Screening mammography: recommendations and controversies. Surg Clin North Am. 2018 Aug; 98(4): 667-75. 10.1016/j.suc.2018.03.003.Tamkovich S.N., Tutanov O.S., Laktionov P.P. Exosomes: generation DOI: 10.1016/j.suc.2018.03.003.TamkovichS.N
- structure, transport, biological activity, and diagnostic application. Biochemistry (Moscow), Supplement Series A: Membrane and Cell Biology. 2016 Oct; 10(3): 163-73. DOI: 10.1134/S1990747816020112
- Yanez-Mo M., Siljander P.R., Andreu Z., Zavec A.B., Borra F.E., Buzas E.I., Buzas K., Casal E., Cappello F., Carvalh o J., Cola E., Silva A.C., Fais S., Falcon-Perez J.M., Ghobrial I.M., Giebel B., Gimona M., Graner M., Gursel I., Gursel M., Heegaard N.H.H., Hendrix A., Kierulf P., Kokubun K., Kosanovic M., Kralj-Iglic V., Kramer-Albers E.-M, Laitinen S., Lasser S., Lener T., Ligeti E., Line A., Lipps G., Llorente A., Lotvall J., Mancek-Keber M., Marcilla A., Mittelbrunn M., Nazarenko I., Hoen E.N.M., Nyman T.A., O'Driscoll L., Olivan M., Oliveira C., Pallinger E., del Portillo H.A., Reventos J., Rigau M., Rohde E., Sammar M., Sanchez-Madrid F., Santarem N., Schallmoser K., Ostenfeld M.S., Stoorvogel W., Stukelj R., Van der Grein S.G., Vasconcelos M.H., Wauben M.H.M., De Wever O. Biological properties of extracellular vesicles and their physiological functions. J Extracell Vesicles. 2015 May; 4: 27066. DOI: 10.3402/jev.v4.27066
- Aghebati-Maleki A., Nami S., Baghbanzadeh A., Karzar B.H., Noorolyai S., Fotouhi A., Aghebati-Maleki L. Implications of exosomes as diagnostic and therapeutic strategies in cancer. J Cell Physiol. 2019 Jun. DOI: 10.1002/jcp.28875
- Clark D.J., Fondrie W.E., Liao Z., Hanson P.I., Fulton A., Mao L., Yang A.J. Redefining the breast cancer exosome proteome by tandem mass tag quantitative proteomics and multivariate cluster analysis. Anal Chem. 2015 Oct; 87(20): 10462-9. DOI: 10.1021/acs.analchem.5b02586


