Полиморфизм генов, ассоциированных с локусами количественных признаков, у кабана ( Sus scrofa L., 1758), обитающего на территории России
Автор: Зиновьева Н.А., Костюнина О.В., Экономов А.В., Шевнина М.С., Домский И.А., Гладырь Е.А., Брем Г.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Биоресурсы: геномное сканирование популяций
Статья в выпуске: 2 т.48, 2013 года.
Бесплатный доступ
Проведены генетические исследования кабанов ( n = 89) по десяти ДНК-маркерам — RYR 1, ESR, FSHB, NCOA 1, BF, MU С4, IGF 2, MC 4 R, POU 1 F 1, ECRF 18/ FUT 1. Отнесение исследуемых особей к различным территориальным кластерам было выполнено на основании расчета коэффициента подобия (Q) при k = 2. Среднее значение Q у животных, обитающих в европейской части и в Западной Сибири, составило 0,984±0,005, у особей из Иркутской области и Хабаровского края — 0,994±0,001. Пять ДНК-маркеров ( RYR 1, ESR, MUC 4, IGF 2, ECRF 18/ FUT 1 ) оказались мономорфными в обоих территориальных кластерах. Установлены незначительные различия в частоте аллелей между особями, входящими в условно западный и восточный кластеры, по ДНК-маркерам FSHB (p A равно соответственно 0,462 и 0,250), BF (p A — 0,020 и 0,143), MC 4 R (p A —0,013 и 0,000) и POU 1 F 1 (p C — 0,000 и 0,111). Достоверные различия между исследуемыми группами отмечены по частотам аллелей NCOA 1 : p A1 — 0,938 у кабанов, отнесенных к условно западному кластеру, и p A1 = 0,000 — к восточному.
Кабаны, днк-маркеры, полиморфизм, muс4
Короткий адрес: https://sciup.org/142133397
IDR: 142133397 | УДК: 639.1:599.731.11:631.523.5:577.21
Текст научной статьи Полиморфизм генов, ассоциированных с локусами количественных признаков, у кабана ( Sus scrofa L., 1758), обитающего на территории России
Кабан — широко распространенный представитель дикой фауны. На основании фенотипических различий вид Sus scrofa L., 1758 был разделен на четыре расы (западную, восточную, индийскую и индонезийскую), которые, в свою очередь, включают 16 (1), а по некоторым данным, около 27 подвидов (2, 3).
В России, по материалам А.А. Данилкина (4), обитает пять подвидов кабана: центрально-европейский ( S. s. scrofa L., 1758), кавказский ( S . s . attila Thorn, 1912), сибирский ( S. s. sibiricus Staffe, 1922), среднеазиатский ( S. s. nigripes Blanf, 1875), уссурийский ( S. s. ussuricus Heude, 1888). Четких границ между вышеназванными подвидами не существует, что связано с естественным расселением животных, сезонными кочевками, обусловленными кормовыми ресурсами, паводками, динамикой высоты снегового покрова, высотными миграциями и т.д. Один из важнейших факторов изменения генетической структуры подвидов — работы по акклиматизации. Например, считалось, что уссурийский подвид наиболее крупный, в результате его пытались расселять в европейской части России, в частности завозили в заповедно-охотничье хозяйство «Завидово» (Тверская обл.) (5, 6). Подобная практика имеет место и в настоящее время.
В последние годы активно ведутся работы по изучению генетического разнообразия популяций диких свиней, в том числе на территории Российской Федерации (7-9), и указанное направление исследований вызывает как научный, так и практический интерес.
Работа выполнена при финансовой поддержке Министерства образования и науки РФ, шифр 2012-1.412-000-1016-008 и 2012-1.4-12-000-1001-010. В проведении исследований использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ГНУ ВИЖ Россельхозакадемии.
Развитие молекулярной генетики открывает новые возможности в характеристике групп животных и их генетической дифференциации. Одно из направлений при исследовании популяций кабанов заключается в оценке степени интрогрессии домашней свиньи вследствие случайной гибридизации. С этой целью применяются различные типы ДНК-маркеров: мтДНК (10), микросателлиты (11), SNP-маркеры (12, 13), ДНК-маркеры, ассоциированные с локусами количественных признаков (14). Как доказательство наличия домашних свиней среди предков кабанов по материнской линии можно рассматривать присутствие азиатского гаплотипа (А) мтДНК, встречающегося в породах домашних свиней с частотой 29 % (10), вследствие исторической интрогрессии в XVII-XIX в.в. от китайских свиней (15), в то время как у «чистых» популяций европейского кабана проявляются только гаплотипы E1 и E2. В качестве одного из критериев интрогрессии домашних свиней в популяции кабана также используют частоту генов, ассоциированных с локусами количественных признаков (QTL). A. Ojeda с соавт. (14) установили, что частота аллеля Q по IGF2 (мутация G3072A), ассоциированного с усиленным ростом мышечной ткани и мясностью, в заводских породах свиней составляет 86 %, в локальных — 3 % и отсутствует у 120 протестированных европейских кабанов. Y. Kuril с соавт. (16) охарактеризовали полиморфизм генов RYR1 , LEP , GH , MYOG , MYF5 и GDF8 в популяции кабанов, обитающей на северо-востоке Польши. Если гены RYR1 , LEP и MYF5 оказались мономорфными, то для MYOG , GH и GDF8 обнаружили полиморфизм с частотой вариантов соответственно 0,42 (аллель А ) и 0,58 (аллель В ), 0,56 (аллель «+») и 0,44 (аллель « - »), 0,63 (аллель С) и 0,37 (аллель Т ). Показано, что в азиатской и европейской популяциях сегрегируют разные аллели MC1R , обусловливающие окрас и ответственные за различия по цвету шкуры между кабанами и домашними свиньями (6), что было положено в основу при изучении интрогрессии в греческой популяции (7).
Цель настоящих исследований заключалась в характеристике полиморфизма генов, ассоциированных с QTL, у разных групп кабанов, обитающих на территории России.
Методика . Биологическим материалом для исследований служили образцы мышечной ткани диких свиней, обитающих на территории России ( n = 89), отобранные в Архангельской области ( n = 3), Башкирии ( n = 3), Владимирской ( n = 3), Волгоградской ( n = 5), Вологодской ( n = 2), Ивановской ( n = 3), Иркутской ( n = 2), Калининградской ( n = 3), Кировской ( n = 8) областях, в Республике Коми ( n = 1), Краснодарском крае ( n = 1), Курганской ( n = 3), Курской ( n = 3), Ленинградской областях ( n = 8), в Республике Марий-Эл ( n = 3), Нижегородской ( n = 4), Омской ( n = 3), Оренбургской ( n = 3), Пензенской ( n = 3), Саратовской ( n = 1), Свердловской ( n = 3), Смоленской ( n = 3), Тамбовской областях ( n = 4), в Республике Татарстан ( n = 1), Тверской области ( n = 3), в Удмуртской Республике ( n = 2), Хабаровском крае ( n = 7) и в Чувашской Республике ( n = 1).
Выделение ДНК проводили на колонках Nexttec и с использованием набора реагентов для выделения ДНК DIAtomTM DNA Prep100 (ООО «Компания «Биоком», Россия) в соответствии с рекомендациями фирмы-изготовителя.
Принадлежность исследуемых животных к генетически дифференцированным кластерам определяли на основании анализа 12 микросателлитов (17) с последующим анализом кластерной структуры исследуемой выборки по методу J.K. Pritchard с соавт. (18). Обработку данных проводили с использованием программного обеспечения Structure (v. 2.3.1) без введения предварительной информации о популяционной принадлежности особей для числа популяций (K-кластеров), равного 2. В качестве ДНК-маркеров QTL использовали гены рианодинового рецептора (RYR1), эстрогенового рецептора (ESR), в-субъединицы фолликулостимулирующего гормона (FSHB), коактиватора стероидных гормонов (NCOA1), пропердина (BF), муцина 4 (MUC4), инсулиноподобного фактора роста 2 (IGF2), рецептора меланокортина 4 (MC4R), гипофизарного транскрипционного фактора (POU1F1), рецептора Escherichia coli (ECRF18/FUT1). При определении полиморфизма по перечисленным генам применяли методики, разработанные в Центре биотехнологии и молекулярной диагностики (Всероссийский НИИ животноводства).
Информацию об аллелях каждого животного суммировали в электронной таблице Microsoft Excel. Полученная матрица генотипов служила основой для статистической обработки результатов, которую осуществляли по стандартным методикам (19, 20).
Результаты . Данные анализа генетической структуры исследуемой выборки кабанов при k = 2 (рис. 1) выявили четкую генетическую дифференциацию, обусловливающую формирование двух кластеров: условно западного (далее первый) и восточного (второй). Первый включает в себя животных из Центральной России, Кавказа, Урала и Западной Сибири, второй — из Хабаровского края и Иркутской области.
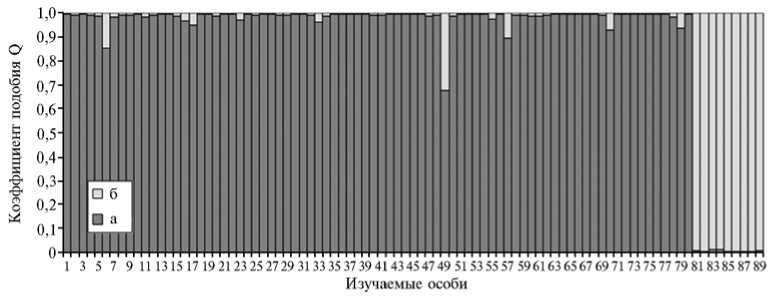
Рис. 1. Генетическая структура изучаемой выборки дикого кабана ( Sus scrofa L., 1758) по 10 ДНК-маркерам QTL : 1-3 — особи из Архангельской области, 4-6 — из Башкирии, 7-9 — Владимирской области, 10-14 — Волгоградской области, 15-16 — Вологодской области, 1719 — Ивановской области, 20-22 — Калининградской области, 23-30 — Кировской области, 31 — Республики Коми, 32 — Краснодарского края, 33-35 — Курганской области, 36-38 — Курской области, 39-46 — Ленинградской области, 47-49 — Республики Марий-Эл, 50-53 — Нижегородской области, 54-56 — Омской области, 57-59 — Оренбургской области, 60-62 — Пензенской области, 63 — Саратовской области, 64-66 — Свердловской области, 67-69 — Смоленской области, 70-73 — Тамбовской области, 74 — Татарстана, 75-77 — Тверской области, 78-79 — Удмуртии, 80 — Чувашии, 81-82 — Иркутской области, 83-89 — Хабаровского края; а — Q 1 , б — Q2. Расчеты выполнены по методу J.K. Pritchard с соавт. (18) для k = 2. Описание маркеров см. в разделе «Методика».
Среднее значение коэффициента подобия (Q) у особей, обитающих на европейской территории и в Западной Сибири, в первом кластере составило Q1 = 0,984±0,005 с индивидуальными вариациями от 0,679 до 0,998, при этом у 78 из 80 исследованных животных значения Q1 оказались выше 0,900 и лишь у двух особей из Республики Марий-Эл и Башкирии составили соответственно 0,679 и 0,855, что позволяет предполагать у них интрогрессию от предков из второго кластера. Среднее значение Q у животных, которые обитали на территории Иркутской области и Хабаровского края, во втором кластере составило Q2 = 0,994±0,001 с вариациями от 0,989 до 0,997. Таким образом, полученные данные позволяют сделать вывод о достоверных генетических различиях изучаемых особей, отнесенных к условно западному и восточному кластерам.
Распределение генотипов по ДНК-маркерам QTL в изучаемой выборке кабанов ( Sus scrofa L., 1758)
|
Генотип, кластер |
1 Частота генотипа, % |
||
|
По маркеру RYR1 |
|||
|
Генотип |
NN |
Nn |
nn |
|
в условно западном кластере |
100,0 |
0,0 |
0,0 |
|
в восточном кластере |
100,0 |
0,0 |
0,0 |
|
По маркеру ESR |
|||
|
Генотип |
ÀÀ |
ÀÂ |
ÂÂ |
|
в условно западном кластере |
100,0 |
0,0 |
0,0 |
|
в восточном кластере |
100,0 |
0,0 |
0,0 |
|
По маркеру FSHB |
|||
|
Генотип |
ÀÀ |
ÀÂ |
ÂÂ |
|
в условно западном кластере |
32,9 |
26,6 |
40,5 |
|
в восточном кластере |
0,0 |
50,0 |
50,0 |
|
По маркеру NCOA1 |
|||
|
Генотип |
A1A1 |
A1A2 |
A2A2 |
|
в условно западном кластере |
87,5 |
12,5 |
0,0 |
|
в восточном кластере |
0,0 |
0,0 |
100,0 |
|
По маркеру BF |
|||
|
Генотип |
ÀÀ |
ÀÂ |
ÂÂ |
|
в условно западном кластере |
1,4 |
1,4 |
97,3 |
|
в восточном кластере |
0,0 |
28,6 |
71,4 |
|
По маркеру MUC4 |
|||
|
Генотип |
CC |
CG |
GG |
|
в условно западном кластере |
100,0 |
0,0 |
0,0 |
|
в восточном кластере |
100,0 |
0,0 |
0,0 |
|
По маркеру IGF2 |
|||
|
Генотип |
|
|
|
|
в условно западном кластере |
100,0 |
0,0 |
0,0 |
|
в восточном кластере |
100,0 |
0,0 |
0,0 |
|
По маркеру MC4R |
|||
|
Генотип |
AA |
AG |
GG |
|
в условно западном кластере |
0,0 |
2,5 |
97,5 |
|
в восточном кластере |
0,0 |
0,0 |
100,0 |
|
По маркеру POU1F1 |
|||
|
Генотип |
CC |
CD |
DD |
|
в условно западном кластере |
0,0 |
0,0 |
100,0 |
|
в восточном кластере |
0,0 |
22,2 |
77,8 |
|
По маркеру ECRF18/FUT1 |
|||
|
Генотип |
AA |
AG |
GG |
|
в условно западном кластере |
0,0 |
0,0 |
100,0 |
|
в восточном кластере |
0,0 |
0,0 |
100,0 |
Мы сравнили частоту генотипов и аллелей по ДНК-маркерам у протестированных животных (табл., рис. 2). Из 10 исследованных ДНК-маркеров пять ( RYR1, ESR, MUC4, IGF2, ECRF18/FUT1 ) оказались мономорфными в обоих территориальных кластерах. Это позволяет предполагать, что мутации, обусловливающие полиморфизм указанных генов, произошли после одомашнивания свиней. Незначительные различия в частоте аллелей ДНК-маркеров между группами особей, входящими в условно западный и восточный кластеры, наблюдались по FSHB (р д — соответственно 0,462 и 0,250), BF (рд — 0,020 и 0,143), MC4R (рд — 0,013 и 0,000) и POU1F1 (pC — 0,000 и 0,111).
Достоверные различия между исследуемыми территориальными группами отмечались по частоте аллелей NCOA1: в первом кластере рд1 = 0,938, во втором кластере рд1 = 0,000. Присутствие аллеля А2 NCOA1 в условно западном кластере кабанов можно рассматривать как следствие интрогрес- сии от китайских свиней в XVII-XIX в.в. (15). В то же время распространение аллеля A2 среди домашних свиней с частотой от 0,722 до 1,000 (21) означает, что нельзя исключать его интродукцию в результате гибридизации с домашними свиньями.
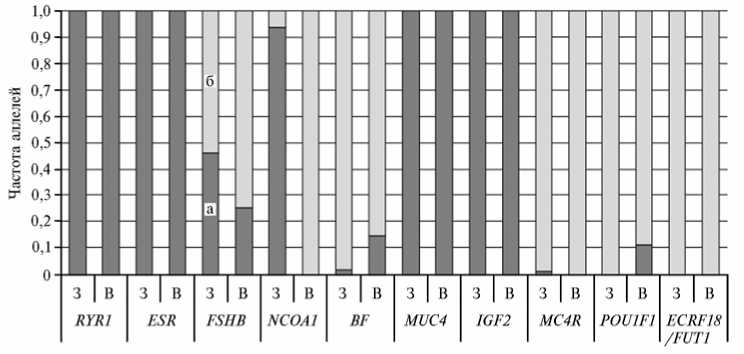
Рис. 2. Распределение аллелей ДНК-маркеров QTL у кабана ( Sus scrofa L., 1758) в западном (3) и восточном (В) территориальных кластерах: а и б — соответственно аллели 1 и 2. Аллель 1 — N по RYR1 , A по ESR , A по FSHB , A1 по NCOA1, A по BF , C по MUC4 , Q по IGF2 , A по MC4R , C по POU1F1 , A по ECRF18/FUT1 ; аллель 2 — n по RYR1 , B по ESR , B по FSHB , A2 по NCOA1 , B по BF , G по MUC4 , q по IGF2 , G по MC4R , D по POU1F1 , G по ECRF18/FUT1 .
Итак, изученные территориальные группы кабана Sus scrofa дифференцированы не только по анонимным ДНК-маркерам, но и по некоторым генам, ассоциированным с локусами количественных признаков у свиней.


