Полиморфизм межмикросателлитных повторов у видов лука
Автор: Домблидес А.С., Домблидес Е.А., Кан Л.Ю., Романов В.С.
Журнал: Овощи России @vegetables
Рубрика: Теория и практика селекции овощных культур
Статья в выпуске: 3 (12), 2011 года.
Бесплатный доступ
В исследование были взяты следующие виды лука: A. altaicum, A. odorum, A. fistulosum, A. nutans, A. schoenoprasum, A. altissimum, A.proliferum, A. tuberosum, A. subhirsutum, A.roylei, A.auctum, A.rupestre, A.vavilovii, ДНК которых была использована для проведения ISSR анализа. Формы, полученные в результате межвидовых скрещиваний: A.cepa x A.nutans и А.сера x A. fistulosum, были в дальнейшем оценены с помощью ДНК маркеров. 5 ISSR праймеров были отобраны из 15 проверенных и использованы для анализа. В итоге было выявлено 76 % полиморфизма у исследуемых образцов. Образцы, полученные в результате гибридизации между видами A. nutans и A. cepa, расположились в дендрограмме ближе к кластеру A. cepa, тогда как вид A. nutans располагался дальше от A.cepa с индексом схожести 0,52 и с индексом 0,49 от гибридных образцов. Виды A. fistulosum и А. altaicum, так же, как и виды A.altissimum и A.schoenoprasum схожи по ISSR спектрам, что не расходится с результатами предыдущих филогенетических исследований.
Issr маркеры, виды лука, полиморфизм
Короткий адрес: https://sciup.org/14024896
IDR: 14024896 | УДК: 635.25:631.527.42
Текст научной статьи Полиморфизм межмикросателлитных повторов у видов лука
Развитие или поиск эффективных ДНК маркеров для лука является основной задачей для контролируемой селекции и получения межвидовых гибридов. ДНК анализ, основанный на ПЦР (такие как RAPD, SSR, AFLP), широко применяли для таксономической классификации и идентификации различных видов, сортов, гибридов и линий лука. RAPD маркеры были использованы рядом исследователей для типирования генотипов лука, выявления интрогрессий, для маркирования хромосом лука шалота (Tanikawa T., и др. 2002; Shigyo M., и др., 1997a). AFLP техника была применена для построения более точных генетических карт на основе межвидовых скрещиваний между A. cepa и А. roylei (A.W. van Heusden, 2000)
ISSR-ПЦР метод основывается на использовании мик-росателлитных праймеров (простые повторы), как правило, в 17-20 нуклеотидных пар (Zietkiewicz E и др. 1994). Простые повторы в геноме являются высокоспецифичным признаком, в том числе таксономическим и филогенетическим. Более того, ограничено количество литературных источников, описывающих применение ISSR-ПЦР метода на генотипах лука.
Данное исследование поможет найти и оценить специфические локусы в геноме различных видов лука, которые могут послужить молекулярными маркерами в роде Allium. На молекулярном уровне необходимо подтвердить «гиб-ридность» полученных растений лука, научно обосновать и определить генетические дистанции как полученных форм, так и их родителей.
Материалы и методы исследований
Для исследований было взяты различные виды и образцы лука из коллекции лаборатории генетики и цитологии ВНИИССОК (табл. 1).
1. Виды и селекционные образцы лука, взятые для исследования
|
Вид |
Количество образцов |
Вид |
Количество образцов |
|
A. altaicum |
4 |
A. tuberosum |
1 |
|
A. odorum |
3 |
A. subhirsutum |
1 |
|
A. fistulosum |
2 |
A.roylei, |
1 |
|
A. nutans |
1 |
A.auctum |
1 |
|
A. schoenoprasum |
1 |
A.rupestre |
1 |
|
A. altissimum |
1 |
A.vavilovii |
1 |
|
A.proliferum |
1 |
A.cepa |
1 |
|
F 1 A.cepa x A.nutans |
2 |
BC 2 F 5 (А.сера x A. fistulosum) |
2 |
|
Апомикт A0BC2F5 |
1 |
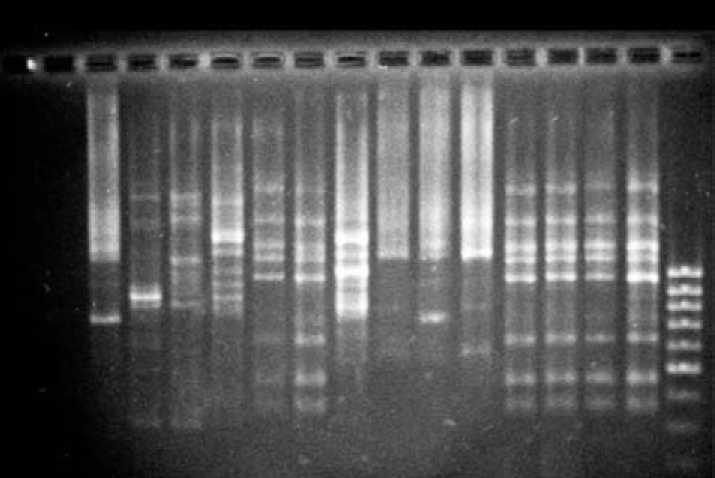
Рис. 1. Спектры геномной ДНК видов лука, полученные в результате ISSR анализа.
Справа налево дорожки 1 – 4 A. altaicum;
5 – 7 A. odorum; 8 A. subhirsutum; 9, 10 A. fistulosum;
11 A. nutans; 12 A. schoenoprasum;
13 A. altissimum; 14 A. tuberosum
Для выделения ДНК использовали метод с использованием CTAB-буфера (Dellaporta, 1983) и двукратной депротеинизацией хлороформом. Полимеразную цепную реакцию проводили с использованием реагентов и праймеров производства компании «СИНТОЛ» Москва.
Для проведения ISSR анализа подбирали оптимальную температуру отжига микросателлитных праймеров на ДНК матрице в диапазоне от 46 до 520С. ISSR фрагменты разделяли в 2% агарозном геле SFR высокого разрешения (Amresco, США) и окрашивали бромистым этидием. Размеры амплифицированных фрагментов определяли в сравнении с маркером молекулярных масс 100 bp DNA ladder (MBI FERMENTAS, Латвия).
Анализ результатов проводили с использованием программ GenStat ver. 6 и TREECON. Коэффициент Жаккарда был использован для построения матрицы генетической схожести.
Результаты исследований
Для исследования генетической изменчивости среди видов лука на данном этапе было отобрано пять ISSR праймеров из пятнадцати протестированных.
Среднее количество продуктов амплификации, приходящихся на 1 праймер, составляло 10,5. Фрагменты были представлены большим спектром длины от 350 до 2000 н.п. 53 ISSR локуса были получены в результате амплификации, из которых 40 были полиморфными, т.е. с использованием пяти ISSR праймеров ((ggat)4, (ag)8yt, (ca)8rg, (ca)8rc, (ga)8yt) удалось выявить 76 % полиморфизма среди представленных видов лука. Обнаруживалось сходство в спектрах у одинаковых видов разной репродукции, однако наблюдались отличия в отдельных спектрах между образцами A. odorum. Все три представителя вида A. altaicum оказались генетически сходными между собой (рис. 1.)
В то же время, было выявлено генетическое родство между видами A. altaicum и A. fistulosum , что подтверждалось другими работами по молекулярной систематике. Как известно, A.altissimum генетически близок к А.schoenoprasum, что также подтвердилось в нашем исследовании с использованием ISSR маркеров. Внешнее сходство A. subhirsutum и A. nutans подтверждалось в наших исследованиях их генетической близостью, однако по данным других авторов, эти два вида лука достаточно далеки друг от друга (Klaas, Friesen, 2002). Чем больше генетическая дистанция между исследуемыми видами, тем меньше у них общих продуктов амплификации. Поэтому, в спектрах геномной ДНК некоторых видов лука можно было определить специфичные продукты амплификации, так, например, у вида A. schoenoprasum с праймером (ag) 8 yt были обнаружены фрагменты 540 и 590 н.п.; у вида A.altissi-mum специфический фрагмент массой 460 н.п., ампли-фицировавшийся с тем же праймером. Выявленные ампликоны, присущие только одному конкретному виду лука, при сравнении с другими видами могут оказаться видоспецифичными.
Маркеры, позволяющие выявлять генетические отличия между различными видами луков, были использованы далее при оценке гибридных комбинаций.
В дендрограмме (рис.2) обращает на себя внимание кластер с формами, полученными в процессе гибридизации и последующим беккроссировани-ем (F5) А.сера и A. fistulosum 92/2 и 92/3, и форма, полученная в результате апомиксиса A0 BC2 F5, все эти генотипы совместно с A.cepa, выступающим как материнская форма, сгруппировались вместе, т.е. были генетически сходными.
Гибриды между A. nutans и A.cepa располагались ближе к кластеру A.cepa . А вид A. nutans , который выступал в качестве отцовской линии, имел коэффициент схожести с кластером A.cepa и гибридной формой 0,52 и 0,49 соответственно.
В данном исследовании было показано, что молекулярные маркеры, полученные на основе полиморфизма меж-микросателлитных локусов, могут быть потенциально использованы для оценки селекционного материала лука, используемого для межвидовых скрещиваний. Универсальность этой техники дает возможность проверять достаточно большое количество комбинаций скрещиваний и форм, полученных в результате беккроссирования.
A.auctum А.сера Fl 92/2 F1.92/3 -
A.apomictei -A.vavilovll -
A.prollferum -
F1A-cepa, A.nutani FIA-cepa, A-nutans -A.nipestre A.altalcum -
A-fistuloeum -A-nutans
A-schoenoprasum A.roy1el
Список литературы Полиморфизм межмикросателлитных повторов у видов лука
- Dellaporta, S.L., Wood, J., Hicks, J.B. A plant DNA minipreparation: Version II. Plant Mol. Biol. Rep. 1983. 1(4). P. 19 -21
- R. Waugh. RAPD Analysis: use for genome characterization, tagging traits and mapping//Plant Molecular Biology -A Laboratory Manual/Ed by Melody S., Clark. Berlin. Heidelberg: Springer-Verlag, 1997.-P. 305-333.
- Tanikawa T., Takagi M., Ichiim (2002) Cultivar identification and genetic diversity in onion (Allium cepa L.) as evaluated by random amplified polymorphic DNA (RAPD) analysis. J Jpn Soc Hort Sci 71:249-251
- Shigyo M., Miyazaki.T, Isshiki S., Tashiro Y. (1997a) Assignment of randomly amplified polymorphic DNA markers to all chromosomes of shallot (Allium cepa L. Aggregatum group). Genes Genet Syst 72:249-252
- Stack S.M., Comings D.E. (1979) The chromosomes and DNA of Allium cepa. Chromosoma 70:161-181
- A.W. Van Heusden J.W. van Ooijen R. Vrielink-van Ginkel W.H.J. Verbeek W.A. Wietsma C. Kik A genetic map of an interspecific cross in Allium based on amplified fragment length polymorphism (AFLP) markers. Theor Appl Genet (2000) 100:118-126
- L.W.D. Raamsdonk, W. Ensink, A.W. Heusden, M. Vrielink-van Ginkel and C. Kik Biodiversity assessment based on cpDNA and crossability analysis in selected species of Allium subgenus Rhizirideum//Theor Appl Genet (2003) 107:1048-1058
- Zietkiewicz E., Rafalski A., and Damian Labuda D. Genome fingerprinting by simple sequence repeats (SSR)-anchored polymerase chain reaction amplification.//Genomics 1994, 20. P. 176-183
- Klaas, M., N. Friesen 2002. Molecular markers in Allium, p 159-186 In: Rabinowitch, H.D. and L. Currah (eds.). Allium crop science: Recent advances CABI Publishing, New York, NY


