Полиморфизм микросателлитных локусов у сортов черной смородины ( Ribes nigrum L.) из коллекции ВНИИСПК
Автор: Пикунова А.В., Князев С.Д., Бахотская А.Ю., Кочумова А.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молекулярная структура генома и селекция
Статья в выпуске: 1 т.50, 2015 года.
Бесплатный доступ
Черная смородина - ведущая ягодная культура в России. Необходимость совершенствования сортимента по целому ряду направлений требует привлечения новых эффективных методов в селекцию. Один из современных подходов при работе с генетическими ресурсами - применение ДНК-маркеров. Впервые в России мы использовали микросателлитные ДНК-маркеры для изучения черной смородины. В настоящей работе проведено генотипирование 27 сортообразцов этой культуры из коллекции Всероссийского НИИ селекции плодовых культур (ВНИИСПК) (в том числе 16 сортов селекции ВНИИСПК) по 14 микросателлитным локусам. Для анализа полиморфизма микросателлитных локусов использовали электрофоретическое разделение ПЦР-продуктов в 6 % денатурирующем полиакриламидном геле (ПААГ) с последующим окрашиванием нитратом серебра. Все локусы, кроме MS06g03, оказались полиморфными (в среднем было амплифицировано 4,9 аллеля на локус). С ДНК некоторых сортообразцов в одном локусе амплифицировалось три фрагмента, что, вероятно, связано с дупликацией этих микросателлитных локусов. Из 68 амплифицированных фрагментов 45 (66 %) были редкими аллелями с частотой £ 0,2. Обнаружено восемь уникальных фрагментов, амплифицированных только на ДНК одного из протестированных сортообразцов. Наблюдаемая гетерозиготность варьировала от 0,259 для локуса g2-H21 до 1 для локуса e4-D03 (в среднем составила 0,608). По сочетанию аллелей в локусе можно было различить от 3 (g2-H21, e1-O21) до 15 (g2-G12) генотипов. Минимальный набор из четырех локусов (e4-D03, g1-M07, g1-E03, g2-B20) позволил дифференцировать все протестированные сортообразцы, то есть для каждого образца был получен уникальный мультилокусный профиль. Коэффициенты попарного генетического сходства варьировали от 0,110 (между сортами Оджебин и Шаровидная) до 0,950 (между сортами Говтва и Ртищевская) и в среднем составили 0,346. Проведен кластерный анализ генетического сходства сортообразцов и построена дендрограмма, в которой выделились несколько кластеров с высокой бутстреп-поддержкой. Изучение потока аллелей через родословные проанализированных сортов показало, что в большинстве случаев распределение аллелей между родственными сортами не противоречило родословным, но были обнаружены и случаи расхождения сведений родословных и SSR-анализа. Так, у сорта Очарование (получен в скрещивании сеянец№1168 ½ сорт Экзотика) в трех локусах (e4-D03, g1-E03, g2-B20) не было общих аллелей с сортом Экзотика. Возможно, что в действительности произошло опыление другой пыльцой, нельзя также исключить ошибку при размножении и пересадке или мутации в указанных локусах. Сорта Оджебин и Бинар (выведен при скрещивании сортов Оджебин х Нарядная) тоже не имели общих аллелей в двух локусах (g1-M07, g1-E03). Полученные результаты свидетельствуют о перспективности применения протестированных маркеров для оценки генетического разнообразия отечественного генофонда черной смородины, а также для разработки методов идентификации и паспортизации ее сортов.
Черная смородина, днк-маркеры, микросателлиты, полиморфизм, идентификация, генофонд
Короткий адрес: https://sciup.org/142133568
IDR: 142133568 | УДК: 634.723.1:575.174.015.3:577.21 | DOI: 10.15389/agrobiology.2015.1.46rus
Текст научной статьи Полиморфизм микросателлитных локусов у сортов черной смородины ( Ribes nigrum L.) из коллекции ВНИИСПК
На долю черной смородины в России приходится половина площадей, занятых под ягодниками (1). На 2013 год в Государственный реестр селекционных достижений, допущенных к использованию, вошло 170 сортов этой важнейшей ягодной культуры (2). Совершенствованием ее сортимента в России занимается около 20 научных учреждений. Селекционные исследования направлены на улучшение биохимического состава ягод, создание продуктивных стабильно плодоносящих сортов, устойчивых к болезням и вредителям, пригодных к механизированной уборке урожая и др. (1). Во Всероссийском НИИ селекции плодовых культур (ВНИИСПК) к настоящему времени в государственное сортоиспытание переданы 26 сортов, из них 12 включены в Государственный реестр сортов, до- пущенных к использованию. Собрана обширная коллекция сортообраз-цов, включающая более 100 интродуцированных сортов и более 700 форм собственной селекции.
Успех селекционных исследований во многом зависит от изучения, подбора и систематизации исходного материала (3). Черная смородина имеет диплоидный набор хромосом ( n = 7), что в определенной степени облегчает изучение ее генома по сравнению с геномами полиплоидных видов. Пионерами в использовании ДНК-маркеров для анализа генетического полиморфизма при работе с генетическими ресурсами черной смородины и других представителей рода Ribes L. стали ученые из Шотландского института селекции сельскохозяйственных культур (Scottish Crop Research Institute, Invergowrie, Dundee, Scotland, Великобритания), применившие RAPD (random amplified polymorphic DNA), AFLP (amplified fragment length polymorphism), ISSR (inter-simple sequence repeat), SNP (single nucleotide polymorphism) и другие маркеры (4, 5). В результате на основании микросателлитных, SNP- и AFLP-маркеров впервые построена генетическая карта черной смородины (6), разработан и применяется метод маркер-опосредованного отбора генотипов, имеющих ген устойчивости к почковому клещу Се (7). В России ДНК-маркирование для оценки генетической коллекции представителей рода Ribes используется сравнительно недавно (8).
В настоящее время наблюдается тенденция к применению высоковоспроизводимых кодоминантных SSR- и SNP-маркеров. SSR (микро-сателлитные) маркеры используются как в фундаментальных исследованиях для оценки генетического полиморфизма, изучения филогенетических отношений, построения генетических карт для групп сцепления, так и в прикладных целях — для проверки родословных, при разработке систем идентификации и паспортизации, поиске маркеров, ассоциированных с хозяйственно полезными признаками, при маркер-опосредованном отборе интересующих генотипов на ранних стадиях развития растений (9). SSR-маркеры применялись для оценки разнообразия генетических коллекций Ribes в Италии и Северной Европе (10, 11).
Мы впервые использовали микросателлитные маркеры для оценки межсортового полиморфизма, определения генетического сходства между сортообразцами, проверки родословных у форм черной смородины из российской коллекции ВНИИСПК, что позволило обсуждать перспективы применения полиморфизма микросателлитных локусов в работе с генетическими ресурсами этой ягодной культуры.
Цель представляемой работы заключалась в изучении полиморфизма микросателлитных локусов у сортов черной смородины из коллекции ВНИИСПК.
Методика. Объектом исследования были 27 сортов черной смородины из коллекции Всероссийского НИИ селекции плодовых культур, в том числе 16 сортов селекции ВНИИСПК — Ажурная, Грация, Гамма, Дачница, Орловский вальс, Монисто, Орловия, Искушение, Чудное мгновение, Очарование, Сластена, Черная вуаль, Экзотика, Блакестон, Зуша, Кипиа-на, а также сорта других оригинаторов — Оджебин, Титания, Шаровидная, Селеченская, Минай Шмырев, Ртищевская, Консорт, Бинар, Говтва, Канахама, Ядреная. Кроме того, сорта Лентяй и Орловская серенада были проанализированы не по всем использованным локусам. Результаты, полученные по этим сортам, учитывались только при анализе соответствия распределения аллелей между родственными сортами и данных по родословным (распределение аллелей между родственными сортами сверяли по всем полиморфным локусам, для которых имелись такие данные).
ДНК выделяли из молодых листьев СТАВ-методом (12).
Всего нами были проанализированы 14 микросателлитных локусов — e1-O01, e1-O21, e3-B02, e4-D03, g1-B02, g1-E03, g1-K04, g1-M07, g2-B20, g2-H21, g2-J08, g2-L17, g2-G12, MS06g03 (13). ПЦР-анализ проводили в реакционной смеси объемом 20 мкл, содержащей 1Х ПЦР буфер, 200 мкМ нуклеотидов, 2 мкМ прямого праймера, 2 мкМ обратного праймера, 0,3 ед. Taq ДНК-полимеразы и 10 нг ДНК, в термоциклере GeneAmp PCR System 9700 («Applied Biosystems», США). Реакцию амплификации осуществляли в следующем режиме: предварительная денатурация — 5 мин при 94 °С; денатурация — 30 с при 94 °С, отжиг праймера (при температуре, подобранной для каждого маркера, см. раздел «Результаты»), синтез ДНК — 30 с при 72 °С (всего 35 циклов); завершающая элонгация — 10 мин при 72 °С.
При анализе полиморфизма микросателлитных локусов использовали электрофоретическое разделение амплификатов в 6 % денатурирующем полиакриламидном геле (ПААТ) в камере Sequi-Gen GT System (38x50 см) («Bio-Rad», США) с последующим окрашиванием нитратом серебра (14). Размеры фрагментов определяли, сравнивая с маркером молекулярных масс 10 bp DNA Ladder («Invitrogen™», США).
Частоту аллеля рассчитывали как сумму частот гомозиготного генотипа и полусумму частот гетерозиготных генотипов, содержащих этот аллель (15). Ожидаемую гетерозиготность вычисляли как He = 1 - p i2 , где p i — частота i -го аллеля. Для оценки наблюдаемой гетерозиготности (Ho) в соответствующем локусе число гетерозиготных образцов делили на общее число образцов. Редким считали аллель с частотой 0,2 и менее. При определении частоты аллеля, вероятности идентичности в соответствии с законом Харди-Вайберга HW P(ID) и вероятности идентичности при анализе сибсов Sib P(ID) использовали программу GENECAP (16). Частоту ноль-аллеля (r) вычисляли по формуле r = (He - HH . )/(1 + Hо . ). Расчет коэффициентов сходства Жаккара (Jaccard similarity coefficient) (17) и построение дендрограммы осуществляли с помощью программы PAST (18). Дендрограмму строили методом кластерного анализа UPGMA (unweighted pair group method with arithmetic mean) (19) с использованием коэффициента Жаккара, число репликаций — 1000.
Результаты . При анализе 14 микросателлитных локусов (табл. 1) у 27 сортообразцов смородины были выявлены имеющие от 1 (MS06g03) до 8 (e4-D03) аллелей, что в среднем составило 4,9 аллеля на локус. При этом все локусы, кроме MS06g03, оказались полиморфными.
Для всех локусов, кроме MS06g03, у черной смородины известно расположение на генетической карте групп сцепления (в частности, локусы, включенные в анализ, расположены в семи различных группах сцепления) (6). Праймеры для локуса MS06g03 были первоначально разработаны на основании сиквенса ДНК яблони и картированы в 10-й группе сцепления в семье от скрещивания сортов Фиеста x Дискавери, MS06g03 рассматривается как мультилокусный маркер — у яблони при его амплификации образуются фрагменты ДНК размером 154-190 п.н. (13), а у смородины у всех сортообразцов наблюдается фрагмент длиной 140 п.н. Вероятно, локус находится в весьма консервативной части генома смородины черной.
В большинстве случаев у каждого генотипа для конкретного локуса было выявлено не более двух фрагментов, но в некоторых локусах у ряда образцов амплифицировались три фрагмента (у сорта Ядреная — в локусе g2-B20, Селеченская — e1-O01, у сортообразцов Гамма, Грация, Орловия, Черная вуаль, Шаровидная, Зуша, Кипиана — в e3-B02). В литературе есть сообщения об амплификации более чем двух аллелей в некоторых микросателлитных локусах у диплоидных форм, что связано с дупликацией микросателлитных локусов на одной и той же или на разных хромосомах (20).
-
1. Обозначения и режимы амплификации микросателлитных локусов, по которым были проанализированы сортообразцы черной смородины ( Ribes nigrum L.) из коллекции Всероссийского НИИ селекции плодовых культур
-
2. Характеристика 13 полиморфных микросателлитных локусов, выявленных у
- 27 сортообразцов черной смородины (Ribes nigrum L.) из коллекции Всероссийского НИИ селекции плодовых культур
На основе микросателлитных профилей у 27 сортообразцов мы проанализировали распределение аллелей для 13 из использованных полиморфных локусов (табл. 2).
|
Локус | |
n |
1 |
Размер |
аллеля |
, п.н. ( |
частота) |
1 Не | |
| Н о |
r |
1 а, Б |
|||
|
e1-O01 |
6 |
138а |
140аб |
142 |
145а |
147 |
149а |
0,737 |
0,769 |
- 0,019 |
0,402, |
||
|
(0,093) |
(0,023) |
(0,419) |
(0,163) |
(0,209) |
(0,093) |
0,101 |
|||||||
|
e1-O21 |
3 |
288 аб |
292 |
294 |
0,423 |
0,407 |
0,011 |
0,639, |
|||||
|
(0,029) |
(0,714) |
(0,257) |
0,402 |
||||||||||
|
e3-B02 |
4 |
188 |
191 |
193а |
194а |
0,655 |
0,800 |
- 0,088 |
0,467, |
||||
|
(0,486) |
(0,257) |
(0,200) |
(0,057) |
0,177 |
|||||||||
|
e4-D03 |
8 |
197а |
201а |
205а |
207аб |
210а |
212а |
217а |
224а |
0,840 |
1,000 |
- 0,087 |
0,342, |
|
(0,180) |
(0,100) |
(0,160) |
(0,020) |
(0,200) |
(0,200) |
(0,100) |
(0,040) |
0,046 |
|||||
|
g1-B02 |
3 |
203 |
205 |
207 |
0,610 |
0,590 |
0,016 |
0,495, |
|||||
|
(0,231) |
(0,513) |
(0,256) |
0,216 |
||||||||||
|
g1-E03 |
7 |
232аб |
235 |
237 |
239а |
245а |
249аб |
270а |
0,726 |
0,593 |
0,077 |
0,416, |
|
|
(0,025) |
(0,400) |
(0,275) |
(0,175) |
(0,075) |
(0,025) |
(0,025) |
0,118 |
||||||
|
g1-K04 |
4 |
285аб |
290 |
295 |
298а |
0,560 |
0,556 |
0,003 |
0,530, |
||||
|
(0,026) |
(0,605) |
(0,237) |
(0,132) |
0,250 |
|||||||||
|
g1-M07 |
7 |
200а |
204а |
207а |
209 |
212а |
217а |
222 аб |
0,739 |
0,704 |
0,020 |
0,405, |
|
|
(0,140) |
(0,140) |
(0,047) |
(0,442) |
(0,140) |
(0,070) |
(0,023) |
0,097 |
||||||
|
g2-B20 |
5 |
147а |
168а |
178 |
181а |
185а |
0,720 |
0,556 |
0,096 |
0,420, |
|||
|
0,189 |
0,135 |
0,432 |
0,189 |
0,054 |
0,119 |
||||||||
|
g2-H21 |
3 |
265а |
267 |
273а |
0,361 |
0,259 |
0,075 |
0,680, |
|||||
|
(0,156) |
(0,781) |
(0,063) |
0,443 |
||||||||||
|
g2-J08 |
4 |
158а |
160 |
162а |
164а |
0,514 |
0,444 |
0,046 |
0,562, |
||||
|
0,083 |
0,667 |
0,167 |
0,083 |
0,274 |
|||||||||
|
g2-L17 |
6 |
150а |
152а |
154а |
161 |
168а |
171а |
0,580 |
0,296 |
0,180 |
0,510, |
||
|
(0,063) |
(0,063) |
(0,094) |
(0,625) |
(0,094) |
(0,063) |
0,200 |
|||||||
|
g2-G12 |
7 |
190аб |
195а |
203а |
207а |
209а |
212 |
215 |
0,997 |
0,926 |
0,035 |
0,355, |
|
|
(0,022) |
(0,178) |
(0,089) |
(0,089) |
(0,156) |
(0,244) |
(0,222) |
0,058 |
||||||
|
Средн< |
ее |
0,652 |
0,608 |
0,192, |
|||||||||
|
0,479 |
|||||||||||||
Примечание. А — Sib P(ID) (вероятность идентичности при анализе сибсов), Б — HW P(ID) (вероятность идентичности в соответствии с законом Харди-Вайберга); n — число аллелей, а — редкие аллели, б — уникальные аллели, амплифицируемые у одного сортообразца из 27 изученных.
Ожидаемая гетерозиготность варьировала от 0,361 в локусе g2-H21
до 0,997 в локусе g2-G12 (в среднем 0,652), в то время как наблюдаемая гетерозиготность — от 0,259 в локусе g2-H21 до 1 в локусе e4-D03 (в среднем 0,608). Таким образом, средние значения для наблюдаемой и ожидаемой гетерозиготности не сильно различались, что обусловлено природой изучаемого объекта. Как известно, перекрестноопыляемые растения, к которым относится черная смородина, характеризуются большей гетерозиготностью по сравнению с самоопыляемыми (15). К тому же вегетативное размножение плодово-ягодных культур позволяет закрепить гетерозиготное состояние генотипа независимо от того, насколько стабильным будет проявление признаков в первом поколении. Поэтому к отличительным особенностям таких культур относится высокая гетерозиготность, которую только усиливает использование метода отдаленной гибридизации и при влечение различных видов в селекцию.
3. Сортоспецифичные фрагменты, амплифи-цируемые в микросателлитных локусах у черной смородины ( Ribes nigrum L.) из коллекции Всероссийского НИИ селекции плодовых культур
|
Сортообразец |
| Локус/размер фрагмента |
|
Орловия |
g1-K04/285 п.н., g1-E03/232 п.н. |
|
Оджебин |
g1-M07/222 п.н. |
|
Монисто |
e4-D03/207 п.н. |
|
Минай Шмырев |
e1-O21/288 п.н. |
|
Бинар |
e1-O01/140 п.н. |
|
Титания |
g1-E03/249 п.н. |
|
Шаровидная |
g2-G12/190 п.н. |
В трех локусах (e1-O01, e3-B02 и e4-D03) наблюдаемая гетерозиготность превысила ожидаемую. В остальных девяти локусах, наоборот, ожидаемая гетерозиготность была выше наблюдаемой, что указывает на наличие гомозиготных генотипов или присутствие неампли-фицируемого ноль-аллеля. Теоретическая частота ноль-аллеля оказалась положительным зна чением для всех локусов, кроме e1-O01, e3-B02, e4-D03, и варьировала от -0,088 до 0,180.
Вероятность идентичности двух генотипов HW P(ID) варьировала от 0,046 по локусу e4-D03 до 0,443 по локусу g2-H21 (в среднем 0,203), вероятность идентичности двух сибсов Sib P(ID) — от 0,342 по локусу e4-D03 до 0,680 по локусу g2-H21 (в среднем 0,489).
Из 68 фрагментов, амплифицированных в 14 локусах, 45 (66 %) соответствовали редким аллелями с частотой, равной или менее 0,2. Также были обнаружены восемь уникальных фрагментов, амплифицированных только на ДНК одного из протестированных сортообразцов (табл. 3).
По сочетанию аллелей в локусе можно было различить от 3 (g2-H21, e1-O21) до 15 (g2-G12) генотипов. Например, в локусе g1-K04, в котором амплифицируется 4 аллеля, наблюдались пять сочетаний аллелей (генотипов), при этом сорт Орловия имел уникальный аллель размером приблизительно 285 п.н. (рис. 1).
Минимальный набор из четырех локусов (e4-D03, g1-M07, g1-E03, g2-B20) позволил различить все протестированные сортообразцы, то есть для каждого образца был получен уникальный мультилокусный профиль.
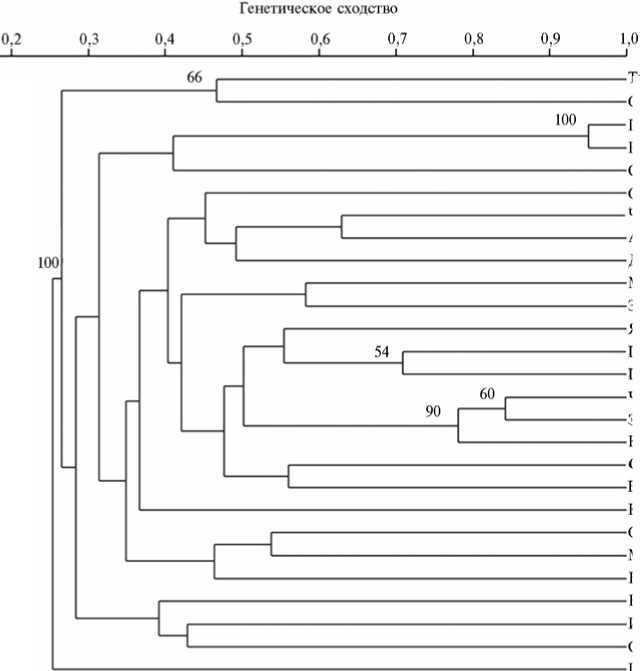
На основе SSR-спектров мы построили дендрограмму сходства для 27 сортообразцов черной смородины (рис. 2). Полученные коэффициенты попарного генетического сходства варьировали от 0,11 (между сортами Оджебин и Шаровидная) до 0,95 (между сортами Говтва и Ртищевская) и в среднем составили 0,346.
В дендрограмме выделились несколько кластеров с высокой бутст-реп-поддержкой (БП). На близком генетическом расстоянии вместе кластеризовались сорта Говтва и Ртищевская (БП 100 %) на несколько большем генетическом расстоянии объединились сорта Чудное мгновение и Экзотика (БП 60 %), у которых материнские формы — это сеянцы из од- ной семьи; к ним примкнул сорт Кипиана (БП 90 %), для которого отцовской формой служил сорт Экзотика. Сорта Гамма и Грация, полученные отбором из той же гибридной семьи, что и сорт Кипиана, кластеризовались отдельно от сортов Экзотика и Кипиана. По данным RAPD-анализа, также наблюдалась кластеризация сортов Гамма и Грация с высоким значением БП, в то время как сорт Кипиана в этот кластер не вошел (8). Вероятно, подобное свидетельствует о том, что сорта Гамма и Грация генетически более сходны друг с другом, чем с сортами Кипиана и Экзотика. Отдельный кластер также сформировали выведенные в Швеции сорта Титания и Оджебин (БП 66 %), имеющие в родословной скандинавский подвид смородины черной.
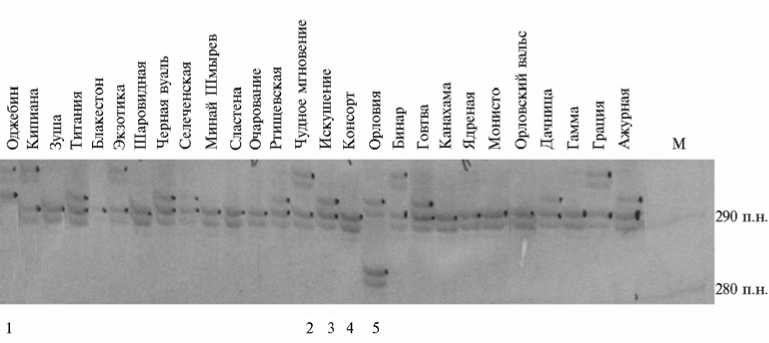
Рис. 1. Электрофореграмма фрагментов амплификации микросателлитного локуса g1-K04 у 27 изученных сортообразцов черной смородины ( Ribes nigrum L.) из коллекции Всероссийского НИИ селекции плодовых культур: 1-5 — условные генотипы по локусу; M — маркер молекулярных масс (10 bp DNA Ladder, «Invitrogen™», США; справа указаны размеры фрагментов).
Анализ потока аллелей через родословные родственных проанализированных сортов показал, что в большинстве случаев распределение аллелей между ними не противоречило родословным. Так, родительский сорт Лентяй и его производные в первом поколении (сорта Ажурная, Орловский вальс, Сластена) имели хотя бы один общий аллель по всем протестированным микросателлитным локусам. То же происходило с сортом Минай Шмырев и с сортами, полученными от него в первом поколении (Зуша, Лентяй, Орловская серенада), а также с сортом Экзотика и потомками этого сорта в первом поколении (сорта Кипиана, Гамма, Грация). Однако у сорта Очарование, для которого по родословной сорт Экзотика служил отцовской формой, в трех локусах (e4-D03, g1-E03, g2-B20) не было общих с ним аллелей. Возможно, что в действительности произошло опыление другой пыльцой, нельзя исключить и ошибку при размножении и пересадке, а также мутации в этих локусах. Сорта Оджебин и Бинар (получен от скрещивания сортов Оджебин х Нарядная) не имели общих аллелей в двух микросателлитных локусах (g1-M07, g1-E03).
При генотипировании микросателлитных локусов у коллекционных сортов черной смородины из Северной Европы в ряде случаев не удалось подтвердить генетическую идентичность одноименных образцов из разных коллекций, например один из 10 сортообразцов сорта Оджебин оказался не идентичен остальным (11). В работе S. Reim с соавт. 11 микросателлитных локусов были использованы при уточнении происхождения у сортов, выведенных в Dresden-Pillnitz (21). В результате в ряде случаев вы- яснили, какой именно сорт участвовал в гибридизации при опылении смесью пыльцы разных сортов, а для некоторых сортов установлена недостоверность родословных как по материнской, так и по отцовской форме.
Титания
Оджебин Говтва Ртищевская Сластена Орловия Черная вуаль Ажурная Дачница Минай Шмырсв Зуша Ядреная Г рация Гамма
Чудное мгновение Экзотика Кипиана Орловский вальс Бинар Консорт Очарование Монисто Блаксстон Канахама Искушение Селеченская Шаровидная
Рис. 2. Дендрограмма сходства 27 сортообразцов черной смородины ( Ribes nigrum L.) из коллекции Всероссийского НИИ селекции плодовых культур, построенная на основании анализа полиморфизма 13 микросателлитных локусов (приведены значения бутстреп-поддержки).
Итак, нами впервые в России проведено генотипирование микросателлитных локусов у черной смородины ( Ribes nigrum L.). Установлен высокий полиморфизм микросателлитных локусов, выявлены редкие и уникальные аллели, что позволило получить уникальный мультилокусный профиль для каждого протестированного образца. Показана возможность проверки родства образцов на основе анализа распределения микросателлитных аллелей. Полученные данные свидетельствуют о перспективности применения протестированных маркеров при оценке генетического разнообразия отечественного генофонда черной смородины, а также при разработке методов идентификации и паспортизации сортов, что может быть использовано для защиты авторских прав селекционеров.
Список литературы Полиморфизм микросателлитных локусов у сортов черной смородины ( Ribes nigrum L.) из коллекции ВНИИСПК
- Князев С.Д., Огольцова Т.П. Селекция смородины черной на современном этапе. Орел, 2004.
- Государственный реестр селекционных достижений, допущенных к использованию. Т. 1. Сорта растений. М., 2013.
- Вавилов Н.И. Теоретические основы селекции. М., 1987.
- Lanham P., Brennan R.M., Hackett C., McNicol R.J. RAPD fingerprinting of blackcurrant (Ribes nigrum L.) cultivars. Theoretical and Applied genetics, 1995, 90: 166-172 ( ) DOI: 10.1007/BF00222198
- Brennan R., Jorgensen L., Woodhead M., Russell J. Future perspectives in blackcurrant breeding. Acta Horticulture, 2002, 585: 39-45.
- Brennan R., Jorgensen L., Hackett C., Woodhead M., Gordon S.L., Russell J. The development of a genetic linkage map of blackcurrant (Ribes nigrum L.) and the identification of regions associated with key fruit quality and agronomic traits. Euphytica, 2008, 161: 19-34 ( ) DOI: 10.1007/s10681-007-9412-8
- Brennan R., Jorgensen L., Gordon S.L., Loades K., Hackett C., Russell J. The development of a PCR-based marker linked to resistance to the blackcurrant gall mite (Cecidophyopsis ribis Acari: Eriophyidae). Theor. Appl. Genet., 2009, 118: 205-211 ( ) DOI: 10.1007/s00122-008-0889-x
- Пикунова А.В., Мартиросян Е.В., Князев С.Д., Рыжова Н.Н. Применение RAPD-анализа для изучения генетического полиморфизма и филогенетических связей у представителей рода Ribes L. Экологическая генетика, 2011, IX(2): 34-44.
- Kalia R.K., Rai M.K., Kalia S., Singh R., Dhawan A.K. Microsatellite markers: an overview of the recent progress in plants. Euphytica, 2011, 177(3): 309-334 ( ) DOI: 10.1007/s10681-010-0286-9
- Cavanna M., Marinoni D.T., Beccaro G.L., Bounous G. Microsatellite-based evaluation of Ribes spp. germplasm. Genome, 2009, 52: 839-848 ( ) DOI: 10.1139/G09-057
- Antonius K., Karhu S., Kaldm H., Lacis G., Rugenius R., Baniulis D., Sasnauskas A., Schulte E., Kuras A., Korbin M., Gunnarsson A., Werlemark G., Ryliskis T.-A.T., Kokk L., Jarve K. Development of the Northern European Ribes core collection based on a microsatellite (SSR) marker diversity analysis. Plant Genetic Resources: Characterization and Utilization, 2012, 10: 70-73 ( ) DOI: 10.1017/S1479262111000980
- Doyle J.J., Doyle J.L. Isolation of plant DNA from fresh tissue. Focus, 1990, 12: 13-15.
- Liebhard R., Gianfranceschi L., Koller B., Ryder C.D., Tarchini R., Van de Weg E., Gessler C. Development and characterization of 140 new microsatellites in apple (Malus * domestica Borkh.). Mol. Breed., 2012, 10: 217-241.
- Маниатис Т., Фрич Э., Сэмбрук Дж. Методы генетической инженерии. Молекулярное клонирование. М., 1984.
- Гаевский Н.А. Знакомство с эволюционной генетикой. Красноярск, 2002.
- Wilberg M.J., Dreher B.P. Genecap: a program for analysis of multilocus genotype data for non-invasive sampling and capture-recapture population estimation. Molecular Ecology Notes, 2004, 4(4): 783-785.
- Jaccard P. Distribution de la flore alpine dans le Bassin des Dranses et dans quelques regions voisines. Bull. Soc. Vaudoise sci. Natur., 1901, 37(140): 241-272.
- Hammer I., Harper D.A.T., Ryan P.D. PAST: Paleontological statistics software package for education and data analysis. Palaeontologia Electronica, 2001, 4(1): 9 (http://palaeo-electronica.org/2001_1/past/issue1_01.htm).
- Sneath P.H.A., Sokal R.R. Numerical taxonomy: theprinciples and practice of numerical classification. San Francisco, 1973.
- Galli Z., Halász G., Kiss E., Heszky L., Dobránszki J. Molecular identification of commercial apple cultivars with microsatellite markers. Horticult. Sci., 2005, 40: 1974-1977.
- Reim S., Flachowsky H., Hanke M.V., Peil A. Verifying the parents of the Pillnitzer apple cultivars. Acta Horticulture, 2009, 814: 319-323.


