Полногеномное исследование ассоциаций SNP с высотой в холке в популяциях локальных и трансграничных пород крупного рогатого скота в России
Автор: Абдельманова А.С., Форнара М.С., Бардуков Н.В., Сермягин А.А., Доцев А.В., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Структура генома и геномные технологии
Статья в выпуске: 6 т.56, 2021 года.
Бесплатный доступ
Рост животного - это классический количественный признак, который влияет на предрасположенность к определенным заболеваниям и связан с продуктивностью сельскохозяйственных животных. Картировано множество локусов количественных признаков (quantitative trait loci, QTLs), влияющих на составляющие роста крупного рогатого скота (КРС), что подтверждает его полигенный детерминизм. В настоящей работе в российских локальных породах установлено преобладание аллелей, связанных с высоким ростом в холке, в трех из четырех выявленных однонуклеотидных полиморфизмов (single nucleotide polymorphisms, SNP). Целью работы была идентификация локусов и характеристика аллельных вариантов, находящихся под давлением отбора и связанных с размерами тела, в популяциях российских локальных и разводимых на территории Российской Федерации трансграничных пород крупного рогатого скота различных направлений продуктивности, по-разному подверженных давлению искусственного отбора и неодинаково распространенных в мире. Исследовали биоматериал (цельную кровь, сперму и ушные выщипы, хранящиеся в УНУ «Банк генетического материала домашних и диких видов животных и птицы» ФГБНУ ФИЦ животноводства - ВИЖ им. академика Л.К. Эрнста) от 670 гол. крупного рогатого скота 13 пород: абердин-ангусской ( n = 39), айрширской ( n = 144), черно-пестрой ( n = 50), голштинской ( n = 184), истобенской ( n = 22), джерсейской ( n = 32), калмыцкой ( n = 27), холмогорской ( n = 26), киргизской ( n = 24), монгольской ( n = 26), тагильской ( n = 26), якутской ( n = 29) и ярославской ( n = 41). Генотипирование проводилось на SNP-чипах разной плотности, GGP Bovine 150K и BovineHD BeadChip («Illumina, Inc.», США), в ходе обработки данных были определены общие для двух чипов SNP, которые использовались для дальнейшего анализа. C помощью программы PLINK 1.9 с использованием фильтров (--geno 0.1), (--mind 0.2), (--maf 0.05) было проведено полногеномное исследование ассоциаций данных генотипирования с признаками физического развития животных. Данные о высоте в холке для исследуемых пород получили из базы данных ФАО. Породы разделили на группы по следующим критериям: в соответствии с ростом (высокорослые, низкорослые), направлением продуктивности (молочные, мясные), степенью давления селекции (примитивные, заводские) и распространенностью в мире (российские, трансграничные). В общей сложности выявили четыре SNP, из которых три были локализованы на 4-й хромосоме (ARS-BFGL-NGS-116590, Hapmap53144-ss46525999, BovineHD0400021479), один - на 14-й хромосоме (BovineHD1400007259). Альтернативные аллели в обнаруженных SNP статистически значимо различаются по частоте встречаемости в разных группах пород животных, а также имеют статистически значимые положительные либо отрицательные корреляции с высотой в холке. Многообразие и разнородность пород, представленных в выборке, позволяют рассматривать выявленные следы селекции как характерные не для одной породы, региона или направления продуктивности, а для группы пород вида Bos taurus taurus , распространение которых от центра одомашнивания шло по Дунайскому пути. Таким образом, выявленные SNP могут использоваться в качестве генетических маркеров в программах селекции на увеличение роста животных и повышение их продуктивности.
Крупный рогатый скот, локальные породы, трансграничные породы, snp-маркеры, днк-чипы, высота в холке
Короткий адрес: https://sciup.org/142231714
IDR: 142231714 | УДК: 636.2:577.2 | DOI: 10.15389/agrobiology.2021.6.1111rus
Текст научной статьи Полногеномное исследование ассоциаций SNP с высотой в холке в популяциях локальных и трансграничных пород крупного рогатого скота в России
Племенная работа по совершенствованию крупного рогатого скота (КРС) имеет важное значение для повышения эффективности скотоводства, поскольку гармонично сложенные, развитые животные имеют высокую продуктивность и пользуются спросом на рынке племенной продукции (1).
Полногеномный анализ ассоциаций (whole-genome associated study, GWAS) позволил идентифицировать множество геномных вариаций, связанных с количественными признаками сельскохозяйственных животных
-
(2) . В последние несколько десятилетий проведены исследования по картированию локусов количественных признаков (quantitative trait loci, QTLs), ответственных за молочную продуктивность (3), устойчивость к болезням (4, 5), воспроизводительные качества (6), рост (7-9), мясные качества (1013) и качества туши КРС (14-16).
Рост животного — это классический количественный признак, привлекающий внимание генетиков на протяжении долгого времени (17-19). Понимание молекулярных основ вариабельности роста между индивидуумами поможет не только получить информацию об общей архитектуре количественных признаков, но также даст представление о механизмах, контролирующих рост организма. Известно, что рост влияет на предрасположенность к определенным заболеваниям и связан с продуктивностью сельскохозяйственных животных. Несмотря на чувствительность к факторам окружающей среды, он обычно передается по наследству, в том числе у человека ( ∼ 85 %) (20).
Доместикация КРС оказала сильное влияние на этот показатель. Рост у вида Bos taurus уменьшился примерно в 1,5 раза от Неолита до Средневековья и снова увеличился только в Раннем Новом времени (21). Вымерший тур ( Bos primigenius ) был намного крупнее своих домашних потомков (высота в холке ∼ 2 м против ∼ 1,1-1,5 м у современного КРС). Коэффициент наследуемости показателя колеблется в пределах ∼ 25-85 % в зависимости от популяции (22, 23). Если механизмы генетической изменчивости способствовали восстановлению роста с Раннего Нового времени, их отпечатки селекции должны быть обнаружены на основе геномных данных современных пород КРС.
В настоящее время картировано множество QTLs, влияющих на составляющие роста КРС, что подтверждает его полигенный детерминизм. Например, сообщалось о картировании 38 и 52 QTLs, объясняющих более 60 % вариаций соответственно роста и массы взрослых особей в американской популяции коров абердин-ангусской породы (24). L. Karim с соавт. (25) исследовали животных голштино-фризской и джерсейской пород для идентификации QTLs, влияющих на признаки, важные для молочного скотоводства. Были проведены измерения для более чем 500 признаков, шесть из которых относились к размерам тела: масса при рождении, масса в 6, 8, 12, 18 и более 24 мес (живая масса), а также высота в холке в 18 мес.
-
I. Randhawa с соавт. (26) сравнили данные однонуклеотидного полиморфизма (single nucleotide polymorphism, SNP) по всему геному у ряда европейских и африканских пород B. taurus с высокой (145-155 см) и низкой (105-133 см) холкой и обнаружили, что наиболее значимый селекционный сигнал, связанный с ростом, картирован на 14-й хромосоме (BTA14) в регионе 24,79-28,25 Mbp (27). Этот отпечаток селекции был недавно подтвержден данными последовательностей полного генома четырех пород B. taurus и ограничен меньшей областью, охватывающей позиции 24,80-25,08 Mbp (28), где расположен ген плеоморфной аденомы 1 ( PLAG1 ).
Ген PLAG1, состоящий из пяти экзонов и четырех интронов и имеющий пять транскриптов, был впервые обнаружен посредством позиционного картирования, когда исследователи изучали плеоморфные аденомы слюнных желез у человека (29). PLAG1 инициирует транскрипцию инсулиноподобного фактора роста 2 (IGF2), митогенного гормона, важного для роста и развития плода, и участвует в генетической изменчивости роста как у человека, так и у КРС (25, 30-32). Опубликовано значительное число работ, в которых исследовалась связь гена PLAG1 с легкостью отела (33), размерами тела (25), живой массой в раннем возрасте (14), характеристиками туши (34), массой при рождении (35) и другими признаками КРС (36). Российские локальные породы представляют большой интерес в качестве резерва биоразнообразия (37, 38), однако остаются неизученными в отношении гена PLAG1 несмотря на проведенные ранее полногеномные исследования (39, 40).
В настоящей работе впервые проведена оценка частоты встречаемости аллелей SNP, связанных с высотой в холке, в популяциях крупного рогатого скота, разводимых на территории России. В российских локальных породах установлено преобладание таких аллелей в трех из четырех выявленных однонуклеотидных полиморфизмов.
Целью работы была идентификация локусов и характеристика аллельных вариантов, находящихся под давлением отбора и связанных с размерами тела, в популяциях российских локальных и разводимых на территории Российской Федерации трансграничных пород крупного рогатого скота различных направлений продуктивности, по-разному подверженных давлению искусственного отбора и неодинаково распространенных в мире.
Мешодика. Исследовали биоматериал (цельную кровь, сперму и ушные выщипы, хранящиеся в УНУ «Банк генетического материала домашних и диких видов животных и птицы» ФГБНУ ФИЦ животноводства — ВИЖ им. академика Л.К. Эрнста) от 670 гол. крупного рогатого скота 13 пород: абердин-ангусской (ANG, n = 39), айрширской (AYR, n = 144), черно-пестрой (BLW, n = 50), голштинской (HLS, n = 184), истобенской (IST, n = 22), джерсейской (JRS, n = 32), калмыцкой (KLM, n = 27), холмогорской (KHL, n = 26), киргизской (KRG, n = 24), монгольской (MNG, n = 26), тагильской (TAG, n = 26), якутской (YKT, n = 29) и ярославской (YRS, n = 41).
ДНК выделяли с помощью коммерческих наборов ДНК-Экстран-1 и ДНК-Экстран-2 (ООО «Синтол», Россия) в соответствии с рекомендациями производителя. Перед генотипированием на SNP-чипах полученные препараты проходили контроль качества: измеряли концентрацию (не менее 15 нг/мкл) двуцепочечной ДНК (флуориметр Qubit 4.0, «Invitrogen, Life Technologies», США), а также соотношение OD 260 /OD 280 (допустимые значения 1,8-2,0), характеризующее чистоту препаратов (спектрофотометр NanоDrop ™ 8000 («Thermo Fisher Scientific, Inc.», США).
Образцы 11 пород генотипировали самостоятельно, генотипы двух пород — абердин-ангусской и джерсейской — были загружены из базы данных WIDDE .
Поскольку генотипирование образцов проводили на SNP-чипах разной плотности — GGP Bovine 150K и BovineHD BeadChip («Illumina, Inc.», США), в процессе обработки данных были определены общие для двух чипов SNP, которые использовались для дальнейшего анализа. Качество генотипирования контролировали в программе PLINK 1.9 (41) с использованием следующих фильтров: успешно генотипированы не менее 90 % локусов (--geno 0.1) не менее чем у 80 % животных (--mind 0.2), частота минорных аллелей не менее 5 % (--maf 0.05). После объединения наборов данных и контроля качества генотипирования для анализа отобрали 115237 SNP. C помощью PLINK 1.9 осуществляли полногеномное исследование ассоциаций (GWAS) однонуклеотидных полиморфизмов с высотой в холке. Поскольку SNP-чипы, на которых проводили генотипирование, были смоделированы с учетом позиций однонуклеотидных полиморфизмов, представленных в сборке генома Bos taurus UMD 3.1.1 , для поиска генов, расположенных вблизи значимо ассоциированных SNP, также использовалась вышеуказанная сборка.
Данные о высоте в холке для исследуемых пород были получены из базы ФАО (DAD-IS, . Поскольку величины показателей варьировали в разных странах, для каждой породы были рассчитаны средние значения без учета страны разведения и пола животного. Затем вычисляли среднее значение высоты в холке для всего массива исследованных пород. Расчеты и визуализацию результатов проводили в программной среде R с использованием собственных скриптов.
На основании данных о размерах тела животных все исследуемые породы были разделены на две группы. Породы, для которых средние значения высоты в холке не превышали средних значений, рассчитанных для всего массива, были отнесены к низкорослым (группа Low). Породы, для которых средние значения показателя превышали средние значения выборки, относили к высокорослым (группа High). Кроме того, исследованные породы подразделяли на группы по следующим критериям: в соответствии с направлением продуктивности (молочные, мясные), степенью давления селекции (примитивные, заводские) и распространенностью в мире (российские, трансграничные).
Достоверность разности частот встречаемости альтернативных аллелей для SNP, показавших значимые ассоциации с признаком высоты в холке, в разных группах животных определяли с использованием критерия χ 2 в программной среде R. При оценке достоверности разницы частот встречаемости альтернативных аллелей между трансграничными и российскими породами из анализа были исключены монгольский и киргизский скот, поскольку эти группы не разводят на территории России в товарных или племенных стадах.
Результаты. Характеристика исследованных пород КРС приведена в таблице 1.
1. Характеристика исследованных российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus )
|
Порода |
Аббревиатура |
Группа |
Географическая классификация по ФАО |
Направление продуктивности |
Селекционное давление |
|
Абердин-ангусская |
ANG |
High |
Трансграничные |
Мясное |
Заводские |
|
Айрширская |
AYR |
High |
Трансграничные |
Молочное |
Заводские |
|
Черно-пестрая |
BLW |
High |
Трансграничные |
Молочное |
Заводские |
|
Холмогорская |
KHL |
High |
Российские |
Молочное |
Заводские |
|
Голштинская |
HLS |
High |
Трансграничные |
Молочное |
Заводские |
|
Ярославская |
YRS |
High |
Российские |
Молочное |
Заводские |
|
Тагильская |
TAG |
High |
Российские |
Молочное |
Заводские |
|
Истобенская |
IST |
High |
Российские |
Молочное |
Заводские |
|
Калмыцкая |
KLM |
Low |
Трансграничные |
Мясное |
Заводские |
|
Джерсейская |
JRS |
Low |
Трансграничные |
Молочное |
Заводские |
|
Киргизский скот |
KRG |
Low |
- |
Мясное |
Примитивные |
|
Монгольский скот |
MNG |
Low |
- |
Мясное |
Примитивные |
|
Якутская |
YKT |
Low |
Российские |
Мясное |
Примитивные |
Примечание. Low — породы, для которых средние значения высоты в холке не превышали средних значений, рассчитанных для всего массива; High — породы, для которых средние значения показателя превосходили средние значения выборки, относили к высокорослым. Прочерки означают, что породы исключены из группы, поскольку не разводятся на территории России.
Полногеномное исследование ассоциаций однонуклеотидных полиморфизмов с показателем высоты в холке (рис. 1) было проведено для 13 пород крупного рогатого скота. Обнаружены статистически значимые (p-value < 1e - 80) ассоциации для некоторых однонуклеотидных полиморфизмов (рис. 2).
Выявлено в общей сложности четыре SNP, из которых три были локализованы на 4-й хромосоме, один — на 14-й хромосоме.
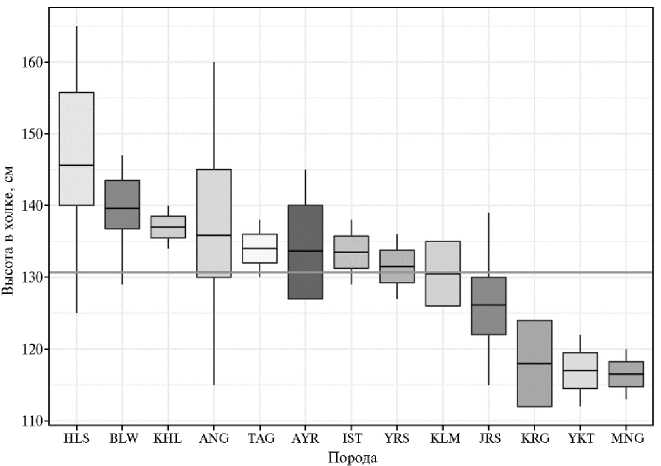
Рис. 1. Средние значения высоты в холке для российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus ): HLS — голштинская, BLW — черно-пестрая, KHL — холмогорская, ANG — абердин-ангусская, TAG — тагильская, AYR — айрширская, IST — ис-тобенская, YRS — ярославская, KLM — калмыцкая, JRS — джерсейская, KRG — киргизский скот, YKT — якутская, MNG — монгольский скот. На диаграммах размаха для каждой породы приведены значения нижнего квартиля, арифметической средней и верхнего квартиля. Горизонтальная линия — среднее значение, рассчитанное для всего массива исследованных пород.
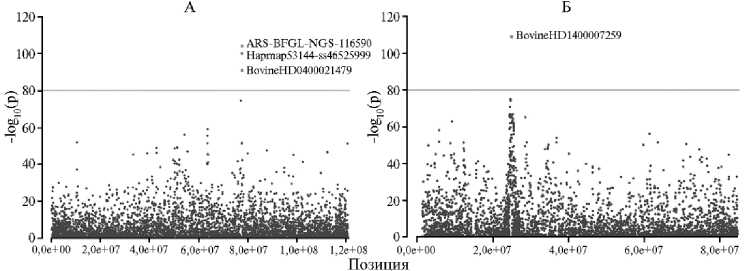
Рис. 2. Локализация однонуклеотидных полиморфизмов, значимо ассоциированных с показателем высоты в холке у российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus ): А — 4-я хромосома, Б — 14-я хромосома; горизонтальные линии — пороговые значения отрицательного логарифма.
-
2. Гены, обнаруженные в регионе локализации однонуклеотидных полиморфизмов, значимо ассоциированных с высотой в холке у российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus )
3. Частоты аллелей в локусах, значимо связанных с признаком высоты в холке, у российских локальных и трансграничных пород крупного рогатого скота (
Bos taurus taurus
)
|
Название SNP Хромосома Позиция Регион, Mb Гены |
Признаки |
|
Hapmap53144-ss46525999 4-я 77555681 77,3-77,8 PURB , MIR4657 , |
Продолжительность бе- |
BovineHD0400021479 4-я 77613816 H2AFV , PPIA , ZMIZ2 , ременности, синтез
|
OGDH , TMED4 , ARS-BFGL-NGS-116590 4-я 77635835 DDX56 , NPC1L1 , NUDCD3 , CAMK2B , YKT6 BovineHD1400007259 14-я 25015640 24,8-25,2 LYN , RPS20 , MOS , PLAG1 , CHCHD7 , SDR16C5 , SDR16C6 |
компонентов молока, белковый и жировой обмен, среднесуточный прирост массы Рост, конституция, потребление корма, фертильность |
Мы провели поиск генов, расположенных на участке ±200 Kb от обнаруженных SNP (табл. 2). В проведенных ранее исследованиях было показано, что регион 24,8-25,2 Mb на 14-й хромосоме содержит гены, для которых установлена связь с размерами тела (42, 43), потреблением корма (44, 45) и фертильностью (46). На 4-й хромосоме в регионе 77,3-77,8 Mb выявлены гены, связанные с обменом веществ (47), составом (48, 49) и синтезом (50) молока, среднесуточным приростом живой массы (51). S.M. Ghoreishifar с соавт. (52) изучали следы селекции в группе из пяти пород КРС, происходящих из северной, средней и южной частей Швеции. Участки генома с наиболее значимыми сигналами отбора обнаружены на BTA5 (105,75106,52 Mbp), BTA1 (2,25-2,52 Mbp) и BTA14 (24,42-25,11 и 25,35-25,73 Mbp). Аннотация генов в этих регионах выявила как уже известные, так и новые гены-кандидаты, ассоциированные с высотной гипоксией ( DCAF8 , PPP1R12A, SLC16A3 , UCP2 , UCP3 , TIGAR ), акклиматизацией к холоду ( AQP3 , AQP7 , HSPB8 ), размером тела и ростом ( PLAG1 , KCNA6 , NDUFA9 , AKAP3 , C5H5112orf1 , RAD51AP1 , FGF6 , TIGAR , CCND2 , CSMD3 ), устойчивостью к болезням и бактериальным инфекциям ( CHI3L2 , GBP6 , PPFIBP1 , REP15 , CYP4F2 , TIGD2 , PYURF , SLC10A2 , FCHSD2 , ARHGEF17 , RELT , PRDM2 , KDM5B ), с репродуктивными качествами ( PPP1R12A , ZFP36L2 , CSPP1 ), удоем и составом молока ( NPC1L1 , NUDCD3 , ACSS1 , FCHSD2 ).
Наши данные согласуются с опубликованными ранее, при этом регионы, описанные в предыдущих работах и расположенные на других хромосомах (53-55), не были выявлены в нашей выборке. Интересно, что в проведенных другими авторами исследованиях, в том числе с использованием контрастных пород, не обнаружено ассоциаций SNP, расположенных в регионе 77,3-77,8 Mb на BTA4, с высотой в холке (56-58).
Для выявленных SNP были рассчитаны частоты встречаемости альтернативных аллелей в каждой из пород (табл. 3).
|
Порода |
Группа |
4-я хромосома |
14-я хромосома |
||||||
|
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
||||||
|
T |
G a |
T a |
] C |
A |
G a |
T |
1 G a |
||
|
HLS |
High |
0,82 |
99,18 |
99,18 |
0,82 |
0,82 |
99,18 |
0,54 |
99,46 |
|
BLW |
High |
1,00 |
99,00 |
99,00 |
1,00 |
1,00 |
99,00 |
0,00 |
100,00 |
|
KHL |
High |
23,08 |
76,92 |
76,92 |
23,08 |
23,08 |
76,92 |
13,46 |
86,54 |
|
ANG |
High |
62,82 |
37,18 |
36,84 |
63,16 |
62,82 |
37,18 |
0,00 |
100,00 |
|
TAG |
High |
26,92 |
73,08 |
73,08 |
26,92 |
26,92 |
73,08 |
9,62 |
90,38 |
|
AYR |
High |
28,47 |
71,53 |
71,53 |
28,47 |
28,47 |
71,53 |
21,88 |
78,13 |
|
IST |
High |
45,00 |
55,00 |
54,55 |
45,45 |
36,36 |
63,64 |
9,52 |
90,48 |
|
YRS |
High |
45,12 |
54,88 |
55,00 |
45,00 |
45,12 |
54,88 |
17,50 |
82,50 |
|
KLM |
Low |
95,83 |
4,17 |
3,85 |
96,15 |
96,15 |
3,85 |
94,23 |
5,77 |
|
JRS |
Low |
95,31 |
4,69 |
4,69 |
95,31 |
95,31 |
4,69 |
100,00 |
0,00 |
|
KRG |
Low |
72,92 |
27,08 |
29,17 |
70,83 |
52,27 |
47,73 |
85,42 |
14,58 |
|
YKT |
Low |
98,28 |
1,72 |
0,00 |
100,00 |
87,50 |
12,50 |
98,28 |
1,72 |
|
MNG |
Low |
100,00 |
0,00 |
6,25 |
93,75 |
89,58 |
10,42 |
100,00 |
0,00 |
Примечание. HLS — голштинская, BLW — черно-пестрая, KHL — холмогорская, ANG — абердин-ангусская, TAG — тагильская, AYR — айрширская, IST — истобенская, YRS — ярославская, KLM — калмыцкая, JRS — джерсейская, KRG — киргизский скот, YKT — якутская, MNG — монгольский скот; Low — породы, для которых средние значения высоты в холке не превышали средних значений, рассчитанных для всего массива; High — породы, для которых средние значения показателя превышали средние значения выборки, относили к высокорослым; a — аллели, связанные с высоким ростом у крупного рогатого скота.
Наиболее существенные различия между частотами альтернативных аллелей наблюдались в группе низкорослых пород: в локусе ARS-BFGL-NGS-116590 частоты аллелей G / T варьировали соответственно от
0 до 27,08 % и от 72,92 до 100 %; в локусе Hapmap53144-ss46525999 частоты аллелей T / C — от 0 до 29,17 % и от 70,83 до 100 %; в локусе BovineHD1400007259 частоты аллелей G / T — от 0 до 14,58 % и от 85,42 до 100 %. У высокорослых пород наибольшая разница в частотах аллелей T / G (соответственно 0 и 100 %) наблюдалась в локусе BovineHD1400007259 у животных черно-пестрой и абердин-ангусской пород.
Оценить статистическую значимость различий между частотами альтернативных аллелей для каждой исследуемой породы в отдельности не удалось в связи с отсутствием в некоторых породах одного из аллелей, поэтому такая оценка была проведена для групп, сформированных по разным признакам (табл. 4).
4. Статистическая значимость различий между частотами альтернативных аллелей в локусах, связанных с признаком высоты в холке, в разных группах российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus )
|
Группа пород |
4-я хромосома |
14-я хромосома |
||
|
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
|
|
T 1 G a 1 p |
T a C 1 p |
A 1 G a 1 p |
T G aпp |
|
|
Высокорослые |
0,204 |
0,796 |
*** |
0,795 |
0,205 *** |
0,201 |
0,799 |
*** |
0,090 |
0,910 |
*** |
|
Низкорослые |
0,349 |
0,651 |
*** |
0,082 |
0,918 ** |
0,309 |
0,647 |
*** |
0,959 |
0,041 |
|
|
Мясные |
0,839 |
0,161 |
0,168 |
0,832 |
0,77 |
0,230 |
0,687 |
0,313 |
*** |
||
|
Молочные |
0,218 |
0,782 |
*** |
0,781 |
0,219 *** |
0,215 |
0,785 |
*** |
0,152 |
0,848 |
*** |
|
Заводские |
0,276 |
0,724 |
*** |
0,721 |
0,279 *** |
0,275 |
0,725 |
*** |
0,177 |
0,823 |
*** |
|
Примитивные |
0,909 |
0,091 |
*** |
0,112 |
0,888 *** |
0,777 |
0,223 |
*** |
0,948 |
0,052 |
|
|
Трансграничные |
0,256 |
0,744 |
*** |
0,740 |
0,260 *** |
0,259 |
0,741 |
*** |
0,187 |
0,813 |
*** |
|
Российские |
0,486 |
0,514 |
* |
0,514 |
0,486 * |
0,448 |
0,552 |
0,306 |
0,694 |
*** |
Примечание. a — аллели, связанные с высоким ростом у крупного рогатого скота.
* p < 0,05, ** p < 0,01; *** p < 0,001, пропуски означают, что статистически значимые различия отсутствуют.
В большинстве случаев частоты альтернативных аллелей в исследованных группах статистически значимо различались. Исключение составила группа мясного скота по всем трем SNP, обнаруженным на BTA4. Кроме того, частоты в SNP BovineHD1400007259 не отличались от теоретически ожидаемых в группе примитивного скота. Такая же ситуация наблюдается в группе низкорослого скота. Это может быть связано с тем, что три из пяти формирующих ее пород, относятся к примитивному скоту (киргизский и монгольский скот, якутская порода). Интересно отметить, что у абердин-ангусской породы, несмотря на то, что она была отнесена к группе крупных пород, в распределении частот аллелей на 4-й хромосоме наблюдались те же тенденции, что и в группе мелких пород.
С высокими значениями высоты в холке связан аллель G в локусе BovineHD0400021479, аллель G в локусе ARS-BFGL-NGS-116590, аллель G в локусе BovineHD1400007259 и аллель Ò в локусе Hapmap53144-ss46525999. Для SNP BovineHD1400007259, расположенного внутри гена PLAG1 , частоты аллеля G , связанного с высоким ростом, согласовывались с полученными в предыдущих исследованиях для голштино-фризской и джерсейской пород (25). Ранее J. Hou с соавт. (58) изучали связь SNP rs109815800 (AC_000171.1: g.25015640, G > T , обозначенный на чипе как BovineHD1400007259) в гене PLAG1 с ростом крупного рогатого скота. Результаты анализа показали, что животные с генотипом T / T были ниже ростом, чем особи с генотипами T / G или G / G (p < 0,001).
Выявлена статистически значимая корреляция между частотами альтернативных аллелей и показателем высоты в холке для всех обнаруженных однонуклеотидных полиморфизмов (табл. 5). Почти во всех группах пород установлены статистически значимые корреляции между частотами аллелей, связанных с высоким ростом в холке, и высотой в холке. Интересно, что для группы низкорослых пород, против ожидания, не выявлено достоверной отрицательной корреляции в локусе BovineHD1400007259.
-
5. Оценка корреляционных взаимодействий между показателем высоты в холке и частотой альтернативных аллелей в локусах, связанных с признаком высоты в холке, в разных группах российских локальных и трансграничных пород крупного рогатого скота ( Bos taurus taurus )
|
Группа пород |
4-я хромосома |
14-я хромосома |
||||||
|
ARS-BFGL-NGS-116590 |
Hapmap53144-ss46525999 |
BovineHD0400021479 |
BovineHD1400007259 |
|||||
|
T / G a |
p |
T a/ C 1 |
p |
A / G a |
p |
T / G a |
p |
|
|
Высокорослые |
0,722 |
*** |
0,719 |
*** |
0,708 |
*** |
0,678 |
*** |
|
Низкорослые |
- 0,172 |
- 0,281 |
- 0,498 |
** |
- 0,063 |
|||
|
Мясные |
0,530 |
*** |
0,456 |
* |
0,090 |
0,767 |
*** |
|
|
Молочные |
0,908 |
*** |
0,908 |
*** |
0,907 |
*** |
0,751 |
*** |
|
Заводские |
0,829 |
*** |
0,827 |
*** |
0,821 |
*** |
0,729 |
*** |
|
Примитивные |
0,962 |
*** |
0,859 |
*** |
0,96 |
*** |
0,975 |
*** |
|
Трансграничные |
0,876 |
*** |
0,875 |
*** |
0,876 |
*** |
0,840 |
*** |
|
Российские |
0,981 |
*** |
0,982 |
*** |
0,989 |
*** |
0,971 |
*** |
|
Вся выборка |
0,838 |
*** |
0,826 |
*** |
0,752 |
** |
0,854 |
*** |
Примечание. a — аллели, связанные с высоким ростом у крупного рогатого скота. Коэффициенты корреляции показаны для аллеля, связанного с высоким ростом в холке.
* p < 0,05, ** p < 0,01; *** p < 0,001, пропуски означают, что статистически значимые различия отсутствуют.
Таким образом, идентифицированы однонуклеотидные полиморфизмы ARS-BFGL-NGS-116590, Hapmap53144-ss46525999, BovineHD0400021479 и BovineHD1400007259, локализованные, согласно сборке генома UMD 3.1.1, на 4-й и 14-й хромосомах и ассоциированные с высотой в холке у крупного рогатого скота, разводимого на территории Российской Федерации. Частоты выявленных SNP в разных группах пород значимо отличались от случайно возможных. Альтернативные аллели в обнаруженных SNP имели статистически значимые положительные либо отрицательные корреляции с показателем высоты в холке. Следовательно, можно сделать вывод о неодинаковом селекционном давлении на эти локусы для разных групп пород. Многообразие и разнородность пород, представленных в выборке, позволяет рассматривать выявленные следы селекции как характерные не для одной породы, региона или направления продуктивности, а для группы пород вида Bos taurus taurus , распространение которых от центра одомашнивания шло по Дунайскому пути.


