Получение и анализ композитных растений томата Solanum lycopersicum L., трансформированных генами рецепторов сигнальных молекул ризобий у гороха
Автор: Рудая Е.С., Долгих Е.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Симбиотические взаимодействия
Статья в выпуске: 3 т.56, 2021 года.
Бесплатный доступ
В основе развития бобово-ризобиального симбиоза лежит сигнальный обмен между партнерами, что обеспечивает их взаимное узнавание, активацию инфекционного процесса и программы органогенеза клубеньков. Интерес представляет изучение возможности приобретения небобовыми растениями способности к рецепции сигнальных молекул ризобий - липохитоолигосахаридных сигналов Nod-факторов и дальнейшей трансдукции сигнала. У гороха ранее были выявлены две рецепторные киназы - SYM10 и K1, которые необходимы для узнавания Nod-факторов при инициации симбиоза с ризобиями. При рецепции Nod-факторов формируется комплекс между этими двумя рецепторными киназами, что приводит к сигнальной трансдукции. В настоящей работе был осуществлен перенос двух генов, кодирующих LysM-РПК SYM10 и K1 у гороха Pisum sativum L., в растения томата Solanumlycopersicum L. с помощью агробактериальной трансформации. У композитных растений, трансформированных генами PsSym10 или PsK1 , впервые выявлена возможность активации экспрессии перенесенных генов рецепторов под влиянием инокуляции типовым штаммом ризобий Rhizobiumleguminosarum bv. viciae CIAM1026. Также показано, что под влиянием рецепторов в генетически трансформированных корнях композитных растений увеличивается экспрессия генов, которые могут регулироваться компонентами «общего» сигнального пути. Целью работы было изучение возможности приобретения растениями S. lycopersicum способности к узнаванию сигнальных молекул ризобий за счет переноса генов, кодирующих рецепторы Nod-факторов у бобового растения P. sativum . Для проведения экспериментов были получены и использованы два вида конструкций в векторе pKm43GW, в которых гены рецепторов PsSym10 или PsK1 были клонированы под промотором гена экстенсина томата pSlEXT1 - pSlEXT1:: PsSym10-3xFLAG ::T35S и pSlEXT1:: PsK1-RFP ::T35S. Молодые проростки томата S. lycopersicum сорта Carmello были трансформированы штаммом Agrobacterium rhizogenes Arqua 1. Трансформированные проростки помещали на агаризованную среду Мурасиге-Скуга (MC) без сахарозы в чашках Петри и культивировали в вертикальном положении в фитотроне до появления каллуса. После этого растения переносили на среду МС c 3 % cахарозы, содержащую 0,3 мг/мл антибиотика цефотаксима, и инкубировали до появления трансгенных корней. Композитные растения переносили в вермикулит, политый 0,5½ средой Fahreus и инкубировали в условиях повышенной влажности в течение 2-3 сут. После этого растения были инокулированы штаммом R. legumi-nosarum bv. viciae CIAM1026, содержащим ген глюкуронидазы uidA ( GUS ). Для анализа использовали трансформированные корни композитных растений томата без инокуляции ризобиями (контроль, 7 сут), а также трансформированные корни через 7 и 21 сут после инокуляции. Количественный анализ экспрессии генов проводили методом количественной ПЦР, совмещенной с обратной транскрипцией (ОТ-ПЦР). При использовании промотора pSlEXT1 наблюдали экспрессию обоих генов PsSym10 и PsK1 в генетически трансформированных корнях растений томата, при этом экспрессия усиливалась под влиянием инокуляции ризобиями. Было показано существенное (примерно в 2,0-2,5 раза) повышение экспрессии гена PsSym10 в ответ на инокуляцию ризобиями как на 7-е, так и на 21-е сут. Уровень экспрессия PsK1 оказался самым высоким через 7 сут после инокуляции в трансформированных корнях композитных растений томата по сравнению с контролем. Чтобы определить, будут ли активироваться компоненты «общего» сигнального пути под влиянием перенесенных рецепторов в композитных растениях томата, были оценены изменения в экспрессии генов S. lycopersicum SlD27 , SlNSP2 , SlRAM1 и SlMAPK6 . Эти гены кодируют каротиноид-изомеразу (DWARF27), регулирующую синтез гормонов стриголактонов, транскрипционные факторы NSP2 и RAM1, а также митоген-активируемую протеинкиназу (MAPK6). Активация экспрессии двух генов SlNSP2 и SlMAPK6 в ответ на инокуляцию может свидетельствовать о влиянии перенесенного гена K1 гороха на восприимчивость растений томата к инокуляции ризобиями.
Бобово-ризобиальный симбиоз, рецептор-подобные киназы, nod-факторы, композитные растения томата, экспрессия генов
Короткий адрес: https://sciup.org/142231363
IDR: 142231363 | УДК: 635.64:573.6.086.83:577.21]:58 | DOI: 10.15389/agrobiology.2021.3.465rus
Текст научной статьи Получение и анализ композитных растений томата Solanum lycopersicum L., трансформированных генами рецепторов сигнальных молекул ризобий у гороха
Способность формировать симбиозы с азотфиксирующими клубеньковыми бактериями порядка Rhizobiales, обобщенно называемыми ри-зобиями, — важное эволюционное приобретение растений, обеспечивающее преимущество в условиях недостатка азота в почве. Однако только представители порядка Fabales и отдельные представители порядка Rosales (некоторые виды Parasponia, сем. Cannabaceae) формируют симбиозы с ри-зобиями (1). Исследования, направленные на расширение круга растений, вступающих в симбиоз с ризобиями, открывают новые перспективы в развитии эффективного земледелия и минимизации использования азотных удобрений. Их важный этап — оценка возможности приобретения небобовыми растениями способности к рецепции сигнальных молекул ризобий и дальнейшей передаче сигнала, что лежит в основе узнавания партнеров.
Липохитоолигосахаридные сигнальные молекулы — Nod-факторы, выделяемые ризобиями, служат ключевыми медиаторами симбиоза у бобовых растений. Именно от узнавания этих молекул растительным партнером зависит органогенез клубеньков и бактериальная колонизация (2). Соединения очень схожей структуры — липохитоолигосахариды Myc-факторы, выделяемые грибами арбускулярной микоризы (АМ) (3), выполняют роль сигнальных молекул, необходимых для развития другого типа симбиоза с грибами AM, который формируют более 80 % наземных растений. Возможность активации ответных реакций при узнавании Nod-факторов может быть связана с наличием у небобовых растений компонентов сигнального пути, общих для передачи сигнала при развитии бобово-ризобиального симбиоза и симбиоза растений с грибами АМ (4, 5). Различия заключаются главным образом в узнавании Nod- и Myc-факторов разными рецепторами и активации специфичных для каждого пути транскрипционных факторов, которые стимулируют свои гены-мишени (6, 7).
В рецепцию Nod- и Myc-факторов вовлечены представители семейства LysM-рецептор-подобных киназ (LysM-РПК), имеющие специфичные LysM-мотивы во внеклеточных доменах. Среди LysM-РПК принято выделять белки класса LYK (LysM-receptor-like kinases) с активным киназным доменом и LYR (LYK related), у которых киназный домен неактивен. Присутствие LysM-мотивов определяет способность таких рецепторов связывать соединения, содержащие остатки N-ацетилглюкозамина (Nod- и Myc-факторы состоят в среднем из 4-5 остатков N-ацетилглюкозамина и содержат специфичную жирную кислоту на не восстанавливающем конце молекулы) (8-10). Некоторые представители семейства LysM-РПК узнают структурно сходные молекулы, имеющие в своем составе остатки N-аце-тилглюкозамина, такие как хитин, пептидогликан муреин и их низкомолекулярные производные (11, 12). Оказалось, что при рецепции как Nod-, так и Myc-факторов у растений должен формироваться комплекс между LYR и LYK белками, что приводит к сигнальной трансдукции (13, 14).
Способность формировать симбиоз с азотфиксирующими ризо-биями выявлена и у ряда представителей небобовых растений, таких как Parasponia andersonii Planch. (сем. Cannabaceae ). У Parasponia была найдена LysM-РПК PanNFP (класс LYR), которая может узнавать сигналы как ри-зобий, так и грибов АМ (15). При этом предполагается, что PanNFP формирует комплексы с разными ко-рецепторами (LYK) при узнавании Nod-и Myc-факторов.
У других небобовых растений, например у томата Solanum lycoper-sicum L., недавно выявлена LysM-РПК SlLYK10 (LYR), которая имеет высокую степень гомологии с рецептором Nod-факторов бобовых растений — MtNFP люцерны слабоусеченной Medicago truncatula Gaertn. (16). SlLYK10 в комплексе с ко-рецептором LysM-РПК SlLYK12 (LYK) связывает Myc-факторы. Более того, введение гена SlLYK10 под сильным промотором в мутанты растений M. truncatula по гену nfp , дефектные по гену рецептора
Nod-факторов, приводило к восстановлению клубенькообразования и формированию функциональных клубеньков (17, 18). Выявленная способность указанного рецептора томата восстанавливать процесс узнавания Nod-факторов у бобовых растений позволила объяснить результаты более ранних исследований, в которых была показана отзывчивость клеток растений томата на добавление Nod-факторов в среду и развитие наиболее ранних ответных реакций на эти сигналы (подщелачивание ростовой среды, деполяризация мембраны) (19). Можно предположить, что способность узнавать Nod-факторы растениями томата связана с активацией комплекса рецепторов Myc-факторов SlLYK10/SlLYK12 из-за сходства структуры сигнальных молекул. Действительно, недавно при изучении LysM-РПК SlLYK10 была показана способность этого рецептора с высокой аффинностью связывать не только Myc-факторы, но и Nod-факторы (18). Такая восприимчивость небобовых растений к сигнальным молекулам ризобий предполагает возможность активации ими компонентов «общего» сигнального пути (ОСП). В связи с этим представляет интерес изучение возможности переноса в небобовые растения генов, кодирующих рецепторы Nod-факторов ризобий с более высокой аффинностью и специфичностью к указанным молекулам, а также оценка отзывчивости растений на эти сигналы. Необходимо выяснить, задействованы ли в передаче сигнала компоненты ОСП и какие транскрипционные факторы, а также гены-мишени при этом активируются.
У гороха Pisum sativum L. были выявлены две LysM-РПК — SYM10 и K1 (LYR и LYK), которые необходимы для узнавания Nod-факторов при инициации симбиоза с ризобиями (20, 21). При этом у мутантов гороха по генам sym10 и k1 практически полностью отсутствуют ответные реакции на инокуляцию ризобиями и добавление Nod-факторов, что свидетельствует о важности этих рецепторов для развития симбиоза (20, 21).
В настоящей работе был осуществлен перенос двух генов, кодирующих LysM-РПК SYM10 и K1 у гороха Pisum sativum L., в растения томата S. lycopersicum с помощью агробактериальной трансформации. У композитных растений, трансформированных генами PsSym10 или PsK1 , впервые выявлена возможность активации экспрессии перенесенных генов рецепторов под влиянием инокуляции типовым штаммом ризобий Rhizobium legu-minosarum bv. viciae CIAM1026. Также показано, что под влиянием рецепторов в генетически трансформированных корнях композитных растений увеличивается экспрессия генов, которые могут регулироваться компонентами ОСП.
Целью работы было изучение возможности приобретения растениями Solanum lycopersicum L. способности к узнаванию сигнальных молекул ризобий за счет переноса генов, кодирующих рецепторы Nod-факторов бобового растения Pisum sativum L.
Ìåòîäèêà. Семена томата сорта Carmello стерилизовали 15 % гипохлоритом натрия NaOCl (0,1 M) в течение 5 мин, далее промывали 6 раз дистиллированной стерильной водой и выдерживали в 10 % H 2 O 2 в течение 2 мин. После этого промывали семена 3 раза большими объемами стерильной воды. Стерилизованные семена помещали в чашки Петри на агаризо-ванную среду Мурасиге-Скуга (МС) без сахарозы (22) и выдерживали в темноте в течение 1 сут при 4 ° С. Далее чашки переносили в условия комнатной температуры и проращивали в темноте в течение 5-7 сут. Молодые проростки переносили в стерильные сосуды (объем 400 мл) для выращивания растений со средой МС, содержащей 3 % сахарозу, и культивировали в условиях фитотрона MLR-352H («Panasonic», Япония) при 21 ° C, 60 % влажности и фотопериоде 16 ч свет/8 ч темнота.
Штамм Rhizobium leguminosarum biovar viciae CIAM1026 культивировали при 28 ° С на агаризованной среде TY (tryptone yeast) с добавлением 0,5 мг/мл стрептомицина. Для стандартных процедур клонирования использовали штаммы Escherichia ñoli XLBlue MRF’ и TOP10 («Thermo Fisher Scientific», США). Для получения композитных растений использовали штамм Agrobacterium rhizogenes Arqua 1, содержащий необходимую конструкцию. A. rhizogenes Arqua 1 культивировали при 28 ° С на агаризованной среде TY.
Для трансформации растений были получены две конструкции — pSlEXT1:: PsSym10 - 3xFLAG ::T35S и pSlEXT1:: PsK1 - RFP ::T35S. Кодирующую полноразмерную последовательность гена PsSym10 без стоп-кодона амплифицировали с использованием в качестве матрицы кДНК (суммарную РНК выделяли из клубеньков гороха сорта Finale, собранных на 21-е сут после инокуляции) (20). Амплификацию проводили с использованием высокоточной Phusion полимеразы («Thermo Fisher Scientific», США) (20). Последовательность гена PsSym10 была слита с последовательностью, кодирующей 3xFLAG, и далее перенесена в вектор pDONR221 («Thermo Fisher Scientific», США). Для работы также использовали ранее полученную конструкцию в векторе pDONR221, которая содержала полноразмерную кодирующую последовательность гена PsK1 (без стоп-кодона), слитую с последовательностью, кодирующей флуоресцентный белок RFP (20).
Вектор pDONR L4-pEXT1-R1r, содержащий промотор гена экстен-сина томата pEXT1 (1121 п.н.), был предоставлен доктором S. Bensmihen (Institut National de la Recherche Agronomique, г. Тулуза, Франция). Вектор с промотором pDONR L4-pEXT1-R1r использовали для мультилокусной гомологичной рекомбинации. Для рекомбинации также применяли векторы pDONR221, содержащие кодирующую последовательность гена PsK1 либо гена PsSym10 , и вектор pENTRY R2-T35S-L3 с терминатором T35S (Ghent University, Бельгия). Необходимые конструкции вводили в вектор доставки pKm43GW (pDEST4-3) с использованием LR-клоназы II («Thermo Fisher Scientific», США). pKm43GW использовали в качестве конечного вектора для клонирования на основе гомологичной рекомбинации. Полученные конструкции в векторе pKm43GW (pKm43GW-pSlEXT1:: PsSym10 - 3xFLAG ::T35S и pKm43GW-pSlEXT1:: PsK1 - RFP ::T35S) вводили в штамм A. rhizogenes Ar-qua1 методом электропорации.
Молодые проростки томата обрезали в области гипокотиля и трансформированы штаммом A. rhizogenes Arqua 1 посредством нанесения бактериальной массы на место поранения. Трансформированные проростки (45 шт.) помещали на агаризованную среду МС без сахарозы (22) в чашку Петри между двумя листами фильтровальной бумаги, смоченной в стерильной дистиллированной воде. Область корней на чашках закрывали фольгой. Чашки помещали в фитотрон и культивировали в вертикальном положении при температуре 21 ° C, 60 % влажности и с фотопериодом 16 ч свет/8 ч темнота в течение 10-14 сут до появления каллуса. После этого растения переносили на среду МС c cахарозой (3 %), содержащую 0,3 мг/мл антибиотика цефотаксима и дополнительно инкубировали в тех же условиях в течение 5-10 сут до появления трансгенных корней. Композитные растения переносили в вермикулит, политый 0,5½ средой Fahreus (0,132 г/л CaCl 2 , 0,12 г/л MgSO 4 •7H 2 O, 0,1 г/л KH 2 PO 4 , 0,075 г/л Na 2 HPO 4 •2H 2 O, 5 мг/л Fe-цитрат, 0,07 мг/л MnCl 2 •4H 2 O, CuSO 4 •5H 2 O, ZnCl 2 , H 3 BO 3 и Na 2 MoO 4 •2H 2 O; pH 7,5), и инкубировали в условиях повышенной влажности (прикрывали прозрачными полиэтиленовыми пакетами) в течение 2-3 сут (21 ° С, 16 ч свет/8 ч темнота). Затем растения инокулировали штаммом
R. leguminosarum bv. viciae CIAM1026, содержащим ген глюкуронидазы uidA ( GUS ), с оптической плотностью суспензии OD 600 = 0,5 (по 2 мл на растение). После культивирования с ризобиями в течение 7 и 21 сут из трансформированных корней растений томата, несущих конструкцию pSlEXT1:: PsSym10-3xFLAG ::T35S или pSlEXT1:: PsK1-RFP ::T35S, выделяли суммарную РНК и синтезировали кДНК. В качестве контроля использовали трансгенные корни растений без инокуляции ризобиями.
В экспериментах суммарную РНК выделяли с использованием реагента PureZol («Bio-Rad Laboratories», США) в соответствии с протоколом производителя. Для удаления геномной ДНК применяли обработку DNAseI («Thermo Fisher Scientific», США). кДНК получали с помощью обратной транскриптазы RevertAid H Minus («Thermo Fisher Scientific», США) с использованием праймера олиго(дТ18) («Sileks», Россия) в соответствии с протоколом производителя.
Анализ экспрессии генов проводили методом количественной ПЦР, совмещенной с обратной транскрипцией (ОТ-ПЦР), на амплификаторе CFX96 Real-Time System («Bio-Rad Laboratories», США) со смесью iQ SYBR Green Super Mix («Bio-Rad Laboratories», США). Условия проведения ПЦР: 30 с при 95 °С, 30 с при 54 °C, 40 с при 72 °C (40 циклов). Все праймеры были разработаны с использованием программы DNAStar и синтезированы в компании Евроген .
Для подсчета усредненных значений относительной экспрессии генов в количественной ОТ-ПЦР и стандартной ошибки среднего (±SEM) использовали встроенные функции программы Microsoft Excel.
Ðåçóëüòàòû. Для трансформации растений томата сорта Carmello (рис. 1) генами PsSym10 и PsK1, кодирующими рецепторы Nod-факторов гороха, использовали штамм Agrobacterium rhizogenes Arqua 1, который содержал два типа конструкций — pSlEXT1:: PsSym10-3xFLAG ::T35S или pSlEXT1:: PsK1-RFP ::T35S).
Рис. 2. Корень композитных растений томата Sola-num lycopersicum L. сорта Carmello, трансформированных конструкцией pSlEXT1:: PsK1-RFP ::T35S, под флуоресцентным бинокуляром («Zeiss», Германия) . Выявлено свечение флуоресцентного белка RFP в клетках.

Рис. 1. Агробактериальная трасформация растений томата Solanum lycopersicum L. сорта Carmello.
В варианте с конструкцией pSlEXT1::PsK1-RFP::T35S, в которой ген рецептора К1 был слит с последовательностью, кодирующей флуоресцентный белок RFP, нам удалось выявить избирательное свечение ге- нетически трансформированных корней (рис. 2). Это свидетельствовало об экспрессии перенесенного гена в трансформированных корнях томата. Растения были трансформированы генами, кодирующими полноразмерные последовательности белков с N-концевыми сигнальными пептидами. Судя по степени свечения RFP, сигнальные пептиды нормально функционировали в растениях томата.
На следующем этапе с помощью метода количественной ПЦР, совмещенной с обратной транскрипцией (ОТ-ПЦР), провели анализ экспрессии генов PsSym10 и PsK1 в корнях композитных растений томата S. lyco-persicum , культивируемых в течение 7 и 21 сут (последовательности праймеров, использованных для ОТ-ПЦР, приведены в таблице). Были использованы трансформированные корни растений томата, несущих конструкцию pSlEXT1:: PsSym10-3xFLAG ::T35S или pSlEXT1:: PsK1-RFP ::T35S, через 7 и 21 сут после инокуляции ризобиями R. leguminosarum bv. viciae CIAM1026, а также трансформированные корни композитных растений томата без инокуляции ризобиями (контроль, 7 сут).
Последовательности праймеров, использованных для проведения количественной ОТ-ПЦР
|
Название гена |
Последовательность праймера |
|
PsK1- F PsK1- R PsSym10- F PsSym10- R SlNSP2- F SlNSP2- R SlMAPK6- F SlMAPK6- R SlD27- F SlD27- R SlRAM1- F SlRAM1- R SlGADH- F SlGADH- R |
5´-CGCGATGTAAAATCAGCAAACATATTG-3´ 5´-CGTCACCATATTGAGCATATTCTGG-3´ 5´-GTACTTCATTGGCGGAGACTG-3´ 5´-CCATAAGTTTCACAAGATTTCCAT-3´ 5´-AAGGCCGATAGGAGACGAAGAAGG-3´ 5´-CCCCACCCCACTCAACCACTC-3´ 5´-CGCGCTTGCTCATCCTTACCTA-3´ 5´-GTGCTGGTATTCGGGATTAAATG-3´ 5´-GCTACCACAGGATTAAGAAACAAG-3´ 5´-CCAACTAGCCAAGGAAAGAAGAT-3´ 5´-GGAAGCGGTCAGGGAAACAGG-3´ 5´-CCAGGAACCGACCCAGGAAATAC-3´ 5´-TGAGAATCAACACACTTCTCCAAGG-3´ 5´-GCATTAAGAATTTCCCCAGAGGTC-3´ |
Анализ показал (рис. 3), что ген PsSym10 экспрессируется только в корнях растений, трансформированных конструкцией pSlEXT1:: PsSym10-3xFLAG ::T35S. Сходным образом экспрессию гена PsÊ1 выявили только в корнях растений, трансформированных конструкцией pSlEXT1:: PsK1-RFP ::T35S (см. рис. 3). Следовательно, после агробактериальной трансформации и переноса генов гороха PsSym10 и PsK1 в ДНК клеток корней томата происходила стимуляция экспрессии этих генов.
Наблюдалось существенное (примерно в 2,0-2,5 раза) увеличение экспрессии гена PsSym10 в ответ на инокуляцию ризобиями как на 7-е, так и на 21-е сут после инокуляции. Повышение уровня экспрессии гена PsK1 оказалось наиболее значительным через 7 сут после инокуляции в корнях у композитных растений томата по сравнению с контрольными. Через 21 сут экспрессия гена PsK1 снижалась, но оставалась более высокой по сравнению с контрольными корнями без инокуляции. Следовательно, оба гена гороха PsSym10 и PsK1 могут активироваться при использовании промотора гена экстенсина pSlEXT1 в композитных растениях томата.
Известно, что промотор гена экстенсина регулируется этиленом (23). Вместе с тем установлено, что при инокуляции бобовых растений ри-зобиями в корнях может повышаться продукция этилена (24). Относительно высокая экспрессия генов гороха при использовании промотора гена экс-тенсина может объясняться повышением содержания этилена в корнях растений под влиянием инокуляции ризобиями. Использование подобного промотора может быть удобным для изучения влияния перенесенных генов бобовых на растения томата при инокуляции ризобиями.
Известно, что компоненты «общего» сигнального пути (ОСП) активируются после связывания сигнальных молекул с рецепторами, локализованными в плазматической мембране клеток корней растений (25). Ранее было показано, что корни растений томата способны воспринимать Nod- факторы. При этом под влиянием сигнальных молекул могут активироваться такие ответные реакции, как изменение потока ионов через мембрану и ее деполяризация (18). Однако оставалось неизвестным, могут ли Nod-факторы активировать компоненты ОСП у небобовых растений.
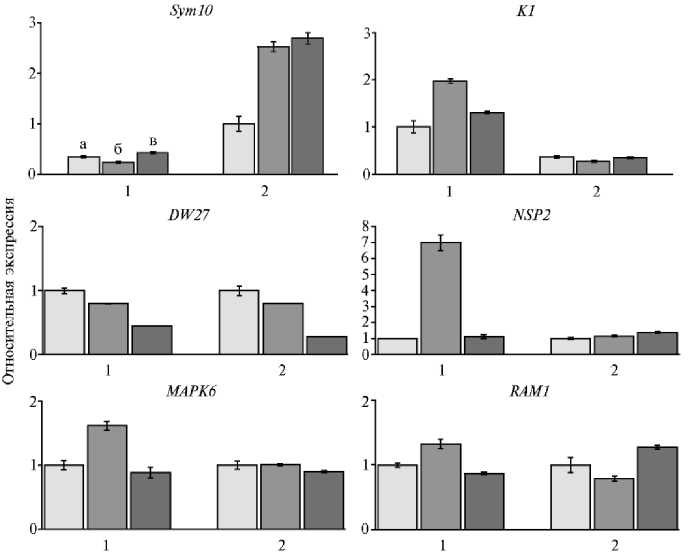
Рис. 3. Относительная экспрессия генов PsSym10 , PsÊ1 , SlD27 , SlNSP2 , SlMAPK6 и SlRAM1 в корнях композитных растений томата Solanum lycopersicum L. сорта Carmello, трансформированных конструкциями pSlEXT1:: PsK1-RFP ::T35S (1) и pSlEXT1:: PsSym10-3xFLAG ::T35S (2) , при инокуляции штаммом Rhizobium leguminosarum bv. viciae CIAM1026: а — контроль (без инокуляции), б — 7-е сут после инокуляции, в — 21-е сут после инокуляции. Уровень транскрипции мРНК изучаемого гена нормализован по отношению к экспрессии гена GADH томата, значения представлены в виде относительной экспрессии генов в варианте с инокуляцией к контролю. Бары представляют стандартные ошибки среднего (±SEM) для трех технических повторностей при анализе экспрессии. В каждом варианте РНК выделяли из 3-5 трансгенных корней.
Мы решили проверить, как будет влиять перенос генов гороха, кодирующих два рецептора к Nod-факторам, на экспрессию маркеров, которые в норме активируются компонентами ОСП, а также выяснить, будут ли активироваться компоненты ОСП под влиянием перенесенных рецепторов в композитных растениях томата при узнавании Nod-факторов ризобий. С этой целью сравнили экспрессию генов, которые считаются маркерами симбиоза растений с грибами АМ (активируются в результате передачи сигнала при формировании симбиоза), в трансформированных корнях томата без инокуляции и при инокуляции ризобиями. Предварительно нами был проведен поиск последовательностей генов у томата, которые могут быть гомологами генов бобовых растений, активируемых при симбиозе с грибами АМ. Прежде всего, это гены β -carotene isomerase DWARF27 (D27), транскрипционного фактора Nodulation Signaling Pathway 2 (NSP2), транскрипционного фактора Required for Arbuscular Mycorrhization (RAM1), а также Mitogen-Activated Protein Kinase (MAPK6) (12). Анализ экспрессии маркерных генов проводили на 7-е и 21-е сут после инокуляции в трансгенных корнях композитных растений томата при инокуляции штаммом R. legumi-nosarum bv. viciae CIAM1026 (см. рис. 3).
В корнях растений томата с конструкцией pSlEXT1:: PsSym10-3xFLAG ::T35S не было выявлено существенных изменений в экспрессии генов SlD27 , SlNSP2 , SlRAM1 и SlMAPK6 (см. рис. 3). У растений, трансформированных конструкцией pSlEXT1:: PsK1-RFP ::T35S, экспрессия SlD27 и SlRAM1 также значительно не изменялась в ответ на инокуляцию, однако экспрессия SlNSP2 и SlMAPK6 заметно увеличивалась (см. рис. 3). Активация этих маркеров может свидетельствовать о влиянии перенесенного гена гороха PsK1 на восприимчивость растений томата к инокуляции ризобиями. В дальнейшем будет проведен более детальный анализ морфологических изменений у растений, трансформированных конструкцией pSlEXT1:: PsK1-RFP ::T35S. Большой интерес может представлять получение и анализ растений, которые будут трансформированы двумя генами PsSym10 и PsÊ1 одновременно (в одной генетической конструкции), поскольку у гороха в связывании Nod-факторов участвуют оба этих рецептора в составе комплекса.
Таким образом, нам удалось получить композитные растения томата сорта Carmello, трансформированные генами гороха PsSym10 и PsK1 , которые кодируют рецепторы Nod-факторов. Интеграция генов Sym10 и K1 в геном томата была проведена с использованием векторных конструкций pSlEXT1:: PsSym10 - 3xFLAG ::T35S и pSlEXT1:: PsK1 - RFP ::T35S и подтверждена методом ПЦР. Под влиянием инокуляции штаммом Rhizobium leguminosarum bv. viciae CIAM1026 в трансгенных корнях композитных растений усиливалась экспрессия как гена PsSym10 , так и гена PsÊ1 . В корнях, трансформированных конструкцией pSlEXT1:: PsK1-RFP ::T35S, избирательно увеличивалась экспрессия генов SlNSP2 и SlMAPK6 , которые активируются под влиянием компонентов «общего» сигнального пути. Активация экспрессии двух генов — SlMAPK6 и SlNSP2 в ответ на инокуляцию может свидетельствовать о влиянии перенесенного гена рецептора гороха на восприимчивость растений томата к инокуляции ризобиями.
Список литературы Получение и анализ композитных растений томата Solanum lycopersicum L., трансформированных генами рецепторов сигнальных молекул ризобий у гороха
- van Velzen R., Holmer R., Bu F., Rutten L., van Zeijl A., Liu W., Santuari L., Cao Q., Sharma T., Shen D., Roswanjaya Y., Wardhani T.A.K., Kalhor M.S., Jansen J., van den Hoogen J., Güngör B., Hartog M., Hontelez J., Verver J., Yang W.C., Schijlen E., Repin R., Schilthuizen M., Schranz M.E., Heidstra R., Miyata K., Fedorova E., Kohlen W., Bisseling T., Smit S., Geurts R. Comparative genomics of the nonlegume Parasponia reveals insights into evolution of nitrogen-fixing rhizobium symbioses. Proceedings of the National Academy of Sciences, 2018, 115(20): E4700-E4709 (doi: 10.1073/pnas.1721395115).
- Murray J.D. Invasion by invitation: rhizobial infection in legumes. Molecular Plant-Microbe In-teractions, 2011, 24(6): 631-639 (doi: 10.1094/MPMI-08-10-0181).
- Cullimore J.V., Ranjeva R., Bono J.-J. Perception of lipo-chitooligosaccharidic Nod factors in legumes. Trends in Plant Science, 2001, 6(1): 24-30 (doi: 10.1016/S1360-1385(00)01810-0).
- Martin F.M., Uroz S., Barker D.G. Ancestral alliances: plant mutualistic symbioses with fungi and bacteria. Science, 2017, 356(6340): eaad4501 (doi: 10.1126/science.aad4501).
- de Bruijn F.J. The common symbiotic signaling pathway (CSSP or SYM). In: The model legume Medicago truncatula /F.J. de Bruijn (ed.). John Wiley & Sons, New Jersey, 2020 (doi: 10.1002/9781119409144.part8).
- Diédhiou I., Diouf D. Transcription factors network in root endosymbiosis establishment and de-velopment. World Journal of Microbiology and Biotechnology, 2018, 34(3): 37 (doi: 10.1007/s11274-018-2418-7).
- Xue L., Klinnawee L., Zhou Y., Saridis G., Vijayakumar V., Brands M., Dörmann P., Gigolash-vili T., Turck F., Bucher M. AP2 transcription factor CBX1 with a specific function in symbiotic exchange of nutrients in mycorrhizal Lotus japonicus. Proceedings of the National Academy of Sci-ences, 2018, 115(39): E9239-E9246 (doi: 10.1073/pnas.1812275115).
- Broghammer A., Krusell L., Blaise M., Sauer J., Sullivan J.T., Maolanon N., Vinther M., Lo-rentzen A., Madsen E.B., Jensen K.J., Roepstorff P., Thirup S., Ronson C.W., Thygesen M.B., Stougaard J. Legume receptors perceive the rhizobial lipochitin oligosaccharide signal molecules by direct binding. Proceedings of the National Academy of Sciences, 2012, 109(34): 13859-13864 (doi: 10.1073/pnas.1205171109).
- Fliegmann J., Canova S., Lachaud C., Uhlenbroich S., Gasciolli V., Pichereaux C., Rossignol M., Rosenberg C., Cumener M., Pitorre D., Lefebvre B., Gough C., Samain E., Fort S., Driguez H., Vauzeilles B., Beau J.M., Nurisso A., Imberty A., Cullimore J., Bono J.J. Lipo-chitooligosaccha-ridic symbiotic signals are recognized by LysM receptor-like kinase LYR3 in the legume Medicago truncatula. ACS Chemical Biology, 2013, 8(9): 1900-1906 (doi: 10.1021/cb400369u).
- Bozsoki Z., Gysel K., Hansen S.B., Lironi D., Krönauer C., Feng F., de Jong N., Vinther M., Kamble M., Thygesen M.B., Engholm E., Kofoed C., Fort S., Sullivan J.T., Ronson C.W., Jensen K.J., Blaise M., Oldroyd G., Stougaard J., Andersen K.R., Radutoiu S. Ligand-recogniz-ing motifs in plant LysM receptors are major determinants of specificity. Science, 2020, 369(6504): 663-670 (doi: 10.1126/science.abb3377).
- Bozsoki Z., Cheng J., Feng F., Gysel K., Vinther M., Andersen K.R., Oldroyd G., Blaise M., Radutoiu S., Stougaard J. Receptor-mediated chitin perception in legume roots is functionally separable from Nod factor perception. Proceedings of the National Academy of Sciences, 2017, 114(38): E8118-E8127 (doi: 10.1073/pnas.1706795114).
- Leppyanen I.V, Shakhnazarova V.Y., Shtark O.Y., Vishnevskaya N.A., Tikhonovich I.A., Dol-gikh E.A. Receptor-like kinase LYK9 in Pisum sativum L. is the CERK1-like receptor that con-trols both plant immunity and AM symbiosis development. International Journal of Molecular Sciences, 2017, 19(1): 8 (doi: 10.3390/ijms19010008).
- Bisseling T., Geurts R. Specificity in legume nodule symbiosis. Science, 2020, 369(6504): 620-621 (doi: 10.1126/science.abd3857).
- He J., Zhang C., Dai H., Liu H., Zhang X., Yang J., Chen X., Zhu Y., Wang D., Qi X., Li W., Wang Z., An G., Yu N., He Z., Wang Y.-F., Xiao Y., Zhang P., Wang E. A LysM receptor heteromer mediates perception of arbuscular mycorrhizal symbiotic signal in rice. Molecular Plant, 2019, 12(12): 1561-1576 (doi: 10.1016/j.molp.2019.10.015).
- Den Camp R.O., Streng A., De Mita S., Cao Q., Polone E., Liu W., Ammiraju J.S.S., Kudrna D., Wing R., Untergasser A., Bisseling T., Geurts R. LysM-type mycorrhizal receptor recruited for rhizobium symbiosis in nonlegume Parasponia. Science, 2011, 331(6019): 909-912 (doi: 10.1126/science.1198181).
- Buendia L., Wang T., Girardin A., Lefebvre B. The LysM receptor-like kinase SlLYK10 regulates the arbuscular mycorrhizal symbiosis in tomato. New Phytologist, 2016, 210(1): 184-195 (doi: 10.1111/nph.13753).
- Liao D., Sun X., Wang N., Song F., Liang Y. Tomato LysM receptor-like kinase SlLYK12 is involved in arbuscular mycorrhizal symbiosis. Frontiers in Plant Science, 2018, 9: 1004 (doi: 10.3389/fpls.2018.01004).
- Girardin A., Wang T., Ding Y., Keller J., Buendia L., Gaston M., Ribeyre C., Gasciolli V., Auriac M.C., Vernié T., Bendahmane A., Ried M.K., Parniske M., Morel P., Vandenbussche M., Schorderet M., Reinhardt D., Delaux P.M., Bono J.J., Lefebvre B. LCO receptors involved in arbuscular mycorrhiza are functional for rhizobia perception in legumes. Current Biology, 2019, 29(24): 4249-4259.e5 (doi: 10.1016/j.cub.2019.11.038).
- Staehelin C., Granado J., Müller J., Wiemken A., Mellor R.B., Felix G., Regenass M., Brough-ton W.J., Boller T. Perception of Rhizobium nodulation factors by tomato cells and inactivation by root chitinases. Proceedings of the National Academy of Sciences, 1994, 91(6): 2196-2200 (doi: 10.1073/pnas.91.6.2196).
- Kirienko A.N., Porozov Y.B., Malkov N. V., Akhtemova G.A., Le Signor C., Thompson R., Saffray C., Dalmais M., Bendahmane A., Tikhonovich I.A., Dolgikh E.A. Role of a receptor-like kinase K1 in pea Rhizobium symbiosis development. Planta, 2018, 248(5): 1101-1120 (doi: 10.1007/s00425-018-2944-4).
- Madsen E.B., Madsen L.H., Radutoiu S., Olbryt M., Rakwalska M., Szczyglowski K., Sato S., Kaneko T., Tabata S., Sandal N., Stougaard J. A receptor kinase gene of the LysM type is involved in legume perception of rhizobial signals. Nature, 2003, 425(6958): 637-640 (doi: 10.1038/na-ture02045).
- Murashige T., Skoog F. A revised medium for rapid growth and bio-assays with tobacco tissue cultures. Physiologia Plantarum, 1962, 15(3): 473-497 (doi: 10.1111/j.1399-3054.1962.tb08052.x).
- Rival P., de Billy F., Bono J.J., Gough C., Rosenberg C., Bensmihen S. Epidermal and cortical roles of NFP and DMI3 in coordinating early steps of nodulation in Medicago truncatula. Devel-opment, 2012, 139(18): 3383-3391 (doi: 10.1242/dev.081620).
- Ligero F., Lluch C., Olivares J. Evolution of ethylene from roots of Medicago sativa plants inoc-ulated with Rhizobium meliloti. Journal of Plant Physiology, 1986, 125(3-4): 361-365 (doi: 10.1016/S0176-1617(86)80158-4).
- Chiu C.H., Paszkowski U. Receptor-like kinases sustain symbiotic scrutiny. Plant Physiology, 2020, 182(4): 1597-1612 (doi: 10.1104/PP.19.01341).


