Построение сетей взаимодействий белковых продуктов генов-кандидатов, ассоциированных с левосторонним смещением сычуга у голштинских коров
Автор: Племяшов К.В., Кузнецова Т.Ш., Крутикова А.А., Семенов Б.С., Беликова А.О., Хусаинова Г.С.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика, геномика, протеомика
Статья в выпуске: 2 т.60, 2025 года.
Бесплатный доступ
Левостороннее смещение сычуга (LDA) - одно из заболеваний молочных коров, приводящее к значительным экономическим убыткам. К его клиническим признакам относятся угнетенное состояние животного, снижение аппетита, атония сычуга и скопление в нем газов, снижение продуктивности. Генетические и метаболические механизмы, обусловливающие эти проявления, в том числе с учетом популяционных различий, остаются недостаточно изученными. В представленной работе мы выполнили анализ популяционной стратификации методом кластеризации геномов животных изучаемых групп, провели полногеномный поиск ассоциаций с использованием регрессионной модели (при этом обнаружили новый, не описанный ранее ассоциированный с LDA полиморфизм) и построили сети белковых взаимодействий продуктов генов-кандидатов, ассоциированных с LDA. Цель работы состояла в многостороннем анализе генетических и физиологических аспектов левостороннего смещения сычуга. Исследование проведено на коровах голштинской породы (средний удой 12274 кг за 305 сут лактации) из трех хозяйств Ленинградской области в 2022-2023 годах. При анализе структуры популяции коров методом ADMIXTURE мы не выявили значительных различий в представленности кластеров в геномах животных опытной и контрольной групп. Отсутствие выраженной стратификации доказывает, что определенные предковые популяции не играют значительной роли в формировании предрасположенности к левостороннему смещению сычуга. С помощь полногеномного поиска ассоциаций при использовании метода SAIGE был обнаружен новый ассоциированный с LDA полиморфизм, расположенный в интроне гена FYN . По результатам функционального аннотирования генов-кандидатов c помощью базы данных STRING (https://string-db.org/) построена сеть белковых взаимодействий их продуктов. Полученная сеть взаимодействий включает как кластеры белков, так и отдельные белки. Мы выявили три кластера, состоящие из заданных нами белков и белков, дополненных базой данных STRING. Первый кластер связан с нарушением функции печени (белки PRKCB, SRP72, ABCB11, SOX4), второй - с метаболизмом кальция (белок GSG1L), третий с воспалительными реакциями (белки IFI44 и FBXL19). Также в сеть вошли не охарактеризованные на сегодняшний день белки, которые напрямую или опосредованно могут быть вовлечены в патогенез левостороннего смещения сычуга у высокопродуктивных коров. В целом с левосторонним смещением сычуга у коров ассоциируется большое число генов, участвующих в жизненно важных метаболических путях. Согласно данным литературы и результатам проведенных нами исследований, в разных популяциях крупного рогатого скота голштинской породы такие гены значительно различаются. Продукты этих генов универсальны и вовлечены в клеточные процессы, связанные с пролиферацией, дифференциацией, миграцией, межклеточной сигнализацией, апоптозом и многими другими функциями клетки. Кроме того, такие белковые продукты могут влиять на экспрессию других генов и некоторые из них при определенных физиологических состояниях клетки играют значительную роль в онкогенезе. Возможно, разнообразие описанных генов связано не только со смещением сычуга, но и с иными сопутствующими заболеваниями, которые нередко диагностируются у коров с левосторонним смещением сычуга. Таким образом, левостороннее смещение сычуга, вероятно, следует определить, как сложное полигенное заболевание, и полученная нами информация дополняет общую базу данных по вариантам генов при этой патологии высокопродуктивных молочных коров и расширяет представления о ее генной архитектуре.
Коровы, голштинская порода, левостороннее смещение сычуга, гены-кандидаты, сети белковых взаимодействий
Короткий адрес: https://sciup.org/142245110
IDR: 142245110 | УДК: 636.2:575.113:616.33-007.41 | DOI: 10.15389/agrobiology.2025.2.287rus
Текст научной статьи Построение сетей взаимодействий белковых продуктов генов-кандидатов, ассоциированных с левосторонним смещением сычуга у голштинских коров
Смещение сычуга — одно из заболеваний крупного рогатого скота, которое может приводить к экономическим убыткам. Смещение может происходить влево, вправо, а также встречается перекрут сычуга. При анализе данных литературных, а также при изучении распространенности заболевания в отечественных популяциях молочного скота левостороннее смещение сычуга (left displaced abomasum, LDA) встречается значительно
∗ Работа выполнена при финансовой поддержке РНФ, грант ¹ 22-16-00143,
чаще по сравнению с двумя другими диспозициями сычуга (1-5).
При проведении обобщенных исследований на голштинской породе наследуемость смещения сычуга варьировала в пределах 0,03-0,53 (6). До 87,4 % коров с LDA имеют сопутствующие заболевания — язву сычуга, бронхопневмонию, кетоз, мастит, метртит/эндометрит, печеночный липи-доз, наличие паразитов, заболевания конечностей, фасциолез и расширение слепой кишки (3). Сообщалось о корреляциях между смещением сычуга и нарушением пищеварения ( r = 0,58) и между смещением сычуга и энтеритом ( r = 0,47) у коров голштинской породы (11).
В первом исследовании по выявлению локусов количественных признаков (QTLs) для LDA у молочного скота (12) был обнаружен полно-геномно значимый QTL на аутосомах Bos taurus BTA1 и BTA3, а также три значимых QTLs соответственно на хромосомах BTA21, BTA23 и BTA24. Были определены гены-кандидаты — INSRR (рецептор, связанный с инсулиновым рецептором) и DRD3 (рецептор дофамина). Те же авторы при проведении скрининга QTL у коров голштинской породы немецкой селекции с диагнозом LDA определили в качестве кандидата ген мотилина MLN на BTA23. Однонуклеотидный полиморфизм (FN298674:g.90T>C), расположенный в первом некодирующем экзоне MLN , влияет на сайт связывания фактора транскрипции NKX2-5, что, по мнению авторов (S. M o m-ke с соавт. (13), может влиять на сократительную функцию сычуга. Также они выявили пониженную на 89 % экспрессию гена мотилина у гомозиготных и гетерозиготных носителей мутации по сравнению с диким типом (13).
У коров как с левосторонним, так и с правосторонним смещением сычуга обнаружено повышение концентрации гормонов перистальтики желудочно-кишечного тракта в сыворотке крови (14). Увеличение концентраций грелина, мотилина, гастрина и снижение уровня лептина у больных животных, вероятно, было связано с необходимостью активации перистальтики отделов пищеварительной системы, в том числе сычуга (14). Отечественные ученые проводили генотипирование быков-производителей голштинской породы (World Wide Sires) по локусу MTL и не обнаружили достоверной связи между индексом риска возникновения смещения сычуга у дочерей и MTL генотипом отца (15).
Авторы, проводившие первые исследования по поиску QTLs, ассоциированных с левосторонним смещением сычуга у коров, получили впоследствии новые данные (16). Из трех однонуклеотидных полиморфизмов (SNPs), ассоциированных с LDA на BTA11, были предложены гены кандидаты IL1RN и AK8 . Анализируя функции этих генов, S. M o mke с соавт. (16) предположили, что метаболизм кальция и инсулина может быть важным патогенетическим фактором LDA у коров и это указывает на вероятную роль генных сетей в патогенезе LDA.
В другом исследовании на голштинских коровах немецкой селекции идентифицировали 6 геномных областей на хромосомах BTA2, BTA8, BTA13, BTA20, BTA24 и BTX, значимо ассоциированных с левосторонним смещением сычуга (17). Эти авторы определили связанные с LDA гены-кандидаты SLC4A3 , EPHA4 , LOC100847275 , HAND2 , LOC781304 , FRMD4A , CDH18 , PIGN , KIAA1468 и LOC100847796 (17).
По результатам анализа голштинских коров китайской селекции наследуемость смещения сычуга составила h2 = 0,108. Восемь QTLs, охватывающих 129 генов в BTA2, BTA4, BTA7, BTA10, BTA14, BTA17 и BTA20, были ассоциированы со смещением сычуга. В качестве кандидатов были названы 10 генов: BMP4 , SOCS4 , GCH1 , DDHD1 , ATG14 , ACBP / DBI , SMO , AHCYL2 , CYP7A1 и CACNA1A (18).
Таким образом, доступные научные публикации по генетическим аспектам смещения сычуга у коров голштинской породы содержат информацию о разных генах-кандидатах в различных популяциях, что свидетельствует о сложном полигенном наследовании этой патологии и необходимости продолжить поиск генов-кандидатов, ассоциированных с LDA у коров молочного направления продуктивности отечественной селекции.
В нашем предыдущем исследовании (19) с помощью полногеномного поиска ассоциаций (genome wide association study, GWAS) и анализа распределения паттернов гомозиготности (runs of homozygosity, ROH) у высокопродуктивных голштинских коров в Ленинградской области мы обнаружили новые суггестивные SNPs, ассоциированные с предрасположенностью к LDA в исследуемой популяции скота и ранее не описанные в связи с указанной патологией. Генетические полиморфизмы выявлены на BTA2, BTA3, BTA6, BTA9, BTA16, BTA23, BTA24, BTA25, BTA27 и BTX. При структурной аннотация и картировании были установлены гены-кандидаты, которые или включали один из выявленных SNPs, или находились вблизи от них. После выполнения функциональной аннотации среди генов-кандидатов были выбраны наиболее перспективные: ABCB11 , SRP72 , PRKCB , RGS18 , GSG1L , FBXL19 , IFI44 и SOX4 . Эти гены-кандидаты кодируют белки, участвующие в широком спектре физиологических процессов. Определение роли этих и других генов в патогенезе левостороннего смещения сычуга в популяциях молочных коров может расширить существующие представления о причинах LDA и помочь в разработке новых методов и препаратов для лечения и профилактики этой патологии.
Наследственные причины возникновения LDA у коров могут быть обусловлены в том числе отдельными генетическими компонентами, которые были внесены в формирующуюся популяцию при селекционной работе. Следовательно, анализ популяционной структуры исследуемого поголовья может дать возможность оценить вклад той или иной предковой группы в генетическую архитектуру предрасположенности высокопродуктивных коров голштинской породы к LDA. Определение подобных компонент имеет практическое значение при отборе и подборе животных для минимизации распространения LDA в поголовье.
В настоящей работе нами выполнен анализ популяционной стратификации методом кластеризации геномов животных изучаемых групп, проведен полногеномный поиск ассоциаций с использованием регрессионной модели и построены сети белковых взаимодействий продуктов генов-кандидатов, ассоциированных с LDA. При этом обнаружен новый, не описанный ранее ассоциированный с LDA полиморфизм.
Цель работы состояла в многостороннем анализе генетических и физиологических аспектов левостороннего смещения сычуга у высокопро- дуктивных коров.
Методика . Сбор биологического материала для исследования проводили в трех хозяйствах Ленинградской области (2022-2023 годы). Образцы крови отбирали у разновозрастной группы высокопродуктивных коров голштинской породы I, II и III лактации, включающей 360 животных co средним удоем 12274 кг за 305 сут лактации, из которых 175 животных относились к контрольной группе и 185 — к опытной группе, включающей особей с диагностированным при клиническом осмотре левосторонним смещением сычуга.
ДНК из проб выделяли по классическому методу фенол-хлороформной экстракции с использованием протеиназы К. Генотипирование проводили с ДНК-чипом GeneSeek® Genomic Profiler™ Bovine 100K («Ne-ogen», США) с плотностью покрытия 95256 SNP. Контроль качества, построение файлов адаптированного расширения (.ped, .bed, .map, .fam) и фильтрацию данных генотипирования осуществляли с помощью программного пакета PLINK 1.90 (20). При фильтрации данных биочипов использовали следующие пороги значений: минимальная частота аллелей < 0,05, порог p-value для распределения Харди-Вайнберга < 1e-6, доля пропущенных вариантов на образец < 0,05, доля пропущенных вариантов на один тип маркера < 0,05.
Для валидации полученных ранее результатов, демонстрирующих генетические ассоциации с LDA у голштинский коров российской селекции (19), и сравнения достоинств существующих подходов полногеномного поиска ассоциаций (GWAS) мы подвергли данные генотипирования анализу с помощью подхода SAIGE (Scalable and Accurate Implementation of GEneralized mixed model), реализованного в пакете SAIGE (v. 1.1.9) (21) на языке программирования R.
При подготовке к осуществлению GWAS для учета скрытого родства между исследованными животными с помощью соответствующей функции пакета SAIGE была построена разреженная матрица генетического родства (GRM). На первом этапе анализа подобрали нулевую логистическую смешанную модель, адекватную матрице родства, данным фенотипа животных и ковариантам (коварианты — возраст животных в месяцах при взятии образца крови и число суток после отела). Затем был выполнен ассоциативный тест с приложением функции SPA (Saddle Point Approximation). Результаты GWAS визуализировали в пакете QQman (v. 0.1.9) в среде Rstudio (v. 2024.09.1+394). При структурной аннотации выявленных ассоциаций пользовались сборкой генома крупного рогатого скота ARS-UCD1.3 с визуализацией при помощи браузера GDV (Genome Data Viewer).
Популяционную структуру выборки анализировали в программе Admixture 1.3 (22) по 82712 маркерам, прошедшим фильтрацию после разрешения неравновесного сцепления. Для определения наиболее вероятного числа предковых групп (K) осуществляли процедуру кросс-валидации (вычисляли CV error) для K от 1 до 7. Полученные данные визуализировали с помощью пакета pophelper (v. 2.9.6) (23) в среде программирования Rstudio (v. 2024.09.1+394).
Биоинформатический анализ взаимодействия белковых продуктов генов, содержащих обнаруженные полиморфизмы, и построение сетей выполняли с использованием базы данных STRING (Search Tool for Retrieval of Interacting Gines/Proteins) и программного обеспечения Cytoscape 3.7.2.
Результаты . При оценке структуры анализируемой популяции голштинских коров (рис. 1) показатель ошибки кросс-валидации (CV) 290
находился на нижней отметке при числе кластеров, равном пяти, что указывает на вклад пяти популяций в формирование генофонда исследованной группы коров голштинской породы в трех хозяйствах Ленинградской области и относительное генетическое разнообразие выборки. При анализе структуры популяции коров мы не выявили значительных различий в представленности кластеров в геномах животных из опытной и контрольной групп. Отсутствие выраженной стратификации доказывает, что опре-дел¸нные предковые популяции не играют значительной роли в формировании предрасположенности к левостороннему смещению сычуга.
Кроме того, различия демонстрируют распределения геномных кла- стеров у животных опытной группы, принадлежащих к каждому хозяйству (см. рис. 1), что также не дает оснований связать представленность опре-
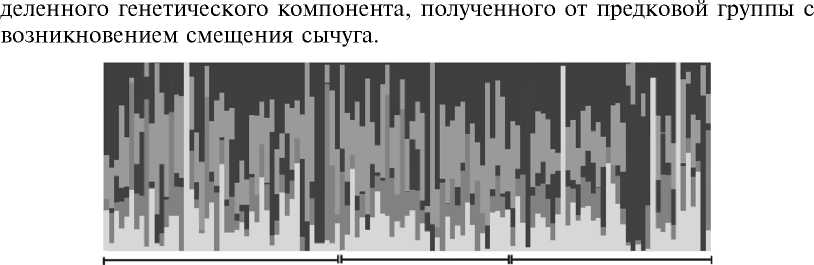
Рис. 1. Генетическая кластеризация высокопродуктивных голштинских коров с левосторонним смещением сычуга из трех хозяйств для числа предковых групп K = 5 (программа Admixture 1.3, n = 120, Ленинградская обл., 2022-2023 годы). Разными цветами обозначены геномные компоненты предковых групп. Горизонтальные отрезки разграничивают животных по хозяйствам.
Хромосома
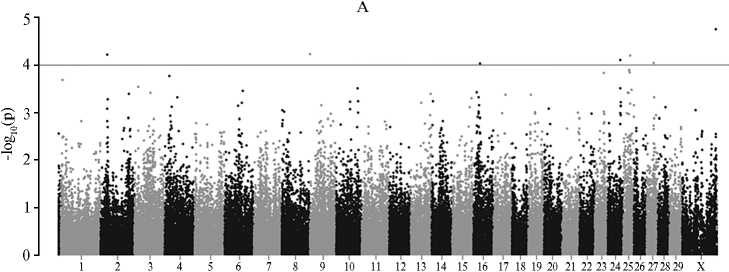
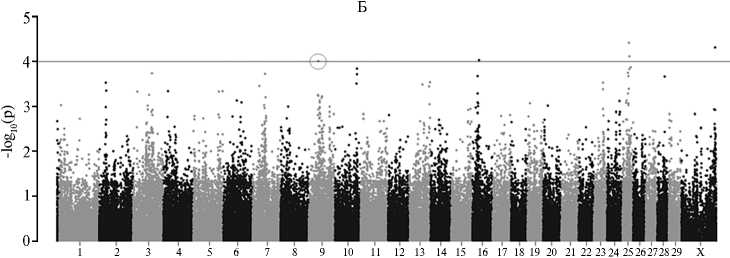
Рис. 2. Распределение точечных мутаций по хромосомам у высокопродуктивных голштинских коров исследуемой выборки по предрасположенности к LDA согласно уровню статистической значимости (манхэттенские графики): А — результаты, полученные ранее с применением mixed linear model (MLM) (19); Б — результаты определения полногеномных ассоциаций с использованием модели Scalable and accurate implementation of generalized mixed model (SAIGE), полученные в настоящем исследовании (n = 360, Ленинградская обл., 2024 год). При SAIGE- анализе обнаружен полиморфизм, для которого в исследованной популяции ранее не была выявлена ассоциация с LDA. Горизонтальная линия — порог достоверности для суггестивных ассоциаций -log10(p) = 1½10-4.
SAIGE представляет собой двухступенчатую реализацию смешанной логистической регрессионной модели для учета родства исследуемых объектов и несбалансированности соотношения случай—контроль в выборке. SAIGE в отличие от смешанных линейных моделей использует функцию правдоподобия SPA, которая продемонстрировала эффективность при работе с бинарными признаками (23). При осуществлении GWAS по левостороннему смещению сычуга у высокопродуктивных голштинских коров из Ленинградской области методом SAIGE было обнаружено 5 однонуклеотидных полиморфизмов, преодолевающих порог суггестивной статистической достоверности, из них 4 (расположенные на BTA16, BTA25 и BTX) были отмечены как статистически значимые для той же патологии в нашем предыдущем исследовании (19). Результаты текущего и предшествующего анализов приведены на рисунке 2. Вновь обнаруженный полиморфизм, расположенный в позиции 9:38708085, находится в интроне гена FYN , белковый продукт которого участвует в регуляции клеточного роста и иммунном ответе (24).
Ранее сообщалось, что применение различных методов GWAS может приводить к выявлению разных наборов полиморфизмов, связанных с одним и тем же признаком (25). Обнаружение в настоящем исследовании нового полиморфизма, не представленного ранее, также указывает на важность сравнения геномных данных, полученных с помощью разных аналитических подходов, для более глубокого понимания патогенеза заболеваний с наследственной компонентой.
При построении сети белковых взаимодействий важно учитывать, какие функции выполняет тот или иной белок, в каких метаболических путях он участвует и какие процессы в клетке регулирует. База данных STRING (26) объединяет все известные и предполагаемые взаимосвязи между белками, включая как физические, так и функциональные взаимодействия, и позволяет вводить пользовательские геномы, на основании которых строит сеть белковых взаимодействий. Существенным преимуществом представляется возможность формировать кластеры белков построенной сети, визуализируя их взаимосвязи. БД STRING (27) включает дополнительные белки, образующие сеть с заданными пользователем белками, на основании консолидированной информации согласно современным представлениям о взаимодействии протеинов у Bos taurus . Такая презентация данных расширяет возможности анализа патогенеза LDA у продуктивных животных.
На основании выявленных нами генов-кандидатов ABCB11 , SRP72 , PRKCB , RGS18 , GSG1L , FBXL19 , IFI44 и SOX4 , ассоциированных у высокопродуктивных коров с LDA, используя базу данных STRING в дополнение к предыдущим исследованиям (19), мы построили сеть белковых взаимодействий продуктов этих генов. На рисунке 3 представлены три основных кластера белковых взаимодействий (отмечены разными цветами), а также белки, не вошедшие в кластеры.
Проведенный анализ кластеров (см. рис. 3) показал следующее. В состав 1-го кластера входит PRKCB (протеинкиназа C-бета 1) — член семейства протеинкиназ С, участвует в путях передачи сигнала и регуляции апоптоза. Однонуклеотидные полиморфизмы (SNPs) в PRKCB ассоциируются с риском воспалительных заболеваний кишечника (28) и возникновением жирового гепатоза печени (29), влияют на пути секреции инсу- лина у пациентов с ожирением (30). У коров с левосторонним смещением сычуга, как правило, наблюдаются повреждения печени и пониженный уровень инсулина (31). Можно предположить, что изменения в гене PRKCB опосредованно влияют на возникновение LDA у коров.
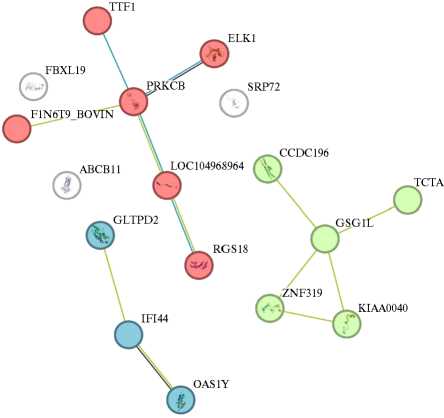
Рис. 3. Сеть белковых взаимодействий генов-кандидатов, ассоциированных с левосторонним смещением сычуга у высокопродуктивных голштинских коров исследуемой выборки, с учетом формируемых кластеров: 1-й кластер — красный цвет, 2-й кластер — зеленый цвет, 3-й кластер — синий цвет (описание кластеров см. в разделе «Результаты») ( n = 360, Ленинградская обл., 20222023 годы).
Также в 1-й кластер входит белок RGS18 из семейства RGS (регуляторы сиг-налинга G-белков). RGS18, подобно всем представителям семейства RGS, ослабляет сигнальную активность G-белков, гидролизуя GTP до GDP в субъ- единице Gα, и модулирует у G-белков функцию нисходящей сигнальной регуляции в разных физиологических условиях (32). Таким образом, продукт гена RGS18 как участник многих процессов также вовлечен в патогенез LAD.
В состав 2-го кластера входит белковый продукт гена GSG1L . Это трансмембраный белок, который представляет собой вспомогательную субъединицу рецептора AMPA. Рецепторы глутамата AMPA-типа служат лиганд-зависимыми кальциевыми каналами, ответственными за большую часть быстрой возбуждающей синаптической передачи сигналов в мозге. Их функционирование и проницаемость для кальция зависят от состава и структуры как субъединиц, так и вспомогательных белков (33). Рецептор AMPA вместе со вспомогательными белками образует сложную молекулярную структуру, проявляющую функциональное разнообразие, и GSG1L может быть важным компонентом нейронных путей (34). У коров левостороннее смещение сопровождается нарушением минерального обмена (гипокальциемия и кетоз) (1), и мутация в гене GSG1L , возможно, способна влиять на метаболизм Са2+ и патогенез заболевания.
Не вошедшие в кластеры белки SRP72, ABCB11 и SOX4 тоже могут быть ассоциированы с поражением печени у коров с LDA. Например, тран-скриптомный подход с использованием прецизионных срезов печени человека, наряду с другими генами, выявил SRP72 как маркерный ген стресса эндоплазматического ретикулума при лекарственно индуцированной гепатотоксичности, приводящей к холестазу или иной патологии печени (36). Кроме того, липидоз печени, который часто встречается у коров с LDA, может сопровождаться симптомами печеночного холестаза. У человека развитие внутрипеченочного холестаза возможно у пациентов с любыми заболеваниями печени, независимо от этиологии (37).
Белок bile salt export pump (BSEP, ABCB11) представляет собой насос-экспортер желчных солей, который по большей части локализован в мембранах желчных канальцев и специфичен в отношении желчных кислот. Мутации в этом белке связывают с прогрессирующим семейным внут-рипеченочным холестазом человека, проявляющимся тяжелыми холестатическими симптомами с раннего детства (38).
Ген SOX4 кодирует одноименный белок, входящий в семейство SOX, является фактором транскрипции, который регулирует стволовость, дифференциацию, образование предшественников и множественные пути развития клеток (39). Показано, что SOX4 индуцируется при гепатобилиарной метаплазии, то есть перепрограммировании гепатоцитов после повреждения печени, и таким образом участвует в координации программ активации и репрессии генов (40).
Ген FBXL19 кодирует белок, принадлежащий к семейству F-box. FBXL19 служит компонентом комплекса лигазы SCF FBXL19 E3, которая регулирует воспалительные реакции, пролиферацию клеток, миграцию, реорганизацию цитоскелета и убиквитин-зависимый катаболизм белка (41).
БД STRING дополнила сеть протеиновых взаимодействий следующими прогнозируемыми функциональными партнерами белковой природы: CCDC196 — предполагаемый домен-содержащий белок со спиральной структурой 196, FAM136A F1N6T9_BOVIN — не охарактеризованный белок, KIAAA0040 ( KIAAA0040 ), MLNR — рецептор мотилина (принадлежит к семейству рецепторов, связанных с G-белком 1), ELK1 — ETS транскрипционный фактор ELK1, ZNF319 — белок цинкового пальца 319, LOC104968964 — субъединица гамма-гуаниннуклеотидсвязывающего белка, гуаниннуклеотидсвязывающие белки (G-белки), NTSR1 — рецептор нейротензина 1 (принадлежит к семейству рецепторов, связанных с F-белком 1), TTF1 — фактор терминации транскрипции. Вместе с белками, представленными на рисунке 3, эти предполагаемые функциональные белки-партнеры, в том числе не охарактеризованные в настоящее время, могут участвовать в патогенезе левостороннего смещения сычуга. Вероятно, они напрямую или опосредованно вовлечены во многие метаболические пути и каскадные реакции, которые на сегодняшний день не определены.
При анализе структуры популяции коров мы не обнаружили значительных различий в представленности кластеров в геномах животных опытной и контрольной групп. Таким образом, отсутствие выраженной стратификации свидетельствует о том, что предковые популяции не играли значительной роли в формировании предрасположенности к левостороннему смещению сычуга.
В результате полногеномного поиска ассоциаций был обнаружен новый полиморфизм, находящийся в интроне гена FYN , принимающего участие в иммунном ответе.
Таким образом, генетическая архитектура левостороннего смещения сычуга у коров голштинской породы носит сложный полигенный характер, ее конструкция формируется за счет вклада множества предковых групп, и гены, составляющие ее, вовлечены во множество метаболических процессов. На основании построенной сети белковых взаимодействий при 294
левостороннем смещении сычуга у высокопродуктивных голштинских коров белковые продукты как минимум четырех генов — PRKCB , SRP72 , ABCB11 и SOX4 вовлечены в процессы, связанные с нарушением функции печени и опосредованно влияют на патогенез заболевания. Белок GSG1L может быть связан с изменением метаболизма кальция, а белки IFI44 и FBXL19 участвуют в воспалительных реакциях. Все эти процессы характерны для коров с левосторонним смещением сычуга. Можно сделать вывод, что с этой патологией у коров ассоциируется большое число различных генов, участвующих в жизненно важных метаболических путях. Не исключено, что разнообразие генов, описанных при указанной патологии, связано не только со смещением сычуга, но и с иными сопутствующими заболеваниями, которые нередко диагностируются у коров с левосторонним смещением сычуга. Полученные нами сведения дополняют общую геномную базу данных по вариантам генов, ассоциированных с левосторонним смещением сычуга у высокопродуктивных молочных коров, расширяя представления о сложности генной архитектуры, которая оказывается затронутой при этом заболевания. В дальнейшей работе мы обратим внимание на прогнозируемые функциональные белки-партнеры, дополненные БД STRING, которые могут оказаться важными участниками метаболических путей и каскадных реакций при левостороннем смещении сычуга у коров.


