ПОВЫШЕНИЕ ДОСТОВЕРНОСТИ МЕТОДА ПЛАВЛЕНИЯ ДНК ПУТЕМ ПРОВЕДЕНИЯ ПОВТОРНЫХ АНАЛИЗОВ
Автор: Д. А. Белов, Ю. В. Белов, А. Н. Зубик, В. Е. Курочкин
Журнал: Научное приборостроение @nauchnoe-priborostroenie
Рубрика: Физика и химия приборостроения
Статья в выпуске: 4 т.32, 2022 года.
Бесплатный доступ
В статье изучена возможность использования повторных анализов методом плавления ДНК для повышения их достоверности, а именно уменьшения погрешности определения температуры плавления ДНК Tm. Экспериментально выявлено статистически значимое различие значений Tm анализируемых образцов в последовательно проведенных анализах, препятствующее повышению достоверности. Показано, что увеличение температуры плавления Tm в проведенной серии экспериментов в среднем на 0.12 град. обусловлено испарением 1.65% воды из пробирки. Экспериментально выявлено изменение массы на 0.8 ± 0.1% от общей массы воды в пробирках в результате теплового режима, аналогичного анализу методом плавления, что частично подтверждает предположение. Рекомендовано при повторных анализах методом плавления ДНК учитывать этот эффект и реализовывать мероприятия, препятствующие испарению воды. Приведены аналитические выражения для относительной оценки изменения концентрации ионов натрия и объема воды в пробе.
Метод плавления ДНК, HRMA, испарение
Короткий адрес: https://sciup.org/142235502
IDR: 142235502 | УДК: 543.426; 543.9 | DOI: 10.18358/np-32-4-i1119
Текст статьи ПОВЫШЕНИЕ ДОСТОВЕРНОСТИ МЕТОДА ПЛАВЛЕНИЯ ДНК ПУТЕМ ПРОВЕДЕНИЯ ПОВТОРНЫХ АНАЛИЗОВ
Современный метод плавления ДНК выполняется после полимеразной цепной реакции в реальном времени (ПЦР-РВ) на детектирующих амплификаторах, при этом для генерации флуоресцентного сигнала используются интеркали-рующие красители, молекулы которых присоединяются к двойной цепи ДНК. При повышении температуры двойная цепь ДНК постепенно распадается на две одноцепочечные молекулы, вызывая отсоединение молекул красителя и спад интенсивности флуоресценции [1].
Графики плавления ДНК (ГП) представляют собой зависимости флуоресцентного отклика пробы от температуры и отличаются друг от друга формой и значением температуры плавления T m , что позволяет определять различия во фрагментах ДНК по их составу, длине и статусу метилирования и применять метод во множестве прикладных задач, таких как геномное сканирование, генотипирование, сопоставление последовательностей и эпигенетические исследования [2].
Значение Tm соответствует температуре, при которой 50% нуклеотидов в молекулах ДНК теряют водородные связи [3], и традиционно определяется как координата точки перегиба ГП, что соответствует значению максимума отрицательной производной ГП по температуре [4]. В ряде задач требуется высокая точность определения температуры Tm, например, для достоверного генотипиро- вания штаммов M. tuberculosis необходимо выявлять различия в значениях Tm менее 0.1 град. [5].
С целью уменьшения погрешности измерения температуры T m применяются известные методики, основанные на фильтрации и/или аппроксимации ГП различными непрерывными функциями, например полиномом третьей степени в ограниченном диапазоне изменения температуры, как в программе ANK_Melting [6], или усовершенствованной сигмоидальной функцией (СФ) [7]. В статьях [8–10] рекомендуется после предварительной нормализации выполнять численное дифференцирование графиков плавления с помощью полиномов Савицкого – Голея второй степени в каждой точке [11].
Метод плавления ДНК является неразрушающим, поэтому одним из дополнительных способов повышения достоверности результатов анализа может быть повторение экспериментов для накопления статистики. В настоящей статье рассматривается возможность применения такого подхода.
ПОЛУЧЕНИЕ И ОБРАБОТКА ИСХОДНЫХ ДАННЫХ
Для получения исходных данных выполнен трехкратный анализ методом плавления ДНК трех известных образцов ампликонов цитокератина (CK-19) на анализаторе нуклеиновых кислот АНК-32, который серийно выпускается в Институте аналитического приборостроения РАН (Россия). Ампликоны были получены в результате 50 циклов ПЦР-РВ с интеркалирующим красителем SYBR Green I ("ПЦР-Микс" М-427, ООО "Синтол", Россия) и специально разработанными праймерами (ООО "ДНК-Синтез", Россия). Образцы 1, 2 и 3 в четырех пробирках каждый анализирова- лись в диапазоне температур 70–95 °C с шагом 0.5 град. и длительностью выдержки на каждом шаге 30 с. Образцы 1, 2 и 3 имеют соответственно содержание нуклеотидов гуанина и цитозина во фрагменте ДНК [G + C], равное 53, 59 и 58% при длине фрагментов Lp, равной 103, 209 и 309 пар нуклеотидов (п. н.). Начальная концентрация ионов натрия во всех пробах равна 0.125 моль/л.
Табл. 1. Значения температур плавления анализируемых образцов (°C)
|
№ эксперимента |
№ образца |
||
|
1 |
2 |
3 |
|
|
1 |
84.37 ± 0.04 |
90.55 ± 0.10 |
90.72 ± 0.08 |
|
2 |
84.53 ± 0.05 |
90.68 ± 0.09 |
90.85 ± 0.06 |
|
3 |
84.65 ± 0.09 |
90.83 ± 0.12 |
90.98 ± 0.06 |
Примечание . Уровень значимости 0.05, объем выборки 4
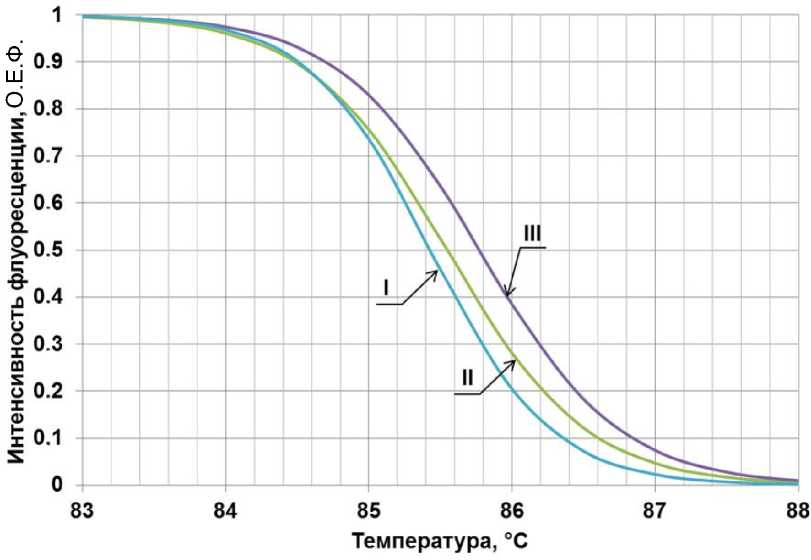
Рис. Графики плавления образца 1 по результатам первого (I), второго (II) и третьего (III) анализов
Значения температур плавления образцов были определены путем применения методики на основе СФ [7]. В табл. 1 приведены средние значения температур плавления трех образцов в трех последовательных анализах.
На рисунке изображены графики плавления от одной пробирки с образцом 1, полученные в результате трех последовательных анализов после нормализации модельной функцией СФ. Графики позволяют наглядно сравнить различия значений T m , соответствующих температуре, при которой интенсивность флуоресценции равна 0.5. Видно, что при каждом следующем анализе значение Tm увеличивается.
На основе вычисленных значений парного t-критерия Стьюдента выявлено статически значимое различие между результатами последовательных экспериментов с уровнем значимости р < 0.05. Эти различия не позволяют использовать повторные анализы методом плавления ДНК для увеличения их достоверности. Актуальным является определение причин изменения значений T m образцов при повторных анализах.
ПРИЧИНЫ ИЗМЕНЕНИЯ ТЕМПЕРАТУР ПЛАВЛЕНИЯ ОБРАЗЦОВ ПРИ ПОВТОРНЫХ АНАЛИЗАХ
В соответствии с работой [12], значение T m определяется по формуле:
T m = 81.5 + 16.6 • lg[Na + ] +
+ 0.41 • [ G + C ] - 675 / L p , (1)
где [Na+] — концентрация ионов натрия, моль/л.
Слагаемое 16.6∙lg [Na+] включено и в другие известные модели [13, 14].
Изменение значений T m может быть обусловлено испарением воды из анализируемой пробы. Так как значения [ G + C ] и L p из формулы (1) остаются постоянными, изменение значений Tm сопровождается увеличением концентрации ионов натрия [Na+].
Сравнение существующих моделей для вычисления температуры плавления ДНК показало значительное расхождение результатов применения моделей с экспериментальными данными [15]. В статьях [16, 17] предложено применять модели, учитывающие зависимость температуры Tm только от факторов, величина которых изменяется в серии экспериментов. Авторы представили формулу (1) в виде упрощенной модели:
T m = T - "Л + 0.41 • [ G + C ]. (2) Lp
Здесь значение параметра Т 1 равно сумме 81.5 + 16.6∙lg([Na+]) .
В табл. 2 приведены вычисленные параметры T 1 и различия параметров T 1 , определенных при повторных анализах. В столбце d103 находятся: значение 0.08 — это разность параметров T 1 при втором и первом анализах образца 1, значение 0.11 этой разницы при третьем и втором анализах и значение 0.19 — при третьем и первом анализах. Аналогично определены изменения параметров T 1 в столбцах d209 и d309 для образцов 2 и 3 (d209 и d309 соответственно).
Изменения параметров T 1 для образцов 1, 2 и 3 после проведения трех анализов направлены в сторону увеличения Т 1 : 0.19; 0.27 и 0.26 град. соответственно. Среднее изменение температуры T m между последовательными анализами составило 0.12 град.
Начальная концентрация натрия во всех пробах равна 0.125 моль/л, что соответствует: 16.6∙lg[0.125] = T 1 – 81.5. Увеличение концентрации натрия на 2% вызывает увеличение параметра T m на 0.14 град. Следовательно, испарение воды в пробирке и увеличение концентрации натрия составляют примерно 2%.
Для проверки этого предположения проанализировано изменение массы заполненных дистиллированной водой тонкостенных пробирок на 0.2 мл (SSI, США) до и после нагрева. 32 пробирки (4 стрипа по 8 шт.) заполняли дистиллированной водой по 25 мкл, закрывали крышки и выдерживали в эксикаторе для удаления мелких капель воды с внешней поверхности пробирок. Нагрев проводили в АНК-32 по программе плавления от 70 °С до 95 °С с шагом 0.5 град. и продолжительностью 30 с на каждой температуре. Взвешивание до и после нагрева, а также до и после заполнения водой
Табл. 2. Различия параметров T 1 при проведении повторных анализов (°С)
|
№ анализа |
103 |
d103 |
209 |
d209 |
309 |
d309 |
|
1 |
69.09 |
0.08 |
69.83 |
0.12 |
69.15 |
0.13 |
|
2 |
69.20 |
0.11 |
69.95 |
0.16 |
69.28 |
0.13 |
|
3 |
69.28 |
0.19 |
70.11 |
0.27 |
69.41 |
0.26 |
(для определения суммарной массы воды) проводили на аналитических весах высшего I специального класса точности (ЗАО "САРТОГОСМ", Россия), дискретность измерений которых составляет d = 0.1 мг, предельно допустимая погрешность e = = 1 мг.
Эксперимент проводили на трех сериях образцов. Итоговое изменение массы 32 пробирок после нагрева составило 6.0 ± 0.5 мг (n = 3), что соответствовало 0.8 ± 0.1% от общей массы воды в пробирках.
Чтобы удостоверится, что полученный результат действительно связан с испарением воды через закрытую крышку пробирки, дополнительную серию из 32 пробирок заполнили водой с минеральным маслом (25 мкл воды и 15 мкл масла). Изменение массы таких пробирок после нагрева было сопоставимо с погрешностью весов и составило 0.5 мкл (0.06% от расчетной массы воды).
ОЦЕНКА ИЗМЕНЕНИЯ КОНЦЕНТРАЦИИ ИОНОВ НАТРИЯ
В соответствии с формулой (1) при неизменных параметрах [ G + C ] и L p изменение температуры плавления образца определяется следующим образом:
A T m = 16.6 - lg( A [Na + ]), (3)
где A [Na+ ] — изменение концентрации ионов натрия в пробе.
Отсюда:
A T m
A [Na + ] = (10 16.6 - 1) - 100%. (4)
Долю испарившейся воды A VH2O оценить по следующей формуле:
V
A V H2o = (1-- H^ ) - 100%.
2 m
10 16.6
можно
С помощью формул (4) и (5) можно более точно оценить значения A [Na + ] и A V HO , соответствующие среднему изменению температуры T m на 0.12 град. в проведенной серии экспериментов: A [Na + ] = 1.68%, A V HzO = 1.65%.
ЗАКЛЮЧЕНИЕ
Использование повторных анализов методом плавления ДНК для увеличения их достоверности, а именно уменьшения погрешности температуры плавления ДНК Tm, затруднено вследствие изме- нения значений Tm с каждым повтором. Для успешного использования описанного подхода необходимо предотвратить процесс испарения воды из пробы, однако полностью исключить испарение является сложной задачей [18].
В проведенной серии экспериментов выявлено среднее увеличение значений T m на 0.12 град., что объясняется увеличением концентрации ионов натрия на 1.68% и уменьшением объема воды на 1.65%. Изменение массы 32 пробирок с водой в результате теплового режима, аналогичного анализу методом плавления, составило 0.8 ± 0.1% от общей массы воды в пробирках, что частично подтверждает предположение.
Выявление различий температур плавления образцов может использоваться для оценки количества испарившейся воды в емкостях, где оценка другими способами, как путем измерения массы пробы, затруднена или невозможна. Примером таких емкостей являются микрофлюидные чипы.
Работа выполнена в ИАП РАН в рамках государственного задания № 075-00761-22-00 Министерства науки и высшего образования РФ.
Список литературы ПОВЫШЕНИЕ ДОСТОВЕРНОСТИ МЕТОДА ПЛАВЛЕНИЯ ДНК ПУТЕМ ПРОВЕДЕНИЯ ПОВТОРНЫХ АНАЛИЗОВ
- 1. Ririe K.M., Rasmussen R.P., Wittwer C.T. Product differentiation by analysis of DNA melting curves during the
- polymerase chain reaction // Anal Biochem. 1997. Vol. 245, is. 2. P. 154–160. DOI: 10.1006/abio.1996.9916
- 2. Montgomery J.L., Sanford L.N., Wittwer C.T. Highresolution DNA melting analysis in clinical research and diagnostics // Expert Rev Mol Diagn. 2010. Vol. 10, is. 2. P. 219–240. DOI: 10.1586/erm.09.84
- 3. Wright T.A., Stewart J.M., Page R.C., Konkolewicz D. Extraction of thermodynamic parameters of protein unfolding using parallelized differential scanning fluorimetry // J Phys Chem Lett. 2017. Vol. 8, is. 3. P. 553–558. DOI: 10.1021/acs.jpclett.6b02894
- 4. Веденов А.А., Дыхне А.М., Франк-Каменецкий М.Д. Переход спираль – клубок в ДНК // Успехи физических наук. 1971. Т. 105, № 11. С. 479–519. DOI: 10.3367/UFNr.0105.197111d.0479
- 5. Pholwat S., Liu J., Stroup S. et al. Integrated microfluidic card with TaqMan probes and high-resolution melt analysis to detect tuberculosis drug resistance mutations across 10 genes // mBio. 2015. Vol. 6, is. 2. e02273. DOI: 10.1128/mBio.02273-14
- 6. Альдекеева А.С., Белов Д.А., Белов Ю.В., Широкорад А.Л. Разработка экспериментальной версии программного обеспечения на основе новой методики определения температуры плавления ДНК // Научное приборостроение. 2019. Т. 29, № 2. С. 22–29. DOI: 10.18358/np-29-2-i2229
- 7. Белов Д.А., Корнева Н.А., Альдекеева А.C., Белов Ю.В., Киселев И.Г. Повышение разрешающей способности генетических анализаторов при определении температуры плавления ДНК // Научное приборостроение. 2016. Т. 26, № 2. С. 17–22. URL: http://213.170.69.26/mag/2016/abst2.php#abst2
- 8. Wittwer C.T., Reed G.H., Gundry C.N., Vandersteen J.G., Pryor R.J. High-resolution genotyping by amplicon melting analysis using LCGreen // Clinical Chemistry. 2003. Vol. 49, is. 6. P. 853–860. DOI: 10.1373/49.6.853
- 9. Gundry C.N., Vandersteen J.G., Reed G.H., et al. Amplicon melting analysis with labeled primers: A closed-tube
- method for differentiating homozygotes and heterozygotes // Clinical Chemistry. 2003. Vol. 49, is. 3. P. 396–406. DOI: 10.1373/49.3.396
- 10. Herrmann M., Durtschi J., Bromley L., Wittwer C., Voelkerding K. Amplicon DNA melting analysis for mutation scanning and genotyping: Cross-platform comparison of instruments and dyes // Clinical chemistry. 2006. Vol. 52, is. 3. P. 494–503. DOI: 10.1373/clinchem.2005.063438
- 11. Press W.H., Teukolsky S.A., Vetterling W.T., Flannery B.P. Numerical recipes in C. 2nd ed., 1992. Cambridge University Press, New York. 994 p.
- 12. Календарь Р.Н., Сиволап Ю.М. Полимеразная цепная реакция с произвольными праймерами // Biopolymers and cell. 1995. Т. 11, № 3-4. С. 55–65. URL: https://www.elibrary.ru/item.asp?id=23336409
- 13. Howley P.M., Israel M.F., Law M.-F., Martin M.A. A rapid method for detecting and mapping homology between heterologous DNAs. Evaluation of polyomavirus genomes // J. Biol. Chem. 1979. Vol. 254, is. 11. P. 4876–4883. DOI: 10.1016/S0021-9258(17)30093-5
- 14. SantaLucia J., Allawi H.T., Seneviratne P.A. Improved nearest-neighbor parameters for predicting DNA duplex stability // Biochemistry. 1996. Vol. 35, is. 11. P. 3555–3562. DOI: 10.1021/bi951907q
- 15. Panjkovich A., Melo F. Comparison of different melting temperature calculation methods for short DNA sequences // Bioinformatics. 2005. Vol. 21, is. 6. P. 711–722. DOI: 10.1093/bioinformatics/bti066
- 16. Курочкин В.Е., Белов Д.А., Белов Ю.В., Зубик А.Н. Определение модельных констант при вычислении температуры плавления ДНК // Научное приборостроение. 2020. Т. 30, № 2. С. 10–16. DOI: 10.18358/np-30-2-i1016
- 17. Belov D.A., Belov Yu.V., Kiselev I.G. Modeling of the DNA melting point dependence on various analysis factors // IEEE 2020 International Multi-Conference on Industrial Engineering and Modern Technologies (FarEastCon). 2020. P. 1–3. DOI: 10.1109/FarEastCon50210.2020.9271634
- 18. Polini A., Mele E., Sciancalepore A.G., et al. Reduction of water evaporation in polymerase chain reaction microfluidic devices based on oscillating-flow // Biomicrofluidics. 2010. Vol. 4, is. 3. Id. 036502. DOI: 10.1063/1.3481776


