Применение бактерий со сверхэкспрессией гена CADA для биоконверсии лизина в кадаверин
Автор: Ахова А.В., Федоненко П.М., Шумков М.С., Ткаченко А.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 1, 2024 года.
Бесплатный доступ
Сконструирован штамм Escherichia coli со сверхэкспрессией гена cadA, кодирующего лизиндекарбоксилазу, и проведена оценка его способности конвертировать лизин в кадаверин в зависимости от рН среды. Клетки, сверхэкспрессирующие лизиндекарбоксилазу, получены на основе штамма E. coli BL21DE3, трансформированного плазмидой pET19b, несущей ген cadA из E. coli MC4100. Бактерии культивировали в 5 мл бульона LB при 37ºС без перемешивания, сверхэкспрессию cadA запускали добавкой 1 мМ изопропил-β-D-тиогалактопиранозида (при ОП600=0.6), через 2 ч. клетки отмывали и переносили в среду с рН 7.4 или 4.0 с добавкой 5 г/л L-лизина гидрохлорида. Количество синтезированного кадаверина определяли методом ТСХ с предварительной дериватизацией дансилхлоридом. Максимальная скорость биоконверсии наблюдалась при культивировании клеток, сверхэкспрессирующих cadA в среде с рН 4.0 (2.8±0.7 ммоль кадаверина/г АСБ в час). Скорость биоконверсии в базовых условиях (рН 7.4, базовая экспрессия) была в 6.8 раз ниже. Конечная концентрация кадаверина при культивировании в среде с рН 7.4 и/или в условиях отсутствия сверхэкспрессии гена cadA составила 0.8-1 мМ, в условиях сверхэкспрессии гена в нейтральной среде накапливалось до 1.2 мМ кадаверина, а в кислой среде - 1.8 мМ.
Кадаверин, лизин, декарбоксилаза, сверхэкспрессия, цельноклеточные биокатализаторы
Короткий адрес: https://sciup.org/147243409
IDR: 147243409 | УДК: 579.22 | DOI: 10.17072/1994-9952-2024-1-54-60
Текст научной статьи Применение бактерий со сверхэкспрессией гена CADA для биоконверсии лизина в кадаверин
Для клонирования и экспрессии рекомбинантных генов в клетках бактерий разработаны специальные генно-инженерные системы. В общем случае, рекомбинантный ген вводится в бактериальную клетку в составе векторной молекулы, которая – с целью повышения копийности – сохраняется в цитоплазме в состоянии эписомы. Генетически модифицированные подобным образом микроорганизмы обычно используют для наработки рекомбинантных белков, но также возможно их применение в качестве цельноклеточных биокатализаторов.
В частности, бактериальные клетки, способные к сверхэкспрессии лизиндекарбоксилазы за счет генетической модификации путем введения векторных молекул, пытаются использовать для биосинтеза кадаверина (1.5-диаминопентана). Особый интерес биосинтез кадаверина представляет потому, что на основе данного диамина могут быть получены пластики с улучшенными свойствами, а эффективный метод химического синтеза этого соединения не разработан [Kind et al., 2014; Huang et al., 2021]. В настоящее время существует два основных направления использования генетически модифицированных микроорганизмов для биосинтеза 1.5-диаминопентана: они либо выступают в качестве цельноклеточных биокатализаторов одностадийной реакции конверсии лизина в кадаверин, либо применяются для синтеза целевого вещества из простых субстратов в процессе ферментации [Ma et al., 2017; Nærdal, 2017].
Одним из первых был получен биокатализатор для конверсии лизина в кадаверин на основе клеток Escherichia coli , сверхэкспрессирующих ген cadA [Nishi et al., 2006], кодирующий индуцибельную изоформу лизиндекарбоксилазы в клетках E. coli , проявляющую максимальную активность при рН 5–6. В дальнейшем на основе клеток, сверхэкспрессирующих cadA , были разработаны биокатализаторы с дополнительными модификациями, направленными на повышение выхода продукта [Kim et al., 2015; Ma et al., 2015]. Также реализованы разработки на основе сверхэкспрессии гена ldcC , кодирующего конститутивную лизиндекарбоксилазу E. coli , максимально активную в среде с нейтральным рН [Oh et al., 2015; Shin et al., 2018].
Основным источником генов лизиндекарбоксилаз для создания биокатализаторов служит E. coli , хотя спектр используемых микроорганизмов постепенно расширяется. Например, созданы генно-модифицированные штаммы E. coli , сверхэкспрессирующие лизиндекарбоксилазы из клеток Klebsiella pneumonia , Ral-stonia eutropha , Pseudomonas aeruginosa и Hafnia alvei [Oh et al., 2015; Kim et al., 2016; Kim et al., 2019].
С целью создания биокатализаторов для синтеза кадаверина из лизина, как правило, применяют генно-инженерные конструкции, обеспечивающие сверхэкспрессию лизиндекарбоксилазы в ответ на добавку индуктора, в частности, сиcтемы на основе плазмид рET и рКЕ [Kim et al., 2015; Oh et al., 2015]. Альтернативным подходом является помещение гена, кодирующего лизиндекарбоксилазу, под контроль сильного конститутивного промотора, что обеспечивает его постоянную сверхэкспрессию [Qian, Xia, Lee, 2011; Ting et al., 2021].
Цель работы – конструирование штамма E. coli , сверхэкспрессирующего ген cadA , и оценка его способности конвертировать L-лизин в кадаверин в зависимости от рН среды.
Материалы и методы
Объекты и условия культивирования
В качестве объекта исследования использован штамм E. coli BL21DE3, трансформированный сконструированной нами плазмидой pET19b_cadA.
Бактериальные клетки, сохраняемые на скошенном агаре LB, переносили в 5 мл бульона LB (рН 7.4) с добавкой 1% глюкозы и культивировали при 37ºС без перемешивания в течение 16–18 ч. В среду для культивирования клеток, несущих плазмиду pET19b_cadA, добавляли 100 мкг/мл ампициллина. Полученную культуру разводили до плотности ОП 600 = 0.1–0.15 в 20 мл свежего бульона LB (рН 7.4) с добавкой (при необходимости) 100 мкг/мл ампициллина и культивировали при 37ºС без перемешивания. По достижении культурой плотности ОП 600 = 0.6 для индукции сверхэкспрессии гена cadA добавляли 1 мМ изопропил-β-D-тиогалактопиранозида (ИПТГ), после чего продолжали культивирование в описанных выше условиях в течение 2 ч. Затем бактериальную культуру центрифугировали (16 000 g, 3 мин.), и бактериальные клетки переносили в бульон LB с рН 7.4 или рН 4.0, содержащий 5 г/л L-лизина гидрохлорида (общий объем культуры составлял 5 мл). Культивирование проводили в описанных выше условиях.
Определение оптической плотности культуры и концентрации абсолютно сухой биомассы
Оптическую плотность измеряли при 600 нм (ОП 600 ) в кювете с длиной оптического пути равной 1 см с использованием спектрофотометра UV-1280 (Shimadzu, Япония). Концентрацию абсолютно сухой биомассы (АСБ) в культуре рассчитывали, используя предварительно полученный калибровочный график зависимости данного показателя и оптической плотности культуры (ОП 600 ).
Конструирование штамма E. coli со сверхэкспресссией гена cadA
Нуклеотидную последовательность гена cadA амплифицировали с применением праймеров cadANdeI 5’-GT CatATG TGTTGGGAGGGGCCTTTTTTAC-3’ и cadABamH 5’- GGATCC GGCAAGCCACTTCCCTTG-3’ с использованием в качестве матрицы хромосомы E. coli MC4100 (сайты рестрикции подчеркнуты; различия с нуклеотидной последовательностью E. coli W3110, GenBank AP009048.1 показаны строчными буквами). Полученный ПЦР-продукт длиной 2 226 пар нуклеотидов клонировали в вектор pGEM-T easy (Promega, США) согласно рекомендациям производителя. pGEM-T easy-вектор со вставкой секвенирова-ли с использованием стандартных праймеров М13F/R, плазмиду c подтверждённой последовательностью гена cadA выделяли из клеток методом щелочного лизиса [Maniatis, Fritsch, Sambrook, 1982] и обрабатывали эндонуклеазами рестрикции NdeI и BamHI (Fermentas, Литва). Полученный ген cadA очищали элек-трофоретически, экстрагировали из агарозного геля с применением набора DNA Extraction Kit (Fermentas, Литва) и лигировали в вектор pET19b (Novagen, США), предварительно также обработанный эндонуклеазами NdeI и BamHI. Полученную конструкцию pET19b_cadA проверяли с помощью ПЦР и секвенирования с использованием стандартных праймеров T7Pro/Term, после чего трансформировали в штамм E. coli BL21DE3 (Invitrogen) для экспрессии рекомбинантных белков.
Количественный анализ кадаверина осуществляли методом тонкослойной хроматографии с предварительной дериватизацией дансилхлоридом [Akhova, Tkachenko, 2022].
Статистический анализ результатов проводили с использованием пакета программ Statistica 6.0 (StatSoft Inc, США). Числовые данные в тексте представлены в виде среднее ± стандартное отклонение, на рисунке столбцы соответствуют среднему значению, отрезки (разбросы) – стандартной ошибке среднего.
Результаты и их обсуждение
В данном исследовании проведена оценка способности бактерий E. coli , характеризующихся сверхэкспрессией гена cadA , кодирующего лизиндекарбоксилазу, осуществлять биконверсию лизина в кадаверин. Для создания продуцента структурная часть гена cadA была слита с Т7 lac -промотором в составе плазмидного вектора pET19b (рис. 1). Полученную плазмиду pET19b_cadA трансформировали в штамм BL21DE3, представляющий собой производное E. coli BL21, в хромосому которого введен фаг λ DE3, несущий ген РНК-полимеразы Т7 под контролем laсUV промотора. Экспрессия последнего запускается добавкой изопропил-β-D-1-тиогалактопиранозида (ИПТГ), что приводит к наработке РНК-полимеразы фага Т7, обеспечивающей считывание целевого гена в составе эписомального вектора. Обязательными условиями сверхэкспрессии гена являлись присутствие плазмиды pET19b_cadA в клетках и добавка ИПТГ в среду культивирования. Источником гена cadA послужил штамм МС4100.
Затем было проведено сравнение способности продуцировать кадаверин клетками с базовым уровнем экспрессии лизиндекарбоксилазы, и клетками, сверхэкспрессирующими ген cadA , а также исследовано влияние рН среды на процесс биоконверсии (рис. 2).
Максимальной скоростью биоконверсии лизина в кадаверин характеризовались клетки, сверхэкс-прессирующие ген cadA, при культивировании на среде с рН 4.0. На второй час скорость биоконверсии в данных условиях составила 2.8±0.7 ммоль кадаверина/г АСБ в час, после чего наблюдалось снижение скорости процесса. В базовых условиях (рН среды 7.4, штамм без плазмиды) скорость биоконверсии равнялась 0.41±0.19 ммоль/г АСБ×ч, т.е. была почти в 7 раз меньше. Сверхэкспрессия гена cadA обеспе- чивала более высокую продукцию кадаверина и при культивировании в нейтральной среде, но скорость продукции была ниже по сравнению с клетками, выращиваемыми при рН 4.0.
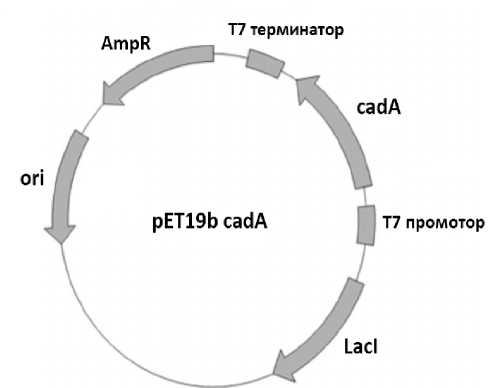
Рис. 1. Схема плазмиды pET19b_cadA.
AmpR – ген, кодирующий β-лактамазу (резистентность к ампициллину); LacI – ген, кодирующий репрессор lac -оперона; ori – район начала репликации вектора
[Diagram of the pET19b_cadA plasmid.
AmpR is a gene encoding β-lactamase (ampicillin resistance); LacI is a gene encoding a repressor of the lac operon; ori is the origin of replication]
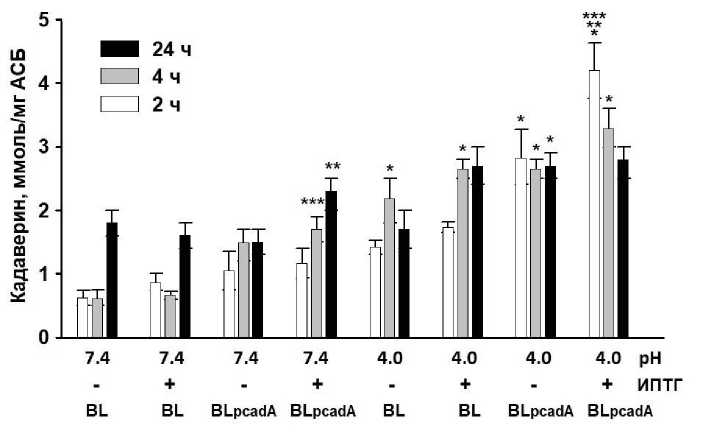
Рис. 2. Влияние рН среды и сверхэкспрессии гена cadA на продукцию кадаверина клетками E. coli BL21DE3.
«BL» – клетки, не несущие плазмиду pET19b_cadA, «BLpcadA» – клетки, несущие плазмиду pET19b_cadA. «+» – добавка 1 мМ ИПТГ (индуктора сверхэкспрессии), «–» – без добавки ИПТГ.
* – статистически значимое отличие от соответствующей культуры, выращиваемой на среде с рН 7.4;
** – статистически значимое отличие от соответствующей культуры без добавки ИПТГ;
*** – статистически значимое отличие от культуры штамма, не несущего плазмиду, при культивировании в аналогичных условиях (t-тест, р ≤0.05, n≥3)
[The effect of the pH of the medium and overexpression of the cadA gene on the production of cadaverine by E. coli BL21 DE3 cells.
“BL” – cells that do not carry the pET19b_cadA plasmid, “BLpcadA” – cells that carry the pET19b_cadA plasmid. “+” – the addition of 1 mM of IPTG (inducer of overexpression), “–” – without the addition of IPTG.
-
* – statistically significant difference from the corresponding culture grown on a medium with a pH of 7.4;
-
* * – statistically significant difference from the corresponding culture without the addition of IPTG;
-
* ** – statistically significant difference from the culture of a strain not carrying a plasmid when cultivated under similar conditions (t-test, p≤0.05, n≥3)]
Смещение рН среды в кислую сторону приводило к ускорению образования кадаверина как в условиях сверхэкспрессии, так и при базовой экспрессии гена cadA (в отсутствие плазмиды, без добавки ИПТГ). Увеличение лизиндекарбоксилазной активности в данном случае можно рассматривать как защитный ответ клетки на кислотный стресс, вызванный снижением рН среды [Meng, Bennett, 1992; Kanjee, Houry, 2013]. Кроме того, можно предположить, что повышение концентрации ионов водорода в среде сопровождалось смещением внутриклеточного рН в область, близкую к оптимальным значениям для лизиндекарбоксилазы CadA, что способствовало повышению скорости катализируемой ею реакции.
Можно также отметить тенденцию к увеличению продукции кадаверина клетками, несущими плазмиду сверхэкспрессии, при выращивании в среде с рН 4.0 без добавки индуктора ИПТГ, однако различия между трансформированными и не трансформированными плазмидой клетками не были статистически значимыми.
Установлено, что при всех вариантах условий культивирования конечная концентрация кадаверина в среде не превышала 2 мМ. При рН среды 7.4 и/или в условиях отсутствия сверхэкспрессии гена cadA итоговая концентрация кадаверина была в интервале 0.8–1 мМ. Сверхэкспрессия гена в нейтральной среде приводила к накоплению 1.2 мМ кадаверина, в кислой среде – к накоплению почти 1.8 мМ. Можно заключить, что данная концентрация является предельной для исследованного организма и лизиндекарбоксилазы CadA, выделенной из клеток E. coli MC4100. Прекращение процесса биоконверсии лизина в кадаверин, несмотря на достаточное количество субстрата в среде, может быть связано с ингибированием фермента по принципу обратной связи. Накопление кадаверина в среде до высоких концентраций оказывает в целом неблагоприятное воздействие на бактериальные клетки. Ранее показано, что добавка в среду культивирования 0.3–0.5 М гидрохлорида кадаверина вызывала частичный лизис клеток в культуре E. coli К12 [Qian, Xia, Lee, 2011]. Однако в процессе декарбоксилирования лизина с образованием кадаверина затрачивается ион водорода, а сам кадаверин, выделяющийся в среду, проявляет свойства основания. Это приводит к защелачиванию среды и смещению рН в область, неоптимальную для проявления активности фермента.
Таким образом, для повышения выхода целевого продукта, по-видимому, необходимо осуществлять культивирование штамма-продуцента в условиях поддержания оптимального значения рН и обеспечивать удаление конечного продукта реакции биоконверсии. Кроме того, решить данную проблему позволит поиск природных лизиндекарбоксилаз, толерантных к высокой концентрации конечного продукта, или получение таких ферментов методами белковой инженерии.
Заключение
Сконструирован штамм E. coli , сверхэкспрессирующий ген cadA , выделенный из E. coli MC4100. Скорость биоконверсии лизина в кадаверин в условиях сверхэкспрессии была в 6.8 раза выше, чем в базовых условиях. Скорость процесса биоконверсии и конечная концентрация кадаверина были выше при постановке реакции при рН 4.0 по сравнению со средой с нейтральным рН. Дальнейшая работа будет направлена на повышение продуктивности процесса биоконверсии с применением полученного продуцента, что может быть осуществлено за счет подбора оптимальных условий протекания реакции декарбоксилирования, в частности, варьирования состава среды культивирования, начальной плотности культуры продуцента, стабилизации продуцента за счет иммобилизации.
Список литературы Применение бактерий со сверхэкспрессией гена CADA для биоконверсии лизина в кадаверин
- Akhova A.V., Tkachenko A.G. Cadaverine biosynthesis in Escherichia coli adaptation to hydrogen peroxide // Applied Biochemistry and Microbiology. 2022. Vol. 58, № 5. Р. 582-589. DOI: 10.1134/S0003683822050039.
- Huang Y. et al. Green chemical and biological synthesis of cadaverine: recent development and challenges // RSC Advances. 2021. Vol. 11, № 39. P. 23922-23942. DOI: 10.1039/d1ra02764f.
- Kanjee U., Houry W.A. Mechanisms of acid resistance in Escherichia coli // Annual Review of Microbiology. 2013. Vol. 67. P. 65-81. DOI: 10.1146/annurev-micro-092412-155708.
- Kim H.J. et al. Optimization of direct lysine decarboxylase biotransformation for cadaverine production with whole-cell biocatalysts at high lysine concentration // Journal of Microbiology and Biotechnology. 2015. Vol. 25, № 7. P. 1108-1113. DOI: 10.4014/jmb.1412.12052.
- Kim H.T. et al. High-level conversion of l-lysine into cadaverine by Escherichia coli whole cell biocata-lyst expressing Hafnia alvei l-lysine decarboxylase // Polymers (Basel). 2019. Vol. 11, № 7. P. 1184. DOI: 10.4014/jmb.1602.02030.
- Kim J.H. et al. Functional study of lysine decarboxylases from Klebsiella pneumoniae in Escherichia coli and application of whole cell bioconversion for cadaverine production // Journal of Microbiology and Biotechnology. 2016. Vol. 26, № 9. P. 1586-1592. DOI: 10.3390/polym11071184. "
- Kind S. et al. From zero to hero - production of bio-based nylon from renewable resources using engineered Corynebacterium glutamicum // Metabolic Engineering. 2014. Vol. 25. P. 113-123. DOI: 10.1016/j.ymben.2014.05.007.
- Ma W. et al. Enhanced cadaverine production from L-lysine using recombinant Escherichia coli co-overexpressing CadA and CadB // Biotechnology Letters. 2015. Vol. 37, № 4. P. 799-806. DOI: 10.1007/s10529-014-1753-5.
- Ma W. et al. Advances in cadaverine bacterial production and its applications // Engineering. 2017. Vol. 3, № 3. P. 308-317. DOI: 10.1016/J.ENG.2017.03.012.
- Maniatis T., Fritsch E.F., Sambrook J.K. Molecular cloning: a laboratory manual. Cold Spring Harbor Laboratory, Cold Spring Harbor, 1982. 545 p.
- Meng S.Y., Bennett G.N. Nucleotide sequence of the Escherichia coli cad operon: a system for neutralization of low extracellular pH // Journal of Bacteriology. 1992. Vol. 174, № 8. P. 2659-2669. DOI: 10.1128/jb.174.8.2659-2669.1992.
- Nsrdal I. et al. L-lysine production by Bacillus methanolicus: genome-based mutational analysis and l-lysine secretion engineering // Journal of Biotechnology. 2017. Vol. 244, P. 25-33. DOI: 10.1016/j.jbiotec.2017.02.001.
- Nishi et al., 2006. US7189543B2 patent.
- Oh Y.H. et al. Development of engineered Escherichia coli whole-cell biocatalysts for high-level conversion of L-lysine into cadaverine // Journal of Microbiology and Biotechnology. 2015. Vol. 42, № 11. P. 14811491. DOI: 10.1007/s10295-015-1678-6.
- Qian Z.G., Xia X.X., Lee S.Y. Metabolic engineering of Escherichia coli for the production of cadaverine: a five carbon diamine // Biotechnology and Bioengineering. 2011. Vol. 108, № 1. P. 93-103. DOI: 10.1002/bit.22918.
- Shin J. et al. Characterization of a whole-cell biotransformation using a constitutive lysine decarboxylase from Escherichia coli for the high-level production of cadaverine from industrial grade l-lysine // Applied Biochemistry and Biotechnology. 2018. Vol. 185, № 4. P. 909-924. DOI: 10.1007/s12010-018-2696-4.
- Ting W.W. et al. Whole-cell biocatalyst for cadaverine production using stable, constitutive and high expression of lysine decarboxylase in recombinant Escherichia coli W3110 // Enzyme and Microbial Technology. 2021. Vol. 148. P. 109811. DOI: 10.1016/j.enzmictec.2021.109811.


