Применение GLAD-ПЦР-анализа для изучения метилирования ДНК в регуляторных областях генов-онкосупрессоров при раке молочной железы
Автор: Сметанникова Наталья Анатольевна, Абдурашитов Мурат Абдурашитович, Акишев Александр Григорьевич, Поздняков Павел Иванович, Дубинин Евгений Викторович, Карпов Андрей Борисович, Вихлянов Игорь Владиславович, Никитин Максим Константинович, Солдатова Светлана Михайловна, Нетесова Нина Александровна
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 6 т.20, 2021 года.
Бесплатный доступ
Аберрантное метилирование ДНК на начальных стадиях канцерогенеза приводит к инактивации ряда генов и показано для многих онкологических заболеваний. Ранее с помощью метода GLAD-ПЦР анализа нами были выявлены аберрантно метилированные сайты R(5mC)GY, которые могут рассматриваться как эпигенетические маркеры при диагностике колоректального рака, рака легкого и рака желудка. При раке молочной железы, по литературным данным, наблюдается метилирование регуляторных участков генов ALX4, BMP2, CCND2, CDH13, CDX1, FOXA1, GALR1, GATA5, GREM1, HIC1, HMX2, HS3ST2, HOXCIO, ICAM5, LAMA1, RARB, RASSF1A, RUNX3, RXRG, RYR2, SFRP2, SOX17, TERT и ZNF613. В настоящей работе было проведено определение аберрантно метилированных сайтов RCGY в регуляторных участках данных генов в препаратах ДНК из тканей рака молочной железы. Анализ образцов ДНК из операционного материала опухолей (n=30) и морфологически неизмененных тканей молочной железы (n=22) показал высокую диагностическую эффективность панели маркеров, включающей сайты R(5mC)GY в составе генов CCND2, BMP2, GALR1, SOX17, HMX2 и HS3ST2: суммарные показатели чувствительности и специфичности составляют 90,0 и 100,0 % соответственно.
Рак молочной железы, гены-онкосупрессоры, метилирование днк, glad-пцр-анализ
Короткий адрес: https://sciup.org/140261351
IDR: 140261351 | УДК: 618.19-006.6:577.21 | DOI: 10.21294/1814-4861-2021-20-6-41-54
Текст научной статьи Применение GLAD-ПЦР-анализа для изучения метилирования ДНК в регуляторных областях генов-онкосупрессоров при раке молочной железы
В 2020 г. рак молочной железы (РМЖ) стал наиболее распространенным злокачественным новообразованием во всем мире: ~2,3 млн выявленных случаев составили 11,7 % от всех впервые зарегистрированных случаев рака. По числу летальных исходов (более 685 тыс.) РМЖ вышел на пятое место в структуре общей смертности от онкологических заболеваний [1]. Основная причина высокой смертности заключается в том, что в 40–70 % случаев диагностируется РМЖ III–IV стадий, что определяет неблагоприятный прогноз [2]. При раннем выявлении (стадии IA–IIB) РМЖ пятилетняя выживаемость достигает 99 % [3].
Наиболее перспективным направлением ранней диагностики РМЖ считается эпигенетический анализ: определение в ДНК опухолевых клеток метильных групп, присоединенных к цитозиновым основаниям в регуляторных участках генов- онкосупрессоров, уровень экспрессии которых при этом значительно снижается. Данная модификация геномной ДНК обнаруживается уже на начальных стадиях канцерогенеза, что имеет решающее значение для раннего выявления онкологических заболеваний, включая РМЖ [4].
GLAD-ПЦР-анализ включает три стадии: 1) гидролиз ДНК ферментом GlaI (G); 2) присоединение к концам гидролизованной ДНК с помощью ДНК-лигазы (L) специфического адаптера (AD); 3) ПЦР в режиме реального времени (степень метилирования сайта R(5mC)GY определяет значение порогового цикла (Cq)) (рис. 1). На последнем этапе используются геномный праймер и флуоресцентный зонд, комплементарные участку ДНК вблизи определяемого сайта GlaI, и второй (гибридный) праймер с нуклеотидной последовательностью 5’-CCTGCTCTTTCATCGGYNN-3’, где пятнадцать 5’-концевых нуклеотидов соответствуют адаптеру, а четыре 3’-концевых нуклеотида комплементарны геномной последовательности в точке гидролиза ДНК ферментом GlaI [6].
Благодаря субстратной специфичности ДНК-эндонуклеазы GlaI метод GLAD-ПЦР позволяет определять сайт R(5mC)GY в заданном положении генома даже в условиях избытка молекул ДНК, где этот сайт неметилирован. GLAD-ПЦР-анализ характеризуется более высокой чувствительностью и воспроизводимостью результатов в сравнении с методиками, основанными на бисульфитной конверсии ДНК (часто сопровождаемой деградацией и значительными потерями исследуемого материала) либо использующими метил-чувствительные эндонуклеазы рестрикции (не способные распознавать частично метилированную ДНК – наиболее характерный паттерн метилирования CpG-островков на самых ранних стадиях канцерогенеза). К важным преимуществам GLAD-ПЦР относятся также значительно меньшая трудоемкость и дороговизна исследования, отличающие применение специфичных метил-связывающих белков [6,11].
Целью исследования явилось применение GLAD-ПЦР-анализа для определения аберрантно метилированных сайтов R(5mC)GY в регуляторных областях генов-онкосупрессоров в препаратах ДНК из тканей РМЖ.
Материал и методы
Материалом для исследования служили образцы ДНК, выделенные из тканей опухолей молочной железы, полученных в ходе оперативного вмешательства у 30 женщин в возрасте от 52 до 78 лет (средний возраст – 63,1 ± 1,3 года, s=6,7). Операционный материал собран в мае – августе 2017 г. в КГБУЗ «Алтайский краевой онкологический диспансер» (г. Барнаул) и КГБУЗ «Областной клинический онкологический диспансер» (г. Кемерово). У большинства пациенток диагностирован инвазивный протоковый рак (n=22) различной степени дифференцировки. В остальных случаях был выявлен инвазивный дольковый (n=7) или смешанный (n=1) рак. У 9 больных установлена I клиническая стадия РМЖ (T1N0M0), у 16 – II стадия (T0–1N1M0, T2–3N0–1M0), у 5 – III стадия (T0–2N2M0, T3N1–2M0, T4N0–2M0, T0–4N3M0).
В зависимости от иммуногистохимических характеристик 3 опухолевых образца были отнесены к люминальному подтипу А, включающему гормонозависимые опухоли, положительные по рецепторам эстрогенов (РЭ) и прогестерона (РП), не имеющие рецепторов эпидермального фактора роста HER2, с низким индексом пролифератив-
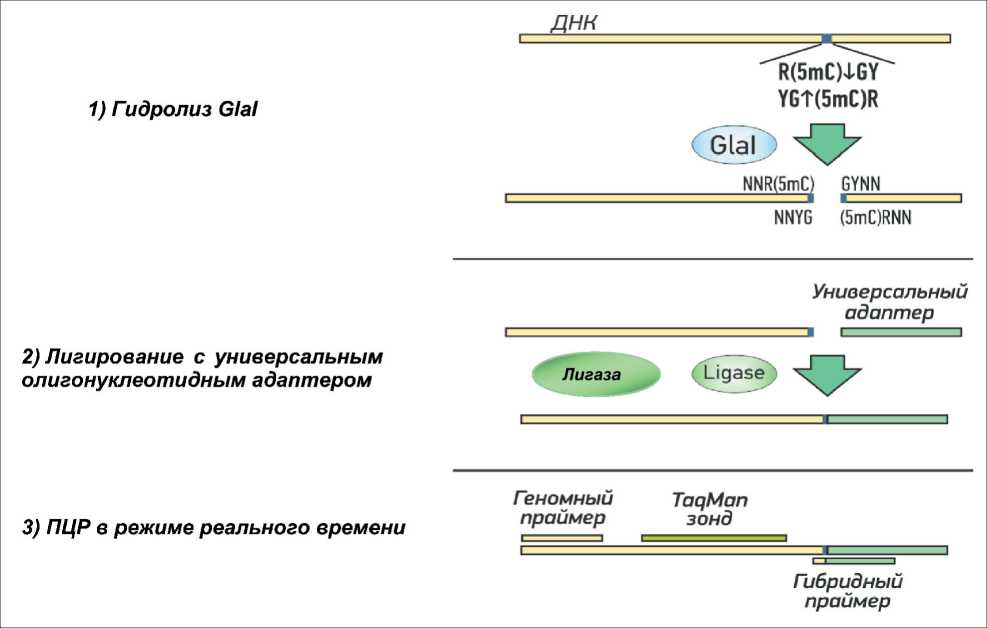
Рис. 1. Схема метода GLAD-ПЦР
Fig. 1. Scheme of the GLAD-PCR method
ной активности (Ki67<20 %). Для 17 образцов определен люминальный подтип B, включающий HER2-негативные опухоли с высоким индексом пролиферативной активности (Ki67≥20 %), РЭ-положительные с низкими или отрицательными значениями РП, либо HER2-позитивные опухоли, РЭ-положительные с любыми значениями Ki67 и РП. Две опухоли были отнесены к HER2-позитивному подтипу (гормононезависимые (РЭ-, РП-) с избытком рецепторов HER2), 8 – к тройному негативному подтипу (РЭ-, РП-, HER2-). В качестве сравнительного контроля были использованы образцы ДНК, выделенные из фрагментов морфологически неизмененных тканей молочной железы, взятых у 22 больных на значительном удалении от опухолевого очага, на линии резекции.
Неоадъювантную химиотерапию больные не получали вследствие малой степени распространенности опухолевого процесса либо наличия сопутствующих заболеваний, расцененных на момент планирования лечения как фактор, способный привести к утяжелению общего состояния. У всех пациенток получено информированное согласие на участие в исследовании.
Каждый тканевой образец помещали в пробирку с раствором RNA-later и хранили в течение суток в холодильнике при температуре +4 °С, затем переносили в морозильную камеру и хранили при температуре -20 °С. Выделение и очистку ДНК проводили методом фенольно-хлороформной экстракции [7].
По результатам анализа опубликованных литературных данных нами был проведен предварительный отбор генов-онкосупрессоров, регуляторные области которых потенциально содержат метилированные сайты RCGY – эпигенетические маркеры РМЖ. При отборе учитывались следующие критерии: присутствие в регуляторной области гена минимум одного сайта RCGY; при наличии нескольких таких сайтов – расстояние между ними не менее 50 п.н., достаточное для одновременной гибридизации на данном участке ДНК геномного праймера и флуоресцентного зонда; возможность подбора для исследуемого участка ДНК праймера и зонда, теоретически не подверженных образованию вторичных структур, ауто- и кросс-димеров, а также гибридизации с последовательностью адаптера; GC-состав исследуемого участка ДНК, не превышающий 76 %; уникальность нуклеотидной последовательности исследуемого участка ДНК (невхождение его в состав повторяющихся элементов генома).
На основании приведенных критериев мы сформировали исходную панель из 24 генов, содержащих потенциально метилированные сайты RCGY: ALX4 [12], BMP2 [13], CCND2 [14], CDH13 [15], CDX1 [16], FOXA1 [17], GALR1 [18], GATA5 [19], GREM1 [14, 20], HIC1 [21], HMX2 [22], HS3ST2 [23], HOXC10 [24], ICAM5 [14], LAMA1
[25], RARB [26], RASSF1A [27], RUNX3 [28], RXRG [29], RYR2 [29], SFRP2 [14, 30], SOX17 [14], TERT [31], ZNF613 [32].
Для исследования потенциальных эпигенетических маркеров РМЖ в составе регуляторных участков генов-онкосупрессоров подбирали специфичные системы праймеров и флуоресцентных зондов (табл. 1). Для этого использовали нуклеотидные последовательности из базы данных GenBank по версии генома человека GRCh38.p13, семейство программ «Vector NTI 11.5» (Invitrogen, США) и онлайн-ресурс «BLAST» .
В ходе предварительных экспериментов в регуляторной области каждого гена выявляли максимально метилированный сайт R(5mC)GY. Для всех сайтов RCGY, расположенных в пределах 200 п.н. от участка гибридизации геномного праймера и флуоресцентного зонда, определяли гибридные праймеры, комплементарные концевым тетрануклеотидам, получаемым после гидролиза последовательности NNR(5mC) ↓ GYNN. Наименьшее значение Cq в ПЦР в режиме реального времени указывало на максимальную степень метилирования сайта R(5mC)GY [6,7].
Для постановки всех этапов GLAD-ПЦР применяли реагенты производства ООО «Сибэнзайм». Ферментативный гидролиз ДНК проводили в течение 30 мин при 30 °С. Реакционная смесь (объемом 21,5 мкл) включала 9,0 нг ДНК исследуемого образца, 1× SE-буфер TMN (10 мМ Трис-HCl (pH 7,9), 5 мМ MgCl 2 , 25 мМ NaCl), 2,0 % диметилсульфоксида (ДМСО), 2,0 мкг бычьего сывороточного альбумина (БСА) и 1,5 ед. акт. эндонуклеазы GlaI. Для лигирования продуктов гидролиза с адаптером (в объеме 30,0 мкл) к каждой пробе добавляли АТР до конечной концентрации 0,5 мМ, универсальный двухцепочечный адаптер (5’-CCTGCTCTTTCATCG-3’/3’-p-GGACGAGAAAGTAGC-p-5’, где p – фосфатная группа) до конечной концентрации 0,5 мкМ и 240 ед. акт. высокоактивной T4-ДНК-лигазы. Реакцию проводили в течение 15 мин при 25 °С. На заключительной стадии в реакционную смесь вносили компоненты ПЦР до следующих концентраций в конечном объеме 60,0 мкл: 1×SE-буфер GLAD (50 мМ Трис-SO 4 (pH 9,0), 30 мМ KCl, 10 мМ [NH 4 ] 2 SO 4 ), 3 мМ MgCl 2 , смесь dNTP до 0.2 мМ каждого, 0,1 мкг/мкл БСА, соответствующую смесь двух праймеров и зонда до 0,4 мкМ каждого, 0,04 ед. акт./мкл SP Taq-ДНК-полимеразы. Для повышения эффективности амплификации GC-богатых участков генов GATA5 , GREM1 , HIC1 , ICAM5 и RUNX3 в ПЦР-смесь дополнительно добавляли ДМСО до концентрации 4 %. Затем по 20,0 мкл полученной смеси переносили в три отдельные микропробирки и проводили ПЦР в режиме реального времени в детектирующем амплификаторе «CXF-96» (Bio-Rad Lab., США) по программе:
Таблица 1/table 1
Геномные праймеры и флуоресцентные зонды, применявшиеся для исследования генов, потенциально содержащих онкомаркеры РМЖ
genomic primers and fluorescent probes used to study genes potentially containing breast cancer tumor markers
|
Генa/ Genea |
Наименование белкового продуктаa/ Protein product namea |
Хромосомная локализация генаa/ Chromosomal gene localizationaa |
Структура геномных праймеров/ флуоресцентных зондовb/ Structure of genomic primers/fluorescent probesb |
|
ALX4 |
ALX homeobox 4 |
11p11.2 |
CGTCAACAACCTCTCATCC FAM-TCCATTTCTTATTTCAGTTTGCCACCA-BHQ1 |
|
BMP2 |
bone morphogenetic protein 2 |
20p12.3 |
CGAGTTGCGGCTGCTCAGC FAM-CCTGAAACAGAGACCCACCCCCAGC-BHQ1 |
|
CCND2 |
cyclin D2 |
12p13.32 |
GAAAGGCAACCCCCCCCAA FAM-CGTCTTGGCCTCAGGTCCCCGC-BHQ1 |
|
CDH13 |
cadherin 13 |
16q23.3 |
GCTGGCTGGCGAGGCAGA FAM-CCTCTCCTCAAAGCCTGGCTCCCAC-BHQ1 |
|
CDX1 |
caudal type homeobox 1 |
5q32 |
CAGCTTCGCTGGCATTCGG FAM-CCCCCTCCAGACTTTAGCCCGGTGC-BHQ1 |
|
FOXA1 |
forkhead box protein A1 |
14q21.1 |
FAM-TCTCCTTCCATCTTGGCCTCGGCTC-BHQ1 AGAGGCTGGGAGCTGGACGA |
|
GALR1 |
galanin receptor 1 |
18q23 |
FAM-TGCAGCAGAGAAGCCCCTGGCACC-BHQ1 GGCGAGAGCTCTTTTGGGAGGC |
|
GATA5 |
GATA binding protein 5 |
20q13.33 |
FAM-CCGAGCCCGCAGCCCCCC-BHQ1 CCCCTCGATGCTGTCCTACCTGT |
|
GREM1 |
gremlin 1, DAN family BMP antagonist |
15q11-13 |
FAM-CTCCGCCCACTCACATCCCTGCC-BHQ1 GAGCGGGTCCTGGGTTGGTT |
|
HIC1 |
hypermethylated in cancer 1 |
17p13.3 |
FAM-CTTGTAGTCGTCGCCGTCGCCGC-BHQ1 GGCTGGGGTCCTCGCTGCTA |
|
HMX2 |
H6 family homeobox 2 |
10q26.13 |
CCAAGGGGTCCAAGGAGTCGG FAM-CCGCAGCCGCCAGGAACCCG-BHQ1 |
|
HS3ST2 |
heparan sulfate-glucosamine 3-O-sulfotransferase 2 |
16p12.2 |
GCCTCCCGGAGGAGTACTATGCC FAM-CACCTTCGTTTCACCGCCCCAAAGC-BHQ1 |
|
HOXC10 |
homeobox protein C10 |
12q13.13 |
GAGGGGGGCAGGGGAGAG FAM-CCCAGGAGGCCCCAGACCATTTC-BHQ1 |
|
ICAM5 |
intercellular adhesion molecule 5 |
19p13.2 |
FAM-CTCCCTCCCAGGCCCCGCCC-BHQ1 AGGGGGAAGAGTCCGCAGCTC |
|
LAMA1 |
laminin, alpha 1 |
18p11.31 |
FAM-CTGACCGCGGCCGCCTCCC-BHQ1 CCACCTTCTCTGCCCACCTCCTA |
|
RARB |
retinoic acid receptor, beta |
3p24 |
TTCAGAGGCAGGAGGGTCTATTC FAM-TCCCAGTCCTCAAACAGCTCGCATGG-BHQ1 |
|
RASSF1A |
Ras association domain family member 1 |
3p21.31 |
ATGTCGGGGGAGCCTGAG FAM-TGCCAGCTCCCGCAGCTCAAT-BHQ1 |
|
RUNX3 |
runt related transcription factor 3 |
1p36.11 |
CCGGGGAGGGAGGTGTGAA FAM-CCGTAGACCCAAGCACCAGCCGC-BHQ1 |
|
RXRG |
retinoid X receptor, gamma |
1q22-q23 |
GCCGCCGTCACCGCTACT FAM-CCACCGCCGTCGCTGCTGC-BHQ1 |
|
RYR2 |
ryanodine receptor 2 (cardiac) |
1q43 |
FAM-TTTCCCCCAAGTCAAGGTGCTGCGAAA-BHQ1 GGGGACCACGGAGGCGACT |
|
SFRP2 |
secreted frizzled related protein 2 |
4q31.3 |
CCAGCCCTCCTCGGATTACCC FAM-CTCCCTTGCTCCCCCCACCCTCC-BHQ1 |
|
SOX17 |
SRY (sex determining region Y)-box 17 |
8q11.23 |
CGCCCTCCGACCCTCCAA FAM-TCCCGGATTCCCCAGGTGGCC-BHQ1 |
|
TERT |
telomerase reverse transcriptase |
5p15.33 |
FAM-CTCCCTGCAACACTTCCCCGCGA-BHQ1 AAAGAGAAATGACGGGCCTGTGTC |
|
ZNF613 |
zinc finger protein 613 |
19q31.41 |
GTGTTGCGGGGAGCTCTCG FAM-CCTCGCTCGGAGTTCGTTTCGCAG-BHQ1 |
Примечание: a – обозначение, хромосомная локализация полное и наименование белковых продуктов генов приводятся согласно указаниям Международного комитета по номенклатуре генов – HUGO Gene Nomenclature Committee [33]; b – структура прямого или обратного геномного праймера приводится соответственно перед или после структуры зонда. В последовательности флуоресцентных зондов FAM – 6-карбоксифлуоресцеин, BHQ1 – Black Hole Quencher 1.
Note: a – designation, chromosomal localization, and the full name of protein products of genes are given according to the instructions of the International Committee on Gene Nomenclature – HUGO Gene Nomenclature Committee [33]; b – the structure of the forward or reverse genomic primer is listed, respectively, before or after the structure of the probe. In the sequence of fluorescent probes FAM – 6-carboxyfluorescein, BHQ1 – Black Hole Quencher 1.
Таблица 2/table 2
Сайты RcgY, отобранные для glad-ПЦР-анализа, их локализация в геноме и структура соответствующих им гибридных праймеров
RcgY sites selected for glad-pcR analysis, their localization in the genome and the structure of the corresponding hybrid primers
|
Ген/Gene |
Сайт/Site |
Координаты сайтаa/ Site coordinatesa |
Структура гибридного праймераb/ Hybrid primer structureb |
|
ALX4 |
ACGT |
chr11:44304756-44304759 |
CCTGCTCTTTCATCGGTСC |
|
BMP2 |
ACGC |
chr20:6770341-6770344 |
CCTGCTCTTTCATCGGTCC |
|
CCND2 |
GCGC |
chr12:4274691-4274693 |
CCTGCTCTTTCATCGGCGG |
|
GALR1 |
GCGC |
chr18: 77249828-77249831 |
CCTGCTCTTTCATCGGCGG |
|
HMX2 |
ACGC |
chr10:123147940-123147943 |
CCTGCTCTTTCATCGGTCT |
|
HS3ST2 |
GCGT |
chr16:22814023-22814026 |
CCTGCTCTTTCATCGGCGG |
|
RARB |
ACGC |
chr3: 25428374–25428377 |
CCTGCTCTTTCATCGGTTC |
|
SOX17 |
GCGC |
chr8: 54458724-54458727 |
CCTGCTCTTTCATCGGCCG |
|
ZNF613 |
GCGC |
chr19: 51887759-51887762 |
CCTGCTCTTTCATCGGCGT |
Примечание: a – координаты сайта даны в соответствии с последней сборкой генома человека GRCh38.p13; b – подчеркнут 3’-концевой тетрануклеотид гибридного праймера, комплементарный последовательности ДНК в точке гидролиза GlaI.
Note: a – Site coordinates are given according to the latest assembly of the human genome GRCh38.p13; b – The 3’-tetranucleotide of the hybrid primer complementary to the DNA sequence at the GlaI hydrolysis point is underlined.
3 мин при 95 о С и 45 циклов – 10 c при 95 °С, 15 с при 61 °С, 20 с при 72 °С. Для устранения влияния возможных начальных флуктуаций на форму кривой амплификации флуоресценцию на первых пяти циклах ПЦР не детектировали.
Скрининг наиболее часто метилированных сайтов R(5mC)GY в составе регуляторных областей генов-онкосупрессоров проводили на ограниченных выборках из 10 препаратов ДНК, исследуя все образцы в трех повторах. Критерием отбора сайта для последующего анализа всей коллекции образцов ДНК опухолевых (n=30) и морфологически неизмененных тканей (n=20) больных РМЖ служило среднее значение Cq ≤ 30, полученное по меньшей мере для одного из 10 образцов. Поскольку степень метилирования сайтов RCGY в регуляторных областях части исследуемых генов не удовлетворяла указанному критерию, из дальнейшей работы были исключены сайты в составе генов CDH13 , CDX1 , FOXA1 , GATA5 , GREM1 , HIC1 , HOXC10 , ICAM5 , LAMA1 , RASSF1A , RUNX3 , RXRG , RYR2 , SFRP2 и TERT . Сайты R(5mC)GY, отобранные для исследования полной коллекции клинических образцов ДНК из тканей больных РМЖ, и соответствующие им гибридные праймеры приведены в табл. 2.
Статистическую обработку экспериментальных данных проводили с использованием программного обеспечения «MedCalc 15.11» (MedCalc Software, Бельгия). Исходя из значений Cq в ПЦР в режиме реального времени, для анализируемых маркеров вычисляли точки отсечения – пороговые величины Cq, дифференцирующие положительные и отрицательные случаи. Результаты работы алгоритма визуализировали с помощью характеристических кривых (ROC-кривые; англ. Receiver Operating
Characteristic Curves) – графиков зависимости процента верно диагностированных положительных случаев (чувствительность) от процента ложноположительных случаев (100-специфичность) для различных возможных значений диагностического теста. Площадь под ROC-кривой (ППК) является интегральным показателем диагностической эффективности маркера (для «идеального» теста ППК=1) [34].
Для отбора оптимальной комбинации маркеров с максимальным суммарным значением ППК, которая дает возможность наиболее результативно дифференцировать образцы ДНК из опухолевых и морфологически неизмененных тканей, применяли метод логистической регрессии. Данный алгоритм позволяет анализировать связи между несколькими независимыми переменными (значения Cq, пропорциональные степени метилирования исследованных сайтов R(5mC)GY) и зависимой переменной (принадлежность образца к опухолевым или морфологически неизмененным тканям).
Результаты и обсуждение
На рис. 2 отображены результаты GLAD-ПЦР-анализа метилированных сайтов RCGY в препаратах ДНК из клинических образцов, на рис. 3 – ROC-кривые, полученные при статистической обработке данных GLAD-ПЦР девяти сайтов R(5mC) GY. В табл. 3 приведены числовые значения параметров, рассчитанных для каждого из исследованных маркеров: показателей чувствительности и специфичности, величины ППК, выраженной в виде доли от всей площади квадрата с указанием стандартной ошибки измерения, и 95 % доверительного интервала.
Характерным эпигенетическим нарушением при канцерогенезе является локальное гиперметилирование так называемых CpG-островков в регуляторных областях генов-онкосупрессоров, которые в норме не содержат метильных групп. В настоящее время показано, что подобное аберрантное метилирование характерно для начальных стадий большинства ненаследственных (спорадических) форм рака, составляющих в среднем более 90 % всех случаев злокачественных новообразований [4]. Описано большое количество генов с разнообразными биологическими функциями, регуляторные области которых характеризуются различиями уровня метилирования ДНК в клетках РМЖ и морфологически неизмененных тканей [14, 32].
GLAD-ПЦР-анализ сайтов R(5mC)GY в генах ALX4 , BMP2 , CCND2 , GALR1 , HMX2 , HS3ST2 , RARB , SOX17 и ZNF613 показывает, что значения Cq для маркеров BMP2 , CCND2 , GALR1 , HMX2 , HS3ST2 и ZNF613 , связанные отрицательной корреляционной зависимостью со степенью метилирования сайта [6], в большинстве образцов ДНК РМЖ в среднем на шесть и более циклов ниже значений Cq в образцах ДНК из морфологически неизмененных тканей. В то же время для маркера ALX4 это различие минимально (менее 1,5 циклов), что обусловливает перекрывание диапазонов стандартных отклонений и делает невозможным использование данного маркера для выявления опухолевых тканей. Для сайтов R(5mC)GY в составе генов SOX17 и RARB разность средних величин Cq для двух групп ДНК-образцов составляет около трех циклов, но в последнем случае отмечается большой разброс индивидуальных значений (рис. 2).
Статистический анализ результатов GLAD-ПЦР (рис. 3 и табл. 3) демонстрирует, что сайты R(5mC)GY в составе генов CCND2 , BMP2 , GALR1 , SOX17 , HMX2 и HS3ST2 характеризуются наиболее сбалансированным соотношением чувствительности и специфичности теста. Полученные значения ППК более 0,8 свидетельствуют, согласно общепринятым критериям оценки данных ROC, о высоком диагностическом потенциале указанных маркеров [34].
Комплексный анализ данных ROC с применением метода логистической регрессии показывает, что комбинация маркеров CCND2 , BMP2 , GALR1 , SOX17 , HMX2 и HS3ST2 , охватывая всю коллекцию исследованных образцов, дает максимальную величину ППК=0,988 и позволяет дифференцировать образцы ДНК из опухолевых и морфологически неизмененных тканей с наибольшей эффективностью. Как видно из табл. 3, суммарные показатели чувствительности и специфичности для панели эпигенетических маркеров РМЖ составляют соответственно 90,0 и 100 %.
Полученные результаты хорошо коррелируют с опубликованными ранее данными по метилированию генов в тканях РМЖ, полученными с применением метода бисульфитной конверсии с последующим проведением метил-специфичной ПЦР [13, 23] либо секвенированием на NGS-платформе [14, 18, 22, 32]. При этом обращает внимание, что для 4 подтипов РМЖ, дифференцируемых по рецепторному статусу опухолей (люминальный А, люминальный В, HER2-позитивный, тройной негативный), обнаружены различия в профиле метилирования генов-онкосупрессоров, включая представленные в табл. 3 гены CCND2, GALR1, SOX17, HMX2, HS3ST2 и ZNF613 [14, 18, 22, 23, 26, 32].
Z. Li et al. показали, что аберрантное метилирование регуляторных участков генов CCND2 и SOX17 ассоциировано с развитием люминальных подтипов А и В [14]. Согласно данным S.K. Kas-sim et al., для люминального подтипа В характерно также гиперметилирование промотора гена HS3ST2 [23]. В клетках РМЖ HER2-позитивного подтипа детектируется аберрантное метилирование генов GALR1 , SOX17 и ZNF613 [14, 18, 32]. По данным C. Stirzaker et al., гиперметилирование промотора, обусловливающее инактивацию гена HMX2 , является отличительным признаком тройного негативного подтипа РМЖ [22].
По результатам ROC-анализа данных GLAD-ПЦР, полученных нами при исследовании сайтов R(5mC)GY в вышеназванных генах, большинство исследованных образцов РМЖ по каждому из маркеров определяются как положительные (табл. 3). Это относится и к генам GALR1 , ZNF613 и HMX2 , аберрантное метилирование которых ассоци-ировано с подтипами РМЖ, представленными в исследованной нами выборке незначительным числом образцов. Отмеченное несоответствие может быть связано с тем, что в предшествующих работах оценивали статус метилирования всего пула CpG-динуклеотидов в составе изучаемых генов, в то время как метод GLAD-ПЦР выявляет сайты R(5mC)GY в конкретных позициях генома человека (табл. 2), метилирование которых может быть общим признаком всех подтипов РМЖ.
Выводы, сделанные в перечисленных исследованиях, основывались на сравнении средних значений степени метилирования ДНК в выборках образцов тканей без определения частотных показателей метилирования или экспрессии генов для групп «РМЖ» и «морфологически неизмененная ткань». Такие данные описаны для генов BMP2 , RARB и ALX4 , также представленных в табл. 3.
M. Du et al. выявили значимое снижение уровня экспрессии мРНК BMP2 в 23 из 32 (71,88 %) исследованных образцов тканей РМЖ, соответствующее высокой частоте (68,75 %) аберрантного метилирования регуляторной области гена [13]. Выполненное нами исследование показало абсолютную диагностическую специфичность сайта RCGY в составе гена BMP2 – все образцы морфо-
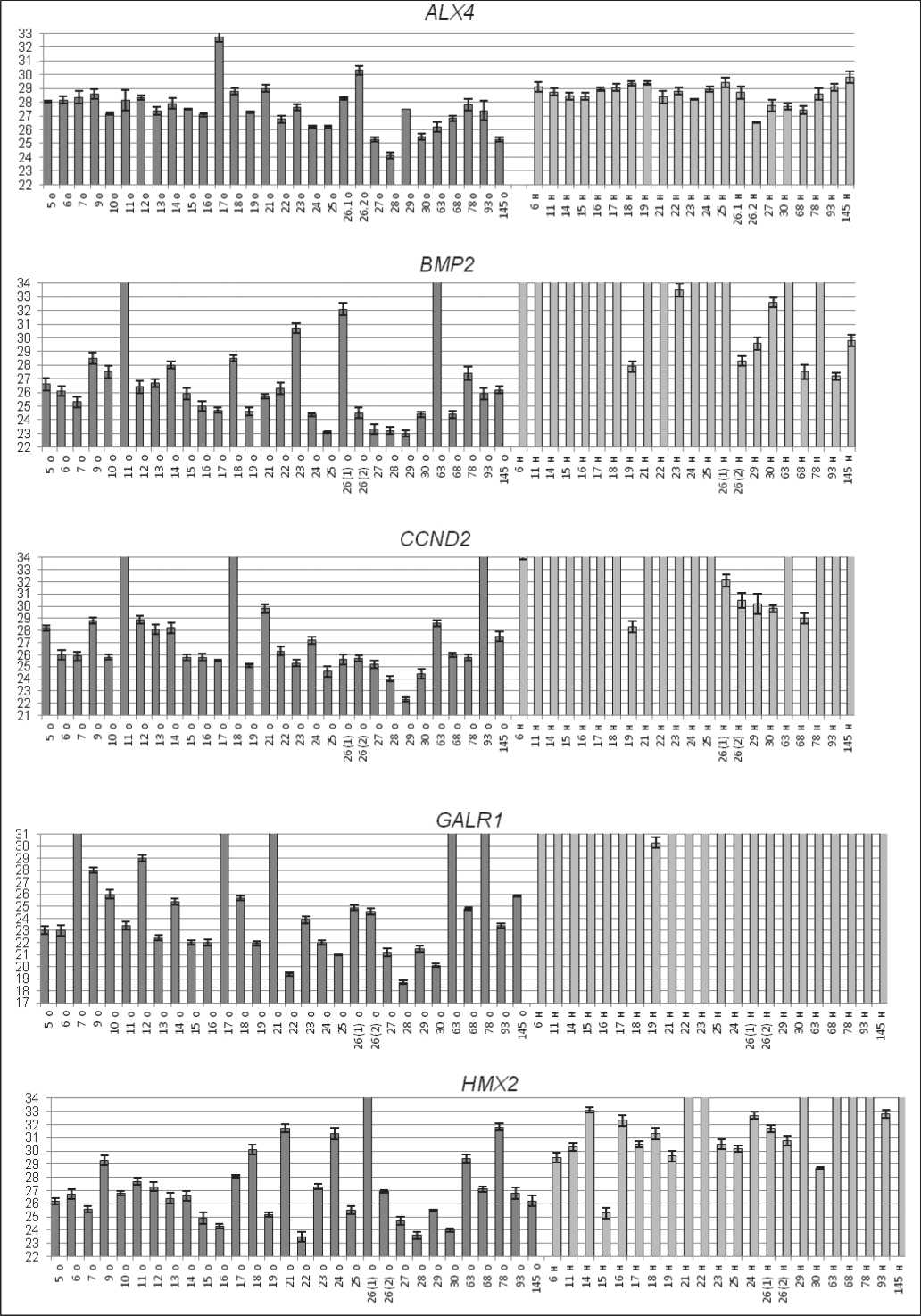
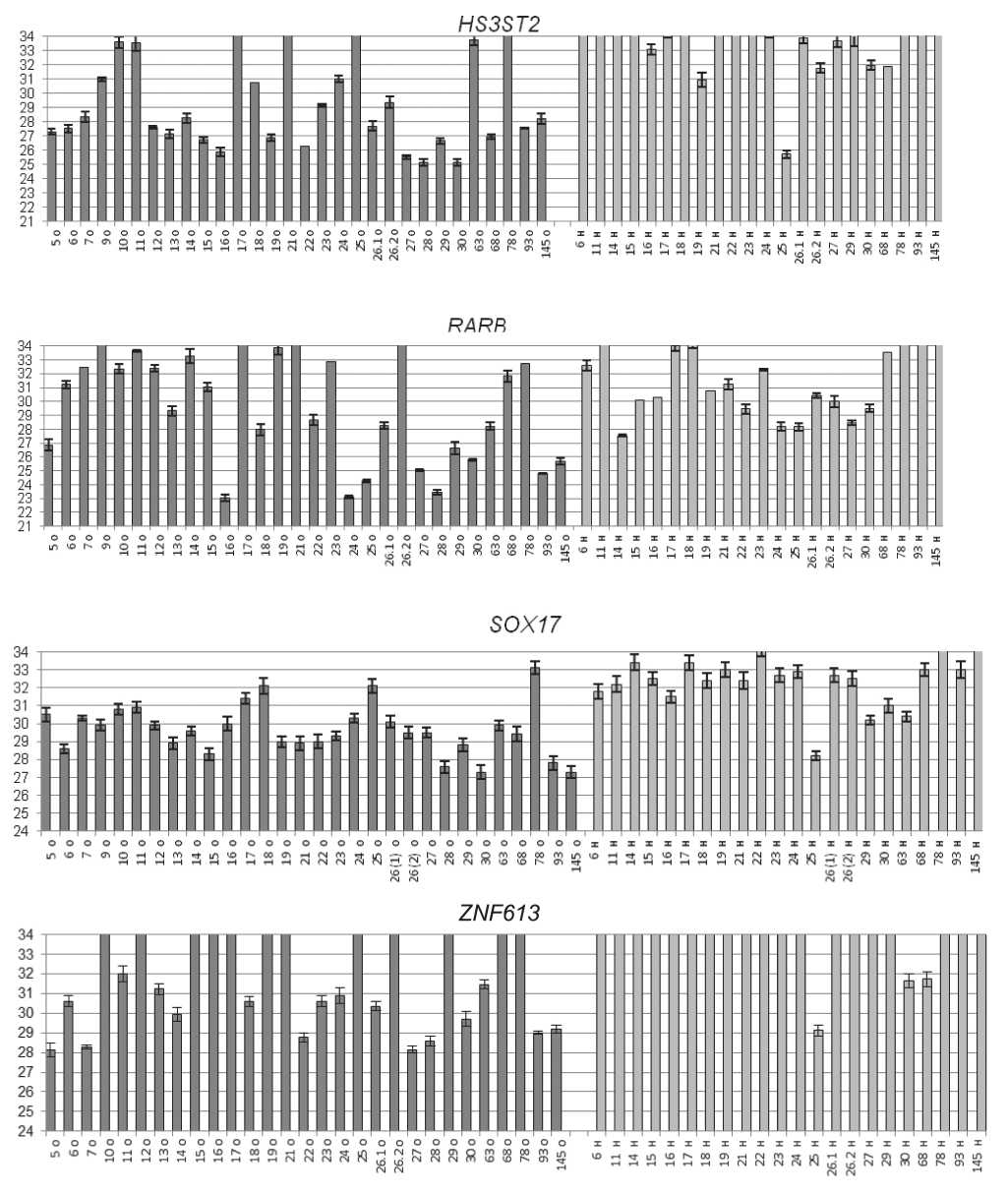
Рис. 2. Результаты GLAD-ПЦР анализа выбранных R(5mC)GY сайтов в препаратах ДНК из образцов тканей больных РМЖ. По оси ординат приведены значения Cq с диапазонами стандартных отклонений. По оси абсцисс указаны номера исследованных образцов тканей (о – опухоль, н – неизмененная ткань на линии резекции)
Fig. 2. Results of GLAD-PCR analysis of selected R (5mC) GY sites in DNA preparations from tissue samples from breast cancer patients. The ordinate shows Cq values with standard deviation ranges. The abscissa shows the numbers of the tissue samples examined (o – tumor, н – unchanged tissue on the resection line)
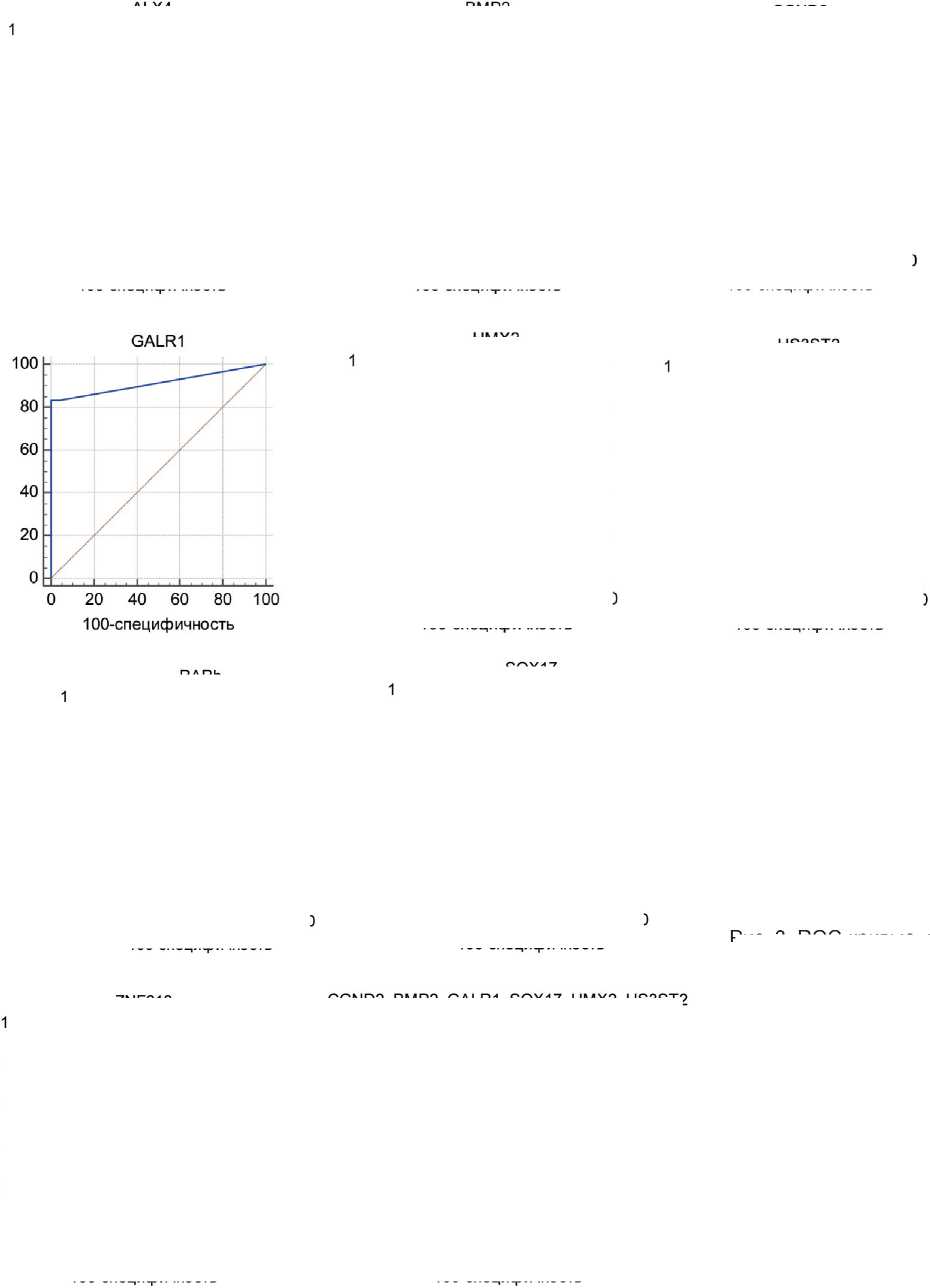
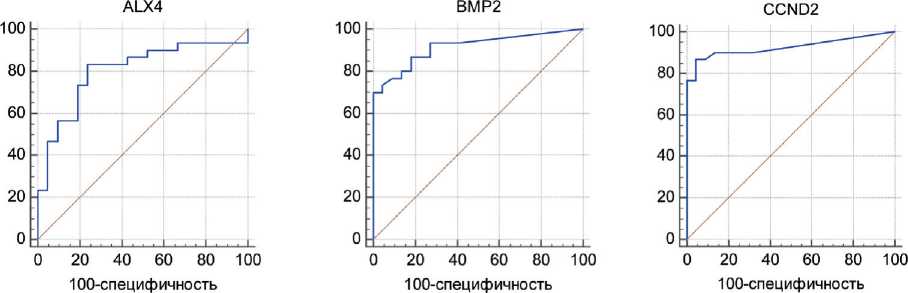
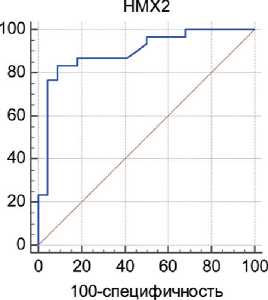
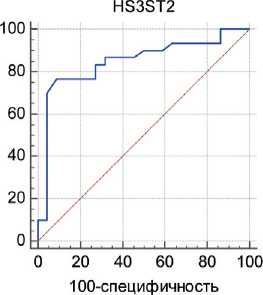
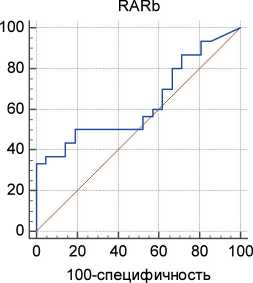
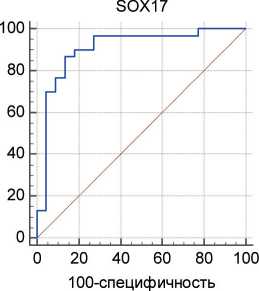
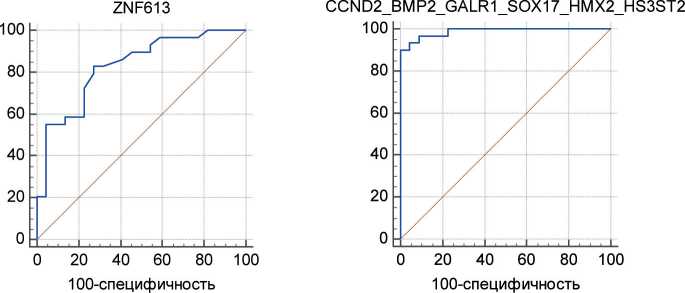
Рис. 3. ROC-кривые, полученные для результатов GLAD-ПЦР-анализа сайтов R(5mC)GY в препаратах ДНК из опухолевых и морфологически неизмененных тканей больных РМЖ. По оси ординат приведены показатели чувствительности (%); по оси абсцисс – процент ложноположительных случаев (100-специфичность) для различных значений диагностических тестов
Fig. 3. ROC curves obtained for the results of GLAD-PCR analysis of R (5mC) GY sites in DNA preparations from tumor and morphologically unchanged tissues of breast cancer patients. The ordinate shows the sensitivity indicators (%), the abscissa shows the percentage of false-positive cases (100-specificity) for different values of diagnostic tests
логически неизмененных тканей определяются как отрицательные (табл. 3).
При исследовании 48 пар образцов тканей, полученных от больных РМЖ, K. Yari et al. показали, что метилированные формы гена RARB обнаруживаются исключительно в опухолевых образцах с частотой 10,4 % [26]. Анализ данных, полученных нами для эпигенетического маркера RARB , также выявил низкий уровень диагностической чувствительности (табл. 3).
J. Yang et al. наблюдали инактивацию гена ALX4 при метилировании промотора во всех трех исследованных клеточных линиях РМЖ и в 69,44 % (75
из 108 образцов) первичных опухолей, тогда как в неизмененных тканях молочной железы этот ген не был метилирован [12]. Однако в проведенном нами исследовании сайта R(5mC)GY в гене ALX4 около трети образцов неизмененных тканей были определены как положительные (табл. 3).
Подобные расхождения могут быть обусловлены, в частности, особенностями морфологических характеристик РМЖ в выборках, исследованных разными авторами. Известно, что гистологически РМЖ представляет собой весьма разнородную группу злокачественных новообразований, которые могут различаться по профилю метилирования
Таблица 3/table 3
Данные Roc-анализа результатов glad-ПЦР, полученных при исследовании сайтов R(5mc) gY в образцах ДНК из опухолевых и морфологически неизмененных тканей больных РМЖ (в порядке уменьшения значений ППК)
data of Roc analysis of glad-pcR results obtained in the study of R(5mc)gY sites in dNa samples from tumor and morphologically unchanged tissues of breast cancer patients (in order of decreasing auc values)
|
Ген/ Gene |
о у ч £ S У ей D D S ^ U S о и U S и =- ^ ° S я 8 н ® И ® S g “ S й 9 ” и 2 К и « S й Л Р О (Й 45 СТО О > g Ч и У Ю G wo о I s » |
ч |
1 М о g S ч ® 5 1 1 « у & to seo ® й 2 Й „ * а 8 5 8 о 2 и |
о 5 & м ® к й 2 и S ^ Ьр g р О О о м О 5 § Эд ti 0J ^ М О р ^ у о н "о У Р4 О tj х у ру о 5 S » ° в 1 ° S g Q 2 ел и Р У Он S ! Ю Р D S О ^ "^ о |
и |
О о Н о |
1 S б PC |
|
CCND2 |
26/30 |
86,7 % |
21/22 |
95,5 % |
0,926 (0,038) |
0,818–0,980 |
|
|
BMP2 |
21/30 |
70,0 % |
22/22 |
100 % |
0,914 (0,039) |
0,803–0,974 |
|
|
GALR1 |
25/30 |
83,3 % |
22/22 |
100 % |
0,913 (0,036) |
0,801–0,973 |
|
|
SOX17 |
26/30 |
86,7 % |
19/22 |
86,4 % |
0,905 (0,048) |
0,791–0,968 |
|
|
HMX2 |
25/30 |
83,3 % |
20/22 |
90,9 % |
0,894 (0,047) |
0,777–0,962 |
|
|
HS3ST2 |
23/30 |
76,7 % |
20/22 |
90,9 % |
0,845 (0,059) |
0,718–0,931 |
|
|
ZNF613 |
24/29 |
82,8 % |
16/22 |
72,7 % |
0,828 (0,058) |
0,697–0,919 |
|
|
ALX4 |
24/29 |
82,8 % |
16/22 |
72,7 % |
0,762 (0,072) |
0,622–0,870 |
|
|
RARB |
9/29 |
31,0 % |
21/22 |
95,5 % |
0,599 (0,081) |
0,452–0,734 |
|
|
Комбинация генов |
|||||||
|
CCND2, BMP2, GALR1, SOX17, HMX2, HS3ST2 |
27/30 |
90,0 % |
22/22 |
100,0 % |
0,988 (0,011) |
0,909–1,000 |
ДНК. Помимо основных форм (неинвазивный/ инвазивный внутрипротоковый и дольковый рак) описан целый ряд особых вариантов РМЖ: папиллярный, тубулярный, аденокистозный, секреторный, апокриновый, метапластический, болезнь Педжета и др., на общую долю которых приходится до 20–25 % всех случаев заболевания [35]. Кроме того, нельзя исключить зависимости статуса метилирования генов от расово-этнической принадлежности пациенток [36].
Список литературы Применение GLAD-ПЦР-анализа для изучения метилирования ДНК в регуляторных областях генов-онкосупрессоров при раке молочной железы
- GLOBOCAN 2020 Database Provides Latest Global Data on Cancer Burden, Cancer Deaths [Internet]. URL: https://ascopost.com/news/december-2020/globocan-2020-database-provides-latest-global-data-oncancer-burden-cancer-deaths/ (cited 19.04.2021).
- Руководство по ранней диагностике рака. Женева: Всемирная организация здравоохранения, 2018. 38 с. [Guide to cancer early diagnosis. Geneva: World Health Organization, 2018. 38 p. (in Russian)].
- Breast Cancer Survival Rates [Internet]. URL: https://www.webmd. com/breast-cancer/guide/breast-cancer-survival-rates (19.04.2021).
- Hesson L.B., Pritchard A.L. Clinical Epigenetics. Springer Singapore, 2019. P. 153–171. doi: 10.1007/978-981-13-8958-0.
- Gao L., Emperle M., Guo Y., Grimm S.A., Ren W., Adam S., Uryu H., Zhang Z.M., Chen D., Yin J., Dukatz M., Anteneh H., Jurkowska R.Z., Lu J., Wang Y., Bashtrykov P., Wade P.A., Wang G.G., Jeltsch A., Song J. Comprehensive structure-function characterization of DNMT3B and DNMT3A reveals distinctive de novo DNA methylation mechanisms. Nat Commun. 2020 Jul 3; 11(1): 3355. doi: 10.1038/s41467-020-17109-4.
- Кузнецов В.В., Акишев А.Г., Абдурашитов М.А., Дегтярев С.Х. Способ определения нуклеотидной последовательности Pu(5mC)GPy в заданном положении протяженной ДНК. Патент РФ № 2525710. Заявл. 13.06.2013. Опубл. 20.08.2014. [Kuznetsov V.V., Akishev A.G., Abdurashitov M.A., Degtyarev S.Kh. Method of detecting nucleotide sequence Pu(5mC)GPy at predetermined position of long-distance DNA. The patent of Russian Federation No 2525710. 20.08.2014. (in Russian)].
- Евдокимов А.А., Нетесова Н.А., Сметанникова Н.А., Абдурашитов М.А., Акишев А.Г., Давидович Е.С., Кузнецов В.В., Ермолаев Ю.Д., Карпов А.Б., Сазонов А.Э., Тахауов Р.М., Дегтярев С.Х. Применение метода GLAD-ПЦР анализа для выявления сайтов метилирования в регуляторных областях генов-онкосупрессоров ELMO1 и ESR1 при колоректальном раке. Вопросы онкологии. 2016; 62(1): 117–21. [Evdokimov A.A., Netesova N.A., Smetannikova N.A., Abdurashitov M.A., Akishev A.G., Davidovich E.S., Kuznetsov V.V., Ermolaev Yu.D., Karpov A.B., Sazonov A.E., Takhauov R.M., Degtyarev S.Kh. Application of GLAD-PCR analysis for the methylation sites detection in the regulatory areas of tumor-suppressor genes ELMO1 and ESR1 in colorectal cancer. Problems in Oncology. 2016; 62(1): 117–21. (in Russian)].
- Evdokimov A.A., Netesova N.A., Smetannikova N.A., Abdurashitov M.A., Akishev A.G. GLAD-PCR Assay of DNA Methylation Markers Associated with Colorectal Cancer. Biol Med (Aligarh) 8: 342. doi: 10.4172/0974-8369.1000342.
- Smetannikova N.A., Evdokimov A.A., Netesova N.A., Abdurashitov M.A., Akishev A.G., Dubinin E.V., Pozdnyakov P.I., Vihlyanov I.V., Nikitin M.K., Topolnitsky E.B., Karpov A.B., Kolomiets S.A., Degtyarev S.K. Application of GLAD-PCR Assay for Study on DNA Methylation in Regulatory Regions of Some Tumor-Suppressor Genes in Lung Cancer. Zhongguo Fei Ai Za Zhi. 2019 Sep 20; 22(9): 551–561. doi: 10.3779/j.issn.1009-3419.2019.09.01.
- Малышев Б.С., Нетесова Н.А., Сметанникова Н.А., Абдурашитов М.А., Акишев А.Г., Дубинин Е.В., Азанов А.З., Вихлянов И.В., Никитин М.К., Карпов А.Б., Дегтярев С.Х. GLAD-ПЦР-анализ сайтов метилирования ДНК в регуляторных областях генов-онкосупрессоров при раке желудка. Acta Naturae. 2020; 12(3): 124–133. doi: 10.32607/actanaturae.11070. [Malyshev B.S., Netesova N.A., Smetannikova N.A., Abdurashitov M.A., Akishev A.G., Dubinin E.V., Azanov A.Z., Vihlyanov I.V., Nikitin M.K., Karpov A.B., Degtyarev S.Kh. GLAD PCR assay of DNA methylation sites in regulatory regions of some tumor-suppressor genes in breast cancer. Acta Naturae. 2020; 12(3): 124–133. (in Russian)]. doi: 10.32607/actanaturae.11070.
- Дубинин Е.В., Евдокимов А.А., Нетесова Н.А., Сметанникова Н.А., Абдурашитов М.А., Акишев А.Г., Давидович Е.С., Дегтярев С.Х., Кузнецов В.В., Михеев В.Н., Карпов А.Б., Малышев Б.С., Федотов В.В. Способ определения метилирования сайтов PuCGPy регуляторных областей генов-онкомаркеров колоректального рака методом GLAD-ПЦР-анализа и набор олигонуклеотидных праймеров и флуоресцентно-меченых зондов для осуществления указанного способа. Патент РФ № 2630669. Заявл. 16.07.2015. Опубл. 10.09.2017. [Dubinin E.V., Evdokimov A.A., Netesova N.A., Smetannikova N.A., Abdurashitov M.A., Akishev A.G., Davidovich E.S., Degtyarev S.Kh., Kuznetsov V.V., Miheev V.N., Karpov A.B., Malyshev B.S., Fedotov V.V. Method of determining the methylation sites PuCGPy regulatory regions of genes-markers of colorectal cancer by GLAD-PCR-analysis and oligonucleotide primers and fluorescent-labelled probes for realizing said method. The patent of Russian Federation No 2630669. 10.09.2017. (in Russian)].
- Yang J., Han F., Liu W., Chen H., Hao X., Jiang X., Yin L., Huang Y., Cao J., Zhang H., Liu J. ALX4, an epigenetically down regulated tumor suppressor, inhibits breast cancer progression by interfering Wnt/β-catenin pathway. J Exp Clin Cancer Res. 2017 Nov; 36(1): 170. doi: 10.1186/s13046-017-0643-9.
- Du M., Su X.M., Zhang T., Xing Y.J. Aberrant promoter DNA methylation inhibits bone morphogenetic protein 2 expression and contributes to drug resistance in breast cancer. Mol Med Rep. 2014 Aug; 10(2): 1051–5. doi: 10.3892/mmr.2014.2276.
- Li Z., Guo X., Wu Y., Li S., Yan J., Peng L., Xiao Z., Wang S., Deng Z., Dai L., Yi W., Xia K., Tang L., Wang J. Methylation profiling of 48 candidate genes in tumor and matched normal tissues from breast cancer patients. Breast Cancer Res Treat. 2015 Feb; 149(3): 767–79. doi: 10.1007/s10549-015-3276-8.
- Yang J., Niu H., Huang Y., Yang K. A Systematic Analysis of the Relationship of CDH13 Promoter Methylation and Breast Cancer Risk and Prognosis. PLoS One. 2016 May 6; 11(5): e0149185. doi: 10.1371/journal.pone.0149185.
- Liu J., Sun X., Qin S., Wang H., DU N., Li Y., Pang Y., Wang C., Xu C., Ren H. CDH1 promoter methylation correlates with decreased gene expression and poor prognosis in patients with breast cancer. Oncol Lett. 2016 Apr; 11(4): 2635–2643. doi: 10.3892/ol.2016.4274.
- Jing X., Liang H., Hao C., Hongxia L., Cui X. Analyses of an epigenetic switch involved in the activation of pioneer factor FOXA1 leading to the prognostic value of estrogen receptor and FOXA1 co-expression in breast cancer. Aging (Albany NY). 2019; 11(18): 7442–7456. doi: 10.18632/aging.102250.
- Lindqvist B.M., Wingren S., Motlagh P.B., Nilsson T.K. Whole genome DNA methylation signature of HER2-positive breast cancer. Epigenetics. 2014 Aug; 9(8): 1149–62. doi: 10.4161/epi.29632.
- Wang D., Yang P.N., Chen J., Zhou X.Y., Liu Q.J., Li H.J., Li C.L. Promoter hypermethylation may be an important mechanism of the transcriptional inactivation of ARRDC3, GATA5, and ELP3 in invasive ductal breast carcinoma. Mol Cell Biochem. 2014; 396(1–2): 67–77. doi: 10.1007/s11010-014-2143-y.
- de Groot J.S., Pan X., Meeldijk J., van der Wall E., van Diest P.J., Moelans C.B. Validation of DNA promoter hypermethylation biomarkers in breast cancer--a short report. Cell Oncol (Dordr). 2014 Aug; 37(4): 297–303. doi: 10.1007/s13402-014-0189-1.
- Cheng G., Sun X., Wang J., Xiao G., Wang X., Fan X., Zu L., Hao M., Qu Q., Mao Y., Xue Y., Wang J. HIC1 silencing in triple-negative breast cancer drives progression through misregulation of LCN2. Cancer Res. 2014 Feb; 74(3): 862–72. doi: 10.1158/0008-5472.CAN-13-2420.
- Stirzaker C., Zotenko E., Song J.Z., Qu W., Nair S.S., Locke W.J., Stone A., Armstong N.J., Robinson M.D., Dobrovic A., Avery-Kiejda K.A., Peters K.M., French J.D., Stein S., Korbie D.J., Trau M., Forbes J.F., Scott R.J., Brown M.A., Francis G.D., Clark S.J. Methylome sequencing in triple-negative breast cancer reveals distinct methylation clusters with prognostic value. Nat Commun. 2015 Feb 2; 6: 5899. doi: 10.1038/ncomms6899.
- Kassim S.K., Shehata H.H., Abou-Alhussein M.M., Sallam M.M., Amin I.I. Laboratory validation of formal concept analysis of the methylation status of microarray-detected genes in primary breast cancer. Tumour Biol. 2017 Jun; 39(6): 1010428317698390. doi: 10.1177/1010428317698390.
- Pathiraja T.N., Nayak S.R., Xi Y., Jiang S., Garee J.P., Edwards D.P, Lee A.V., Chen J., Shea M.J., Santen R.J., Gannon F., Kangaspeska S., Jelinek J., Issa J.P., Richer J.K., Elias A., McIlroy M., Young L.S., Davidson N.E., Schiff R., Li W., Oesterreich S. Epigenetic reprogramming of HOXC10 in endocrine-resistant breast cancer. Sci Transl Med. 2014 Mar 26; 6(229): 229ra41. doi: 10.1126/scitranslmed.3008326.
- Симонова О.А., Кузнецова Е.Б., Поддубская Е.В., Кекеева Т.В., Керимов Р.А., Троценко И.Д., Танас А.С., Руденко В.В., Алексеева Е.А., Залетаев Д.В., Стрельников В.В. Гены ламининов, конститутивно и аномально метилированные при раке молочной железы. Молекулярная биология. 2015; 49(4): 667–677. [Simonova O.A., Kuznetsova E.B., Poddubskaya E.V., Kekeeva T.V., Kerimov R.A., Trotsenko I.D., Tanas A.S., Rudenko V.V., Alekseeva E.A., Zaletayev D.V., Strelnikov V.V. DNA methylation in the promoter regions of the laminin family genes in normal and breast carcinoma tissues. Molecular Biology. 2015; 49(4): 598–607. (in Russian)].
- Yari K., Rahimi Z. Promoter Methylation Status of the Retinoic Acid Receptor-Beta 2 Gene in Breast Cancer Patients: A Case Control Study and Systematic Review. Breast Care (Basel). 2019; 14(2): 117–123. doi: 10.1159/000489874.
- Hagrass H.A., Pasha H.F., Shaheen M.A., Abdel Bary E.H., Kassem R. Methylation status and protein expression of RASSF1A in breast cancer patients. Mol Biol Rep. 2014; 41(1): 57–65. doi: 10.1007/s11033-013-2837-3.
- Garattini E., Bolis M., Garattini S.K., Fratelli M., Centritto F., Paroni G., Gianni’ M., Zanetti A., Pagani A., Fisher J.N., Zambelli A., Terao M. Retinoids and breast cancer: from basic studies to the clinic and back again. Cancer Treat Rev. 2014 Jul; 40(6): 739–49. doi: 10.1016/j.ctrv.2014.01.001.
- Wu H.C., Southey M.C., Hibshoosh H., Santella R.M., Terry M.B. DNA Methylation in Breast Tumor from High-risk Women in the Breast Cancer Family Registry. Anticancer Res. 2017; 37(2): 659–664. doi: 10.21873/anticanres.11361.
- Lin I.H., Chen D.T., Chang Y.F., Lee Y.L., Su C.H., Cheng C., Tsai Y.C., Ng S.C., Chen H.T., Lee M.C., Chen H.W., Suen S.H., Chen Y.C., Liu T.T., Chang C.H., Hsu M.T. Hierarchical clustering of breast cancer methylomes revealed differentially methylated and expressed breast cancer genes. PLoS One. 2015 Feb 23; 10(2): e0118453. doi: 10.1371/journal.pone.0118453.
- Masood S., El-Gabry E., Zhang C., Wang Z. DNA Methylation of the hTERT Gene in Breast Cancer Revisited: Diagnostic and Clinical Implications. Lab Med. 2016 Nov; 47(4): 293–299. doi: 10.1093/labmed/lmw043.
- Silva T.C., Coetzee S.G., Gull N., Yao L., Hazelett D.J., Noushmehr H., Lin D.C., Berman B.P. ELMER v.2: an R/Bioconductor package to reconstruct gene regulatory networks from DNA methylation and transcriptome profiles. Bioinformatics. 2019; 35(11): 1974–1977. doi: 10.1093/bioinformatics/bty902.
- HUGO Gene Nomenclature Committee. The resource for approved human gene nomenclature [Internet]. URL: http://www.genenames.org (cited 2021 Apr 19).
- Логистическая регрессия и ROC-анализ – математический аппарат [Интернет]. URL: https://loginom.ru/blog/logistic-regressionroc-auc (дата обращения: 19.04.2021). [Logistic regression and ROC analysis – mathematical apparatus [Internet]. URL: https://loginom.ru/blog/logistic-regression-roc-auc (cited 19.04.2021). (in Russian)].
- «Золотой стандарт» диагностики и лечения рака молочной железы 2021. Российское общество онкомаммологов. Версия 2.0. 176 с. [Интернет]. URL: http://www.abvpress.ru/project/ www.abvpress.ru/KP_POOM_12.10.20.pdf (дата обращения: 19.04.2021). [«Gold Standard» for breast cancer diagnosis and treatment 2021. Russian society of oncomammologists. Version 2.0. 176 p. [Internet]. URL: http://www.abvpress.ru/project/www.abvpress.ru/KP_POOM_12.10.20.pdf (cited 19.04.2021). (in Russian)].
- Xia Y.Y., Ding Y.B., Liu X.Q., Chen XM., Cheng S.Q., Li L.B., Ma M.F., He J.L., Wang Y.X. Racial/ethnic disparities in human DNA methylation. Biochim Biophys Acta. 2014; 1846(1): 258–62. doi: 10.1016/j.bbcan.2014.07.001.


