Применение нейронных сетей для семантической сегментации изображений глазного дна
Автор: Парингер Рустам Александрович, Мухин Артем Владимирович, Ильясова Наталья Юрьевна, Демин Никита Сергеевич
Журнал: Компьютерная оптика @computer-optics
Рубрика: Обработка изображений, распознавание образов
Статья в выпуске: 4 т.46, 2022 года.
Бесплатный доступ
Развитие нейросетевых алгоритмов произвело революцию во многих областях, а особенно в тех, что связаны с интеллектуальным анализом изображений. Особую сложность представляют собой задачи обработки биомедицинских данных, которым свойственны проблемы несбалансированности, малого объема и некачественной разметки. В данной работе производится исследование возможности использования нейронных сетей для решения задачи семантической сегментации изображений глазного дна. Для оценки применимости нейронных сетей для решения данной задачи было произведено сравнение их результатов с результатами сегментации изображений с помощью текстурных признаков. В результате оказалось, что нейронные сети превосходят в точности текстурные признаки по метрикам precision (~25 %) и recall (~50 %). Нейронные сети могут быть применены для решения задач биомедицинской сегментации изображений с предварительным применением алгоритмов балансировки и аугментации данных.
Свертка, нейронные сети, сверточные сети, сегментация, глазное дно
Короткий адрес: https://sciup.org/140295019
IDR: 140295019 | DOI: 10.18287/2412-6179-CO-1010
Текст научной статьи Применение нейронных сетей для семантической сегментации изображений глазного дна
В настоящее время актуальной задачей является повышение эффективности лазеркоагуляции сетчатки при лечении диабетического макулярного отека (ДМО) с применением современных цифровых технологий обработки изображений глазного дна, а также методов интеллектуального анализа неструктурированных данных [1]. Оптимизированная лазеркоагуляция обеспечивает максимально равномерное воздействие лазерной энергии на пигментный эпителий поражённой сетчатки, что увеличивает безопасность лечения и его эффективность, а также позволяет избежать применения более дорогостоящих методов лечения [2, 3].
При этом большое значение имеет высокоточное распознавание патологических и анатомических структур глазного дна с целью формирования зон лазерного воздействия и персонализированного плана оптимального расположения коагулятов в области ДМО [4–5]. Это увеличит качество лазерного лечения и объективной оценки объёма и локализации патологических структур, позволяющей прогнозировать результаты лечения и своевременно изменять тактику лечения диабетической ретинопатии.
В последнее время большинство задач интеллектуального анализа данных решается с помощью нейросетевых алгоритмов [6, 7]. Появление нейронных сетей произвело революцию в задачах обработки изображений. Особенно успешно нейронные сети применяются для решений задач классификации и сегментации изображений [8, 9]. Так, в работе [10] с их помощью решается задача диагностики диабетического макулярного отёка, а в работе [11] определяется поза человека.
В биомедицине нейронные сети нашли своё частое применение при решении задач семантической сегментации, например, для определения области поражения легких вирусом SARS‑CoV‑2 [12] или для нахождения раковых опухолей головного мозга человека [13].
Однако применение нейронных сетей в биомедицине сопряжено с рядом специфичных для этой области проблем. Чаще всего из-за соблюдения политики конфиденциальности и трудоёмкости процедуры разметки, требующей высокой квалификации от специалиста, создать достаточно большой набор данных необходимого качества крайне затруднительно [14]. Влияние проблемы недостаточного объёма данных можно нивелировать благодаря применению различных техник аугментации данных [15]. Так, в задачах, связанных с обработкой биомедицинских изображений, особенно действенной оказывается эластичная аугментация [16] данных. Другая проблема, свойственная биомедицинским данным, – проблема выраженного дисбаланса классов [17]. Решение проблемы в случае, когда несбалансированными оказываются множество классов, является нетривиальным, однако существуют алгоритмы, нивелирующие данную проблему [18, 19]. Наиболее комплексная проблема – низкое качество разметки данных [20], определить и устранить которую на этапе обучения алгоритма труднее всего.
В настоящей работе рассматривается применение нейронных сетей для сегментации изображений глазного дна. Особенностями данной задачи является тот факт, что исходные данные являются несбалансированными, число изображений мало, а разметка недостаточно точно совпадает с действительным расположением объектов.
Существует ряд работ, посвященных решению данной задачи, однако они в основном узкоспециализированы и рассматривают лишь сегментацию изображений на один класс, например, класс кровяных сосудов [21] или экссудатов [22]. В данной же работе рассматривается задача сегментации изображений глазного дна на несколько классов, актуальная для создания технологии поддержки принятия решений врачом при диагностике и лечении диабетического макулярного отека [23].
1. Архитектуры нейронных сетей
Применение нейронных сетей для решения задачи семантической сегментации изображений обусловлено рядом причин. Нейросетевые алгоритмы обладают хорошей обобщающей способностью; являются более точными (превзошли любые другие подходы) во множестве задач интеллектуального анализа данных; способны учитывать весь контекст изображения [24].
Однако их применимость ограничивается особенностями обучающего набора данных.
Поэтому для применения нейросетевых алгоритмов первоочередной задачей является подготовка данных и нивелирование проблем несбалансированности данных и небольшого объема данных.
Одной из первых архитектур нейронных сетей, успешно примененных для решения задачи семантической сегментации биомедицинских изображений, является архитектура U-Net [25]. Данная архитектура относится к классу полносверточных нейронных сетей. Данная сеть состоит из двух частей: энкодера, выделяющего признаки исходного изображения, и декодера, восстанавливающего карту сегментации по признакам, выделенным энкодером. Для эксперимента, представленного в настоящей статье, использовались построенные на основе U-Net сети: ResNetUnet, DenseNetUnet и XceptionUnet, где в качестве энкодера использовалась одна из сетей ResNet, DenseNet или Xception соответственно. Веса предобученных сетей использовались для инициализации энкодера и были зафиксированы на время обучения.
-
1.1. ResNetUnet
Архитектура ResNetUnet использует в качестве энкодера признаков сеть ResNet-101 [26], предобу-ченную на наборе данных ImageNet [27]. К особенностям архитектуры ResNet можно отнести её глубину. Это была первая архитектура, позволившая исследователям обучать нейронные сети более чем с 20 слоями. Также в данной архитектуре впервые были использованы skip-connection (модификация архитектуры, позволяющая сигналу проходить через сеть, пропуская один или несколько слоев) для предотвращения проблемы затухающего градиента (рис. 1).
|
1024x1024x3 |
Image |
Result |
|
Block 1-2... |
| 1x1 Conv, 8,/1 I |U24xl024x64 |
|
|
512x512x128 |
Block 3... |
UnetBlock 5 |512x512x256 |
|
____________+------------------ |
||
| ConvDepthWise |
| BatchNorm | 1x6
UnetBlock
1x1 Conv
MaxPool...
|
256x256x128 I |
|||
|
ConvBlock 0, /1 |
|||
|
UnetBlock 4 |
|||
|
IdenBlock 1-2 |
256x256x512 |
||
|
ConvBlock 3 |
|||
|
128x128x512 |
| UnetBlock 3 | |
||
|
| IdenBlock 4-6 | |
128x128x768 |
||
|
1---- — |
|||
|
ConvBlock 7 |
|||
|
64x64x10... |
г |
UnetBlock 2 |
|
|
I IdenBlock 8-22 1 |
' 64x64x12... |
||
|
ConvBlock 23 |
|||
|
32x32x20... |
|||
|
IdenBlock 24-25 |
UnetBlock 1 I |
||
ReLU
Resize, x2 |
|
IdenBlock |
|
|
1x1 Conv |
|
|
BatchNorm |
|
|
I ReLU I |
|
|
, ; , |
|
|
3x3 Conv |
|
|
I BatchNorm | |
о |
|
I ReLU I |
о |
|
, ; , |
|
|
1x1 Conv |
3 |
|
BatchNorm |
|
|
I Sum H |
|
Block
| BatchNorm |
ReLU
ConvBlock
1x1 Conv, /2
BatchNorm
ReLU
3x3 Conv
BatchNorm
ReLU
1x1 Conv
BatchNorm
| 1x1 Conv,/2 |
| BatchNorm |
Рис. 1. Архитектура ResNetUnet
-
1.2. DenseNetUnet
Архитектура DenseNetUnet использует в качестве энкодера признаков сеть DenseNet-169 [28], которая была предобучена на наборе данных ImageNet [27].
Архитектура DenseNet хорошо обучается на небольших наборах данных и предлагает соединять все слои с помощью skip-connection в пределах одного строительного блока сети (рис. 2).
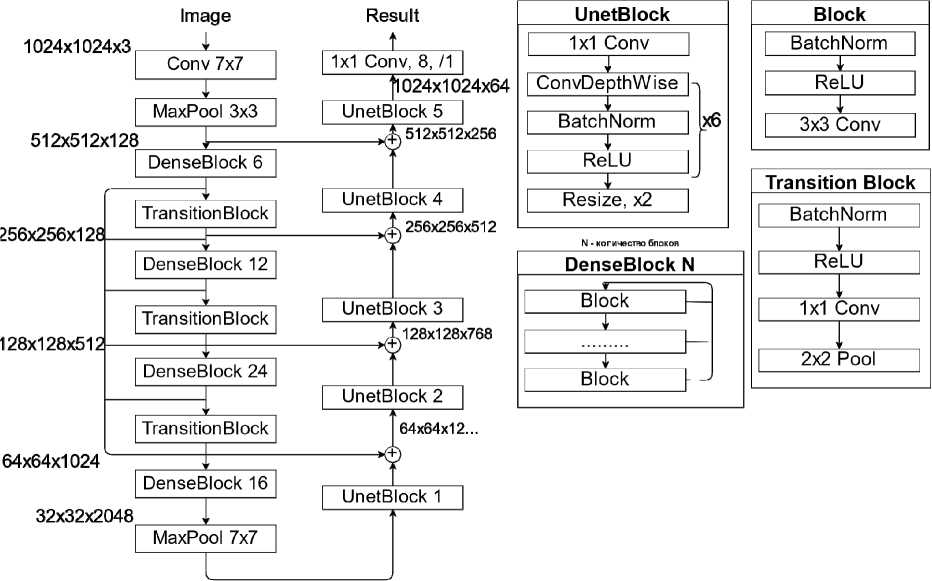
Рис. 2. Архитектура DenseNetUnet
1.3. XceptionUnet
2. Текстурные признаки
И последняя архитектура, XceptionUnet, использует в качестве энкодера признаков сеть Xception-65 [29], которая была предобучена на наборе данных ImageNet [27]. К особенностям данной архитектуры нейронной сети относится использование комбинации pointwise (точечная свертка с размером ядра 1×1) и depthwise (пространственная свертка, независимо применяемая к каждому каналу) сверток вместо классической свертки. Данная замена позволяет сократить число обучаемых параметров без влияния на точность сети. На рис. 3 схематично представлена архитектура XceptionUnet.
Одним из классических способов решения задачи сегментации изображений является подход, основанный на использовании текстурных признаков. Исследования, в которых применяются текстурные признаки для сегментации изображений, по-прежнему актуальны.
Image
024x1024x3 Г
Result
512x512x32
Block 1...
I 1x1 Conv, 8,7T t 1024x1 024x64
| UnetBlock 5 | f 512x512x160
EntryFlow
MiddleFlow
Block 2...
512x512x64 J
| EntryFlow 1
256x256x128 I
| UnetBlock 4 | t 256x256x384
| EntryFlow 2
128x128x256 П
| UnetBlock 3 | J 128x128x512
| EntryFlow 3
64x64x728 Д
| MiddleFlow 4-20
| UnetBlock 2 | t 64x64x984
| EntryFlow 21
32x32x1024
| ExitFlow 22
32x32x2048
| UnetBlock 1 |
, A 32x32x3072
| ConvDepthWise
I BatchNorm
ReLU
| 1x1 Conv, 72 |
ConvDepthWise
BatchNorm
ReLU
I 1x1 Conv
I BatchNorm
ReLU
| Repeat 2 times | ConvDepthWise,/2 | BatchNorm |
ReLU
1x1 Conv
| BatchNorm
| BatchNorm |
Sum I
1x1 Conv
BatchNorm
ReLU
Repeat 2 times
ConvDepthWise
BatchNorm
ReLU
1x1 Conv
BatchNorm
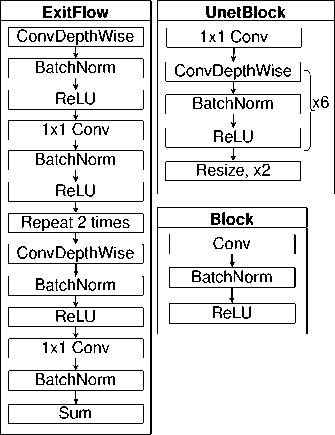
Рис. 3. Архитектура XceptionUnet
Сегментация изображений с помощью текстурных признаков выполняется в несколько этапов [30]:
1. Фрагментация изображения. Этап, на котором изображение разбивается на квадратные области, например, размером 12×12 пикселей.
2. Вычисление текстурных признаков для каждого фрагмента. На данном этапе для каждого фрагмента вычисляются текстурные признаки, например, с помощью ПО Mazda [31]. Этот этап является наиболее длительным. Так, вычисление небольшого набора из пары десятков признаков для одного изображения
размера 1024×1024 пикселей может занимать несколько часов при использовании вычислений на центральном процессоре современного многоядерного компьютера.
3. Классификация фрагментов на основе вычисленных значений текстурных признаков. На данном этапе производится классификация одного из пикселей фрагмента изображения по вектору значений вычисленных текстурных признаков. Чаще всего классификатор строится с использованием алгоритма деревьев решений.
3. Описание эксперимента
Текстурные признаки хорошо изучены и успешно применяются для решения множества задач [30, 32]. Однако их использование занимает много времени, что делает их внедрение в медицинскую практику затруднительным. Помимо этого, текстурные признаки обладают недостаточной обобщающей способностью для решения комплексных задач.
Для проведения эксперимента использовался набор данных, предоставленный офтальмологами в рамках выполнения работ по гранту РФФИ, состоящий из 115 изображений глазного дна, которые были размечены на 8 классов: оптический диск (OD), макула (M), кровяные сосуды (BV), твердые экссудаты (HE), мягкие экссудаты (SE), новые коагуляты (NC), пигментированные коагуляты (PC), геморрагия (H). Для данных изображений также были вычислены текстурные признаки.
При исследовании данного набора изображений было выяснено, что классы новых и пигментированных коагулятов являются редкими и присутствуют менее чем в 10% изображений. Также редким классом является класс мягких экссудатов, он присутствует лишь в 37% изображений. Наличие трех редких классов обозначает проблему несбалансированности исходного набора данных. Влияние данной проблемы было нивелировано благодаря использованию алгоритма балансировки данных, который учитывает несбалансированность множества классов [33].
Данный алгоритм не увеличивает исходный набор данных, а лишь изменяет частоту выбора изображений, содержащих тот или иной класс. Проблема небольшого объема данных была нивелирована использованием аугментации: поворот на случайный угол, отражения, эластичная деформация. В результате применения аугментации исходный набор данных был расширен в 30 раз.
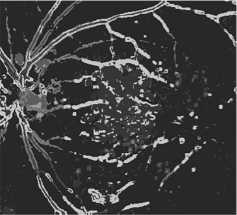
Помимо этого, был выявлен ряд ошибок в разметке данных. Пример некачественной разметки для класса кровяных сосудов представлен на рис. 4.
Нейронные сети построены и обучены с помощью библиотеки TensorFlow [34]. Использование предобу-ченных энкодеров позволяет осуществлять обучение нейронных сетей значительно быстрее. При обучении использовались следующие параметры:
- Размер входа: 1024×1024×3.
- Количество эпох: 12.
- Функция ошибки: FocalLoss [35].
- Оптимизатор: Adam [36].
- Скорость обучения: 0,003.
4. Результаты
Использование полного набора текстурных признаков для сегментации изображений неэффективно, поэтому был произведен отбор признаков согласно индивидуальному критерию информативности дискриминантного анализа [37]. Отбор признаков по данному критерию является классическим способом поиска информативных признаков.
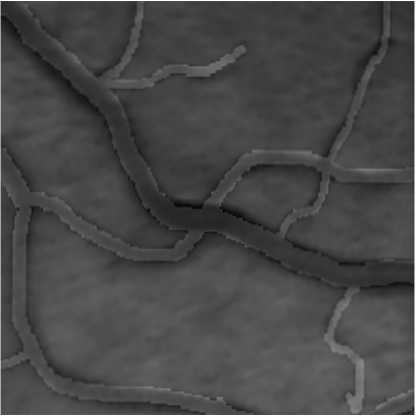
Рис. 4. Пример некачественной разметки кровяных сосудов
Выбор именно такого метода отбора признаков также обусловлен свойствами дискриминантного анализа. Его критерии позволяют выбрать такие признаки, что наилучшим образом разделяют пространство объектов. Классификация пикселей по значениям текстурных признаков выполнялась с помощью дерева решений.
Для оценки качества сегментации изображений использовались метрики: precision, recall, f1-score . Достоверность результатов эксперимента обеспечена использованием k-fold кросс-валидации [38]. Для нейронных сетей набор данных был разбит на три части ( k =3), а для текстурных признаков – на пять ( k =5). Полученные по всем частям значения метрик усреднялись.
В табл. 1 представлены результаты эксперимента. Столбец TF ( Textural features ) соответствует результатам, полученным с помощью текстурных признаков. В таблице avg и w . avg – среднее и взвешенное среднее соответственно. Так, по метрике precision видно, что нейронные сети превосходят текстурные признаки по большинству классов. Однако, согласно precision текстурные признаки способны отделять классы кровяных сосудов и твердых экссудатов от других лучше, чем нейронные сети. Согласно метрике recall нейронные сети более чувствительны, что может говорить о лучшей обобщающей способности нейронных сетей. Также вероятнее всего нейронные сети способны находить объекты, которые были пропущены в процессе разметки. Метрика f-score демонстрирует результаты, схожие с метрикой precision .
Визуализация результатов сегментации представлена на рис. 5. Здесь видно, что нейронная сеть справляется с задачей существенно лучше по сравнению с текстурными признаками. Данный факт можно объяснить лучшей обобщающей способностью нейронных сетей и их устойчивостью к различным условиям съемки глазного дна.
Также в результате эксперимента было замечено, что сегментация одного изображения на ЦПУ нейронной сетью занимает порядка нескольких се- кунд. В свою очередь, для сегментации изображения с помощью текстурных признаков уходит значительно больше времени (больше часа).
Табл. 1. Результаты эксперимента
|
Precision |
Recall |
F1-score |
||||||||||
|
DenseNet |
ResNet |
Xception |
TF |
DenseNet |
ResNet |
Xception |
TF |
DenseNet |
ResNet |
Xception |
TF |
|
|
OD |
0,66 |
0,66 |
0,60 |
0,50 |
0,94 |
0,94 |
0,97 |
0,79 |
0,78 |
0,77 |
0,73 |
0,61 |
|
M |
0,53 |
0,56 |
0,51 |
0,45 |
0,81 |
0,81 |
0,87 |
0,26 |
0,63 |
0,65 |
0,64 |
0,33 |
|
BV |
0,25 |
0,26 |
0,27 |
0,52 |
0,92 |
0,92 |
0,92 |
0,57 |
0,40 |
0,41 |
0,41 |
0,55 |
|
HE |
0,41 |
0,40 |
0,50 |
0,89 |
0,82 |
0,82 |
0,71 |
0,59 |
0,54 |
0,53 |
0,55 |
0,71 |
|
SE |
0,33 |
0,72 |
0,54 |
0,00 |
0,56 |
0,20 |
0,52 |
0,00 |
0,38 |
0,27 |
0,48 |
0,00 |
|
NC |
0,53 |
0,40 |
0,43 |
0,00 |
0,06 |
0,05 |
0,13 |
0,00 |
0,11 |
0,07 |
0,15 |
0,00 |
|
PC |
0,23 |
0,37 |
0,27 |
0,00 |
0,37 |
0,28 |
0,40 |
0,00 |
0,28 |
0,26 |
0,28 |
0,00 |
|
H |
0,33 |
0,37 |
0,33 |
0,39 |
0,85 |
0,78 |
0,83 |
0,42 |
0,47 |
0,50 |
0,46 |
0,41 |
|
avg |
0,41 |
0,47 |
0,43 |
0,34 |
0,67 |
0,60 |
0,67 |
0,33 |
0,45 |
0,43 |
0,46 |
0,33 |
|
w. avg |
0,42 |
0,48 |
0,44 |
0,33 |
0,63 |
0,56 |
0,64 |
0,31 |
0,45 |
0,43 |
0,46 |
0,31 |
Исходное изображение
Исходная разметка Исходное изображение Исходная разметка
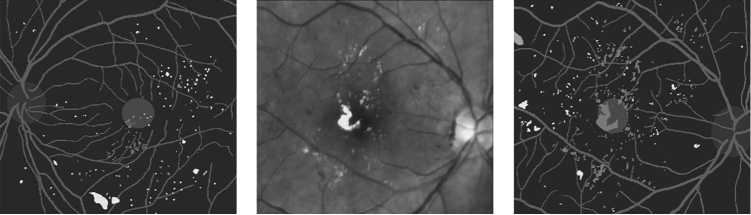
Текстурные признаки
Нейронная сеть Текстурные признаки Нейронная сеть
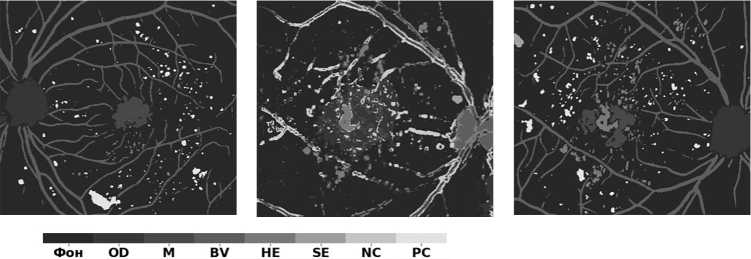
Рис. 5. Примеры сегментации изображений глазного дна, полученных в результате эксперимента
Заключение
В данной статье представлены результаты применения нейросетевых алгоритмов для решения задачи семантической сегментации изображений глазного дна с выраженным дисбалансом классов. Исходя из анализа результатов было выяснено, что нейронные сети превосходят в точности текстурные признаки. Более того, нейронные сети могут быть применены для сегментации изображений глазного дна, которые были получены при различных условиях съемки, в отличие от текстурных признаков. Использование предобученных нейронных сетей и их дообучение на малом наборе данных совместно с использованием алгоритма балансировки и техник аугментации позволили разработать достаточно точные алгоритмы семантической сегментации изображений глазного дна по малому набору данных.
Алгоритм сегментации изображений глазного дна, основанный на использовании нейронной сети с архитектурой XceptionUnet, может быть использован в системах поддержки принятия решений врачом-диагностом при необходимости работы в условиях реального времени за счёт использования графического ускорителя пользовательского уровня.
Работа выполнена при финансовой поддержке РФФИ (грант № 19-29-01135), Министерства науки и высшего образования РФ в рамках выполнения работ по государственному заданию ФНИЦ «Кристаллография и фотоника» РАН.
Список литературы Применение нейронных сетей для семантической сегментации изображений глазного дна
- Kermany DS, Goldbaum M, Cai W. Identifying medical diagnoses and treatable diseases by image-based deep learning. Cell 2018; 172(5): 1122-1131.
- Kozak I, Luttrull JK. Modern retinal laser therapy. Saudi J Ophthalmol 2015; 29(2): 137-146.
- Gafurov SD, Katakhonov ShM, Holmonov MM. Features of the use of lasers in medicine. European science 2019; 3(45): 92-95.
- Ilyasova NYu, Shirokanev AS, Kupriyanov AV, Paringer RA. Technology of intellectual feature selection for a system of automatic formation of a coagulate plan on retina. Computer Optics 2019: 43(2): 304-315. DOI: 10.18287/2412-6179-2019-43-2-304-315.
- Ilyasova NYu, Shirokanev AS, Kirsh DV, Demin NS, Zamytskiy EA, Paringer RA, Antonov AA. Identification of prognostic factors and predicting the therapeutic effect of laser photocoagulation for DME treatment. Electronics 2021; 1(12): 1420. DOI: 10.3390/electronics10121420.
- Li Z, Liu F, Yang W, Peng S, Zhou J. A survey of convolutional neural networks: analysis, applications, and prospects. IEEE Trans Neural Netw Learn Syst 2021: 1-21.
- Samek W, Montavon G, Lapuschkin S, Anders CJ, Müller KR. Explaining deep neural networks and beyond: A review of methods and applications. Proc IEEE 2021; 109(3): 247-278.
- Rawat W, Wang Z. Deep convolutional neural networks for image classification: A comprehensive review. Neural comput 2017; 29(9): 2352-2449.
- Guo Y, Liu Y, Georgiou T, Lew MS. A review of semantic segmentation using deep neural networks. Int J Multimed Inf Retr 2018; 7(2): 87-93.
- Singh RK, Gorantla R. DMENet: diabetic macular edema diagnosis using hierarchical ensemble of CNNs. Plos one 2020; 15(2): e0220677.
- Cao Z, Hidalgo G, Simon T, Wei SE, Sheikh Y. OpenPose: realtime multi-person 2D pose estimation using Part Affinity Fields. IEEE Trans Pattern Anal Mach Intell 2019; 43(1): 172-186.
- Apostolopoulos ID, Mpesiana TA. Covid-19: automatic detection from x-ray images utilizing transfer learning with convolutional neural networks. Phys Eng Sci Med 2020; 43(2): 635-640.
- Ismael SA, Mohammed A, Hefny H. An enhanced deep learning approach for brain cancer MRI images classification using residual networks. Artif Intell Med 2020; 102: 101779.
- Arellano AM, Dai W, Wang S, Jiang X. Ohno-Machado L. Privacy policy and technology in biomedical data science. Annu Rev Biomed Data Sci 2018; 1: 115-129.
- Shorten C, Khoshgoftaar TM. A survey on image data augmentation for deep learning. J Big Data 2019; 6(1): 60.
- Castro E, Cardoso JS, Pereira JC. Elastic deformations for data augmentation in breast cancer mass detection. 2018 IEEE EMBS Int Conf on Biomedical & Health Informatics (BHI) 2018: 230-234.
- Ishwaran H, O'Brien R. Commentary: the problem of class imbalance in biomedical data. J Thorac Cardiovasc Surg 2021; 161(6): 1940.
- Charte F, Rivera AJ, del Jesus MJ, Herrera F. MLSMOTE: Approaching imbalanced multilabel learning through synthetic instance generation. Knowl-Based Syst 2015; 89: 385-397.
- Pereira RM, Costa YM, Silla CN Jr. MLTL: A multi-label approach for the Tomek Link undersampling algorithm. Neurocomputing 2020; 383: 95-105.
- Hao D, Zhang L, Sumkin J, Mohamed A, Wu S. Inaccurate labels in weakly-supervised deep learning: Automatic identification and correction and their impact on classification performance. IEEE J Biomed Health Inform 2020; 24(9): 2701-2710.
- Tian C, Fang T, Fan Y, Wu W. Multi-path convolutional neural network in fundus segmentation of blood vessels. Biocybern Biomed Eng 2020; 40(2): 583-595.
- Kaur J, Mittal D. A generalized method for the segmentation of exudates from pathological retinal fundus images. Biocybern Biomed Eng 2018; 38(1): 27-53.
- Bhagat N, Grigorian RA, Tutela A, Zarbin MA. Diabetic macular edema: pathogenesis and treatment. Surv Ophthalmol 2009; 54(1): 1-32.
- Gabbasov R, Paringer R. Influence of the receptive field size on accuracy and performance of a convolutional neural network. 2020 Int Conf on Information Technology and Nanotechnology (I TNT) 2020: 1-4. DOI: 10.1109/ITNT49337.2020.9253219.
- Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation. Int Conf on Medical Image Computing and Computer-Assisted Intervention 2015: 234-241.
- He K, Zhang X, Ren S, Sun J. Deep residual learning for image recognition. Proc IEEE conf on Computer Vision and Pattern Recognition 2016: 770-778.
- Krizhevsky A, Sutskever I, Hinton GE. Imagenet classification with deep convolutional neural networks. Adv Neural Inf Process Syst 2012; 25: 1097-1105.
- Iandola F, Moskewicz M, Karayev S, Girshick R, Darrell T, Keutzer K. Densenet: Implementing efficient convnet descriptor pyramids. arXiv preprint 2014. Source: https://arxiv.org/abs/1404.1869.
- Chollet F. Xception: Deep learning with depthwise separable convolutions. Proc IEEE conf on Computer Vision and Pattern Recognition 2017: 1251-1258.
- Ilyasova N, Paringer R, Kupriyanov A, Kirsh D. Intelligent feature selection technique for segmentation of fundus images. 2017 Seventh Int conf on Innovative Computing Technology (INTECH) 2017: 138-143.
- MaZda Web Site. Source: http://www.eletel.p.lodz.pl/programy/mazda/index.php.
- Wu J, Poehlman S, Noseworthy MD, Kamath MV. Texture feature based automated seeded region growing in abdominal MRI segmentation. 2008 Int Conf on BioMedical Engineering and Informatics 2008; 27(2): 263-267.
- Mukhin A, Kilbas I, Paringer R, Ilyasova N. Application of the gradient descent for data balancing in diagnostic image analysis problems. 2020 Int Conf on Information Technology and Nanotechnology (ITNT) 2020: 1-4. DOI: 10.1109/ITNT49337.2020.9253278.
- TensorFlow. Source: https://www.tensorflow.org.
- Lin TY, Goyal P, Girshick R, He K, Dollar P. Focal loss for dense object detection. Proc IEEE int conf on Computer Vision 2017: 2980-2988.
- Kingma DP, Ba J. Adam: A method for stochastic optimization. arXiv preprint 2014. Source: https://arxiv.org/abs/1412.6980.
- Tang H, Maitre H, Boujemaa N, Jiang W. On the relevance of linear discriminative features. Inf Sci 2010; 180(18): 3422-3433.
- Stone M. Cross-validatory choice and assessment of statistical predictions. Journal of the Royal Statistical Society: Series B (Methodological) 1974; 36(2): 111-133.


