Программа определения и анализа ассоциациированности видов в экологических сообществах
Автор: Селезнев Д.Г.
Журнал: Самарская Лука: проблемы региональной и глобальной экологии @ssc-sl
Рубрика: Краткие сообщения
Статья в выпуске: 3 т.32, 2023 года.
Бесплатный доступ
Описана разработанная автором методика выделения ассоциаций видов, которая отличается простотой реализации, устойчивостью и воспроизводимостью результатов. Недостатком методики является использование только качественных данных о наличии или отсутствии вида в пробе, а не его количественных характеристик.
Видовые ассоциации, статистическая методика, общая сила связности
Короткий адрес: https://sciup.org/148327557
IDR: 148327557 | УДК: 574.9 | DOI: 10.24412/2073-1035-2023-10498
Текст краткого сообщения Программа определения и анализа ассоциациированности видов в экологических сообществах
Важной 1 частью анализа качественных экологических данных является выявление видовых ассоциаций, обусловленных прямыми (трофические, топические, фориче-ские и др.) и косвенными (общность биото-пических условий, характер питания, защита от хищников) межвидовыми отношениями (Odum, Barrett, 1971). С этой целью было разработано несколько специализированных матричных индексов, для определения уровня значимости которых предлагается использовать одну из девяти рандо-мизационных процедур построения теоретического распределения индекса (Gotelli, 2000). Однако в экологических исследованиях задача определения видовых ассоциаций решается сравнительно редко из-за неочевидности математического аппарата и сложности его реализации.
Автором была разработана статистическая методика выделения положительных и отрицательных ассоциаций видов, позволяющая избежать использования матричных индексов и рандомизационной процедуры определения их уровня значимости. Для этого на бинарной матрице встречаемости видов в экологических пробах ис- пользуются дискретные гипергеометрическое или биномиальное распределения, дающие оценку вероятности наблюдаемой совместной встречаемости пар видов. Критическая область функции распределения (confidence intervals of distribution) позволяет принять одно из трех решений: пара видов отрицательно либо положительно ассоциирована или их совместная встречаемость в пробах носит случайный характер. В качестве альтернативы критической области возможно использование байесовского вывода (Bayesian inference), где в качестве оцениваемого параметра может быть взята любая равномерно распределенная наблюдаемая характеристика, например, общее число проб, а в качестве генеративной функции – функция вероятности дискретного распределения.
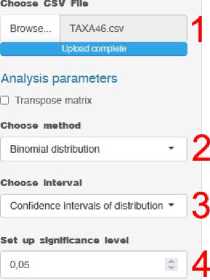
Предложенная методика реализована в виде web-приложения, размещенного на сервере Института биологии внутренних вод по адресу (рис. 1). Приложение написано на языке R в среде Shiny (Chang et al., 2023) и позволяет проанализировать бинарную пользовательскую матрицу формата «виды – пробы» с использованием двух дискретных распределений с заданной критической областью или с помощью байесовских доверительных интервалов (credible intervals of Bayesian inference). Исходная матрица может быть транспонирована для определения ассоциаций между пробами по видовому со- ставу или, другими словами, определения фаунистического сходства. Полученные результаты в виде списка ассоциированных видов или проб могут быть сохранены в формате CSV для дальнейшего анализа.
Species association analysis
® All 301 pairs
О 248 positively associated pairs
О 53 negatively associated pairs
Calculate
Method
i Download

Result
Graph Graph analysis
Search:

Graph parameters
S Species names abbreviations
Nodes position method
Kamada-Kawai (fixed)
Nodes clustering method
Multi-level optimization of modularity

Draw and analyze graph
entries
Рис. 1. Интерфейс web-приложения Species association analysis. Цифрами обозначены: 1 – загрузка пользовательского файла, 2 – выбор статистического распределения, 3 – выбор метода определения видовых ассоциаций, 4 – пороговый уровень значимости, 5 – выбор алгоритма расположения (layout) узлов графа, 6 – выбор алгоритма кластеризации узлов графа Fig. 1. Interface of the Species association analysis web application. The numbers indicate: 1 – loading a user file, 2 – choosing a statistical distribution, 3 – choosing a method for determining species associations, 4 – threshold level of significance, 5 – choosing an algorithm for the layout of graph nodes, 6 – choosing an algorithm for clustering graph nodes
Для визуализации результатов анализа с помощью пакета igraph (Csardi, Nepusz, 2006) приложение строит неориен-тируемый граф, где в качестве узлов представлены виды из списка ассоциированных, а в качестве ребер – выявленные ассоциации между ними. Размер маркера узла пропорционален частоте встречаемости вида, а толщина ребра пропорциональна силе свя- зи через настраиваемые коэффициенты (рис. 2).
Взаимное расположение узлов на графе может быть определено с помощью трех алгоритмов (выпадающий список 5 на рис. 1), одним из которых является метод многомерного шкалирования (MDS). Этот метод часто строит не оптимальный с визуальной точки зрения граф, но расположе- ние вершин в нем несет дополнительную информацию о представленных фаунистических комплексах. На рис. 2 в кластер I входят реофильные пресноводные виды, в кластер II – лимнофильные пресноводные, в кластер III – лимнофильные эвригалин-
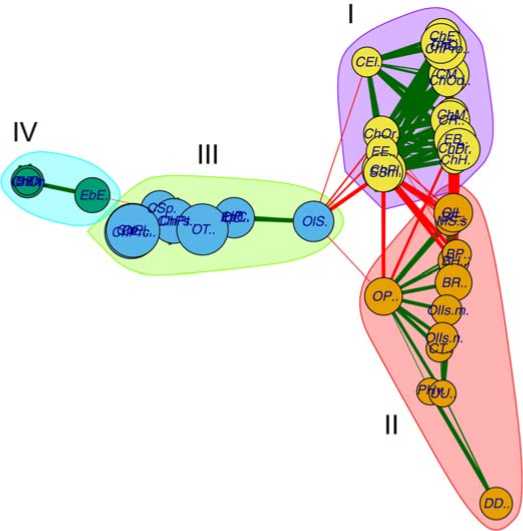
Рис. 2. Граф положительно ассоциированных видов макрозообентоса средних и малых рек бассейна Нижней Волги (Golovatyuk et al., in press). Выделение ассоциаций: биномиальное распределение с 5% верхней критической областью. Расположение вершин методом MDS Fig. 2. Graph of positively associated macrozoobenthos species of medium and small rivers of the Lower Volga basin [Golovatyuk et al. In press]. Identification of associations: binomial distribution with 5% upper critical region. Location of vertices using MDS method
ные, в кластер IV – лимнофильные гало- фильные виды. Таким образом, на графе по оси абсцисс расположен градиент солености воды, а по оси ординат – градиент скорости течения.
Группировка узлов в кластеры также осуществляется с помощью различных алгоритмов, использующих принцип максимизации модулярности графа (выпадающий список 6 на рис. 1). Отдельно можно выделить алгоритм Optimal community structure, добивающийся максимально возможной модулярности средствами целочисленного программирования, но имеющий экспоненциальную сложность (Brandes et al., 2008). Если в визуализацию включены отрицательно ассоциированные виды, задачей алгоритма кластеризации является максимизация отрицательных и минимизация положительных связей между кластерами. Эту задачу успешно решает доработанный алгоритм Affiinity propagation, корректно обрабатывающий отрицательные веса ребер (Traag, Bruggeman, 2008), тогда как остальные алгоритмы считают их нулевыми.
В приведенном рис. 2 кластеризация по- ложительно ассоциированных видов осуществлялась методом многоуровневой оптимизации модулярности (Muli-level optimization of modularity) (Blondel et al., 2008). Кроме указанных выше солености и скорости течения, распределенных по ортогональным осям графа, алгоритм кластеризации учел температуру воды и тип грунта, не имея о них явных данных. Так от кластера I к кластеру IV температура воды в летний период последовательно повышается с 16,9 °C до 27°C, а грунт меняется от гравийно-галечного через заиленный песок и глину до черного ила в кластере IV.
В web-приложении на вкладке «Graph analysis» выделенные кластеры анализируются по плотности связей и их средней силе в кластере. Интегральная характеристика, названная «общей силой связности» (total connectivity strength) объединяет относительное число и среднюю силу связей в кластере в единую нормированную характеристику, позволяющую сравнивать кластеры между собой.
Предложенная методика отличается простотой реализации (для проверки степени ассоциированности видов возможно использование MS Excel 2010+), а также устойчивостью и воспроизводимостью ре
СПИСОК
Список литературы Программа определения и анализа ассоциациированности видов в экологических сообществах
- Blondel V.D., Guillaume J.-L., Lambiotte R., Lefebvre E. Fast unfolding of communities in large networks // Journal of Statistical Mechanics: Theory and Experiment. 2008. P10008. P. 1-12. DOI: 10.1088/1742-5468/2008/10/P10008
- Brandes U., Delling D., Gaertler M., Gorke R., Hoefer M., Nikoloski Z., Wagner D. On Modularity Clustering // IEEE Transactions on Knowledge and Data Engineering. 2008. Vol. 20, iss. 2. P. 172-188. DOI: 10.1109/TKDE.2007.190689
- Chang W., Cheng J., Allaire J., Sievert C., Schloerke B., Xie Y., Allen J., McPherson J., Dipert A., Borges B. shiny: Web Application Framework for R. R package version 1.7.4.1. 2023. [https://CRAN.R-project.org/package=shiny].
- Csardi G., Nepusz T. The igraph software package for complex network research // International journal of complex systems. 2006. Vol. 1695 (5). P. 1-9. [https://igraph.org].
- Golovatyuk L.V., Seleznev D.G., Kurina E.M. Analysis of macrozoobenthos species associations in the eastern european plain under conditions of changing climatic zones // Hydrobiologia. (In press).
- Gotell N.J. Null model analysis of species co-occurrence patterns // Ecology. 2000. Vol. 81, iss. 9. P. 2606-2621. [2606:NMAOSC]2.0.CO;2. DOI: 10.1890/0012-9658(2000)081
- Odum E.P., Barrett G.W. Fundamentals of Ecology. Third edition. Philadelphia: Saunders, 1971. 574 p.
- Traag V.A., Bruggeman J.Community detection in networks with positive and negative links //Physical Review E. 2008. Vol. 80, iss. 3. P036115. DOI: 10.1103/PhysRevE.80.036115


