Разложение хлорбензойных кислот штаммами Rhodococcus ruber P25 и Microbacterium oxydans B51 в условиях кометаболизма
Автор: Егорова Дарья Олеговна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 3, 2022 года.
Бесплатный доступ
Штаммы-деструкторы полихлорированных бифенилов (ПХБ) Rhodococcus ruber P25 и Microbacterium oxydans В51 осуществляют разложение основных интермедиатов аэробной бактериальной деструкции ПХБ - хлорбензойных кислот (ХБК). Установлено, что при культивировании штамма M. oxydans В51 на бифениле в качестве источника углерода происходит увеличение колониеобразующих единиц на четыре порядка за 4 сут., а при культивировании на 2ХБК - на три порядка за 3 сут. Уровень деструкции субстрата при этом составил 99.0-99.5%. При культивировании в условиях кометаболизма (в среде присутствуют ХБК и бифенил) удельная скорость роста штаммов M. oxydans В51 и R. ruber P25 увеличивалась в 1.53-1.58 раза, а эффективность деструкции 2ХБК и 4ХБК составляла 98.9-99.2%. Основные метаболиты бактериальной трансформации бифенила и ХБК в среде не зафиксированы, однако установлено накопление ионов хлора (через 5 сут. культивирования концентрация составляла 64.98-98.05% от максимально возможного). Полученные результаты свидетельствуют об эффективной деструкции ХБК штаммами M. oxydans В51 и R. ruber P25 в присутствии дополнительного источника углерода.
Хлорбензойные кислоты, rhodococcus, microbacterium, бифенил, кометаболизм
Короткий адрес: https://sciup.org/147239672
IDR: 147239672 | УДК: 579.222 | DOI: 10.17072/1994-9952-2022-3-218-225
Текст научной статьи Разложение хлорбензойных кислот штаммами Rhodococcus ruber P25 и Microbacterium oxydans B51 в условиях кометаболизма
For citacion: Egorova D. O. [Degradation of chlorobenzoic acids by strains Rhodococcus ruber P25 and Microbacterium oxydans B51 under cometabolism]. Bulletin of the Perm University. Biology. Iss. 3 (2022): рр. 218-225. (In Russ.).
Хлорбензойные кислоты (ХБК) являются химически стабильными соединениями и оказывают токсический, тератогенный, мутагенный и канцерогенный эффекты при проникновении в организм животных и человека [Федоров, 1993]. Данные свойства ХБК послужили основанием для их включения в перечень вредных и опасных соединений, утвержденный министерством здравоохранения и медицинской промышленности РФ1 Однако производство и применение ХБК не запрещены, в связи с чем хлорбензоаты продолжают использоваться при создании пластмасс, растворителей и некоторых гербицидов. Проникновение ХБК в окружающую среду происходит как с продукцией, так и с отходами производств. Следует отметить, что хлорбензойные кислоты являются основным интермедиатом аэробной бактериальной деструкции полихлорированных бифенилов (ПХБ), что приводит к их накоплению на территориях, загрязнённых ПХБ. Основой биохимической трансформации ПХБ является ферментативный комплекс аэробного окисления незамещенного бифенила. В результате поэтапного действия бифенил 2,3-диоксигеназы, дигидро-дигидрокси-бифенил дегидрогеназы, 2,3-дигидрокси 1,2-диоксигеназы и гидролазы происходит окисление молекулы бифенила/ПХБ до образования бензойной/ХБК кислот [Parales, Resnik, 2006; Kolar et al., 2007; Ilori et al., 2008; Chang et al., 2013; Agulló, 2019; Jia et al., 2019]. Накопление ХБК в среде приводит к ингибированию процесса бактериальной утилизации полихлорированных бифенилов [Statford et al., 1996; Cho et al., 2002].
Бензойная и хлорбензойные кислоты подвержены аэробной бактериальной деструкции, однако их трансформация обусловлена действием специализированных ферментных систем. Начальное окисление бензойной кислоты осуществляет фермент бензоат 1,2-диоксигеназа, кодируемый геном benA [Parales, Resnik, 2006]. Анализ международной базы GenBank показал, что данный ген широко распространен у представителей родов Acinetobacter , Burkholderia, Corynebacterium, Kokuria, Rhodococcus и Pseudomonas , в том числе описан у штаммов-деструкторов ПХБ [Denef, 2006]. Разложение ХБК обусловлено действием различных групп ферментов, кодируемых специфическими генами: 4ХБК – разложение осуществляют ферменты, кодируемые fcb -генами; 3ХБК – генами tfd , cba , cbn , 2ХБК -–генами clc и ohb [Tsoi et al., 1999; Hickey et al., 2001; Providenti, Wyndham, 2001; Radice et al., 2007; Xu et al., 2017]. Результаты проведенного анализа доступных баз данных и литературы свидетельствуют о том, что данные гены менее распространены, чем benA , а у штаммов-деструкторов ПХБ практически не встречаются.
В ранее проведенных исследованиях установлено, что штамм Rhodococcus ruber P25 осуществляет деструкцию ПХБ и 4ХБК, содержит гены деструкции бифенила, а также гены деструкции бензойной кислоты и 4ХБК [Плотникова и др., 2012; Shumkova et al., 2015]. Штамм Microbacterium oxydans В51 осуществляет разложение хлорированных бифенилов и обладает ферментативным комплексом, позволяющим утилизировать 2ХБК [Рыбкина и др., 2003; Solyanikova et al., 2015]. Известно, что в присутствии в среде нескольких источников углерода бактериальные штаммы разлагают наиболее доступный субстрат, а остальные соединения используются для метаболизма по остаточному принципу. В случае штаммов-деструкторов ПХБ, бифенил и образующаяся в результате его окисления бензойная кислота являются предпочтительным субстратом в сравнении с хлорбензойными кислотами. Однако, как было сказано выше, накопление ХБК может иметь негативные последствия для процесса аэробной бактериальной деструкции ПХБ. В связи с этим представляет научный и практический интерес изучение процессов разложения бифенила и ХБК при их одновременном присутствии в среде культивирования штаммов-деструкторов ПХБ.
Цель настоящего исследования ‒ изучить возможность использования хлорбензойных кислот в качестве источника углерода штаммами Rhodococcus ruber P25 и Microbacterium oxydans В51 в присутствии бифенила.
Материалы и методы исследований
Бактериальные штаммы
Штамм Rhodococcus ruber P25 (=ИЭГМ 896) выделен из загрязненной почвы химического производства (г. Пермь, Россия). Характеризуется способностью разлагать широкий спектр ароматических соединений, в том числе полихлорированных бифенилов [Плотникова и др., 2012]. Штамм Microbacterium ox-ydans В51 выделен из почвы, загрязненной отходами соледобывающего производства (г. Березники, Пермский край, Россия), и осуществляет разложение моно-, ди- и трихлорированных бифенилов, а также ряда хлорбензойных кислот [Рыбкина и др., 2003; Solyanikova et al., 2015].
Среда культивирования, реактивы
Минеральная среда К1, состава (г/л): K 2 HPO 4 *3H 2 O ‒ 3.2, NaH 2 PO 4 *2H 2 O ‒ 0.4, (NH 4 ) 2 SO 4 ‒ 0.5, MgSO 4 *7H 2 O ‒ 0.15, Ca(NO 3 ) 2 ‒ 0.01. Для получения плотных питательных сред вносили агар-агар до конечной концентрации 1.5%.
В работе использовали аналитически чистые химические реактивы, бифенил (>98%), 4ХБК (>98%), 2ХБК (>98%) фирмы Sigma-Aldrich (Steinheim, Germany).
Периодическое культивирование, анализ ростовых параметров
Бактериальную культуру помещали в колбы Эрленмейера объемом 250 мл, содержащих 90 мл среды К1. В качестве источника углерода в колбы вносили: бифенил до конечной концентрации 0.5 г/л; 2ХБК – 0.5 г/л; бифенил и 2ХБК или 4ХБК – 0.5 г/л; бифенил и 2ХБК или 4ХБК – 0.75 г/л. Культивирование проводили на термостатируемой круговой качалке (Environmental Shaker-Incubator ES 20/60, “BioSan”, Латвия) при 120 об/мин и температуре 28ºС в течение 5 сут. Каждые 24 ч. измеряли оптическую плотность культуры на спектрофотометре BioSpec-mini (Shimadzu, Япония) при длине волны 600 нм; высевали на агаризованную среду К1 с соответствующим субстратом для подсчета колониеобразующих единиц (КОЕ/мл), а также отбирали образцы культуральной среды для анализа концентрации субстрата и возможных метаболитов. Удельную скорость роста рассчитывали по классической формуле
μ = (LnCx-LnC0)/ (tx-t0), где Cx – концентрация культуры в высшей точке роста, C0 – концентрация культуры в начальный момент роста, t0 и tx – время в начале и в конце логарифмической фазы роста культуры.
Аналитические исследования
Количественный анализ бифенила выполняли с помощью газового хроматографа “Agilent 6890N” с масс-селективным детектором и кварцевой капиллярной колонкой НР-5MS (длина 30 м, диаметр 0.25 мм) (Agilent Technology, США) при программировании температуры согласно [Hernandez et al., 1997].
Расчет содержания бифенила в каждом исследуемом образце осуществляли методом внутренней нормализации. На основании полученных расчетных площадей пиков оценивали содержание бифенила после процесса биодеструкции.
Наличие ХБК, бензойной кислоты и ионов хлора определяли спектрофотометрически и методом ВЭЖХ. Культуральную жидкость очищали от бактериальных клеток центрифугированием (9660 g в течение 3 мин. на центрифуге miniSpin (Eppendorf, Германия). Наличие ионов хлора в супернатанте определяли на спектрофотометре UV-Visible BioSpec-mini (Shimadzu, Япония) при Х макс от 460 до 540 нм через 5 мин. после внесения в супернатант 5%-ного азотнокислого серебра. Количественную оценку проводили согласно калибровочной кривой. Содержание в супернатанте бензойной и хлорбензойных кислот определяли с помощью хроматографа LC-20A (Shimadzu, Япония) с колонкой Discovery C18 (150 х 4.6 мм или 250 х 4.6 мм) (Supelco, Sigma-Aldrich, США) и УФ-детектором при 205 нм. Анализ проводили в системе ацетонитрил : 0.1%-ый Н 3 РО 4 (70:30), идентификацию веществ осуществляли с помощью сравнения времени удержания на колонке исследуемых и стандартных соединений. Количество образовавшихся продуктов оценивали по величине площади и высоты пиков на хроматограмме относительно данных величин стандартных соединений.
Скорость деструкции ХБК
Скорость деструкции субстрата рассчитывали по формуле
V = (С0 – Ci)/(ti – t0), где C0 – концентрация ХБК в начальный момент времени, мг/л, Сi ‒ концентрация ХБК в конечный момент времени, мг/л, ti – конечный момент времени, сут, t0 – начальный момент времени, сут.
Статистический анализ
Все эксперименты проводили в трехкратной повторности. Полученные данные обрабатывали с использованием стандартных пакетов компьютерных программ Microsoft Excel и Statistica 6.0.
Результаты и их обсуждение
Установлено, что штамм M. oxydans В51, эффективно использует в качестве ростового субстрата бифенил и 2ХБК (рис. 1).
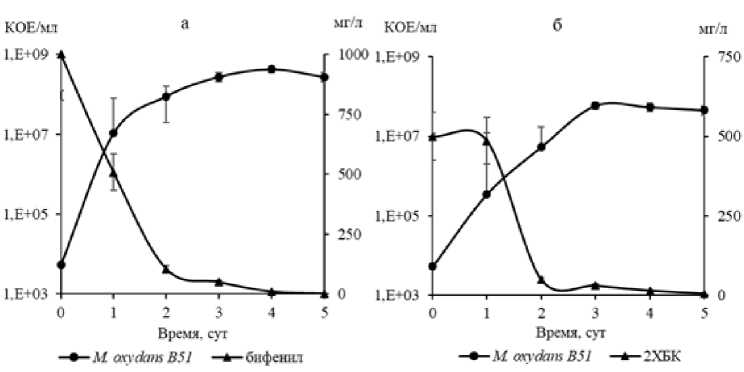
Рис. 1. Рост штамма M. oxydans B51 и изменение концентрации субстрата при культивировании на бифениле (а) и 2ХБК (б)
[Growth of M. oxydans B51 strain and change in substrate concentration during cultivation on biphenyl (a) and 2CBA (б)]
Рост бактериальной культуры коррелировал с убылью субстрата (коэффициент корреляции 0.967– 0.981). Следует отметить, что в обоих случаях в среде не были зафиксированы аналитически значимые количества возможных интермедиатов. На основании полученных данных можно утверждать, что штамм M. oxydans В51 осуществляет полную утилизацию бифенила и 2ХБК без накопления промежуточных соединений в случае, если данные субстраты являются единственным источником углерода.
Ранее было показано, что штамм R. ruber P25 способен расти на бифениле или 4ХБК [Плотникова и др., 2012].
В результате проведенных экспериментов по периодическому культивированию установлено, что штаммы M. oxydans В51 и R. ruber P25 осуществляют разложение 2ХБК и 4ХБК в присутствии в среде бифенила (рис. 2, 3).
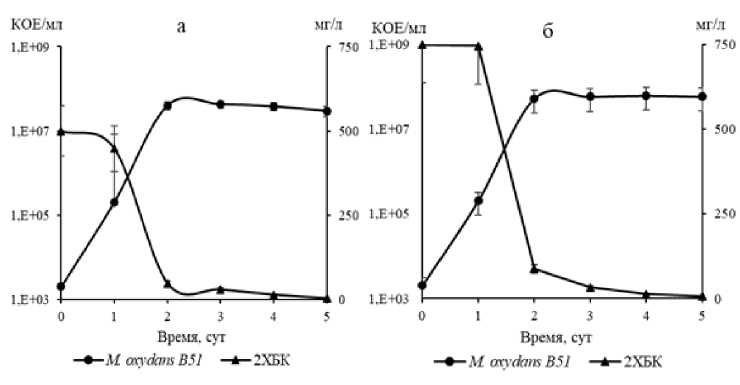
Рис. 2. Рост штамма M. oxydans B51 на бифениле и 2ХБК при начальной концентрации 500 мг/л (а) и 750 мг/л (б)
[Growth of the M. oxydans B51 strain on biphenyl and 2CBA at an initial concentration of 500 mg/L (a) and 750 mg/L (б)]
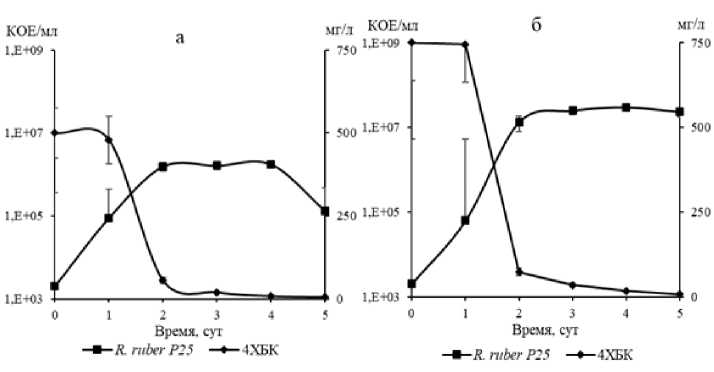
Рис. 3. Рост штамма R. ruber P25 на бифениле и 4ХБК при начальной концентрации 500 мг/л (а) и 750 мг/л (б)
[Growth of the strain R. ruber P25 on biphenyl and 4CBA at an initial concentration of 500 mg/l (a) and 750 mg/l (б)]]
Наблюдалась обратная линейная корреляция между ростом бактериальных культур и изменением концентрации присутствовавших в среде хлорбензойных кислот (коэффициент корреляции составлял 0.989–0.992). Увеличение начальной концентрации 2ХБК в полтора раза не приводило к повышению максимальной плотности культуры штамма M. oxydans B51, тогда как в случае штамма R. ruber P25 аналогичное увеличение концентрации 4ХБК привело к разнице в максимальной концентрации колониеобразующих единиц на один порядок. Эффективность деструкции ХБК в присутствии бифенила составляла 98.9–99.2%. Достоверных различий по данному параметру в случае, когда ХБК являлись единственным источником углерода в ростовой среде, не выявлено. Следует отметить, что несмотря на присутствие в среде бифенила, накопление бензойной кислоты, как его метаболита, зафиксировано не было.
Анализ литературных данных показал, что штаммы M. oxydans B51 и R. ruber P25 осуществляют деструкцию 2ХБК и 4ХБК как в присутствии бифенила, так и без него, эффективнее, чем ранее описанные штаммы-деструкторы ХБК родов Achromobacter , Acinetobacter , Bacillus , Burkholderia , Hydrogenophaga , Microbacterium , Ralstonia , Pseudoaminobacteria , Pseudomonas , Staphylococcus [Ilori et al., 2008; Adebusoye, Miletto, 2011; Xu et al., 2017].
Рост штаммов сопровождался выделением ионов хлора (табл. 1). Высокие значения ионов хлора в среде свидетельствуют об активно протекавшем процессе дехлорирования молекул хлорбензойных кислот.
Таблица 1
Выделение ионов хлора в процессе роста штаммов на ХБК в присутствии бифенила
[Release of chlorine ions during the growth of strains on CBA in the presence of biphenyl]
|
ХБК |
Концентрация, г/л |
Время, сут. |
Концентрация хлора, % |
|
Rhodococcus ruber P25 |
|||
|
4ХБК |
0.5 |
0 |
0 |
|
5 |
98.05 |
||
|
0.75 |
0 |
0 |
|
|
5 |
64.98 |
||
Microbacterium oxydans B51
|
2ХБК |
0.5 |
0 |
0 |
|
2 |
75.5 |
||
|
5 |
83.9 |
||
|
0.75 |
0 |
0 |
|
|
2 |
58.4 |
||
|
5 |
75.5 |
Расчет ростовых параметров показал, что в присутствии бифенила и хлорбензойной кислоты удельная скорость роста штаммов повышается (табл. 2).
При культивировании на одинаковых концентрациях ХБК внесение бифенила в среду приводит к повышению удельной скорости роста бактериальных клеток в 1.53–1.58 раза. Изменение концентрации
ХБК в присутствии бифенила оказывает влияние только на ростовые процессы штамма R. ruber P25, удельная скорость роста которого увеличивается в 1.32 раза. Можно предположить, что присутствие двух субстратов способствует увеличению скорости роста штаммов за счет большей концентрации доступного углерода.
Таблица 2
Удельная скорость роста (сут-1) штаммов M. oxydans B51 и R. ruber P25 [Specific growth rate (day-1) of strains M. oxydans B51 and R. ruber P25]
|
Субстрат |
M. oxydans B51 |
R. ruber P25 |
|
Бифенил (500 мг/л) |
2.81 |
2.30 |
|
ХБК (500 мг/л) |
3.11 |
2.16 |
|
ХБК (500 мг/л) + бифенил (500 мг/л) |
4.94 |
3.32 |
|
ХБК (750 мг/л) + бифенил (500 мг/л) |
5.08 |
4.39 |
Проанализирована скорость деструкции хлорбензоатов исследуемыми штаммами как в присутствии бифенила, так и без внесения бифенила в среду культивирования (табл. 3). Установлено, что наличие бифенила не влияет на скорость деструкции 2ХБК штаммом M. oxydans B51, но оказывает положительный эффект на скорость деструкции 4ХБК штаммом R. ruber P25.
Таблица 3 Скорость деструкции ХБК ((мг/л)сут-1) штаммами M. oxydans B51 и R. ruber P25
[The rate of destruction of CBA ((mg/l) day-1) by strains M. oxydans B51 and R. ruber P25]
|
Субстрат |
M. oxydans B51 |
R. ruber P25 |
|
ХБК |
99.1±0.1 |
87.5±0.1 |
|
ХБК (500 мг/л) + бифенил |
99.3±0.2 |
98.9±0.1 |
|
ХБК (750 мг/л) + бифенил |
148.6±0.2 |
148.5±0.2 |
В работе [Yasir et al., 2021] отмечается, что бифенил может оказывать положительный эффект на би-одеградативную активность штаммов в отношении ПХБ. Однако сведений о стимулирующем эффекте бифенила в отношении бактериальной деструкции ХБК в литературе не выявлено.
Заключение
В результате проведенных исследований установлено, что штаммы M. oxydans B51 и R. ruber P25 используют в качестве источника углерода 2ХБК и 4ХБК соответственно в присутствии в среде дополнительного субстрата – незамещенного бифенила. Анализ ростовых параметров культур показал, что в условиях кометаболизма не происходит снижения эффективности деструкции хлорбензойных кислот. Таким образом, штаммы M. oxydans B51 и R. ruber P25 могут быть использованы в качестве агентов в природоподобных технологиях, направленных на удаление хлорбензоатов из окружающей среды, в том числе в районах, загрязнённых бифенилом и его производными.
Список литературы Разложение хлорбензойных кислот штаммами Rhodococcus ruber P25 и Microbacterium oxydans B51 в условиях кометаболизма
- Плотникова Е.Г. и др. Особенности разложение 4-хлорбифенила и 4-хлорбензойной кислоты штаммом Rhodococcus ruber Р25 // Микробиология. 2012. Т. 81, № 2. С. 159-170. DOI: 10.1134/S0026261712020117.
- Рыбкина Д.О. и др. Новый аэробный грамположительный микроорганизм с уникальными свойствами деструкции орто- и пара-хлорированных бифенилов // Микробиология. 2003. Т. 72, № 6. C. 759765.
- Федоров Л.А. Диоксины как экологическая опасность: ретроспектива и перспективы. М.: Наука, 1993. 266 с.
- Adebusoye S.A., Miletto M. Characterization of multiple chlorobenzoic acid-degrading organisms from pristine and contaminated system: mineralization of 2,4-dichlorobenzoic acid // Bioresource Technology. 2011. Vol. 102. P. 3041-3048. https://doi.org/10.1016/j.biortech.2010.10.026.
- Agullo L. Genetics and biochemistry of biphenyl and PCB biodegradation // Rojo F. (ed.) Aerobic Utilization of Hydrocarbons, Oils, and Lipids. Handbook of Hydrocarbon and Lipid Microbiology. Springer, 2019. P. 595-622.
- Chang Y.-C. et al. Isolation of biphenyl and polychlorinated biphenyl-degrading bacteria and their degradation pathway // Applied Biochemistry and Biotechnology. 2013. Vol. 170. P. 381-398. https://doi.org/10.1007/s12010-013-0191-5.
- Cho Y.-C. et al. Enhancement of microbial PCB dechlorination by chlorobenzoates, chlorophenols and chlorobenzenes // FEMS Microbiology Ecology. 2002. Vol. 42. P. 51-58. https://doi.org/10.1111/j.1574-6941.2002.tb00994.x.
- Denef V.J. Genetic and genomic insights into the role of benzoate-catabolic pathway redundancy in Burkholderia xenovorans LB400 // Applied and Environmental Microbiology. 2006. Vol. 72. P. 585-595. https://doi.org/10.1128/AEM.72.L585-595.2006.
- Hernandez B.S. et al. Terpene-utilizing isolates and their relevance to enhanced biotransformation of poly-chlorinated biphenyls in soil // Biodegradation. 1997. Vol. 8. P. 153-158. https://doi.org/10.1023/A:1008255218432.
- Hickey W.J. et al. Cloning, nucleotide sequencing, and functional analysis of a novel, mobile cluster of biodegradation genes from Pseudomonas aeruginosa strain JB2 // Applied and Environmental Microbiology. 2001. Vol. 67. P. 4603-4609. https://doi.org/10.1128/AEM.67.10.4603-4609.2001.
- Ilori M.O. et al. Aerobic mineralization of 4,4'-dichlorobiphqnyl and 4-chlorobenzoic acid by a novel natural bacterial strain that grows poorly on benzoate and biphenyl // World Journal of Microbiology and Biotechnology. 2008. Vol. 24. P. 1259-1265. https://doi.org/10.1007\s11274-007-9597-y.
- Jia Y. et al. Identification and characterization of a meta-cleavage product hydrolase involved in biphenyl degradation from Arthrobacter sp. YC-RL1 // Applied Microbiol Biotechnology. 2019. Vol. 103, № 16. P. 6825-6836. https://doi.org/10.1007/s00253-019-09956-z.
- Kolar A.B. et al. PCB-degrading potential of aerobic bacteria enriched from marine sediments // International Biodeterioration and Biodegradation. 2007. Vol. 60. P. 16-24. https://doi.org/10.16/jibiod.2006.11.004.
- Parales R.E., Resnick S.M. Aromatic Ring Hydroxylating Dioxygenases // Ramos JL., Levesque R.C. (Eds) Pseudomonas. Boston: Springer, 2006. P. 287-340. https://doi.org/10.1007/0-387-28881-3_9.
- Providenti M.A., Wyndham R.C. Identification and functional characterization of CbaR, a MarR-like modulator of the cbaABC-encoded chlorobenzoate catabolism pathway // Applied and Environmental Microbiology. 2001. Vol. 67. P. 3530-3541. https://doi.org/ 10.1128/AEM.67.8.3530-3541.2001.
- Radice F. et al. Cloning of the Arthrobacter sp. FG1 dehalogenase genes and construction of hybrid pathways in Pseudomonas putida strains // Applied Microbiology and Biotechnology. 2007. Vol. 75. P. 1111-1118. https://doi.org/10.1007/s00253-007-0906-z.
- Shumkova E.S. et al. Draft genome sequence of Rhodococcus ruber strain P25, an active polychlorinated biphenyl degrader // Genome Announcements. 2015. Vol. 3. Article e00990-15. https://doi.org/10.1128/genomeA.00990-15.
- Solyanikova I.P. et al. Peculiarities of the degradation of benzoate and its chloro- and hydroxy-substituted analogs by actinobacteria // International Biodeterioration & Biodegradation. 2015. Vol. 100. P. 155-164. https://doi.org/10.1016/jibiod.2015.02.028.
- Stratford J. et al. Influence of chlorobenzoates on the utilisation of chlorobiphenyls and chlorobenzoate mixtures by chlorobiphenyl/chlorobenzoate-mineralising hybrid bacterial strains // Archives of Microbiology. 1996. Vol. 165. P. 213-218. https://doi.org/10.1007/BF01692864.
- Tsoi T.V. et al. Cloning and expression of the Arthrobacter globiformis KZT1 fcbA gene encoding dehalogenase (4-chlorobenzoate-4-hydroxylase) in Escherichia coli // FEMS Microbiology Letters. 1991. Vol. 81. P. 165-170. https://doi.org/10.1016/0378-1097(91)90298-o.
- Xu C. et al. Degradation of three monochlorobenzoate isomers by different bacteria isolated from a contaminated soil // International Biodeterioration & Biodegradation. 2017. Vol. 120. P. 192-202. https://doi.org/10.1016/jibiod.2017.02.020.
- Yasir M.W. et al. Biotreatment potential of co-contaminants hexavalent chromium and polychlorinated biphenyls in industrial wastewater: individual and simultaneous prospects // Science of the Total Environment. 2021. Vol. 779. Article 146345. https://doi.org/10.1016/j.scitotenv.2021.146345.


