Разработка HybProb молекулярного маркера на аллель L4 для маркер-опосредованной селекции перца сладкого Capsicum spp. на платформе Real-Time
Автор: Гавриш Сергей Федорович, Будылин Михаил Вячеславович, Верба Вадим Михайлович
Журнал: Овощи России @vegetables
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 6 (56), 2020 года.
Бесплатный доступ
Актуальность. Одним из условий создания современного высокотехнологичного гибрида сладкого перца является наличие в нём устойчивости к вирусу мягкой крапчатости (PMMoV), относящемуся к роду Tobamovirus. Данный вирус способен распространяться вместе с семенами, что может приводить к серьезному экономическому ущербу. Основным методом контроля распространения данного заболевания является возделывание устойчивых F1 гибридов перца. На сегодняшний день единственным универсальным источником устойчивости сладкого перца ко всем штаммам PMMoV является аллель гена L - L4. Однако до сих пор не существует надёжной маркерной системы, которая могла бы быть интегрирована в MAS. Методы. На основе данных из базы NCBI нами была сгенерирована молекулярная диагностическая система на основе гибридизационных зондов. Валидация была проведена на семи образцах рода Capsicum из мировых банков гермплазмы. После валидации для тестирования нового маркера использовали расщепляющиеся поколения F2 и F3 ряда селекционных образцов. Результаты. Используя экспериментальную маркерную систему Fret4 нами было протестировано 7 эталонных образцов гермплазмы перца с полным соответствием пиков расплава заявленным аллелям гена L (L0/L1/L3/L4). Далее при помощи маркерной системы был проведён MAS на 10 сортопопуляциях общим числом в 10 тысяч растений для выявления гена L4. В результате нам удалось создать высокоточную и экономически эффективную маркерную систему. Данная маркерная система, основанная на гибридизационных зондах легко интегрируется в MAS, позволяя быстро и надёжно определять аллельное состояние L4 при селекции сладкого перца на устойчивость к Тобамовирусам.
Ген устойчивости
Короткий адрес: https://sciup.org/140250355
IDR: 140250355 | УДК: 635.649:631.52 | DOI: 10.18619/2072-9146-2020-6-67-72
Текст научной статьи Разработка HybProb молекулярного маркера на аллель L4 для маркер-опосредованной селекции перца сладкого Capsicum spp. на платформе Real-Time
2020;(6):67-72.
Relevance. One of the conditions for creating a modern high-tech hybrid of sweet pepper is the presence of resistance to the Pepper mild mottle virus ( PMMoV ). This virus belongs to the genus Tobamovirus . This virus can spread through seeds, causing enormous damage to the sweet pepper crop. The main method of controlling of virus is the cultivation of F 1 hybrids of pepper with genetic resistance. To date, the L4 gene is able to confer resistance to all known strains of this virus. However, there is still no reliable marker system that could be integrated into the MAS.
Methods. Based on data from the NCBI, we generated a molecular diagnostic system based on hybridization probes. Validation was performed on seven pepper accessions from the world's germplasm banks. After validation, the new marker was successfully tested on 2540 plants of F 2 and F 3 generation.
Results. Using the experimental marker system Fret4, we tested 7 reference samples of pepper germplasm with full correspondence of the melt peaks to the declared alleles of the L gene (L0/L1/L3/L4). Then, using a marker system, MAS was carried out on 10 populations of an number of 10 thousand plants to identify the L4 allele. As a result, we were able to create a highly accurate and cost-effective marker system for the most important pepper resistance gene. This marker system based on hybridization probes is easily integrated into the MAS, allowing fast and reliable determination of the L4 allele state in sweet pepper breeding to Tobamovirus.
В ирус мягкой крапчатости перца ( ВМКП, PMMoV ) относится к роду Tobamovirus , наиболее известным представителем которого является вирус табачной мозаики ( TMV) , и считается одним из наиболее опасных вирусных патогенов рода Capsicum spp. [1,2]. Распространен практически во всех частях света: Австралии [3], Северной Америке [4] , Юго-Восточной Азии [5,6], Северной и Восточной Африке [7,8], Украине [9], Европе [10,11], Перу [12]. Ярко выраженные симптомы поражения у растений проявляются на плодах в виде часто расположенных бугорков разного размера более темных по окраске, чем окружающие ткани. Подобные деформации плода и изменения окраски его поверхности заметно снижают товарные качества плодов. При поражении молодых растений может проявляться сильное отставание в росте, вплоть до его полной остановки. Высокая вредоносность вируса мягкой крапчатости обусловлена его природной стойкостью и разнообразием способов переноса с одного растения на другое: на одежде, рабочем инструменте, через поливную систему, зараженную почву, семена, пыльцу [1,13–15]
Помимо ухудшения товарного качества плодов наибольшую проблему вызывает сложность деактивации вируса в зараженных семенах. Для этого ранее предлагались химические и термические способы [16–19]. Показано, что химические способы не являются достаточно эффективными, особенно если обработка проводится позднее, чем через 3 месяца после заготовки семян с плодов, так как за это время вирус успевает проникнуть в глубокие слои семени [16]. Малоэффективной является и тепловая обработка семян из-за высокой температуры деакцтивации вируса – 95°C [20]. Наиболее эффективной в этом случае остается соблюдение мер фитосанитарного контроля и генетическая устойчивость, которая не гарантирует получения безвирусных семян, но позволяет уменьшить масштабы распространения вируса. Так, например, в исследованиях [21], в результате искусственного заражения в устойчивых линиях не были обнаружены вирусные белки, в то время как они были найдены в линиях, не имеющих генетической устойчивости. В результате реакции сверчувствительности было ограничено распространение вируса в клетках устойчивых растений.
В роде Capsicum spp. за устойчивость к тобамови-русам отвечает серия аллелей L1, L2, L3, L4 доминантного гена L, который локализован в теломерной области хромосомы 11 [22]. Аллели L1 и L2 отвечают за устойчивость к вирусу табачной мозаики, вирусу мозаики томата ( ToM V), а также менее распространенным: вирусу крапчатости сладкого перца ( BpeMV ), вирусу зеленой мягкой табачной мозаики ( TMGMVI ) и ряду других. За устойчивость к вирусу мягкой крапчатости, который представлен двумя патотипами P1,2 и
P 1,2,3 [17], отвечают аллели L3 и L4 [23]. Генотипы, содержащие аллель L3, устойчивы к патотипу P 1,2 , но восприимчивы к P 1,2,3 . Аллель L4 дает устойчивость к обоим патотипам, а также к вирусу табачной мозаики, вирусу мозаики томата и ряду других вирусов рода Tobamovirus [1,14]. Проявление устойчивости, обусловленной наличием этих аллелей в генотипе растения, характеризуется реакцией сверхчувствительности, приводящей к быстрой гибели поражённых клеток [12]. Впервые аллель L4 был обнаружен в линии PI 260429 ( Capsicum chacoense Hunz.) в 1982 году [23], а первый коммерческий гибрид перца F 1 Himes с геном L4 создан в 1998 году [24]. Несмотря на то, что штаммы вируса мягкой крапчатости, которые способны поражать устойчивые L4 растения были обнаружены в Японии [25] и Израиле [26], в настоящее время аллели L3 и L4 широко используются в селекционных программах по всему миру [10]. Полагая, что генетическая вариативность вируса мягкой крапчатости не очень высокая даже при сравнении шатммов из разных регионов мира [6] и широкий спектр устойчивостей к большинству вирусов из рода Tobamovirus , который дает аллель L4, позволяет говорить о необходимости его интрогрессии в селекционные линии для получения гибридов F 1 с комплексной устойчивостью к Тобамовирусам.
При ведении классической селекции, основанной на фенотипических признаках, основной трудностью является идентификация и фиксация аллели L4 в гомозиготном состояния в расщепляющихся популяциях. Очевидно, что использование искусственного заражения — довольно опасный (с фитосанитарной точки зрения) способ, так как деактивировать вирус в семенах полученных линий не представляется возможным. Единственным способом в этом случае является использование молекулярных маркеров.
Оптимальным инструментом для интрогрессии аллели L4 в родительские линии, используемые для производства F 1 гибридов, является маркер-опосре-дованная селекция (MAS). Однако для того, чтобы потенциал этого метода использовался в полной мере, требуется разработка маркерной системы, которая одновременно была бы надёжной и имела формат, пригодный для высокопропускной платформы Real-Time ПЦР. Ранее для идентификации аллели L4 использовались различные молекулярные маркеры для агарозного и полиакриламидного гелей, такие как RAPD [10,14], ALFP [1], CAPS и SCAR [27]. Маркеры, основанные на SNP мутациях, были разработаны корейскими учёными [28]. Мы разработали абсолютно новый молекулярный маркер, основанный на SNP мутации, детектируемой посредством гибридизационных зондов HybProb, полностью совместимых с высокопропускным анализом на платформе Real-Time ПЦР.
Таблица 1. Перечень линий, использованных для валидации маркера Table 1. List of lines used for marker validation
|
Таксономическая группа |
Каталожный номер |
Название линии |
Аллели L |
|
C. chacoense Hunz. |
CGN23208 |
SA 185 |
L4 |
|
C. chacoense Hunz. |
CGN21477 |
PI 260429 |
L4 |
|
C. chacoense Hunz. |
CGN22155 |
PI 213917 |
L3 |
|
C. chacoense Hunz. |
CGN19196 |
I 7208 |
L3 |
|
C. frutescens L. |
CGN21546 |
Tabasco |
L2 |
|
C. annuum L. |
CGN19226 |
Mospa |
L1 |
|
C. annuum L. |
CGN21472 |
Alwin |
L0 |
Материалы и методыСелекционный материал
На первом этапе данной работы в качестве основных контролей были использованы линии, предоставленные Нидерландским центром генетических ресурсов (Netherlands Centre of Genetic Resource, CGN), указанные в таблице 1.
Дальнейшее тестирование маркеров проводилось на селекционном материале, полученном научными сотрудниками НИИСОК. Устойчивые и неустойчивые контроли дополнительно тестировались на устойчи-вость/восприимчивость путем искусственного заражения с использованием линий-дифферециаторов, приведенных в таблице 1. Линия Л-B4694-911 использовалась в качестве контроля с аллелью L4 в гомозиготном состоянии. Данная линия была получена в 2007-2012 гг путем многократного заражения расщепляющейся по аллели L4L4 селекционной сортопопуля-ции B4694 ( C. annuum ) штаммом вируса мягкой крапчатости перца PV-0093 (DSMZ). Линия Л-Белозерка использовалась в качестве неустойчивого контроля с аллелью (L0L0) в гомозиготном состоянии. Гибридная комбинация F 1 Л-B4694-911 х Л-Белозерка — в качестве устойчивого контроля с гетерозиготным состоянием аллели L4 (L4L0) (рис. 2). Для скрининга использовались селекционные сортопопуляции различных поколений (F 3 -F 4 и линии), расщепляющиеся по аллели L4: F 3 Центурион, F 4 Эмбери, полученные с помощью искусственного заражения линии ((Дименций х 05СПЕ4609)-2)-3121 и ((Дименций х 05СПЕ4609)-2)-3212. В каждом селекционном образце анализировали 96 растений, каждое растение – индивидуально.
Для демонстрации работы маркера Fret4 (табл. 3) использовали селекционный материал трех сортоти-пов: Венгерский (конусовидная (треугольная) форма плода), Блочный (прямоугольная форма плода), Капия (узкотреугольная форма плода). Использовали селекционные линии поколений F 3 -F 5 . В качестве контролей применяли: Л-Белозерка, Бутуз – неустойчивые конт-роли, линия ((Дименций х 05СПЕ4609)-2)-3121, ((Дименций х 05СПЕ4609)-2)-3212, а также ряд гибридов иностранного происхождения. Все растения выращивали в пленочных теплицах.
Тестирование растений проводили дважды:
-
1. в начале вегетационного периода (май) – все растения в каждом селекционном номере
-
2. в конце вегетационного периода (сентябрь) – только отобранные селекционером растея, у которых по результатам ПЦР анализа было гетерозиготное (L4L0) или гомозиготное ( L4L4) состояние аллели L4.
Выделение ДНК
Выделение ДНК проводили при помощи усовершенствованных методик [29,30]. 0,2 г образца было гомогенизировано с 300 мкл экстракционного буфера (0,4 М LiCl, 25mM EDTA, 0,2 M Tris–HCL (pH 9), 2,5 % PVP-40) на гомогенизаторе Tissue Lyser II (Qiagen, Germany). Затем следовала очистка хлороформ/изо-амилом 24/1 с последующим улавливанием ДНК при помощи диоксида кремния (силика) с добавлением 96% этанола и 6M NaI. Далее силику осаждали, промывали 70% спиртом и элюировали ДНК в MQ воду.
Дизайн маркерной системы
В качестве основы для дизайна маркерной системы были использованы последовательности аллелей L сладкого перца из базы данных NCBI [31] (L3 AB523370; L1 AB523372; L1a, AB523373; L1c, AB523374; L2 AB523375; L2b AB523376; L4 AB523377) [32]. На основе последующего биоинформатического анализа аллелей L были выбраны однонуклеотидные полиморфизмы для создания маркерных систем на основе гибридизационных зондов.
ПЦР амплификация
ПЦР реакционная смесь была рассчитана на 10 мкл 1 х буфер, 2 mM MgCl 2 , 0,1 mM dNTPs, 0,5 U Taq полимераза (Fermentas, Waltham, MA, USA) 3–10 μM праймеров и 10 нг ДНК. Условия реакции были следующие: первичная денатурация 95 °C 5 мин, с последующими 40 циклов 95 °C 10 сек, 62 °C 15 сек, и 72 °C 5 сек. Затем проводилось плавление ПЦР продукта при следующих условиях 95 °C 1 мин, 42 °C 2 с поднятием температуры 0,04 °C / сек, до 95 °C, проводя снятие флуоресценции по 5 раз/°C. Амплификация проводилась на приборе LightCycler 480 II. Обработка результатов проводилась при помощи программного обеспечения LightCycler® 480 SW 1.5.1.
Результаты
В ходе проведённого исследования нами был создан кодоминантный маркер, дискриминирующий аллель L4 из всех аллельных вариантов гена L. В настоящее время Fret4 не имеет аналогов среди маркеров на аллель L4, упоминаемых в иностранных источниках. В своей статье учёные из Турции [10] указали, что при валидации всех доступных на тот момент маркеров на L4, достоверно информативным оказался только доминантный маркер AP-7/AP-8 [14]. Однако данный маркер является доминантным и не предоставляет информации об остальных аллелях Ln, что делает его малопригодным для использования в MAS. Другие маркеры, основанные на SNP, в том числе представленные корейскими учёными [28] не обнару-
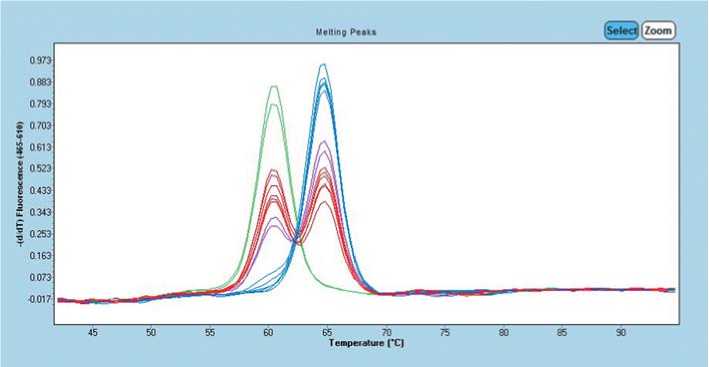
Рис. 1. Melting curve analysis с маркерной системой Fret4, расщепляющейся по аллели L4 тестовой сортопопуляции C. аnnuum L. в поколении F 2 . Пик расплава для аллели L4 располагается на 65°C (синий цвет), пик расплава всех остальных аллелей гена L находится на 60°C (зелёный цвет), сочетание обоих пиков расплава на 60°C и 65°C (красный цвет) соответствует гетерозиготе между аллелью L4 и любой другой аллелью гена L
Fig.1. Melting curve analysis with the Fret4 marker system, splitting at the L4 allele of the test variety population C. annuum L. in the F 2 generation
Таблица 2. Результаты тестирования маркера Fret4 на селекционном материале, 2014 год Table 2. Results of testing the Fret4 marker on breeding material, 2014
|
Название селекционного образца |
Поколение |
Аллельное состояние гена L на момент начала скрининга |
Число проанализированных образцов |
Результат скрининга, аллельное состояние гена/число растений |
|
Л-Белозерка (контроль) |
Лин |
L0L0 |
96 |
L0L0 – 96 |
|
Л-B4694-911 (контроль) |
Лин |
L4L4 |
96 |
L4L4 – 96 |
|
F1 B4694-911 х Л-Белозерка (контроль) |
F 1 |
L4L0 |
96 |
L4L0 – 96 |
|
Центурион |
F 3 |
L? |
1920 (20 семей х 96 растений) |
L4L0 – 1054 L0L0 – 468 L4L4 – 398 |
|
Эмбери |
F 4 |
L? |
621 (9 семей х 96 растений) |
L4L4 – 542 L4L0 – 54 L0L0 – 25 |
|
((Дименций х 05СПЕ4609)-2)-3121 |
Лин |
L? |
96 |
L4L0 – 30 L0L0 – 21 L4L4 – 45 |
|
((Дименций х 05СПЕ4609)-2)-3212 |
Лин |
L? |
96 |
L4L4 – 96 |

AB

C
D
Рис. 2. Пораженный вирусом мягкой крапчатости неустойчивый контроль Л-Белозерка (слева) и реакция сверхчувствительности на устойчивой линии (справа) (А), устойчивый гибрид F 1 B4694-911 х Л-Белозерка (контроль) (Tm:0-3) (B), устойчивая линия ((Дименций х 05СПЕ4609)-2)-3121 (С) и линия ((Дименций х 05СПЕ4609)-2)-3212 (D)
Fig.2. Unstable control L-Belozerka infected with virus (left) and hypersensitivity reaction on the resistant line (right) (A), resistant hybrid F 1 B4694-911 x L-Belozerka (control) (Tm:0-3) (B), resistant line ((Dimension x 05SPE4609) -2) -3121 (C) and line ((Dimension x 05SPE4609) -2) -3212 (D)
Таблица 3. Результаты использования маркера Fret4 на селекционном материале, 2020 год Table 3. Results of using the Fret4 marker on breeding material, 2020
|
Пленочные теплицы (1 этап отбора) |
Всего образцов протестировано |
Образцов с аллелью L4 (R1 и H2) после массового скрининга |
|
Блочный+Ламуйо |
364 |
277 |
|
Венгерский |
134 |
73 |
|
Капия |
5 |
0 |
|
Пленочные теплицы (2 этап отбора) |
Всего образцов протестировано |
Образцов с аллелью L4 (R и H) после визуальной оценки |
|
Блочный+Ламуйо |
277 |
H – 55 R – 23 |
|
Венгерский |
73 |
H – 13 R – 13 |
|
Капия |
0 |
0 |
1 — устойчивый генотип с гомозиготным состоянием аллелей L4L4
2 — устойчивый генотип с гетерозиготным состоянием аллелей L4L0
жили заявленной корреляции с L4 при проверке на линиях-дифференциаторах и были отвергнуты при выборе мишени для генерации маркерной системы. В свою очередь результаты, полученные с применением маркера Fret4, показали, что он может быть широко использован в селекционных программах с целью успешного мониторинга интрогрессии L4 на каждом этапе линейной и беккросной селекции.
После успешной валидации на линиях-дифференциаторах, маркер Fret4 был протестирован при массовом скрининге селекционных популяций различных поколений (F 3 -F 6 и линии) C. annuum L., расщепляющихся по аллели L4. В общей сложности был проведен скрининг 10 сортопопуляций и линий (более 10 тыс. растений) Результаты скрининга некоторых наиболее ценных из них приведены в таблице 2.
Как видно из таблицы 2 маркер Fret4 позволяет контролировать аллельное состояние гена в каждом поколении и дифференцировать все растения на устойчивые и неустойчивые. Он подтвердил свою работоспособность при анализе контролей с различным состоянием аллеля L4 (Л-Белозерка, Л-B4694-911, F 1 B4694-911 х Л-Белозерка), а также позволил уточнить данные для вышеперечисленных образцов и определить дальнейшую стратегию селекции для каждого из этих образцов.
В результате скрининга нами было установлено, что в линии ((Дименций х 05СПЕ4609)-2)-3212 аллель L4 находится в гомозиготном состоянии. По результатам скрининга были отобраны устойчивые растения в других селекционных образцах.
Результаты использования маркера в условиях практической селекционной работы приведены в таблице 3.
На первом этапе на молодых растениях до момента начала визуальной оценки селекционеры проводили массовый скрининг всех линий F5-F6, и результаты заносились в селекционный журнал. На втором этапе работы – отбирали визуально лучшие растения, не зная результатов ПЦР анализа (слепой метод), а затем полученные результаты сопоставлялись и корректировались с учетом полученных данных ПЦР анализа. Такая методика оценки связана с некоторыми особенностями устойчивых линий, у которых могут про- являться ненужные с точки зрения селекции признаки. Поэтому в дальнейшей селекционой работе приоритет будут иметь устойчивые линии (L4L4 и L4L0), отобранные на основании визуальной оценки.
Обсуждение
После дальнейшей доработки линии (табл. 2) были включены в селекционную работу. В 2014 году на основе устойчивой линии ((Дименций х 05СПЕ4609)-2)-3212 был получен первый гибрид F 1 64-13 (Tm:0-3), который в настоящее время продается в Японию. В 2016-2017 годах на основе линий ((Дименций х 05СПЕ4609)-2)-3121 получены гибриды блочного типа F 1 255-16 и F 1 444-17 с зеленой окраской плода в технической спелости, F 1 240-16 – с желто-белой окраской плода в технической спелости, которые в настоящее время проходят испытания в открытом и защищенном грунте Крымского селекционного центра компании Гавриш, а также в ряде фермерских хозяйств и тепличных комбинатов. В 2019 году на основе устойчивой линии Центурион-1422 (получена из Центурион F 3 ) получены первые гибриды венгерского типа, и в 2020 году проведено их предварительное сортоиспытание.
Линии, полученные из F 4 Эмбери (сортотип Блочный), имеющие крупные плоды кубовидной формы (до 200 г) с темно-зеленой окраской в технической и красной в биологической спелости будут использованы при создании гибридов, предназначенных для выращивания в пленочных теплицах таких стран как Турция и Иордания, а также ряда европейских стран.
Выводы
В результате работы была подтверждена надёжность маркера Fret4 и его воспроизводимость. По результатам проведённого исследования можно судить о том, что нами была создана инновационная маркерная система, подходящая для маркер-опосре-дованной селекции на платформе Real-Time ПЦР. Помимо информативной составляющей, использование маркера Fret4 является экономически выгодным в сравнении со всеми имеющимися аналогами. Его применение не требует трудоёмких манипуляций с агарозным гелем. Сама концепция формата анализа с использованием гибридизационных зондов позволяет удешевить стоимость синтеза флуоресцентных олигов, затрачиваемых на детекцию аллельного состояния гена, таких как системы гидролизных зондов TaqMan или же KASP (LGC Genomics).
Сочетание результатов визуальной оценки селекционного материала с данными полученными с помощью маркера Fret4 начиная с поколения F2 позволяет принимать решения о целесообразности дальнейшей работы с каждым конкретным селекционным образцом: увеличивать объем изучаемого материала в расщепляющихся по аллелю L4 ценных популяциях и уменьшать – в случае гомозиготного состояния аллеля и высокой общей оценки сортопопуляции, концентрируясь на стабилизации хозяйственно ценных признаков (урожайность, качество плода); исключать из работы неустойчивые образцы; уделять большее внимание устойчивым семьям в поколении F3; принимать решение о включении селекционного образца в работу по определенному направлению, требующему наличие гена устойчивости. Молекулярный анализ селекционного материала с помощью маркера Fret4 позволяет оптимизировать площади, занятые под селекцию и избежать трудоемкого и небезопасного с фитосани-тарной точки зрения процесса искусственного заражения растений, вести контролируемую в каждом поколении селекцию по данному гену с тем чтобы гарантировано получить устойчивые линии к пятому-шестому поколению. На этапе активного жизненного цикла гибрида F1 регулярный скрининг родительских линий позволяет поддерживать гомозиготность по аллелю L4 .
Об авторах:
-
• Литература / References
-
1. Kim H.J., Han J.-H., Yoo J.H., Cho H.J., Kim B.-D. Development of a Sequence Characteristic Amplified Region Marker linked to the L4 Locus Conferring Broad Spectrum Resistance to Tobamoviruses in Pepper Plants. Molecules & Cells (Springer Science & Business Media BV). 2008;25(2).
-
2. Scholthof K.-B.G. et al. Top 10 plant viruses in molecular plant pathology. Molecular plant pathology. Wiley Online Library, 2011;12(9): 938–954.
-
3. Pares R.D., Gunn L.V. The role of non-vectored soil transmission as a primary source of infection by pepper mild mottle and cucumber mosaic viruses in glasshouse-grown capsicum in Australia. Journal of Phytopathology. Wiley Online Library, 1989;126(4):353–360.
-
4. Adkins S., Lamb E.M., Roberts P.D., Gooch M.D., Breman L., Shuler K.D. Identification of Pepper mild mottle virus in commercial bell pepper in Florida. Plant disease. Am Phytopath Society, 2001;85(6):679–679.
-
5. Kenyon L., Kumar S., Tsai W.-S., Hughes J. d’A. Virus diseases of peppers (Capsicum spp.) and their control. Adv Virus Res. 2014;(90):297–354.
-
6. Yu M., Zhou T., Wu Y., An M. Complete genome sequence of a pepper mild mottle virus isolate from northeast China. Genome announcements. Am Soc Microbiol, 2018;6(9).
-
7. Mnari-Hattab M., Ezzaier K. Biological, serological, and molecular characterization of Pepper mild mottle virus (PMMoV) in Tunisia. Tunisian Journal Plant Protection. 2006;(1):1–12.
-
8. Waweru B.W., Kilalo D.C., Miano D.W., Kimenju J.W., Rukundo P. Diversity and economic importance of viral diseases of pepper (Capsicum spp.) in Eastern Africa. Journal of Applied Horticulture. 2019;21(1).
-
9. Shamraichuk V., Shevchenko T., Bysov A., Emad A. Novel tobamovirus isolated from vegetable crops in Ukraine. Bulletin of Taras Shevchenko National University of Kyiv. 2013;65(3).
-
10. Özkaynak E., Devran Z., Kahveci E., Doğanlar S., Başköylü B., Doğan F., İşleyen M., Yüksel A., Yüksel M. Pyramiding multiple genes for resistance to PVY, TSWV and PMMoV in pepper using molecular markers. Verlag Eugen Ulmer, 2014.
-
11. Larregla S., Ojinaga M. Evaluation of PMMoV resistance in a pseudoF2 generation for the introgression of L4 resistance gene in sensitive pepper landraces of northern Spain. APSNET , 2020.
-
12. Vélez-Olmedo J.B., Fribourg C.E., Melo F.L., Nagata T., de Oliveira A.S., Resende R.O. Pepper mild mottle virus isolates from Peru induce severe symptoms in susceptible pepper plants and belong to the P 1, 2 pathotype. Tropical Plant Pathology. Springer, 2020. P.1–5.
-
13. Salamon P., Kaszta M. Investigation on the transmission of some tobamoviruses by pollen and seed in pepper ( Capsicum annuum L.). International Journal of Horticultural Science. 2000;6(3):127–131.
-
14. Matsunaga H., Saito T., Hirai M., Nunome T., Yoshida T. DNA markers linked to Pepper mild mottle virus ( PMMoV ) resistant locus (L4) in Capsicum. Journal of the Japanese Society for Horticultural Science. The Japanese society for horticultural science, 2003;72(3):218–220.
-
15. Choi G.-S., Kim J.-H., Kim J.-S., Kim H.-R. Tobamoviruses of green peppers
growing on hydroponic systems. Research in Plant Disease. The Korean Society of Plant Pathology, 2004;10(3):194–197.
-
-
16. Demski J.W. Tobacco mosaic virus in seedborne in pimiento peppers. Plant Disease. 1981;65(9):723–724.
-
17. Rast A.T.B. Pepper tobamoviruses and pathotypes used in resistance breeding. Capsicum Newslett. 1988;(7)”20–23.
-
18. Wetter C., Conti M., Altschuh D., Tabillion R., Van Regenmortel M.H.V. Pepper mild mottle virus, a tobamovirus infecting pepper cultivars in Sicily. Phytopathology. St. Paul, 1984;74(4):405–410.
-
19. Svoboda J., Červená G., Rodová J., Jokeš M. First report of pepper mild mottle virus in pepper seeds produced in the Czech Republic. Plant Protection Science. Institute of Agricultural and Food Information, 2006;42(1):34–37.
-
20. ICTV. Virus taxonomy: 2014 release. ICTV London , 2014.
-
21. Hsu C., Lin S., Wang Y., Chan Y., Lee L., Barchenger D.W., Kenyon L., Kumar S. Resistance to Tomato mosaic virus (ToMV)in sweet pepper ( Capsicum annuum L.). XXX International Horticultural Congress IHC2018: II International Symposium on Plant Breeding in Horticulture 1282. 2018. P.31–36.
-
22. Chaim A.B., Grube R.C., Lapidot M., Jahn M., Paran I. Identification of quantitative trait loci associated with resistance to cucumber mosaic virus in Capsicum annuum. Theor Appl Genet. 2001;102(8):1213–1220.
-
23. Boukema I.W. Resistance to TMVin Capsicum chacoense Hunz.is governed by allele of the L-locus. Capsicum Newsl. 1984;(3):47–48.
-
24. Sagi Z.S., Salamon P. Breeding white, sweet pepper hybrids armed with the tobamovirus resistance allele L4. Avignon, France: INRA, 1998. P.173.
-
25. Genda Y., Kanda A., Hamada H., Sato K., Ohnishi J., Tsuda S. Two amino acid substitutions in the coat protein of Pepper mild mottle virus are responsible for overcoming the L4 gene-mediated resistance in Capsicum spp. Phytopathology. 2007;97(7):787–793.
-
26. Antignus Y., Lachman O., Pearlsman M., Maslenin L., Rosner A. A New Pathotype of Pepper mild mottle virus (PMMoV) Overcomes the L4 Resistance Genotype of Pepper Cultivars. Plant Dis. 2008;92(7):1033–1037.
-
27. Lee J.-D., Han J.-H., Yoon J.-B. A set of allele-specific markers linked to L locus resistant to Tobamovirus in Capsicum spp. Horticultural Science & Technology. Korean Society of Horticultural Science, 2012;30(3):286–293.
-
28. Yang H.-B., Liu W.Y., Kang W.-H., Jahn M., Kang B.-C. Development of SNP markers linked to the L locus in Capsicum spp. by a comparative genetic analysis. Molecular breeding. Springer, 2009;24(4):433.
-
29. Hong Y.K. DNA extraction conditions from Porphyra perforata using LiCl. Journal of Applied Phycology. 1995;7(2):101–107.
-
30. Foissac X. Polyvalent detection of Fruit tree Tricho. Capillo and Res. 2001;(45):33–35.
-
31. National Center for Biotechnology Information. URL: https://www.ncbi.nlm.nih.gov/ (accessed: 14.10.2020).
-
32. Tomita R., Sekine K.-T., Mizumoto H., Sakamoto M., Murai J., Kiba A., Hikichi Y., Suzuki K., Kobayashi K. Genetic basisfor the hierarchical interaction between Tobamovirus spp. and L resistance gene alleles from different pepper species. Molecular plant-microbe interactions. Am Phytopath Society, 2011;24(1):108–117.
Список литературы Разработка HybProb молекулярного маркера на аллель L4 для маркер-опосредованной селекции перца сладкого Capsicum spp. на платформе Real-Time
- Kim H.J., Han J.-H., Yoo J.H., Cho H.J., Kim B.-D. Development of a Sequence Characteristic Amplified Region Marker linked to the L4 Locus Conferring Broad Spectrum Resistance to Tobamoviruses in Pepper Plants. Molecules & Cells (Springer Science & Business Media BV). 2008;25(2).
- Scholthof K.-B.G. et al. Top 10 plant viruses in molecular plant pathology. Molecular plant pathology. Wiley Online Library, 2011;12(9): 938-954.
- Pares R.D., Gunn L.V. The role of non-vectored soil transmission as a primary source of infection by pepper mild mottle and cucumber mosaic viruses in glasshouse-grown capsicum in Australia. Journal of Phytopathology. Wiley Online Library, 1989;126(4):353-360.
- Adkins S., Lamb E.M., Roberts P.D., Gooch M.D., Breman L., Shuler K.D. Identification of Pepper mild mottle virus in commercial bell pepper in Florida. Plant disease. Am Phytopath Society, 2001;85(6):679-679.
- Kenyon L., Kumar S., Tsai W.-S., Hughes J. d'A. Virus diseases of peppers (Capsicum spp.) and their control. Adv Virus Res. 2014;(90):297-354.
- Yu M., Zhou T., Wu Y., An M. Complete genome sequence of a pepper mild mottle virus isolate from northeast China. Genome announcements. Am Soc Microbiol, 2018;6(9).
- Mnari-Hattab M., Ezzaier K. Biological, serological, and molecular characterization of Pepper mild mottle virus (PMMoV) in Tunisia. Tunisian Journal Plant Protection. 2006;(1):1-12.
- Waweru B.W., Kilalo D.C., Miano D.W., Kimenju J.W., Rukundo P. Diversity and economic importance of viral diseases of pepper (Capsicum spp.) in Eastern Africa. Journal of Applied Horticulture. 2019;21(1).
- Shamraichuk V., Shevchenko T., Bysov A., Emad A. Novel tobamovirus isolated from vegetable crops in Ukraine. Bulletin of Taras Shevchenko National University of Kyiv. 2013;65(3).
- Özkaynak E., Devran Z., Kahveci E., Doganlar S., Baçkoylü B., Dogan F., îçleyen M., Yüksel A., Yüksel M. Pyramiding multiple genes for resistance to PVY, TSWV and PMMoV in pepper using molecular markers. Verlag Eugen Ulmer, 2014.
- Larregla S., Ojinaga M. Evaluation of PMMoV resistance in a pseudoF2 generation for the introgression of L4 resistance gene in sensitive pepper landraces of northern Spain. APSNET, 2020.
- Vélez-Olmedo J.B., Fribourg C.E., Melo F.L., Nagata T., de Oliveira A.S., Resende R.O. Pepper mild mottle virus isolates from Peru induce severe symptoms in susceptible pepper plants and belong to the P 1, 2 pathotype. Tropical Plant Pathology. Springer, 2020. P.1-5.
- Salamon P., Kaszta M. Investigation on the transmission of some tobamoviruses by pollen and seed in pepper (Capsicum annuum L.). International Journal of Horticultural Science. 2000;6(3):127-131.
- Matsunaga H., Saito T., Hirai M., Nunome T., Yoshida T. DNA markers linked to Pepper mild mottle virus (PMMoV) resistant locus (L4) in Capsicum. Journal of the Japanese Society for Horticultural Science. The Japanese society for horticultural science, 2003;72(3):218-220.
- Choi G.-S., Kim J.-H., Kim J.-S., Kim H.-R. Tobamoviruses of green peppers growing on hydroponic systems. Research in Plant Disease. The Korean Society of Plant Pathology, 2004;10(3):194-197.
- Demski J.W. Tobacco mosaic virus in seedborne in pimiento peppers. Plant Disease. 1981;65(9):723-724.
- Rast A.T.B. Pepper tobamoviruses and pathotypes used in resistance breeding. Capsicum Newslett. 1988;(7)"20-23.
- Wetter C., Conti M., Altschuh D., Tabillion R., Van Regenmortel M.H.V. Pepper mild mottle virus, a tobamovirus infecting pepper cultivars in Sicily. Phytopathology. St. Paul, 1984;74(4):405-410.
- Svoboda J., Červená G., Rodová J., Jokeš M. First report of pepper mild mottle virus in pepper seeds produced in the Czech Republic. Plant Protection Science. Institute of Agricultural and Food Information, 2006;42(1):34-37.
- ICTV. Virus taxonomy: 2014 release. ICTVLondon, 2014.
- Hsu C., Lin S., Wang Y., Chan Y., Lee L., Barchenger D.W., Kenyon L., Kumar S. Resistance to Tomato mosaic virus (ToMV) in sweet pepper (Capsicum annuum L.). XXX International Horticultural Congress IHC2018: II International Symposium on Plant Breeding in Horticulture 1282. 2018. P.31-36.
- Chaim A.B., Grube R.C., Lapidot M., Jahn M., Paran I. Identification of quantitative trait loci associated with resistance to cucumber mosaic virus in Capsicum annuum. Theor Appl Genet. 2001;102(8):1213-1220.
- Boukema I.W. Resistance to TMV in Capsicum chacoense Hunz. is governed by allele of the L-locus. Capsicum Newsl. 1984;(3):47-48.
- Sagi Z.S., Salamon P. Breeding white, sweet pepper hybrids armed with the tobamovirus resistance allele L4. Avignon, France: iNRa, 1998. P.173.
- Genda Y., Kanda A., Hamada H., Sato K., Ohnishi J., Tsuda S. Two amino acid substitutions in the coat protein of Pepper mild mottle virus are responsible for overcoming the L4 gene-mediated resistance in Capsicum spp. Phytopathology. 2007;97(7):787-793.
- Antignus Y., Lachman O., Pearlsman M., Maslenin L., Rosner A. A New Pathotype of Pepper mild mottle virus (PMMoV) Overcomes the L4 Resistance Genotype of Pepper Cultivars. Plant Dis. 2008;92(7):1033-1037.
- Lee J.-D., Han J.-H., Yoon J.-B. A set of allele-specific markers linked to L locus resistant to Tobamovirus in Capsicum spp. Horticultural Science & Technology. Korean Society of Horticultural Science, 2012;30(3):286-293.
- Yang H.-B., Liu W.Y., Kang W.-H., Jahn M., Kang B.-C. Development of SNP markers linked to the L locus in Capsicum spp. by a comparative genetic analysis. Molecular breeding. Springer, 2009;24(4):433.
- Hong Y.K. DNA extraction conditions from Porphyra perforata using LiCl. Journal of Applied Phycology. 1995;7(2):101-107.
- Foissac X. Polyvalent detection of Fruit tree Tricho. Capillo and Res. 2001;(45):33-35.
- National Center for Biotechnology Information. URL: https://www.ncbi.nlm.nih.gov/ (accessed: 14.10.2020).
- Tomita R., Sekine K.-T., Mizumoto H., Sakamoto M., Murai J., Kiba A., Hikichi Y., Suzuki K., Kobayashi K. Genetic basis for the hierarchical interaction between Tobamovirus spp. and L resistance gene alleles from different pepper species. Molecular plant-microbe interactions. Am Phytopath Society, 2011;24(1):108-117.


