Разработка и валидация SNP-панели низкой плотности для характеристики генетического разнообразия популяций северного оленя (Rangifer tarandus)
Автор: Харзинова В.Р., Денискова Т.Е., Доцев А.В., Соловьева А.Д., Романенко Т.М., Лайшев К.А., Федоров В.И., Охлопков И.М., Reyer H., Wimmers K., Brem G., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Северное оленеводство
Статья в выпуске: 6 т.54, 2019 года.
Бесплатный доступ
Северный олень ( Rangifer tarandus ) - ценный член арктических экосистем и основной вид сельскохозяйственных животных Российского Севера, для которого необходимо проводить анализ генетической структуры и выполнять дифференциацию дикой и домашней форм, пород и популяций с помощью современных методов молекулярно-генетического анализа. Применение ДНК-чипов на основе параллельного генотипирования сотен тысяч SNP-маркеров - эффективный, но дорогостоящий подход для изучения генома северного оленя. В настоящей работе впервые для северного оленя были отобраны однонуклеотидные полиморфизмы, отвечающие критериям SNP-маркеров для создания пользовательского ДНК-чипа. Нашей целью был выбор оптимального числа SNP-маркеров, позволяющих проводить популяционно-генетические исследования северного оленя без потери биоинформационного контента, и сравнительный анализ информативности отобранных и всех полиморфных SNP-маркеров. Исследования проводили на диких и домашних северных оленях в 2019 году. Дикая популяция (WLD, n = 83) включала оленей, обитающих в западной части полуострова Таймыр, и представителей лено-оленекской и сундрунской субпопуляций с территории Республики Саха (Якутия). Группа домашних оленей состояла из животных ненецкой породы, обитающих на территории Ненецкого автономного округа (NEN, n = 100) и Мурманской области (MUR, n = 19), а также эвенской и эвенкийской пород из Республики Саха (Якутия) (YAK, n = 19). Все животные были генотипированы с использованием ДНК-чипа высокой плотности BovineHD BeadChip, который содержал 777962 SNPs («Illumina, Inc.», США). После проведения контроля качества и применения фильтров в анализе осталось 4456 полиморфных SNP-маркеров. В программе TRES по методу Delta было отобрано 368 наиболее информативных SNP-маркеров. Биоинформационную обработку полученных данных проводили в программах Admixture 1.3, PLINK 1.9 и пакетах R (ggplot2, adegenet 1.3-1, pophelper, diveRsity). Показано, что среди отобранных 368 SNPs преобладали маркеры с высокой частотой минорного аллеля (70 % с MAF ≥ 0,3), тогда как из 4456 маркеров около 50 % имели MAF ≤ 0,1. При сравнении результатов анализа главных компонент (PCA), дискриминантного анализа главных компонент (DAPC) и кластерного анализа не было обнаружено потери информационной ценности 368 SNPs по сравнению с использованием 4456 маркеров. Сопоставляя степень генетических различий на основе попарных значений FST между изучаемыми группами северного оленя, мы показали схожесть характера межпопуляционных связей, оцененного с помощью 4456 и 368 SNP-маркеров. Таким образом, отобранная панель SNP-маркеров может рассматриваться как информативный, универсальный и дешевый вариант при создании пользовательского ДНК-чипа для характеристики дикой и домашней форм северного оленя.
Северный олень, snp-маркеры, днк-чипы
Короткий адрес: https://sciup.org/142226276
IDR: 142226276 | УДК: 636.294:575.174:575.113:577.2.08 | DOI: 10.15389/agrobiology.2019.6.1167rus
Текст научной статьи Разработка и валидация SNP-панели низкой плотности для характеристики генетического разнообразия популяций северного оленя (Rangifer tarandus)
Северный олень ( Rangifer tarandus ) представляет собой уникальный вид животных, внутри которого сосуществуют дикая и домашняя формы — ценные члены арктических экосистем (1, 2). Обе формы нуждаются в разработке рациональных программ по сохранению генетического разнообразия. Стремительное совершенствование и возрастающая доступность технологий секвенирования нового поколения (next generation sequencing, NGS) привели к тому, что с каждым годом увеличивается число видов животных, для которых становится известна нуклеотидная последовательность полного генома (3, 4). За последние несколько лет наблюдается рас-
При проведении исследований использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФНЦ животноводства — ВИЖ им. академика Л.К. Эрнста. Исследования выполнены при поддержке РНФ, проект 16-16-10068-П.
цвет исследований по расшифровке полного генома северного оленя, которые привели к презентации трех версий сборки генома.
В 2017 году группа китайских исследователей представила первый вариант генома домашнего северного оленя (5). В качестве образца ДНК была взята кровь от одной самки эвенкийской породы из популяции, завезенной в горы Внутренней Монголии на территории Китая, а секвенирование проведено на платформе HiSeq 4000 («Illumina, Inc.», США). В соответствии с этой версией сборки, общий размер генома домашнего северного оленя составил 2,64 Gb. Были расшифрованы 21555 протеин-ко-дирующих генов и полностью реконструированы 3803 гена (92,6 % от размера генома). Однако позднее обнаружилось, что эта сборка генома фрагментирована и имеет высокий процент отсутствующих данных (6). В 2019 году был анонсирован геном карибу — североамериканского сородича Rangifer tarandus (6) с размером 2,205 Gb. Согласно данным R.S. Taylor с соавт. (6), геном карибу включает 33177 протеин-кодирующих генов, в том числе 3820 (93,1 %) полностью реконструированных. Тем не менее, поскольку дикая и домашняя формы вида Rangifer tarandus четко дифференцируются по SNP-маркерам (8), описанная сборка генома тоже может быть не актуальной для домашних северных оленей. И, наконец, группа скандинавских исследователей (7) провела глубокое секвенирование генома домашнего оленя с использованием приборов HiSeq2500 и HiSeq 4000 («Illumina, Inc.», США). Однако полная нуклеотидная последовательность этой сборки, общий размер которой составляет 2,66 Gb и включает 26785 протеин-кодирующих генов, отсутствует как в базах данных NCBI (National Center for Biotechnology Information), так и на других платформах. Как следствие, несмотря на значительные успехи в расшифровке генома северного оленя, этот вид остается в категории немодельных.
Негативные антропогенные факторы и непредсказуемые изменения климата в зоне обитания северного оленя приводят к необратимым критическим изменениям в биоразнообразии этого вида (9, 10). В настоящее время продолжаются поиск и внедрение различных подходов, которые позволили бы оценить генетическое разнообразие, изучить структуру популяций и надежно дифференцировать внутривидовые группы (11).
Однонуклеотидные полиморфизмы (single nucleotide polymorphism, SNP) становятся приоритетным и наиболее популярным типом ДНК маркеров для изучения генетической структуры популяций (12). Применение ДНК-чипов позволяет проводить одновременное генотипирование сотен тысяч маркеров. Тем не менее основная задача большинства исследований сводится к определению породного, группового или индивидуального происхождения животного (13). В этом случае более оправдано использовать только те SNP-маркеры, которые могут наиболее точно дифференцировать релевантных особей при более низкой стоимости и большей скорости процедуры, чем полногеномное генотипирование (13, 14).
В настоящей работе впервые для северного оленя были отобраны однонуклеотидные полиморфизмы, отвечающие критериям SNP-маркеров для создания пользовательского ДНК-чипа: преобладание маркеров с частотой минорного аллеля выше 0,3, сохранение биоинформационной ценности и универсальность для применения в популяционно-генетических исследованиях как домашней, так и дикой форм вида R. tarandus.
Нашей целью был выбор оптимального числа SNP-маркеров, позволяющих проводить популяционно-генетические исследования северного оленя без потери биоинформационного контента, и сравнительный анализ 1168
информативности отобранных и всех полиморфных SNP-маркеров.
Методика. Исследования проводили на диких и домашних северных оленях в 2019 году. Дикая популяция (WLD, n = 83) включала оленей, обитающих в западной части полуострова Таймыр, и представителей лено-оленекской и сундрунской субпопуляций с территории Республики Саха (Якутия). Группа домашних оленей состояла из животных ненецкой породы, обитающих на территории Ненецкого автономного округа (NEN, n = 100) и Мурманской области (MUR, n = 19), а также эвенской и эвенкийской пород из Республики Саха (Якутия) (YAK, n = 19).
Геномную ДНК выделяли с использованием наборов ДНК-Экстран2 (ЗАО «Синтол», Россия) согласно протоколу фирмы-производителя. Качество и целостность ДНК проверяли с помощью электрофореза в 1 % агарозном геле. Концентрацию ДНК измеряли на флуориметре Qubit 2.0 («Invitrogen, Inc.», США). Чистоту ДНК устанавливали посредством оценки соотношения поглощения А260/А280 на спектрофотометре NanоDrop8000 («ThermoFisher Scientific», США).
Всех изучаемых особей северного оленя генотипировали с использованием ДНК-чипа высокой плотности Illumina BovineHD Genotyping BeadChip, который содержал 777962 SNPs («Illumina, Inc.», США). Генотипы получали в программе GenomeStudio 2 («Illumina, Inc.», США). Качество чтения (GenCall, GC) и степень кластеризации SNP (GenTrain, GT) оценивали посредством установления фильтров с пороговыми значениями 0,5 для обоих показателей (15). В программе PLINK v. 1.90 (16) из анализа были исключены SNP, находящиеся на половых хромосомах, характеризующиеся частотой минорного аллеля (MAF) ниже 5 % (-maf 0,05), отклонявшиеся от равновесия Харди-Вайнберга при p <10-6 (-hwe 1e-6), находящиеся в неравновесии по сцеплению (--indep-pairwise 50 5 0.5), а также те, для которых были генотипированы менее 90 % индивидуумов (--geno 0,1). После применения всех фильтров в анализе осталось 4456 полиморфных SNP-маркеров.
Следующим этапом стал отбор наиболее информативных SNP-маркеров для включения в пользовательскую панель. Селекцию маркеров проводили в программе TRES (Toolbox for Ranking and Evaluation of SNPs) (13) по методу Delta (17) в соответствии с уравнением:
5 = I P iA — P jA I , где p i A — частота аллеля A в i -й популяции, p j A — частота того же аллеля в j -й популяции.
Затем локусы оценивали на основе значений δ и ранжировали от наивысшего до наименьшего значения, равного 0,2. В результате было отобрано 368 SNP-маркеров.
Визуализацию графиков распределения SNP-маркеров по группам в зависимости от величины MAF осуществляли с помощью программной среды R (18). Анализ главных компонент (principal component analysis, PCA) проводили в программе PLINK 1.9 с визуализацией в R пакете ggplot2 (19). Дискриминантный анализ главных компонент (discriminant analysis of principal components, DAPC) для изучаемых групп северного оленя выполняли с использованием R пакета adegenet 1.3-1 (20, 21), кластерный анализ — в программе Admixture 1.3 (22) с последующей визуализацией результатов в R пакете «pophelper» (23). Попарные значения FST (24) рассчитывали в пакете R «diveRsity» (25). Все биоинформационные анализы выполняли на основании 4456 и 368 SNP-маркеров для того, чтобы оценить эффективность применения предлагаемой панели (368 маркеров) по срав- нению с полным набором полиморфных SNP-маркеров (4456 маркеров), идентифицируемых с помощью ДНК-чипа высокой плотности Illumina BovineHD Genotyping BeadChip в популяциях северного оленя.
Результаты. Сравнение распределения отобранных SNP по группам в зависимости от частоты минорного аллеля (рис. 1) выявило, что преобладающее количество SNP в панели из 4456 маркеров находилось в группе MAF 0,1 (около 49,3 %), тогда как количество маркеров в остальных группах снижалось от 800 в группе MAF 0,2 (17,9 %) до 250 (5,6 %) в группе MAF 0,5 (см. рис. 1, А). При использовании панели из 368 маркеров распределение SNPs кардинально отличалось (см. рис. 1, Б). Так, группа MAF 0,1 составляла лишь 4,1 % от общего числа маркеров, включенных в панель. Наибольшее число SNPs (57,1 %) было представлено группами MAF 0,3 и MAF 0,4. В группу MAF 0,5 входило около 15,8 % всех маркеров панели.
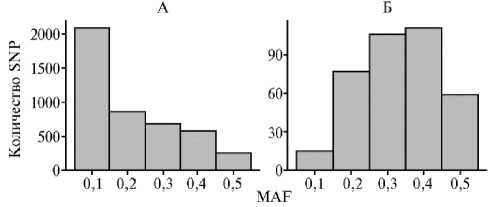
Рис. 1. Распределение SNP-маркеров по группам (2019 год) в зависимости от частоты минорного аллеля (MAF) при сравнении двух панелей маркеров: А — полный набор полиморфных маркеров (4456 SNP), идентифицируемых с помощью ДНК-чипа высокой плотности Illumina BovineHD Genotyping BeadChip в популяциях домашнего и дикого северного оленя ( Rangifer tarandus ); Б — 368 SNP, отобранных для создания пользовательского ДНК-чипа.
В целом, анализ главных компонент, выполненный для изучаемых групп северного оленя на основе 4456 и 368 SNP маркеров, не выявил значительных различий в характере пространственного формирования кластеров (рис. 2, А, Б). Так, первая главная компонента (PC1) четко отделяла группы MUR и NEN от группы WLD, в то время как популяция YAK, рас- полагавшаяся практиче- ски на PC1, была дифференцирована второй главной компонентой (PC2) от трех групп северного оленя. Тем не менее величина генетической изменчивости, объясняемой PC1 и PC2, при использовании панели из 4465 SNP маркеров (см. рис. 2, А) оказалась в несколько раз ниже по сравнению со второй панелью (см. рис. 2, Б): 11,8 против 36,28 % для PC1 и 3,21 против 10,32 % для PC2.
Метод DAPC был использован при изучении закономерностей генетического разнообразия, характерных для исследованных групп северного оленя (рис. 2, В, Г). Линейный дискриминант 1 (LD 1) четко дифференцировал группы NEN, MUR и YAK от группы WLD. Группа YAK была наиболее дистанцированной по линейному дискриминанту 2 (LD 2). Кроме того, группы NEN, MUR и WLD располагались практически на оси LD 2. Значительных отличий в информативности двух изучаемых систем мы также не обнаружили.
При сопоставлении степени генетических различий между изучаемыми группами северного оленя (табл.) была показана схожесть межпопуляционных связей, оцененных с помощью 4456 и 368 SNP-маркеров. Так, наибольшую генетическую близость выявили между группами MUR и NEN, а максимальную дифференциацию — между WLD и NEN.
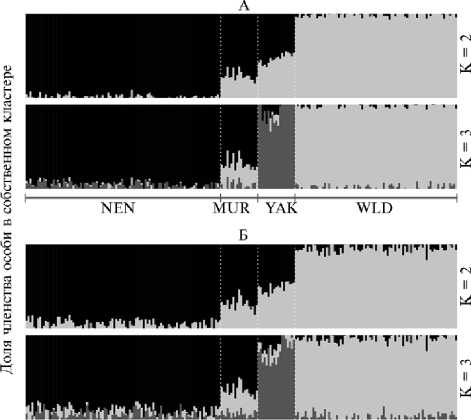
На рисунке 3 представлены результаты адмикс-анализа для изучаемых групп северного оленя на основании 4456 (см. рис. 3, А) и 368 (см. рис. 3, Б) SNP-маркеров для числа предполагаемых кластеров 2 и 3. При
К = 2 с помощью обеих панелей были получены схожие результаты кластеризации. Популяции NEN и WLD были четко дифференцированы и образовывали собственный кластер (светло-серый для WLD и черный для NEN), в то время как популяции MUR и YAK представляли собой смешанные кластеры. При К = 3 группа YAK выделялась в свой кластер, консолидированность особей внутри которого выглядела более однозначно при использовании набора, включающего все полиморфные SNP-маркеры (см. рис. 3, А). Однако различия в паттерне распределения групп животных при К = 3 были незначительными.
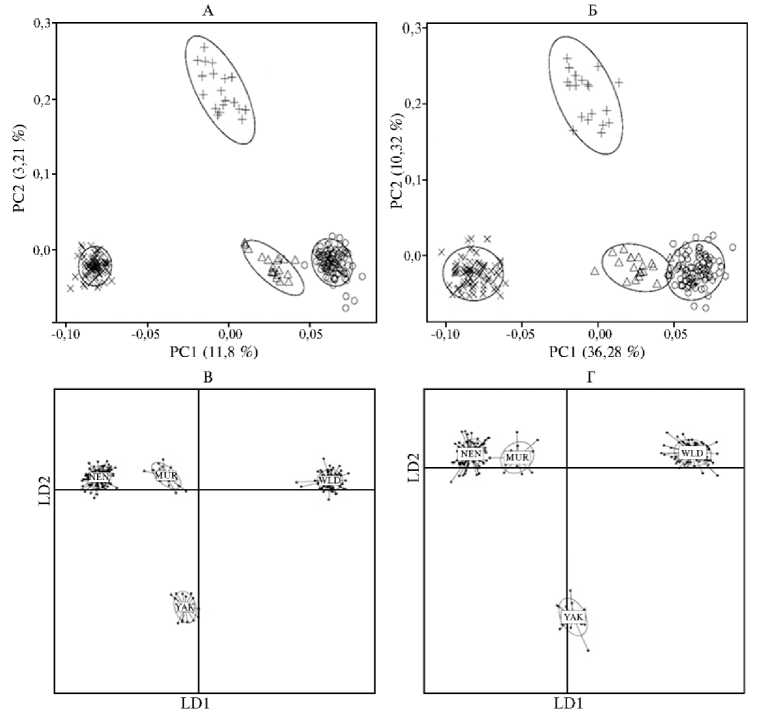
Рис. 2. Результаты анализа главных компонент (PCA) (А, Б) и дискриминантного анализа главных компонент (DAPC) (В, Г) , выполненного для четырех популяций северного оленя ( Rangifer tarandus ) на основе данных двух панелей SNP: А, В — полный набор полиморфных маркеров (4456 SNP), идентифицируемых с помощью ДНК-чипа высокой плотности Illumina Bovi-neHD Genotyping BeadChip; Б, Г — 368 SNP, отобранных для создания пользовательского ДНК-чипа; WLD, X — дикая популяция (полуостров Таймыр, Республика Саха—Якутия), NEN, о — домашние олени ненецкой породы (Ненецкий автономный округ), MUR, △ — домашние олени ненецкой породы (Мурманская обл.), YAK, + — домашние олени эвенской и эвенкийской пород (Республика Саха—Якутия) (2019 год).
Эффективность применения на оленях ДНК-чипов, разработанных для крупного рогатого скота (КРС), была продемонстрирована в зарубежных (26, 27) и отечественных исследованиях (8, 28). Тем не менее среди всех прогенотипированных SNP только около 5 % полиморфны и непосредственно используются в анализе: 1068 для чернохвостого и белохвостого оленей (26), 1532 для представителей рода Cervus (27) и 512 для якутского домашнего оленя (28) при использовании Bovine SNP50 BeadChip;
8357 и 8145 — соответственно для домашних и диких северных оленей из 777962 SNPs на чипе BovineHD BeadChip (8).
Несмотря на успешность применения вышеперечисленных ДНК-чипов для характеристики генетической структуры популяций оленей, оценки их генетического разнообразия и взаимосвязей, существуют несколько факторов, препятствующих широкому использованию этого подхода в рутинном тестировании. Во-первых, следует принимать во внимание высокую стоимость ДНК-чипов. Во-вторых, практически 95 % контента ДНК-чипа отсекается в качестве неинформативного для северных оленей. Кроме того, детальные геномные исследования не всегда нужны в практическом оленеводстве.
Значения FST, рассчитанные по 4456 и 368 SNP-маркерам для четырех популяций северного оленя ( Rangifer tarandus ) (2019 год)
|
Популяция |
NEN |
MUR |
YAK |
WLD |
|
NEN |
0,025 |
0,184 |
0,218 |
|
|
MUR |
0,012 |
0,142 |
0,138 |
|
|
YAK |
0,043 |
0,040 |
0,215 |
|
|
WLD |
0,060 |
0,042 |
0,048 |
П р и м еч а ни е. WLD — дикая популяция (полуостров Таймыр, Республика Саха—Якутия), NEN — домашние олени ненецкой породы (Ненецкий автономный округ), MUR — домашние олени ненецкой породы (Мурманская обл.), YAK — домашние олени эвенской и эвенкийской пород (Республика Саха— Якутия); FST — индекс фиксации. Над диагональю представлены значения, рассчитанные по 368 SNP-маркерам, под диагональю — по 4456 SNP-маркерам.
NEN MUR ' YAK WLD
Рис. 3. Результаты сравнительного адмикс-анализа четырех популяций северного оленя ( Rangifer ta-randus ) (2019 год) для двух панелей SNP: А — полный набор полиморфных маркеров (4456 SNP), идентифицируемых с помощью ДНК-чипа высокой плотности Illumina BovineHD Genotyping BeadChip, Б — 368 SNP, отобранных для создания пользовательского ДНК-чипа. K — число предполагаемых кластеров. WLD — дикая популяция (полуостров Таймыр, Республика Саха—Якутия), NEN — домашние олени ненецкой породы (Ненецкий автономный округ), MUR — домашние олени ненецкой породы (Мурманская обл.), YAK — домашние олени эвенской и эвенкийской пород (Республика Саха—Якутия).
В этом случае весьма перспективна разработка пользовательского ДНК-микрочипа, которая способствуют уменьшению затрат анализа без существенного снижения его биологической ценности (29, 30). Подобные SNP-панели, содержащие ограниченное число информативных маркеров, созданы для модельных сельскохозяйственных животных — овец (31, 32), крупного рогатого скота (33) и лошадей (34).
В нашем исследовании было показано, что 368 SNP-маркеров позволяют воспроизвести информативность всех обнаруженных полиморфных маркеров (4456), что, в целом, согласуется с полученными ранее данными на других видах млекопитающих. По сообщению M.P. Heaton с со-авт. (33), всего 32 SNP-маркера достаточно для определения происхождения с точностью 99,99 % в популяциях мясного скота (даже в случае с сильно смешанными стадами). J.W Kijas с соавт. (31) предложили исполь- зовать SNP-панели из 88 маркеров при подтверждении достоверности отцовства для более чем 80 пород овец, а для тех же целей в коневодстве необходим 101 маркер (34). Кроме того, панель из 163 маркеров достаточна для определения породной и индивидуальной принадлежности в широком спектре пород овец (32).
Частота минорного аллеля (MAF) — важный параметр для селекции SNP-маркеров при последующей разработке информативных пользовательских ДНК-чипов. С помощью биоинформационного подхода TRES (13) из 4456 полиморфных SNPs нами были выбраны 368 маркеров. Произведенные расчеты продемонстрировали сдвиг отобранных однонуклеотидных полиморфизмов в сторону повышения MAF с 0,1 до 0,30,4. Наблюдаемая тенденция согласовалась с таковой для методики, которую использовали J.W. Kijas с соавт. (31) и M.P. Heaton с соавт. (32) при разработке панелей происхождения для овец (панели состояли соответственно из 88 и 163 маркеров). По данным J.W. Kijas с соавт. (31), выбор SNP-маркеров со смещением в сторону значений MAF ≥ 0,3 для включения в панель способствует повышению ее универсальности, то есть позволяет успешно применять ее для расширенного породного и популяционного состава изучаемого вида. Отобранные в нашей работе 368 маркеров полностью отвечают указанному критерию.
В нашей работе селекция маркеров проводилась на основе SNP-маркеров на ДНК-чипе, разработанном для крупного рогатого скота, в соответствии с локализацией по хромосомам, характерной для КРС. Поскольку выравнивание нуклеотидных последовательностей генома северного оленя проводилось по геному КРС как референтному (6) (без разбивки на хромосомы северного оленя), можно предположить, что представленная SNP-панель не будет значительно уступать по информативности тем, которые разрабатываются на основе версии сборки генома северного оленя, предложенной R.S. Taylor с соавт. (6).
Таким образом, отобрано минимально необходимое число полиморфных SNP-маркеров, позволяющих без потери биоинформационного содержания проводить генетические исследования в популяциях северных оленей. Для оценки информативности панели из 368 SNP-маркеров полученные на ее основе данные сравнивали с результатами, рассчитанными при использовании 4456 SNP-маркеров, выявленных с помощью ДНК-чипа высокой плотности. Среди маркеров, включенных в тестируемую панель, зафиксировано явное смещение в сторону однонуклеотидных полиморфизмов с частотой минорного аллеля более 0,3, что соответствует международным критериям создания панелей для различных видов сельскохозяйственных животных. Все использованные биоинформационные подходы (анализ главных компонент, дискриминантный анализ главных компонент, кластерный анализ и расчет попарных генетических дистанций FST) не выявили потери эффективности панели из 368 SNP-маркеров. Успешное тестирование представленной панели проводилось как в популяциях домашних северных оленей трех пород (ненецкая, эвенская, эвенкийская), так и в группе диких таймырских и якутских оленей, что свидетельствует об универсальности отобранных SNP-маркеров. Отобранные однонуклеотидные полиморфизмы могут быть рекомендованы для включения в состав первого пользовательского ДНК-чипа, предназначенного для геномной характеристики дикой и домашней форм северного оленя.
Список литературы Разработка и валидация SNP-панели низкой плотности для характеристики генетического разнообразия популяций северного оленя (Rangifer tarandus)
- Баскин Л.М. Северный олень. Управление поведением и популяциями. Оленеводство. Охота. М., 2009.
- Лайшев К.А., Южаков А.А. Стабильность оленеводства - в рациональном использовании биоресурсов и инновационных решениях. Научный вестник Ямало-Ненецкого автономного округа, 2017, 1(94): 45-48.
- Zhang J., Chiodini R., Badr A., Zhang G. The impact of next-generation sequencing on genomics. Journal of Genetics and Genomics, 2011, 38(3): 95-109 ( ). DOI: 10.1016/j.jgg.2011.02.003
- Kchouk M., Gibrat J.F., Elloumi M. Generations of sequencing technologies: from first to next generation. Biol. Med. (Aligarh), 2017, 9(3): 1000395 ( ). DOI: 10.4172/0974-8369.1000395
- Li Z., Lin Z., Ba H., Chen L., Yang Y., Wang K., Qiu Q., Wang W., Li G. Draft genome of the reindeer (Rangifer tarandus). GigaScience, 2017, 6(12): 1-5 ( ). DOI: 10.1093/gigascience/gix102
- Taylor R.S., Horn R.L., Zhang X., Golding G.B., Manseau M., Wilson P.J. The caribou (Rangifer tarandus) genome. Genes, 2019, 10(7): 540 ( ).
- DOI: 10.3390/genes10070540
- Weldenegodguad M., Pokharel K., Ming Y., Honkatukia M., Peippo J., Reilas T, Røed K.H., Kantanen J. OP149 sequencing of reindeer (Rangifer tarandus) genomes: insights into evolution, domestication, and adaptation. Proc. 37th International Conference on Animal Genetics. Lleida, Spain, 2019: 42.
- Kharzinova V.R., Dotsev A.V., Deniskova T.E., Solovieva A.D., Fedorov V.I., Layshev K.A., Romanenko T.M., Okhlopkov I.M., Wimmers K., Reyer H., Brem G., Zinovieva N.A. Genetic diversity and population structure of domestic and wild reindeer (Rangifer tarandus L. 1758): a novel approach using BovineHD BeadChip. PLoS ONE, 2018, 13(11): e0207944 ( ).
- DOI: 10.1371/journal.pone.0207944
- Yannic G., Pellissier L., Ortego J., Lecomte N., Couturier S., Cuyler C., Dussault C., Hundertmark K.J., Irvine R.J., Jenkins D.A., Kolpashikov L., Mager K., Musiani M., Parker K.L., Røed K.H., Sipko T., Þórisson S.G., Weckworth B.V., Guisan A., Bernatchez L., Côté S.D. Genetic diversity in caribou linked to past and future climate change. Nature Climate Change, 2014, 4: 132-137 ( ).
- DOI: 10.1038/nclimate2074
- Южаков А.А. Проблемы экологии и мониторинг оленеводства на Ямале. Мат. V Межд. науч. конф. "Экология древних и традиционных обществ". Тюмень, 2016: 269-274.
- Ogden R. Unlocking the potential of genomic technologies for wildlife forensics. Molecular Ecology Resources, 2011, 11(Suppl. 1): 109-116 ( ).
- DOI: 10.1111/j.1755-0998.2010.02954.x
- Helyar S.J., Hemmer-Hansen J., Bekkevold D. Taylor M.I., Ogden R., Limborg M.T., Cariani A., Maes G.E., Diopere E., Carvalho G.R., Nielsen E.E. Application of SNPs for population genetics of nonmodel organisms: new opportunities and challenges. Molecular Ecology Resources. 2011, 11(Suppl. 1): 123-136 ( ).
- DOI: 10.1111/j.1755-0998.2010.02943.x
- Kavakiotis I., Triantafyllidis A., Ntelidou D., Alexandri P., Megens H.J., Crooijmans R.P.M.A., Groenen M.A.M., Tsoumakas G., Vlahavas I. TRES: identification of discriminatory and informative SNPs from population genomic data. Journal of Heredity, 2015, 106(5): 672-676 ( ).
- DOI: 10.1093/jhered/esv044
- Wilkinson S., Wiener P., Archibald A.L., Law A., Schnabel R.D., McKay S.D., Taylor J.F., Ogden R. Evaluation of approaches for identifying population informative markers from high density SNP chips. BMC Genetics, 2011, 12: 45 ( ).
- DOI: 10.1186/1471-2156-12-45
- Fan J.B., Oliphant A., Shen R., Kermani B.G., Garcia F., Gunderson K.L., Hansen M., Steemers F., Butler S.L., Deloukas P., Galver L., Hunt S., Mcbride C., Bibikova M., Rubano T., Chen J., Wickham E., Doucet D., Chang W., Campbell D., Zhang B., Kruglyak S., Bentley D., Haas J., Rigault P., Zhou L., Stuelpnagel J., Chee M.S. Highly parallel SNP genotyping. Cold Spring Harb. Symp. Quant. Biol., 2003, 68: 69-78 ( ).
- DOI: 10.1101/sqb.2003.68.69
- Chang C.C., Chow C.C., Tellier L.C., Vattikuti S., Purcell S.M., Lee J.J. Second-generation PLINK: rising to the challenge of larger and richer datasets. GigaScience, 2015, 4(1): s13742-015-0047-8 ( ).
- DOI: 10.1186/s13742-015-0047-8
- Shriver M.D., Smith M.W., Jin L., Marcini A., Akey J.M., Deka R., Ferrell R.E. Ethnic-affiliation estimation by use of population-specific DNA markers. American Journal of Human Genetics, 1997, 60(4): 957-964.
- R Core Team (2018). R: A language and environment for statistical computing. R Foundation for Statistical Computing, Vienna, Austria. Режим доступа: https://www.R-project.org/. Без даты.
- Wickham H. ggplot2 - Elegant graphics for data analysis. Springer-Verlag, NY, 2009 ( ).
- DOI: 10.1007/978-0-387-98141-3
- Jombart T. adegenet: a R package for the multivariate analysis of genetic markers. Bioinformatics, 2008, 24(11): 1403-1405 ( ).
- DOI: 10.1093/bioinformatics/btn129
- Jombart T., Ahmed I. adegenet 1.3-1: new tools for the analysis of genome-wide SNP data. Bioinformatics, 2011, 27(21): 3070-3071 ( ).
- DOI: 10.1093/bioinformatics/btr521
- Alexander D.H., Novembre J., Lange K. Fast model-based estimation of ancestry in unrelated individuals. Genome Research, 2009, 19(9): 1655-1664 ( ).
- DOI: 10.1101/gr.094052.109
- Francis R.M. POPHELPER: an R package and web app to analyse and visualize population structure. Molecular Ecology Resources, 2017, 17(1): 27-32 ( ).
- DOI: 10.1111/1755-0998.12509
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38(6): 1358-1370 ( ).
- DOI: 10.2307/2408641
- Keenan K., McGinnity P., Cross T.F., Crozier W.W., Prodöhl P.A. diveRsity: an R package for the estimation of population genetics parameters and their associated errors. Methods in Ecology and Evolution, 2013, 4(8): 782-788 ( ).
- DOI: 10.1111/2041-210X.12067
- Haynes G.D., Latch E.K. Identification of novel single nucleotide polymorphisms (SNPs) in deer (Odocoileus spp.) using the BovineSNP50 BeadChip. PLoS ONE, 2012, 7(5): e36536 ( ).
- DOI: 10.1371/journal.pone.0036536
- Kasarda R., Moravčíková N., Trakovická A., Krupová Z., Ondrej K. Genomic variation across cervid species in respect to the estimation of red deer diversity. Acta Veterinaria-Beograd, 2017, 67(1): 43-56 ( ).
- DOI: 10.1515/acve-2017-0005
- Харзинова В.Р., Доцев А.В., Соловьева А.Д., Федоров В.И., Охлопков И.М., Виммерс К., Рейер Х., Брем Г., Зиновьева Н.А. Популяционно-генетическая характеристика домашнего северного оленя в республике Якутия на основании полногеномного SNP анализа. Сельскохозяйственная биология, 2017, 52(4): 669-678 ( ).
- DOI: 10.15389/agrobiology.2017.4.669rus
- Lorenz M.G.O., Cortes L.M., Lorenz J.J., Liu E.T. Strategy for the design of custom cDNA microarrays. Biotechniques, 2003, 34(6): 1264-1270 ( ).
- DOI: 10.2144/03346mt02
- Neerincx P.B.T., Rauwerda H., Nie H., Groenen M.A.M., Breit T.M., Leunissen J.A.M. OligoRAP - an Oligo Re-Annotation Pipeline to improve annotation and estimate target specificity. BMC Proc., 2009, 3(Suppl. 4): S4 ( ).
- DOI: 10.1186/1753-6561-3-S4-S4
- Kijas J.W., McEwan J., Clarke S., Henry H., Maddox J., McCulloch R., Driver F., Ilic K., Heaton M. Development of a SNP panel for parentage assignment in sheep. International Sheep Genomics Consortium, 2012. Режим доступа: http://sheephapmap.org/news/PAG_2012_ParentagePoster.pdf. Без даты.
- Heaton M.P., Leymaster K.A., Kalbfleisch T.S., Kijas J.W., Clarke S.M., McEwan J., Maddox J.F., Basnayake V., Petrik D.T., Simpson B., Smith T.P., Chitko-McKown C.G., International Sheep Genomics Consortium. SNPs for parentage testing and traceability in globally diverse breeds of sheep. PLoS ONE, 2014, 9(4): e94851 ( ).
- DOI: 10.1371/journal.pone.0094851
- Heaton M.P., Harhay G.P., Bennett G.L., Stone R.T., Grosse WM, Casas E., Keele J.W., Smith T.P.L., Chitko-McKown C.G., Laegreid W.W. Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle. Mammalian Genome, 2002, 13(5): 272-281 ( ).
- DOI: 10.1007/s00335-001-2146-3
- Holl H.M., Vanhnasy J., Everts R.E., Hoefs-Martin K., Cook D., Brooks S.A., Carpenter M.L., Bustamante C.D., Lafayette C. Single nucleotide polymorphisms for DNA typing in the domestic horse. Animal Genetics, 2017, 48(6): 669-676 ( ).
- DOI: 10.1111/age.12608


