Разработка методики молекулярно-генетической паспортизации штаммов сельскохозяйственных микроорганизмов с помощью AFLP-фингерпринтинга
Автор: Сафронова В.И., Чижевская Е.П., Андронов Е.Е.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Санитария, экология, микробиология
Статья в выпуске: 6 т.47, 2012 года.
Бесплатный доступ
Объектом исследования были 16 штаммов из Ведомственной коллекции полезных микроорганизмов сельскохозяйственного назначения Россельхозакадемии (ВКСМ), относящиеся к родам Bacillus, Lactobacillus, Rhizobium и Bradyrhizobium. Целью работы была разработка методики генотипической паспортизации штаммов с помощью AFLP-фингерпринтинга, обеспечивающего детекцию нуклеотидного полиморфизма и мелких геномных перестроек. В результате исследований разработан протокол проведения AFLP-фингерпринтинга, начиная от выделения ДНК из клеток микроорганизмов и заканчивая компьютерной обработкой данных. Показано, что метод AFLP-фингерпринтинга обладает высокой штаммовой специфичностью, разрешающей способностью и воспроизводимостью результатов. Метод может быть использован для получения индивидуальных генетических паспортов микроорганизмов, депонируемых в ВКСМ, в целях защиты авторских прав на коммерческие штаммы.
Микробиологическая коллекция, генетическая паспортизация коммерческих штаммов микроорганизмов, aflp-фингерпринтинг
Короткий адрес: https://sciup.org/142133353
IDR: 142133353 | УДК: 631/635:579.64:57.088
Текст научной статьи Разработка методики молекулярно-генетической паспортизации штаммов сельскохозяйственных микроорганизмов с помощью AFLP-фингерпринтинга
К важными задачам коллекционной работы относится идентификация и паспортизация культур микроорганизмов, которые необходимы для безопасного использования микробиологических ресурсов в биотехнологической отрасли (включая растениеводство, животноводство и пищевую промышленность), а также защиты авторских прав на коммерческие штаммы. Для этих целей в настоящее время привлекаются как методы, основанные на изучении физиолого-биохимических свойств штаммов, так и современные молекулярно-генетические методы, например BOX, ERIC, REP-PCR и AFLP-фингерпринтинг (1, 2). Различные варианты фингерпринтин-га основаны на изучении геномной ДНК микроорганизмов для выявления индивидуальных особенностей штаммов, которые могут быть использованы при создании штамм-специфичных паспортов. AFLP-фингерпринтинг (amplified fragment length polymorphism) — один из наиболее перспективных приемов молекулярно-генетической паспортизации микроорганизмов. Метод заключается в анализе полиморфизма длин рестрицированных и ам-плифицированных фрагментов ДНК (3, 4). Его преимущество — очень высокая чувствительность, которая позволяет генерировать индивидуальные профили штаммов и различать их в пределах одного вида. Ранее с помощью AFLP-фингерпринтинга были успешно проведены исследования генетических различий между близкородственными штаммами клубеньковых бактерий, относящихся к разным родам (5-7).
В связи с этим наша цель заключалась в разработке методики генетической паспортизации штаммов, депонированных в Ведомственной коллекции полезных микроорганизмов сельскохозяйственного назначения Россельхозакадемии, с помощью AFLP-фингерпринтинга.
Методика . Объектами исследования были 16 практически ценных штаммов из коллекции ВКСМ: Rhizobium leguminosarum (348, 700, 261 и
Работа выполнена при поддержке Министерства образования и науки РФ в рамках ФЦП «Исследования и разработки по приоритетным направлениям развития научно-технического комплекса России на 2007-2013 годы» (Государственный контракт № 16.518.11.7095).
712), Rhizobium galegae 912, Bradyrhizobium japonicum 630, Bradyrhizobium sp. 820, Bacillus thuringiensis (626, 634, 603, 640 и 642), а также штаммы Lactobacillus plantarum (616, 621, 631 и 613). Штаммы клубеньковых бактерий культивировались при 28 ° C на маннитно-дрожжевом агаре (YMA), Bacillus thuringiensis — на мясопептонном агаре (МПА), молочнокислые бактерии — на жидком сусле (8).
Общую ДНК клубеньковых бактерий выделяли по следующей методике: 1,5 мл ночной культуры клеток ризобий центрифугировали при 14000 об/мин в течение 2 мин. Надосадочную жидкость удаляли, а клетки ресуспендировали в 500 мкл буфера TE (Трис-HCl — 10 ммоль/л, EDTA — 5 ммоль/л, pH 8,0). К суспензии добавляли лизоцим (1 мг/мл), инкубировали 5 мин при комнатной температуре, затем вносили SDS до концентрации 0,5 % и протеиназу К до концентрации 0,05 мг/мл и инкубировали при 37 ° C в течение 1 ч. Экстракцию проводили смесью фенола, хлороформа и изоамилового спирта (24:24:1). ДНК осаждали двумя объемами этанола 5 мин при комнатной температуре. Раствор центрифугировали 2 мин при 14 000 об/мин и растворяли осадок в 100 мкл дистиллированной воды. В случае выделения ДНК из бактерий рода Bacillus и Lactobacillus вводили стадию предварительной обработки протеиназой К в концентрации 0,05 мг/мл при 60 ° C в течение 1 ч, при необходимости для разрушения клеток суспензию встряхивали со стеклянными шариками в гомогенизаторе FastPrep24 («MP Biomedicals», США) при максимальной мощности в течение 1 мин. Окончательную очистку проводили, как описано выше.
Полученную геномную ДНК (50 нг) использовали для одновременной реакции рестрикции и лигирования. В состав реакционной смеси входили по 2,5 ед. рестриктаз MseI и EcoRI («MBI Fermentas», Литва), 1 ед. лигазы фага Т4 («MBI Fermentas», Литва), два олигонуклеотидных адаптера — для сайта EcoRI (adEco1 CTCGTAGACTGCGTACC и adEco2 AAT-TGGTACGCAGTCTAC) и для сайта MseI (adMse1 GACGAGAGTCCTG-AG и adMse2 TACTCaGgACTCAT) в количестве 5 пмоль каждого. Реакцию проводили в буфере для лигазы («MBI Fermentas», Литва) при 37 ° C в течение 16 ч.
В финальной амплификации, результатом которой были геномные фингерпринты, в качестве матрицы использовали 1 мкл реакционной смеси, полученной на предыдущей стадии, и селективные праймеры (по одному для каждого адаптера, флуоресцентно меченного красителем FAM): Mse_a GA-TGAGTCCTGAGTAAA, Mse_cg GATGAGTCCTGAGTAACG или Mse_ca GATGAGTCCTGAGTAACA — для сайта MseI; Eco_0 GACTGC-GTACCAATT или Eco_a GACTGCGTACCAATTCA — для сайта EcoRI (по 10 пмоль каждого). Для поиска оптимального сочетания испытывали все возможные комбинации MseI/EcoRI селективных праймеров. Предварительную оценку результата проводили в агарозном 3 % геле, финальную — в условиях автоматического капиллярного электрофореза на генетическом анализаторе ABI 3500xl («Applied Biosystems», США). В смесь для электрофоретического разделения добавляли также интервальный маркер молекулярной массы GeneScan-600 LIZ Size Standard («Applied Biosystems», США).
При сопоставлении фингерпринтов использовали программу FPQuest («Bio Rad», США). Исходным материалом для анализа служили файлы, полученные после электрофоретического разделения фрагментов и содержащие кривые, которые соответствовали фрагментам AFLP (FAM) и стандарту молекулярной массы (LIZ). Файлы предварительно экспортировали в TIFF-формат.
Степень сходства между фингерпринтами оценивали по корреля- ции Пирсона, ориентированной скорее на поиск общего сходства между кривыми, чем на поиск фрагментов одинакового размера.
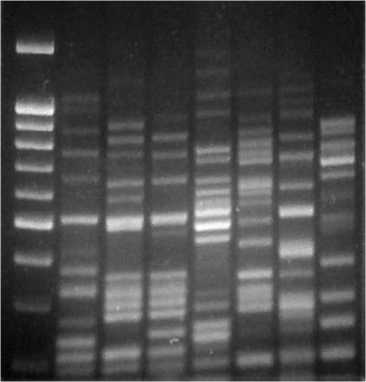
Рис. 1. Предварительный электрофорез в 3 % агарозном геле AFLP-фрагментов ДНК ризо-бий, полученных с использованием селективных праймеров Mse_ca/Eco_a.
Резуёътаты . Из всех проанализированных штаммов мы выделили геномную ДНК, концентрация и степень очистки которой были достаточными для проведения AFLP. Реакции рестрикции и лигирования с адаптерами дали отличный результат, так как при использовании рестрик-ционно-лигазной смеси в качестве матрицы на завершающем этапе AFLP-ПЦР для всех проанализированных микроорганизмов получили высокоспецифичные профили. В результате изучения разных комбинаций селективных праймеров была отобрана пара Mse_ca/Eco_a как дающая оптимальное соотношение количества и специфичности фрагментов (рис. 1).
Обработка полученных данных в программе FPQuest позволила эф фективно выравнять фингерпринты, вычислить степень их родства и провести кластерный анализ, включающий статистическую поддержку кластеризации. На первом этапе была построена дендрограмма родства, объеди няющая все полученные данные, включая различные штаммы и разные объемы смеси фрагментов, использованные при электрофорезе (1; 0,5; 0,25 или 0,1 мкл) с целью подбора оптимального соотношения для обеспечения эффективного разделения и элиминации эффектов, связанных с перегрузкой или недостаточным количеством внесенного материала (рис. 2).
Анализ полученных данных показал, что за исключением штаммов, имеющих высокий уровень сходства (штаммы 621, 616, 631 и 613 Lactobacillus ; 626/634 и 640/642 Bacillus thuringiensis ), фингерпринты группируются строго по штаммам, демонстрируя очень высокое сходство между профилями, соответствующими разным количествам AFLP-смеси и принадлежащим одному и тому же штамму. На основании сопоставления результатов было продемонстрировано, что в проводимом эксперименте оптимальное для нанесения в генетический анализатор количество соответствует 0,1 мкл смеси AFLP. Поэтому для финального сравнения выбрали именно эти образцы с наименьшей концентрацией (рис. 3). Их анализ выявил очевидные различия в наборах фрагментов ДНК, что свидетельствует об очень высокой чувствительности метода, который позволяет получать уникальные профили для каждого штамма и дискриминировать их в пределах одного вида. Тем не менее, несмотря на очень высокую специфичность метода, кластерный анализ AFLP-профилей позволяет достаточно четко группировать штаммы в соответствии с их филогенетическим родством. На дендрограмме (см. рис. 3) штаммы ризобий (348, 912, 630, 820, 700, 261 и 712), штаммы Bacillus (626, 634, 603, 640 и 642), а также штаммы Lactobacillus (616, 621, 631 и 613) образуют обособленные группы. Среди проанализированных клубеньковых бактерий пять штаммов — 348 ( Rhizobium leguminosarum bv. trifolii ), 912 ( Rhizobium galegae ), 630 ( Bradyrhizobium japonicum ), 820 ( Bradyrhizobium sp.), 700 ( Rhizobium leguminosarum bv. phaseoli ) оказались вне кластеров (уровни сходства менее 30 %) . Другие два штамма
Pearson correlation (Opt: 1,00%) [0,0 %-100,0 %]
ABI
ABI
lllllllllllllll llllllll
■ill пип и mi
11 III
11 III
I II
I II
I II
I II I I I I I
Illi I Illi lllll
lllll
I
ii i
I I
d
{
II lllllllllllllllllll i iiiiiii min ill..... i iiiiiiiiiiiiiiiini i i пни и mi ii mi i
I II
II II III
Illi
i mil и и i hi
III II llll 11II II
I
III
1111111*11111111
I lllll.............I I lllll llllllll Illi
Illi
I
I
II
I I
и
II II llll
II IIII
II
II Illllll
II I I I I I
III
626-3 6264
6344 634-1 634-2 626-1 626-2 634-3 603-1 603-2 603-3 6034
6404 6424 642-2
640-2
640-3
III
III
III
ISHII! I III llllll lllllllllllll 11II III
II11III ......
Illllll II
■III III
I
II
iiiiiiiniiiieio и i
lllll
lllllllllllllllllll

■III HI
■lllll III ■llllll II11
I II
IBII
lllll I ■ I II II I II I I I I
II Illllll
II Illllll
1 llllll
I III
642-3
642-1
I I
I III
II llll ■ III
llllll I llllll I llll I
II l■llll 111
III !■—llll II llllllll III
■
ii
III llllll*
ill ■пинmini
Hinn iiiiiiii
inn i
llllll I «III
III.............. IIIllllIII..... lllllllllllllll llll llll llll
Illi II lllll I 111 III llllll
III I lllll I II II Illi
I I
I I
II II
i
11 11
I I
ii
III
I II
I
in ini
i hi ii in
I
in и
I 11 IIIIII
I I I llll I
II 111
llllll lllll I II I Illi I
II I III
ii 11 ini mm ini in i пин и num 11 ш и i n i inni и mi inimii i mini 11in и in
in i i и ini in inn mini 11 in
I I I II II I I 11 II ■ I llllllll I llllll II III I
i in
■i nimi I lllll II I II
II
III III lllllllllllll I llll II I llll II I
I I IIllll I I llll I II I
I 111
I lllllllllllllll IIIHIIIIII llllll II 11
III III IIIHIIIIII lllllllllllll lllll
I 1 1
llllll II Illi IIIHIIIIII I Illi llllll IIIIIIlllllIII
I lllll I lllll II III II llllllll II Illi llll III llllllll II lllllllllllllllll 11II llll II I
III
d
II
mi mi i huh iniiiiiiii i in ini i ii ii min ini
III IIIllllllI III llllll III II I II I llllll
III
Illi IIIJ lllllllllllllll I III Illi II I II II lllll
II
d
lllll
II 111
I llll
iiii Mill
I И 1 1
lllll llllll
Illi
Illi
III
Illi Illi
Illi I
II II
I II I
III llll
III
III
III
I II lllll I Illi I
min i i imui i
Mil Illi
llll llllllll
III lllll
I III
I III
II
II
640 1 621-1 621-3 613-1 613-3 616-1 616-2 613-2 621-2 631-2 631-3 631-1 6134 6314 616-3 6164 6214 820-1 820-3 8204 820-2 912-1 912-2 912-3 9124 348-2 348-3 348-1 3484 630-1 630-3 6304 630-2 700-2 700-3 700-1 7004 261-1 261-3 2614 261-2 712-1 712-3 712-2
Рис. 2. Сопоставление всех данных (все штаммы микроорганизмов и все количества), полученных при разделении AFLP-фрагментов на генетическом анализаторе (ABI 3500xl, «Applied Biosystems», США): 1 - 1 мкл, 2 — 0,5 мкл, 3 — 0,25 мкл, 4 — 0,1 мкл.
ризобий (261 и 712 Rhizobium leguminosarum bv. viciae ), как и штаммы Bacillus и Lactobacillus , могут быть отнесены к отдельным группам, поскольку степень сходства более 50 % достаточна для статистически достоверного
разделения кластеров (9, 10).

IIIIIDIIIIIIIIIIIIIIIIIII шнiiiши । mu iiiiiiiiiiiiiiiiiiiiiiniii mu min iiiiii in min нт и mi inn iiiiiniiiiiiiiiiiiiiii iiiiiiiiiiiiiiiiiiiiii iiiiiiiiiiiiiii in mi iiiiiiiiiiiiiiiiiiiiii
348-4 912-4 630-4 820-4 700-4 261-4 712-4 626-4 634-4 603-4 640-4 642-4 616-4 621-4 631-4 613-4
Pearson correlation (Opt: [0,0 %-100,0 %] ABI
1,00%) ABI
Рис. 3. Сопоставление данных AFLP-фингерпринтинга, соответствующих внесению 0,1 мкл смеси, для всех проанализированных штаммов.
Таким образом, метод AFLP-фингерпринтинга продемонстрировал высокую разрешающую способность при проведении как внутривидовой, так и межвидовой дифференциации генотипов. Метод характеризуется высокой воспроизводимостью, наличием ряда эффективных подходов к анализу полученных данных и может с успехом применяться в растениеводстве, ветеринарии и в пищевой промышленности для молекулярно-генетической характеристики практически ценных штаммов микроорганизмов. Результаты исследования будут использованы для стандартизации процедуры проведения комплексной доказательной паспортизации штаммов сельскохозяйственных микроорганизмов, депонированных в Ведомственной коллекции полезных микроорганизмов сельскохозяйственного назначения Рос-сельхозакадемии (ВКСМ).


