Разработка методов автоматического извлечения знаний из текстов научных публикаций для создания базы знаний Solanum tuberosum
Автор: Сайк О.В., Деменков П.С., Иванисенко Т.В., Колчанов Н.А., Иванисенко В.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Картофелеводство: наука и технологии
Статья в выпуске: 1 т.52, 2017 года.
Бесплатный доступ
В настоящее время в мире существуют сотни научных журналов, публикующих результаты исследований в различных областях биологии растений и агробиологии. Сотни тысяч международных патентов содержат сведения по агробиотехнологии. Число статей и патентов со временем растет в экспоненциальной прогрессии. Например, изучению важнейшей сельскохозяйственной культуры Solanum tuberosum L. посвящено более 1,5 млн публикаций. Анализ такого огромного количества экспериментальных фактов, представленных в текстовых источниках (научных публикациях и патентах), требует применения автоматизированных методов извлечения знаний (text-mining). Интеллектуальные методы автоматического анализа текстов уже широко применяются в биологии и медицине для извлечения информации о свойствах и функции молекулярно-генетических объектов. Основанные на таких методах системы осуществляют экстракцию представленных в документах знаний, их интеграцию и представление в формализованном виде в соответствии с онтологией предметной области, и это отличает их от таких систем, как Google, Яндекс и др., где для поиска документов используются ключевые слова. Среди известных систем интеллектуального извлечения знаний из научных публикаций можно выделить STRING, LMMA, ConReg, GeneMania и др. Ранее впервые в России нами была разработана система интеллектуального извлечения знаний в области биомедицины ANDSystem, которая содержит более 10 млн фактов о молекулярно-генетических взаимодействиях для человека и животных из более чем 25 млн научных публикаций. Для извлечения знаний в ANDSystem используются специальные семантико-лингвистические правила, позволяющие распознавать в естественноязыковых текстах взаимодействия между соответствующими объектами - белками, генами, метаболитами, лекарства, микроРНК, биологическими процессами, заболеваниями и др. Однако задача автоматизации извлечения знаний из текстов по биологии растений, агробиологии и агробиотехнологиям до сих пор не решена, несмотря на ее актуальность. Целью настоящей работы была адаптация методов, представленных в системе ANDSystem, для автоматического извлечения знаний по растениеводству и создание на этой основе базы знаний SOLANUM TUBEROSUM, содержащей информацию по генетике, маркерам, селекции, семеноводству, диагностике возбудителей заболеваний, средствам защиты и технологиям хранения картофеля. Онтология базы знаний включает данные словарей более чем по 20 типам объектов (молекулярно-генетические объекты - белки, гены, метаболиты, микроРНК, биологические процессы, биомаркеры и др.; сорта картофеля и их фенотипические признаки; болезни и вредители картофеля; биотические и абиотические факторы окружающей среды; агробиотехнологии возделывания, биотехнологии переработки и хранения картофеля и др.). Описание отношений между этими объектами, включая молекулярные, регуляторные и ассоциативные взаимодействия, содержит более 25 типов связей. Для извлечения информации о взаимодействиях в сумме создано более 5 тыс. семантических шаблонов. Значения точности и полноты извлечения знаний с помощью разработанных правил, оценка которых осуществлялась с привлечением экспертного ручного анализа выборок текстов, составили соответственно более 65 % и 70 %. На основе разработанных подходов предполагается создание полномасштабной версии базы знаний SOLANUM TUBEROSUM.
База данных, методы автоматического извлечения знаний из текстов
Короткий адрес: https://sciup.org/142214015
IDR: 142214015 | УДК: 633.491:004.65:[631.5+632.9 | DOI: 10.15389/agrobiology.2017.1.63rus
Текст научной статьи Разработка методов автоматического извлечения знаний из текстов научных публикаций для создания базы знаний Solanum tuberosum
В настоящее время изучение молекулярно-генетических систем начинает занимать основное место в геномных, протеомных, метаболомных и транскриптомных исследованиях в разных областях биологии, включая растениеводство (1-4). Особое значение новые подходы начали приобретать при изучении взаимосвязи генотип—фенотип. Реконструкция и анализ генных сетей приходят на смену традиционным подходам, основанным на поиске отдельных генов, отвечающих за формирование фенотипических признаков растений, включая комплексные хозяйственно ценные
Работа поддержана бюджетным проектом ИЦиГ СО РАН в рамках КПНИ по приоритетному направлению «Картофелеводство».
признаки, такие как резистентность к заболеваниям и вредителям, устойчивость к воздействию абиотических факторов, урожайность (5-8). Важнейшим источником информации о молекулярно-генетических взаимодействиях, происходящих на внутриклеточном, межклеточном и организменном уровнях организации растений, служат базы данных, обобщающие результаты экспериментов, научные публикации и патенты. Число публикаций увеличивается каждый год в экспоненциальной прогрессии. Даже при простом поисковом запросе по ключевому слову «potato» в системах Web of Science и Google Patents ответ включает информацию о более чем 60 тыс. статей и 900 тыс. патентов. Многие из них содержат данные о молекулярно-генетических взаимодействиях. Быстрое накопление новых знаний, представленных в научных публикациях и базах данных, существенным образом связано с развитием экспериментальных высокопроизводительных омиксных технологий (геномных, транскриптомных, протеомных и метаболомных). Применение высокопроизводительных технологий секвенирования позволило расшифровать геном картофеля.
В базе данных NCBI Genomes представлена версия генома картофеля GCA_000226075.1 SolTub_3.0 (9, 10), включающая аннотацию 37966 белков. В другой базе данных NCBI Gene приведена информация о последовательностях и функциях 33037 генов картофеля.
Для установления взаимодействий между биологическими объектами часто используют экспериментальные методы прямого анализа белок-белковых взаимодействий (дрожжевые двугибридные системы), тран-скриптомный анализ (дифференциальная экспрессия и коэкспрессия генов) и т.д. В базе GEO представлены данные более чем 1300 экспериментов по экспрессии генов картофеля, полученные с использованием транскриптомных технологий. Например, в работе Y. Ou с соавт. (11), представленной в GEO (GSE43237), проводился полногеномный анализ мишеней микроРНК в клубнях, хранящихся в холоде. В результате были выявлены 53 известных и 59 новых микроРНК, а также 70 генов-мишеней, потенциально вовлеченных в реакцию на низкие температуры хранения. В другой работе (12), также представленной в базе GEO (GSE56333), с помощью высокопроизводительного секвенирования изучали влияние инфекции Y-вируса картофеля на устойчивость картофеля к личинкам колорадского жука.
В мире активно развиваются базы данных, содержащие информацию о молекулярно-генетических взаимодействиях, полученную в результате анализа фактографических баз данных и научных публикаций. В частности, в базе PlantCyc (13-16) содержится информация о молекулярно-генетических сетях более чем для 22 видов растений, включая картофель. База данных PotatoCyc (раздел PlantCyc по картофелю) содержит сведения о 558 биологических путях, 5790 ферментах, 3122 реакциях и 2413 метаболитах. Однако эта база данных создана на основе ручного анализа научных публикаций, что гарантирует высокое качество данных, но неизбежно ведет к задержке в представлении фактов, опубликованных в научных статьях.
Современное число публикаций и патентов — это так называемые big data (сверхбольшие объемы данных), эффективная обработка которых требует применения автоматического анализа текстов (text-mining). Технология автоматического извлечения знаний из научных публикаций наиболее активно развивается в биомедицине (17-21). Среди широко распространенных систем анализа текстов по указанной тематике можно выделить STRING (22-24), LMMA (25) и ConReg (26). Система STRING включает 64
описания белок-белковых взаимодействий, которые подтверждены экспериментально либо спрогнозированы различными методами (включая предсказания по близости расположения генов в геноме, филогенетическим профилям, коэкспрессии). В системе STRING используется информация, экстрагированная из баз данных, а также полученная из публикаций с использованием методов автоматического анализа текстов. Система LMMA предназначена для реконструкции биологических сетей на основе интеграции данных литературы о молекулярных взаимодействиях и сведений по генной экспрессии, полученных в микрочиповых экспериментах. Она базируется на оценках статистической значимости совстречаемости биологических терминов в текстах из PubMed. ConReg — это плагин для системы Cytoscape (27), ориентированный на исследование генетической регуляции у эукариотических модельных организмов. Здесь данные по генетической регуляции взяты из разных баз и дополнены информацией о предсказанных сайтах связывания транскрипционных факторов, а также сведениями, извлеченными с помощью автоматического анализа текстов PubMed.
Ранее нами была разработана система ANDSystem для автоматической экстракции медико-биологических знаний из текстов PubMed с помощью методов семантических шаблонов (28-30). Система ANDSystem включает модуль лингвистического анализа, который в автоматическом режиме осуществляет извлечение из произвольного текстового потока фактографической информации, относящейся к определенной предметной (проблемной) области согласно заданной онтологии. Модуль лингвистического анализа состоит из трех основных частей: морфологического анализатора, проблемно ориентированной онтологии и семантического анализатора. Морфологический анализатор реализует следующие функции: дескрипторная разметка текста (распознавание в тексте включаемых в онтологию концептов, в том числе терминологических словосочетаний); лемматизация; частеречная разметка. Проблемно ориентированная онтология формирует концептуальную модель проблемной области. Семантический анализатор реализует функции концептуального поиска в тексте документа и интерфейс пользователя. Работа системы обеспечивается двумя основными словарями: грамматический словарь поддерживает лемматизацию, частеречную разметку и распознавание словосочетаний по линейному контексту; онтология поддерживает семантический анализ, включая элементы ограниченного логического вывода. Кроме того, используется толковый словарь (слово либо словосочетание как концепт), интегрированный в онтологию.
В настоящей работе проведена адаптация и настройка методов системы ANDSystem для автоматического извлечения знаний по генетике, маркерам, омиксным ресурсам, селекции, семеноводству, диагностике возбудителей заболеваний, средствам защиты и технологиям хранения картофеля с целью создания базы знаний SOLANUM TUBEROSUM. Настройка системы ANDSystem включала создание онтологии предметной области и семантико-лингвистических правил (шаблонов) для анализа естественноязыковых текстов и извлечения знаний, формализованных согласно заданной онтологии. Важная составляющая онтологии предметной области — словари объектов, информация о взаимодействиях, между которыми извлекается из текстов с использованием шаблонов. Созданная онтология базы знаний SOLANUM TUBEROSUM содержит словари более чем для 20 типов объектов. В качестве молекулярно-генетических объектов рассмотрены белки, гены, метаболиты, микроРНК, биологические процессы, биомаркеры и др. Отдельными словарями представлены сорта картофеля и их фенотипические признаки, включая болезни картофеля. Большой раз- дел онтологии посвящен вредителям картофеля, а также биотическим и абиотическим факторам окружающей среды. Онтология также содержит словари агробиотехнологии возделывания, биотехнологии переработки и хранения картофеля. Анализ качества извлечения знаний с помощью созданных шаблонов показал хорошую точность (65 %) и полноту (70 %).
Онтологическая модель.
Под онтологией нами понимается набор
O =
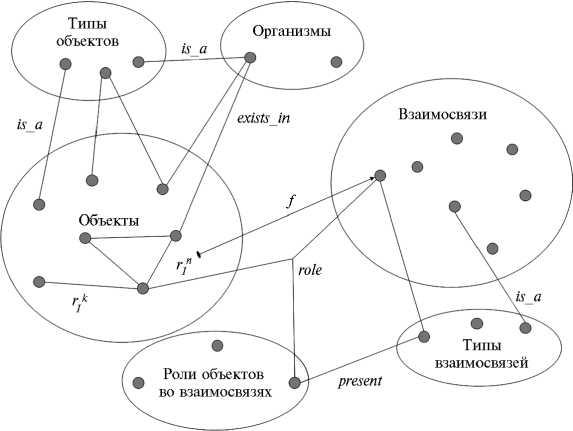
Рис. 1. Графическое представление онтологии ассоциативных семантических сетей, использованное для проектирования базы знаний SOLANUM TUBEROSUM.
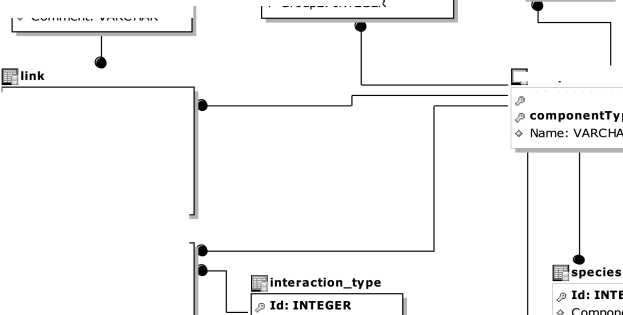
Структура базы знаний. С использованием разработанной онтологической модели представления данных была спроектирована база знаний SOLANUM TUBEROSUM. Она включает базу данных, содержащую молекулярно-генетическую информацию, информацию о технологиях, заболеваниях, факторах среды, полученную в результате анализа текстов научных публикаций, патентов и баз данных. Кроме того, в базе знаний представлены методы, которые использовались для извлечения знаний из текстов, и методы, предназначенные для анализа молекулярногенетических сетей, имеющихся в базе данных. При создании базы знаний была использована реляционная СУБД MySQL 5.6. База данных содержит 18 таблиц, описывающих следующие разделы: Plant, Potato Pathogens and 66
Pests, Environment, Technology, Bioinformatics, Associative networks (рис. 2).
Раздел Plant. Раздел Plant предназначен для описания молекулярногенетических данных. В текущей версии базы знаний в этом разделе при-
^"dbases
23 Id: INTEGER
-
♦ Name: VARCHAR
-
♦ XrefName: VARCHAR
-
♦ wwwLink: VARCHAR
о interaction_db: INTEGER
-
4 Comment: VARCHAR
^groups
^keywords
4 Componentld: INTEGER
4 ComponentType: INTEGER
4 Groupl: INTEGER
4 Group2: INTEGER
,p Componentld: INTEGER
^ ComponentType: INTEGER
23 Keyword: VARCHAR
У entity
23 Id: INTEGER
4 Name: VARCHAR
4 Comment: VARCHAR
I ’component
У interaction
4 Name: VARCHAR
4 Comment: VARCHAR
23 Linkld: INTEGER
4 Componentld: INTEGER
4 ComponentType: INTEGER
4 Databaseld: INTEGER
4 Id: VARCHAR
4 Comment: VARCHAR
23 Id: INTEGER
4 ComponentType: INTEGER
4 Typeld: INTEGER
4 Intertant_count: INTEGER
4 Comment: VARCHAR
:: INTEGER
'component-Synonym
23 Componentld: INTEGER
23 ComponentType: INTEGER
23 Synonym: VARCHAR
У interaction concern
:: INTEGER
4 ScientificName: VARCHAR
4 CommonName: VARCHAR
4 Atcc: VARCHAR
4 Description: VARCHAR
^component-Species
23 Componentld: INTEGER
23 ComponentType: INTEGER
23 Speciesld: INTEGER
23 Interactionld: INTEGER
23 Componentld: INTEGER
23 ComponentType: INTEGER
4 Speciesld: INTEGER
4 Roleld: INTEGER
4 Directionld: INTEGER
Э-
31interaction concern direction
23 Id: INTEGER
4 Name: VARCHAR
4 Direction: INTEGER
4 Color: VARCHAR
4 Comment: VARCHAR
^interaction-concern-role 123 Id: INTEGER
4 Name: VARCHAR
4 Comment: VARCHAR
Рис. 2. Структура основных таблиц реляционной базы данных в разработанной базе знаний SOLANUM TUBEROSUM.
ведена информация по картофелю, дополненная сведениями по семи модельным растениям ( Solanum lycopersicum , Nicotiana tabacum , Arabidopsis tha-liana , Oryza sativa Indica Group, Oryza sativa Japonica Group, Zea mays , Triticum aestivum ). Молекулярно-генетические данные включают словари названий и их синонимов для генов (более 140000 терминов), белков (более 19000 терминов), метаболитов (более 42594 терминов), микроРНК (более 10000 терминов), генетических биомаркеров (более 20 терминов) и биологических процессов (более 100000 терминов). Отдельные словари представляют сорта картофеля (206 сортов), селекционно значимые качества, хозяйственно ценные признаки и потребительские свойства (более 1300 терминов). Созданы специальные словари, описывающие более 100 физиологических (фенотипических) признаков картофеля и болезней.
Раздел Potato Pathogens and Pests. В нем содержатся словари молекулярно-генетических объектов для 24 возбудителей заболеваний и вредителей картофеля. Молекулярно-генетические данные, как и в разделе Plant, представлены генами (3451 ген), белками (476 белков), метаболитами и биологическими процессами. Отдельными словарями описаны маркеры резистентности к средствам защиты растений, а также молекулярные мишени для химических средств защиты растений.
Раздел Environment. Здесь представлены словари для двух типов объектов — биотических и абиотических факторов окружающей среды (соответственно более 100 и более 50 терминов).
Раздел Technology. Следует отметить, что в качестве самостоятельных объектов в разработанной базе знаний, наряду с молекулярно-генетическими объектами и факторами окружающей среды, представлены различные технологии селекции, возделывания, защиты и диагностики забо-67
леваний картофеля, переработки и хранения. В текущей версии базы знаний описано более 100 технологий.
Раздел Associative networks. В качестве информационной модели предметной области использована ассоциативная семантическая сеть, имеющая вид ориентированного двудольного графа, вершины которого соответствуют объектам предметной области, а дуги задают отношения между ними. Для описания взаимодействий между молекулярно-генетическими объектами использованы отношения следующих типов: 1-й — физические взаимодействия, то есть образование короткоживущих или постоянных молекулярных комплексов; 2-й — химические взаимодействия (каталитические реакции и процессы) по типу субстрат—фермент—продукт, в которых участвуют белки (ферменты) и низкомолекулярные соединения (метаболиты), в том числе реакции протеолитического расщепления одного белка (субстрата) другим белком (протеолитическим ферментом), по-странсляционные модификации белков (фосфорилирование, гликозилирование и т.д.); 3-й — регуляторные взаимодействия, включая регуляцию экспрессии генов транскрипционными факторами, регуляцию активности или функции белка другими белками, регуляцию (или осуществление) транспорта одних белков другими белками, регуляцию стабильности или деградации одних белков другими белками или метаболитами (регуляторные события также будут подразделяться по эффекту, оказываемому одним объектом на другой, то есть усиление или ослабление процесса); 4-й — коэкс-прессия (одновременная экспрессия нескольких генов), которая была вызвана общими регуляторными механизмами, активизирующими экспрессию при изменяющихся условиях в клетке; 5-й — ассоциативные связи (в эту категорию входят неклассифицированные связи между молекулярногенетическими объектами, а также связи между молекулярно-генетическими объектами и объектами, соответствующими понятиям селекции, феномики и семеноводства, фитопатологии, диагностики, средствам защиты, агробиотехнологиям возделывания и биотехнологиям переработки и хранения картофеля). Взаимоотношения между понятиями селекции, феномики и семеноводства, заболеваниями, приемами диагностики и средствами защиты, технологиями основаны на различного типа регуляторных связях (положительная и отрицательная регуляция), а также связях, описывающих вовлечение, применение, ассоциативные связи и др.
Раздел Bioinformatics. С базой знаний тесно связан специально разработанный раздел, в котором представлены биоинформатические методы анализа экспериментальных данных по молекулярным механизмам функционирования анализируемых биологических систем, приоритезации генов, предсказанию маркеров, планированию экспериментов и др. Анализ осуществляется на основании экспериментальных данных, вводимых пользователем, а также данных по сети молекулярно-генетических взаимодействий, автоматически извлекаемых из раздела Associative networks.
В настоящее время активно развивается направление биоинформа-тического анализа экспериментальных данных, связанное с задачей приоритезации генов при выявлении среди них наиболее важных для изучаемых биологических процессов (включая ответ на биотические и абиотические факторы окружающей среды), фенотипических (физиологических) признаков, заболеваний и т.д. (31, 32). Для этого в систему SOLANUM TUBEROSUM интегрированы методы известного пакета программ GUILD , основанные на анализе структуры графа генных сетей (33). На рисунке 3 приведен пример приоритезации генов, специфически контролирующих метаболизм крахмала, которые могут представлять интерес в качестве кандидатов для селекционной работы. В описываемом случае критериями для приоритезации служило число связей генов-кандидатов с реперными генами в реконструированной генной сети, ассоциированной с метаболизмом крахмала.
А
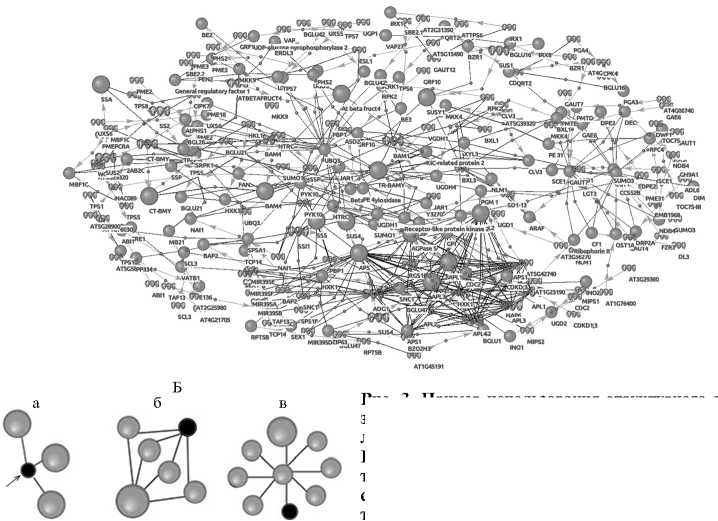
Рис. 3. Пример использования структурного анализа графов генной сети, ассоциированной с метаболизмом крахмала в разработанной базе знаний SO-LANUM TUBEROSUM (А) , для поиска новых потенциально важных для селекции генов, специфически контролирующих метаболизм крахмала, по критериям оценки приоритета (Б) : а — S = 0,8 (максимальный приоритет), ген-кандидат (отмечен черным кружком) напрямую взаимодействует с тремя известными ключевыми участниками метаболизма крахмала (реперные гены, представлены кружками большего размера); б — S = 0,0346, ген-кандидат связан с реперным геном через четыре гена-посредника; в — S = 0,0115 (минимальный приоритет), ген-кандидат связан с реперным геном через хаб. Выявленный ген-кандидат с наибольшим приоритетом — potato starch branching enzyme 22.1 (отмечен стрелкой).
Еще один класс биоинформатических методов, реализованный в SOLANUM TUBEROSUM, основан на оценках обогащенности биологических процессов генами, идентифицированными в эксперименте (например, при транскриптомном анализе). Такие методы широко используются в известных компьютерных системах, предназначенных для интерпретации экспериментальных данных, например DAVID (34), PANTHER (35, 36), GORILLA (37, 38) и др.
Извлечение знаний с помощью семантико-лингвистических шаблонов. В системе ANDSystem тексты распознаются модулем лингвистического анализа, на вход которому подается текстовый поток фактографической информации, относящейся к определенной предметной (проблемной) области. Проблемно ориентированная онтология, реализованная в SOLANUM TUBEROSUM, формирует концептуальную модель знаний. В модуле лингвистического анализа используются морфологический и семантический анализаторы. Морфологический анализатор выполняет дескрипторную разметку текста (распознавание в тексте включаемых в онтологию концептов, в том числе терминологических словосочетаний), лемматизацию (приведение слова к нормальной форме), частеречную разметку. Семантический анализатор осуществляет концептуальный поиск знаний в обработанном морфологическим анализатором тексте с помщью семантико-лингвистических шаблонов. Для лемматизации и частеречной разметки используется грамматический словарь. Функциональная схема системы извлечения знаний о взаимодействиях между объектами онтологии в базе знаний SOLANUM TUBEROSUM представлена на рисунке 4.
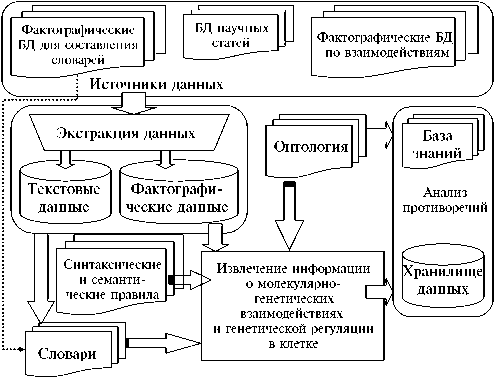
Рис. 4. Функциональная схема системы извлечения знаний о взаимодействиях между объектами онтологии в разработанной базе знаний SOLANUM TUBEROSUM.
Исходными данными при этом служат внешние источники информации, включающие три группы фактографических баз данных, используемых для составления словарей, извлечения знаний о молекулярно-генетических объектах и извлечения знаний о молекулярных взаимодействиях в клетке и генных сетях, а также базы библиографических данных (для извлечения знаний с помощью семантико-лингвистических шаблонов о взаимодействиях между объектами онтологии базы знаний SOLANUM TUBEROSUM).
Семантико-лингвистические шаблоны представляют собой структурированные записи с информацией о типах объектов, словарях, правилах анализа текстов или регулярных выражениях и метаописание семантики взаимодействия. Структура шаблона включает следующие основные группы полей: Регулярное выражение, Словари, Взаимодействия, Атрибуты объекта, Атрибуты взаимодействия. Регулярное выражение определяет порядок расположения в анализируемом предложении имен объектов и специальных слов-связок, указывающих на заданный тип взаимодействий между конкретными объектами. Структура регулярного выражения представляет собой последовательность идентификаторов словарей объектов и словарей слов-связок. В качестве разделителя между идентификаторами словарей используется символ « - ». В регулярном выражении также может указываться допустимое число слов, не являющихся именами объектов, которые могут располагаться между названиями объектов в предложении. Кроме того, регулярное выражение может содержать отрицание. Всего нами было разработано около 5000 таких семантико-лингвистических шаблонов, используемых в ANDSystem для извлечения знаний из текстов научных публикаций.
В качестве примера рассмотрим шаблон для извлечения связей между генами и фенотипами организма (см. рис. 5, A), адаптированный для базы знаний SOLANUM TUBEROSUM. В этом шаблоне в качестве объектов используются словари ГЕН, ФЕНОТИП, ОРГАНИЗМ и ТЕХНОЛОГИЯ, а в качестве слов-связок — словари «regulation» и «identifica-tion». Из регулярного выражения следует, что объект 1 (некий ген из словаря ГЕН) вовлечен в регуляцию объекта 2 (некий фенотип из словаря ФЕНОТИП). Можно видеть, что у объектов и у взаимодействий между 70
этими объектами есть свои атрибуты. В рассматриваемом примере объекты 3 и 4 — это соответственно организм и технология для объекта 1. В то же время объект 3 представляет собой атрибут взаимодействия для объекта 1, указывающий на организм, в котором оно осуществляется.
А
Структура семантических шаблонов в ANDSystem
Регулярное выражение:
Объект 1 Ключевое слово 1 Объект 2 Объект 3 Ключевое слово 2 Объект 4
Ген-/г_<пго/гег/_1п_ге£м/а//оп_о/-ФЕНОТИП-ОРГАНИЗМ-!Уега1(/1с<1П,оп_/>1'-ТЕХНОЛОГИЯ
V J
/Словари:
Объект 1 название из словаря генов GENE
Объект 2 название из словаря фенотипов PHENOTYPE
Объект 3 название из словаря организмов ORGANISM
Объект 4 название из словаря технологий TECHNOLOGY ключевое слово 1 ключевые слова, описывающие взаимодействие, из словаря REGULATION ключевое слово 2 ключевые слова, описывающие взаимодействие, из словаря IDENTIFICATION Взаимодействие:
Взаимодействие 1: <объект 1> isinvolvedinregulationof -объект 2>
Тип взаимодействия: regulation
Атрибуты объекта:
Атрибут 1: <объекг 3> is_a_organism Jor <объект 1 >
Атрибут 2: <объект 4> is_a_methodJor <объект 1>
Атрибуты взаимодействия:
Атрибут 1: Собьект 3> is a organismJor Взаимодействие 1
Б
Предложение:
Hie chromosomal location of the major gene Ryadg controlling extreme resistance to potato virus ¥ (PVY) in Solatium tuberosum subsp. andigena was identified by RFLP analisis of a diploid potato population.
Название объекта 1: gene Ryai|g
Название объекта 2: resistance to potato virus Y (PVY)
Название объекта 3: Solatium tuberosum subsp. andigena
Название объекта 4: RFLP analisis
Взаимодействие:
Взаимодействие 1: gene Ryadg is_involved_in_regulation_of resistance to potato virus Y (PVY) Тип взаимодействия: regulation
Атрибуты объекта:
Атрибут 1: Solanum tuberosum subsp. andigena is_a_organismJor gene Ryadg
Атрибут 2: RFLP analisis is_a_methodJor gene Ryadg
Атрибуты взаимодействия:
Атрибут 1: Solanum tuberosum subsp. andigena is_a_organismJor Взаимодействие 1
Рис. 5. Примеры структуры семантико-лингвистического шаблона, используемого в ANDSystem (А) , и компьютерной выдачи результатов его отработки (Б) при извлечении информации о взаимодействиях объектов в предложении из статьи J.H. Hamalainen с соавт. (39) в базе знаний SO-LANUM TUBEROSUM.
По существу, шаблон содержит всю информацию о типах объектов и типах их взаимоотношений без уточнения названий конкретных объектов. В результате отработки шаблона из текстов идентифицируются конкретные имена объектов, которые удовлетворяют заданному регулярному выражению. В случае применения рассмотренного шаблона к предложению «The chromosomal location of the major gene Ryadg controlling extreme resistance to potato virus Y (PVY) in Solanum tuberosum subsp. andigena was identified by RFLP analysis of a diploid potato population» (39) (см. рис 5, Б) выдача ответа имеет следующий вид. В рассматриваемом случае объекту 1 соответствует ген Ryadg, объекту 2 — фенотип resistance to potato virus Y (PVY), объекту 4 — организм Solanum tuberosum subsp. andigena , а объекту 5 — технология RFLP analysis.
Таким образом, создана начальная версия базы знаний для хране- ния информации по генетике, селекции, семеноводству, диагностике возбудителей заболеваний, средствам защиты и технологиям хранения картофеля, для чего разработана соответствующая онтология (включает словари понятий по генетике, селекции, феномике и семеноводству, агробиотехнологиям возделывания и биотехнологиям переработки и хранения картофеля, заболеваниям, вредителям, диагностике и средствам защиты, факторам окружающей среды и др.). Выполнена настройка методов ANDSystem для извлечения знаний из текстов научных публикаций, патентных и фактографических баз данных в предметной области, задаваемой созданной онтологией, и адаптация ранее разработанных пользовательских интерфейсов. С помощью этой системы планируется провести масштабный автоматический анализ текстов научных публикаций и патентных баз данных. Также предполагается значительно расширить объемы словарей базы знаний за счет извлечения новых имен объектов в процессе анализа.
Список литературы Разработка методов автоматического извлечения знаний из текстов научных публикаций для создания базы знаний Solanum tuberosum
- Fiehn O. Metabolomics -the link between genotypes and phenotypes. Plant Mol. Biol., 2002, 48: 155-171 ( ) DOI: 10.1023/A:1013713905833
- Kristensen T.N., Pedersen K.S., Vermeulen C.J., Loeschcke V. Research on inbreeding in the «omic» era. Trends Ecol. Evol., 2010, 25(1): 44-52 ( ) DOI: 10.1016/j.tree.2009.06.014
- Weckwerth W. Green systems biology -from single genomes, proteomes and metabolomes to ecosystems research and biotechnology. J. Proteomics, 2011, 75(1): 284-305 ( ) DOI: 10.1016/j.jprot.2011.07.010
- Kumar A., Pathak R.K., Gupta S.M., Gaur V.S., Pandey D. Systems biology for smart crops and agricultural innovation: filling the gaps between genotype and phenotype for complex traits linked with robust agricultural productivity and sustainability. OMICS: A Journal of Integrative Biology, 2015, 19(10): 581-601 ( ) DOI: 10.1089/omi.2015.0106
- Lachowiec J., Queitsch C., Kliebenstein D.J. Molecular mechanisms governing differential robustness of development and environmental responses in plants. Ann. Bot., 2016, 117(5): 795-809 ( ) DOI: 10.1093/aob/mcv151
- Lee T., Kim H., Lee I. Network-assisted crop systems genetics: network inference and integrative analysis. Curr. Opin. Plant Biol., 2015, 24: 61-70 ( ) DOI: 10.1016/j.pbi.2015.02.001
- Hammer G., Cooper M., Tardieu F., Welch S., Walsh B., van Eeuwijk F., Chapman S., Podlich D. Models for navigating biological complexity in breeding improved crop plants. Trends Plant Sci., 2006, 11(12): 587-593 ( ) DOI: 10.1016/j.tplants.2006.10.006
- Vanhaeren H., Inzé D., Gonzalez N. Plant growth beyond limits. Trends Plant Sci., 2016, 21(2): 102-109 ( ) DOI: 10.1016/j.tplants.2015.11.012
- Potato Genome Sequencing Consortium. Genome sequence and analysis of the tuber crop potato. Nature, 2011, 475(7355): 189-195 ( ) DOI: 10.1038/nature10158
- Rensink W.A., Iobst S., Hart A., Stegalkina S., Liu J., Buell C.R. Gene expression profiling of potato responses to cold, heat, and salt stress. Funct. Integr. Genomics, 2005, 5(4): 201-207 ( ) DOI: 10.1007/s10142-005-0141-6
- Ou Y., Liu X., Xie C., Zhang H., Lin Y., Li M., Song B., Liu J. Genome-wide Identification of microRNAs and their targets in cold-stored potato tubers by deep sequencing and degradome analysis. Plant Mol. Biol. Rep., 2015, 33(3): 584-597 ( ) DOI: 10.1007/s11105-014-0771-8
- Petek M., Rotter A., Kogovšek P., Baebler Š., Mithöfer A., Gruden K. Potato virus Y infection hinders potato defence response and renders plants more vulnerable to Colorado potato beetle attack. Mol. Ecol., 2014, 23(21): 5378-5391 ( ) DOI: 10.1111/mec.12932
- Chae L., Kim T., Nilo-Poyanco R., Rhee S.Y. Genomic signatures of specialized metabolism in plants. Science, 2014, 344(6183): 510-513 ( ) DOI: 10.1126/science.1252076
- Dreher K. Putting the plant metabolic network pathway databases to work: going offline to gain new capabilities. In: Plant metabolism: methods and protocols. Ser. Methods in Molecular Biology/G. Sriram (ed.). Springer Science+Business Media, NY, 2014, V. 1083: 151-171 ( ) DOI: 10.1007/978-1-62703-661-0_10
- Chae L., Lee I., Shin J., Rhee S.Y. Towards understanding how molecular networks evolve in plants. Curr. Opin. Plant Biol., 2012, 15(2): 177-184 ( ) DOI: 10.1016/j.pbi.2012.01.006
- Zhang P., Dreher K., Karthikeyan A., Chi A., Pujar A., Caspi R., Karp P., Kirkup V., Latendresse M., Lee C., Mueller L.A. Creation of a genome-wide metabolic pathway database for Populus trichocarpa using a new approach for reconstruction and curation of metabolic pathways for plants. Plant Physiol., 2010, 153(4): 1479-1491 ( ) DOI: 10.1104/pp.110.157396
- Gonzalez G.H., Tahsin T., Goodale B.C., Greene A.C., Greene C.S. Recent advances and emerging applications in text and data mining for biomedical discovery. Brief. Bioinform., 2016, 17(1): 33-42 ( ) DOI: 10.1093/bib/bbv087
- Wu H.Y., Chiang C.W., Li L. Text mining for drug-drug interaction. In: Biomedical Literature Mining. Ser. Methods in molecular biology/V.D. Kumar, H.J. Tipney (eds.). Springer Science+Business Media, NY, 2014, V. 1159: 47-75 ( ) DOI: 10.1007/978-1-4939-0709-0_4
- Piedra D., Ferrer A., Gea J. Text mining and medicine: usefulness in respiratory diseases. Archivos de Bronconeumología (Engl. Ed.), 2014, 50(3): 113-119 ( ) DOI: 10.1016/j.arbr.2014.02.008
- Fluck J., Hofmann-Apitius M. Text mining for systems biology. Drug Discov. Today, 2014, 19(2): 140-144 ( ) DOI: 10.1016/j.drudis.2013.09.012
- Krallinger M., Erhardt R.A., Valencia A. Text-mining approaches in molecular biology and biomedicine. Drug Discov. Today, 2005, 10(6): 439-445 ( ) DOI: 10.1016/S1359-6446(05)03376-3
- Szklarczyk D., Franceschini A., Wyder S., Forslund K., Heller D., Huerta-Cepas J., Simonovic M., Roth A., Santos A., Tsafou K.P., Kuhn M. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucl. Acids Res., 2014, 28: gku1003 ( ) DOI: 10.1093/nar/gku1003
- Von Mering C., Huynen M., Jaeggi D., Schmidt S., Bork P., Snel B. STRING: a database of predicted functional associations between proteins. Nucl. Acids Res., 2003, 31(1): 258-261 ( ) DOI: 10.1093/nar/gkg034
- Snel B., Lehmann G., Bork P., Huynen M.A. STRING: a web-server to retrieve and display the repeatedly occurring neighborhood of a gene. Nucl. Acids Res., 2000, 28(18): 3442-3444 ( ) DOI: 10.1093/nar/28.18.3442
- Li S., Wu L., Zhang Z. Constructing biological networks through combined literature mining and microarray analysis: a LMMA approach. Bioinformatics, 2006, 22(17): 2143-2150 ( ) DOI: 10.1093/bioinformatics/btl363
- Pesch R., Böck M., Zimmer R. ConReg: Analysis and visualization of conserved regulatory networks in eukaryotes (In: German Conference on Bioinformatics, 2012). Dagstuhl research Online Publication Server, 2012, 26: 69-81 ( ) DOI: 10.4230/OASIcs.GCB.2012.69
- Shannon P., Markiel A., Ozier O., Baliga N.S., Wang J.T., Ramage D., Amin N., Schwikowski B., Ideker T. Cytoscape: a software environment for integrated models of biomolecular interaction networks. Genome Res., 2003, 13: 2498-2504 ( ) DOI: 10.1101/gr.1239303
- Demenkov P.S., Ivanisenko T.V., Kolchanov N.A., Ivanisenko V.A. ANDVisio: a new tool for graphic visualization and analysis of literature mined associative gene networks in the ANDSystem. In Silico Biology, 2012, 11(3, 4): 149-161 ( ) DOI: 10.3233/ISB-2012-0449
- Ivanisenko V.A., Saik O.V., Ivanisenko N.V., Tiys E.S., Ivanisenko T.V., Demenkov P.S., Kolchanov N.A. ANDSystem: an Associative Network Discovery System for automated literature mining in the field of biology. BMC Syst. Biol., 2015, 9(Suppl. 2): S2 ( ) DOI: 10.1186/1752-0509-9-S2-S2
- Saik O.V., Ivanisenko T.V., Demenkov P.S., Ivanisenko V.A. Interactome of the hepatitis C virus: literature mining with ANDSystem. Virus Res., 2016, 218: 40-48 ( ) DOI: 10.1016/j.virusres.2015.12.003
- Yu B. Role of in silico tools in gene discovery. Mol. Biotechnol., 2009, 41(3): 296-306 ( ) DOI: 10.1007/s12033-008-9134-8
- Li J., Lin X., Teng Y., Qi S., Xiao D., Zhang J., Kang Y. A Comprehensive evaluation of disease phenotype networks for gene prioritization. PloS ONE, 2016, 11(7): e0159457 ( ) DOI: 10.1371/journal.pone.0159457
- Guney E., Oliva B. Exploiting protein-protein interaction networks for genome-wide disease-gene prioritization. PloS ONE, 2012, 7(9): e43557 ( ) DOI: 10.1371/journal.pone.0043557
- Huang D.W., Sherman B.T., Lempicki R.A. Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources. Nat. Protoc., 2008, 4(1): 44-57 ( ) DOI: 10.1038/nprot.2008.211
- Thomas P.D., Kejariwal A., Guo N., Mi H., Campbell M.J., Muruganujan A., Lazareva-Ulitsky B. Applications for protein sequence-function evolution data: mRNA/protein expression analysis and coding SNP scoring tools. Nucl. Acids Res., 2006, 34(Suppl 2): W645-W650 ( ) DOI: 10.1093/nar/gkl229
- Mi H., Poudel S., Muruganujan A., Casagrande J.T., Thomas P.D. PANTHER version 10: expanded protein families and functions, and analysis tools. Nucl. Acids Res., 2015, 44(D1): D336-D342 ( ) DOI: 10.1093/nar/gkv1194
- Eden E., Lipson D., Yogev S., Yakhini Z. Discovering motifs in ranked lists of DNA sequences. PLoS Comput. Biol., 2007, 3(3): e39 ( ) DOI: 10.1371/journal.pcbi.0030039
- Eden E., Navon R., Steinfeld I., Lipson D., Yakhini Z. GOrilla: a tool for discovery and visualization of enriched GO terms in ranked gene lists. BMC Bioinformatics, 2009, 10: 48 ( ) DOI: 10.1186/1471-2105-10-48
- Hämäläinen J.H., Watanabe K.N., Valkonen J.P.T., Arihara A., Plaisted R.L., Pehu E., Miller L., Slack S.A. Mapping and marker-assisted selection for a gene for extreme resistance to potato virus Y. Theor. Appl. Genet., 1997, 94(2): 192-197.


