Разработка мультиплексной панели микросателлитов для оценки достоверности происхождения и степени дифференциации популяций северного оленя Rangifer tarandus
Автор: Харзинова В.Р., Гладырь Е.А., Федоров В.И., Романенко Т.М., Шимит Л.Д., Лайшев К.А., Калашникова Л.А., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Геномное сканирование, контроль происхождения
Статья в выпуске: 6 т.50, 2015 года.
Бесплатный доступ
Северный олень ( Rangifer tarandus ), единственный представитель рода Rangifer, принадлежит к числу наиболее интересных объектов исследования генетического разнообразия. Создание панелей STR (short tandem repeats) маркеров - один из приемов изучения генетической структуры популяций и оценки достоверности происхождения животных. Целью настоящей работы стала разработка мультилокусной панели анализа STR маркеров и оценка ее информативности для контроля достоверности происхождения и изучения биоразнообразия российских популяций северного оленя. В качестве биологического материала для исследований использовали образцы тканей (пробы уха) северного оленя эвенской (EVN, n = 44), эвенкийской (EVK, n = 44), ненецкой пород ( n = 45) и оленей тувинской популяции (TUV, n = 35). ДНК выделяли с использованием колонок фирмы «Nexttec» (Германия) согласно рекомендациям фирмы-изготовителя. Полиморфизм девяти STR (NVHRT76, RT9, NVHRT24, RT30, RT1, RT6, RT27, NVHRT21, RT7) определяли по собственным методикам с использованием ДНК-анализатора ABI3130xl («Applied Biosystems», США). Статистическую обработку данных проводили с использованием пакета MS Excel 2007 с плагином GenAIEx v. 6.5, программного обеспечения MSA 4.05, Phylip, v. 3.5c, Treev32 и Structure, v. 2.3.4. Исследуемые популяции северного оленя характеризовались относительно высокой степенью генетического разнообразия. Среднее число аллелей на локус составило 6,11±0,56 у TUV; 6,67±0,50 - у NEN; 8,00±0,76 - у EVN и 8,89±0,65 - у EVK. Наименьшим числом эффективных аллелей на локус характеризовалась популяция TUV (3,37±0,47), максимальным - EVK (4,89±0,46), популяции EVN и NEN занимали промежуточное положение (соответственно 4,42±0,53 и 3,90±0,38). Число аллелей в отдельных локусах варьировало от 4 (в локусах NVHRT21 и NVHRT24 у TUV) до 12 (в локусе RT7 у EVK и в локусе RT1 у EVN). Вероятность совпадения генотипов (PI) по девяти локусам составила от 1,8½10 -9 у NEN до 5,9½10 -11 у EVK, что подтвердило высокую информативность предложенной панели в контроле достоверности происхождения. Расчет среднего значения коэффициента членства Q в i -м кластере для наиболее вероятного числа кластеров k = 3 и k = 4 (Q i/k) показал неоднородность генетической структуры всех исследованных популяций. Наибольшей генетической обособленностью характеризовались популяции TUV (Q 2/3 = 0,899±0,034, Q 3/4 = 0,883±0,035) и NEN (Q 3/3 = 0,885±0,031, Q 4/4 = 0,813±0,038). Изучаемые популяции пород EVN и EVK были дифференцированы в меньшей степени, и четкой кластеризации между ними не обнаружили. Расчет значения R st (AMOVA) показал, что общая молекулярная изменчивость была на 11,4 % обусловлена различиями между популяциями и на 88,6 % - индивидуальными различиями между животными (p st при парном сравнении были выявлены сходные тенденции. Популяция TUV характеризовалась наибольшей удаленностью от остальных (D Nei = 0,283-0,502, F st = 0,299-0,452), при этом она оказалась наиболее дифференцирована от NEN и более близка к EVN. Минимальные генетические различия отмечались между EVN и EVK (D Nei = 0,068, F st = 0,032). Полученные результаты подтвердили высокую функциональную емкость разработанной STR панели как для анализа достоверности происхождения, так и для изучения биоразнообразия российских популяций северного оленя.
Породы северного оленя, микросателлиты, биоразнообразие
Короткий адрес: https://sciup.org/142133639
IDR: 142133639 | УДК: 636.294:575.174:575.113:577.2.08:51-76 | DOI: 10.15389/agrobiology.2015.6.756rus
Текст научной статьи Разработка мультиплексной панели микросателлитов для оценки достоверности происхождения и степени дифференциации популяций северного оленя Rangifer tarandus
Исследование генетической структуры видов и пород сельскохозяйственных животных с помощью молекулярно-генетических методов — важная составляющая в системе сохранения и использования биологических ресурсов (1), однако если у основных видов домашних животных она охарактери-
∗ Исследования выполнены при поддержке Российского научного фонда, проект ¹ 14-36-00039. При проведении исследований было использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ВИЖ им. академика Л.К. Эрнста.
зована достаточно полно, то многие виды, имеющие значение только для отдельных регионов или коренных народов, остаются малоизученными.
Северный олень ( Rangifer tarandus ) — важнейший элемент экосистем Севера и неотъемлемая часть жизни и культуры коренных народов (2). Этот вид — единственный представитель рода Северные олени ( Rangifer ), относящегося к семейству Оленевые подотряда Жвачные, и один из немногих видов животных, у которых дикая форма сосуществует с домашней. Большинство ученых считают, что северный олень образует 12-15 подвидов, в том числе 5-7 подвидов на территории России. Так, по одним представлениям, все олени европейского Севера принадлежат к подвиду R. tarandus tarandus L. — европейский северный олень (3-5). Согласно другим, в европейском ареале обитают 2 подвида: R. t. fennicus L o nn (европейский лесной северный олень) и R. t. tarandus L. (европейский тундровый северный олень) (6-9).
Процессы породообразования в северном оленеводстве имеют много общего с генезисом пород других видов одомашненных животных — лошадей, овец, яков, верблюдов и т.д. В основе породной специализации лежит народная традиционно сложившаяся система селекции и полное отсутствие коммерческой мотивации, которая позднее появилась при выведении многих заводских пород в Европе и Северной Америке (10). Общепринято выделять четыре аборигенных породы полуодомашненных северных оленей, которые созданы северными народностями России и различаются направлением продуктивности, экстерьерно-конституциональными особенностями и приспособленностью к конкретным природно-климатическим условиям, — ненецкую, чукотскую, эвенкийскую и эвенскую (11).
Оленей ненецкой породы разводят на севере европейской части России и за Уралом, в низовьях Оби и Енисея. В настоящее время эта порода самая многочисленная и процветающая. Чукотские олени обитают на территории Чукотского и Камчатского полуостровов и северо-востока Якутии. Эвенкийскую породу разводят в таежной зоне Сибири и Дальнего Востока от реки Енисей до побережья Охотского моря и на острове Сахалин. В породе выделяют несколько внутрипородных групп. Горно-таежные районы Якутии, Магаданская и Камчатская области — ареал эвенской породы (по своему типу занимает промежуточное положение между чукотской и эвенкийской). С эвенской породой ведется работа по консолидации, изучению генетической структуры и биологии. У северных оленей также выделяют несколько породных групп, обладающих экстерьерно-конституциональными особенностями, но пока не получивших статуса пород. Так, представляют интерес животные тувинской популяции (Тоджинский р-н, Республика Тыва), которые выделяются крупностью. Ряд ученых относят их к самостоятельной породной группе тувино-тофаларского типа (12).
В настоящее время племенная работа в оленеводстве ведется традиционными методами, при этом наиболее полное представление о структуре популяций можно получить на основе анализа по комплексу различных биологических параметров, включая генетические.
Изучение генетического разнообразия северного оленя всегда вызывало повышенный интерес. Исследования начались еще в 1960-х годах, когда в популяционной генетике стал широко применяться простой и быстрый метод электрофореза в агаровом, крахмальном и акриламидном геле. Позже появились сообщения об исследовании полиморфизма трансферринов сыворотки крови (13-16). С развитием молекулярной генетики стало возможным применение более тонких методов, позволяющих проводить анализ непосредственно на уровне ДНК и имеющих больший потенциал по сравнению с маркерными системами, используемыми ранее (17, 18).
Информативность ДНК-анализа во многом определяется типом и количеством используемых ДНК-маркеров. В этой связи актуально создание мультиплексных панелей анализа ДНК-маркеров для изучения генетических особенностей видов и пород животных. Среди многообразия маркерных систем наибольшее распространение получили микросателлиты, известные также как короткие тандемные повторы (short tandem repeats, STR). Благодаря высокой степени полиморфизма, менделевскому типу наследования и равномерному распределению по всему геному STR панели используются на всех основных видах сельскохозяйственных животных (19). Они находят применение в контроле достоверности происхождения, определении степени инбридинга, оценке чистопородности, характеристике биоразнообразия и степени генетической дифференциации пород и внутрипород-ных генетических структур (20-23). В настоящее время имеется множество публикаций, в которых показана прикладная значимость STR в характеристике генофонда северного оленя (24-32), однако исследования российских пород и популяций северного оленя до настоящего времени не проводились.
Цель представленной работы заключалась в разработке мультиплексной STR панели для оценки достоверности происхождения и степени дифференциации российских аборигенных популяций северного оленя.
Методика . Исследования проводили на оленях ( Rangifer tarandus ) эвенской (EVN, ФГУП «Ючюгейское», Республика Саха, n = 44), эвенкийской (EVK, НАОК «Таба», Республика Саха, n = 44), ненецкой пород (NEN, СПК «Индига», Ненецкий автономный округ, n = 45) и тувинской популяции (TUV, Тоджа, Республика Тыва, n = 35). В качестве биологического материала использовали образцы тканей уха. Выделение ДНК осуществляли с помощью колонок Nexttec («Nexttec™ Biotechnologie GmbH», Германия) согласно рекомендациям фирмы-изготовителя.
По результатам анализа специальной литературы были отобраны 9 STR маркеров, в том числе 3 локуса, разработанных для северных оленей, — NVHRT21, NVHRT24, NVHRT76 (18), и 6 локусов, разработанных для оленей карибу, — RT1, RT6, RT7, RT9, RT27, RT30 (33), сформированные в одну мультиплексную панель. ПЦР проводили в конечном объеме 15 мкл. В пробирки вносили по 14 мкл реакционной смеси, состоящей из 1,5 мкл 10½ ПЦР-буфера, 1,5 мкл 2 мМ раствора dNTPs, 0,15 мкл 100 мМ МgСl2, 2,54 мкл 10 мМ смеси праймеров, 0,2 мкл (1 ед.) Таq-полимеразы («Диалат Лтд.», Россия), 8,01 мкл бидистиллированной воды, и добавляли 1 мкл (50-100 нг) исследуемой геномной ДНК. Состав ПЦР-буфера: 16,6 мМ (NH4)2SO4, 67,7 мМ Трис-НС1 (рН = 8,8), 0,1 объема Tween 20. После начальной денатурации (94 °С, 5 мин) выполняли 30 циклов амплификации (95 °С, 1 мин; 55 °С, 30 с; 72 °С, 1 мин). Режим отжига рассчитывали с учетом температуры плавления праймеров. Фрагменты исследовали на генетическом анализаторе ABI3130xl («Applied Biosystems», США). Размеры аллелей определяли с помощью программного обеспечения Gene Mapper v. 4 («Applied Biosystems», США). Для обработки результатов анализа формировали матрицу генотипов в формате Microsoft Excel.
Для характеристики аллелофонда определяли среднее число аллелей (Na) и число эффективных аллелей (Ne) в расчете на локус, степень наблюдаемой (Ho) и ожидаемой гетерозиготности (He), информационный индекс Шеннона (I) и индекс фиксации (Fis) (34). Информативность разработанной STR панели для контроля достоверности происхождения оценивали по показателям вероятности совпадения генотипов индивидуумов (PI) (35) и вероятности исключения в качестве родителей (P1, P2, P3) (36, 37).
Генетическую структуру популяций оценивали с помощью кластерного анализа по методу J.K. Pritchard с соавт. (38) без введения предварительной информации о популяционной принадлежности индивидуумов. 758
Для каждой из групп рассчитывали среднее значение коэффициента членства Q в i -м кластере для общего числа кластеров k (Qi/k). Долю межпопуляционной изменчивости в общей изменчивости определяли по показателю Rst (AMOVA) (39). Степень генетической дифференциации популяций оценивали на основании значений Fst (40) и генетических дистанций по M. Nei (41).
Статистическую обработку данных проводили с использованием пакета MS Excel 2007 с плагином GenAIEx v. 6.5.1 (42), программного обеспечения Phylip (v. 3.5c) (43) и Structure 2.3.4 (38).
Результаты. Выбор микросателлитных локусов для включения в STR панель осуществляли с учетом их полиморфного характера, а также исходя из возможности их амплификации в одной реакции (близкие температуры плавления праймеров) и анализа на ДНК-анализаторе при использовании специфических флуоресцентных меток (спектры для каждого из красителей не перекрываются). В результате нами была предложена STR панель, позволяющая проводить одновременный анализ 9 локусов (рис. 1).
|
92-110 пн. |
116-128 п.н. |
J [ |
147-155 п.н. |
190-220 п.н. |
222-240 п.н. |
|
|
NVHRT76 |
RT9 |
NVHRT24 |
RT30 |
RT1 |
||
|
1 1_________1_________l_ |
1 |
1 |
1 1 1 |
1 |
1 |
_1______1350 |
|
. 105-117 |
135-155 |
, . 159-167 , |
216-234 । |
|||
|
п.н. |
П.Н. |
J । пн | |
П.Н. |
|||
|
RT6 |
RT27 |
NVHRT21 |
RT7 |
Рис. 1 . Схема мультиплексной тест-системы, разработанной для анализа 9 STR у северного оленя Rangifer tarandus . Фрагменты, меченные специфическими флуоресцентными красителями FAM и HEX, показаны соответственно серым и черным цветом.
Исследованные породы характеризовались относительно высоким генетическим разнообразием: среднее число аллелей на локус составило 6,11±0,56 у TUV, 6,67±0,50 — у NEN, 8,00±0,76 — у EVN и 8,89±0,65 — у EVK. Такая же закономерность наблюдалась в отношении числа эффективных аллелей: наименьшее значение этого показателя отмечено в группе TUV (3,37±0,47 аллелей на локус), максимальное — у EVK (4,89±0,46 аллелей на локус), группы EVN и NEN занимали промежуточное положение (соответственно 4,42±0,53 и 3,90±0,38). Число аллелей в отдельных локусах варьировало от 4 (в локусах NVHRT21 и NVHRT24 у TUV) до 12 (в локусе RT7 у EVK и в локусе RT1 у EVN). Расчет Ho и He показал, что во всех группах наблюдался существенный дефицит гетерозигот (от 10 до 16 %). Это подтверждалось также высокими положительными значениями индекса Fis. Вместе с тем значения информационного индекса Шеннона указывали на достаточно высокое генетическое разнообразие в трех из четырех исследованных групп (I ≥ 1,5), за исключением TUV (табл. 1).
1. Генетическое разнообразие изучаемых пород северного оленя Rangifer tarandus по локусам микросателлитов
|
Порода/географи-ческая группа |
Степень гетерозиготности |
H o - H e |
I |
F is |
|
|
H o |
H e |
||||
|
EVN |
0,63±0,04 |
0,74±0,03 |
- 0,11 |
1,608±0,108 |
0,15±0,05 |
|
EVK |
0,68±0,04 |
0,78±0,02 |
- 0,10 |
1,752±0,089 |
0,13±0,04 |
|
NEN |
0,56±0,05 |
0,72±0,03 |
- 0,16 |
1,517±0,089 |
0,21±0,08 |
|
TUV |
0,55±0,06 |
0,67±0,04 |
- 0,12 |
1,363±0,111 |
0,18±0,07 |
|
В среднем |
0,61±0,03 |
0,73±0,02 |
- 0,12 |
1,560±0,053 |
0,17±0,03 |
П р и м еч а ни е. EVN, EVK, NEN, TUV — соответственно эвенская, эвенкийская, ненецкая породы и тувинская популяция (описание см. в разделе «Методика»); Ho — наблюдаемая гетерозиготность He — ожидаемая гетерозиготность, I — индекс Шеннона, Fis — индекс фиксации.
При оценке информативности разработанной STR панели вероятность совпадения генотипов (PI) составила от 1,84½10-9 у NEN до
5,9½10-11 у EVK, то есть была практически исключена. Значения показателей вероятности исключения (P1, P2, P3), рассчитанные по результатам анализа с использованием STR панели, были минимальными у TUV и максимальными у EVK и варьировали от 99,61 до 99,97 % для P1 (исключение одного из родителей, если генотипы обоих известны), от 95,36 до 99,30 % для P2 (исключение в качестве родителя, если известен генотип только одного родителя) и от 99,94 до > 99,99 % для P3 (исключение обоих родителей). Полученные данные подтверждают высокую информативность ДНК-анализа для оценки достоверности происхождения у северного оленя и функциональную емкость разработанной STR панели.
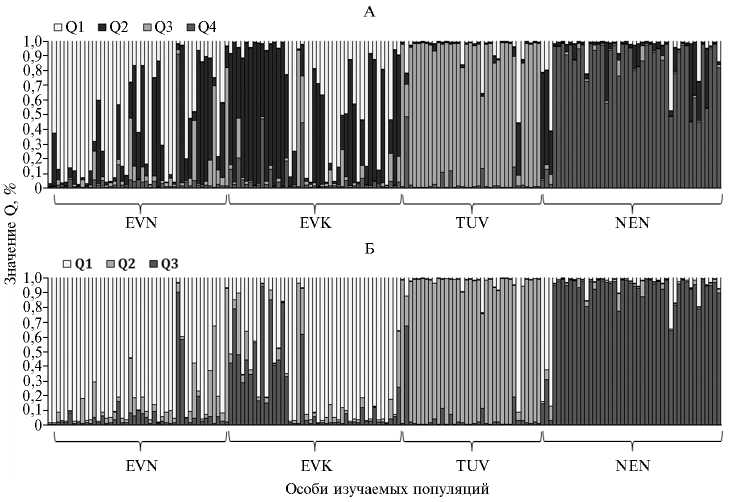
Рис. 2. Результаты анализа генетической структуры изучаемых популяций северного оленя Rangifer tarandus для числа кластеров k = 3 (A) и k = 4 (Б) : EVN, EVK, NEN, TUV — соответственно эвенская, эвенкийская, ненецкая породы и тувинская популяция (описание см. в разделе «Методика»); Q1-Q4 — значение коэффициента членства Q в соответствующем кластере. Значения Q рассчитаны по J.K. Pritchard с соавт. (38).
Результаты кластерного анализа для наиболее вероятного числа кластеров k = 3 (рис. 2, А) и k = 4 (см. рис. 2, Б) показали неоднородность генетической структуры исследованных популяций. Наибольшей генетической обособленностью характеризовались группы TUV (Q2/3 = 0,899±0,034, Q3/4 = 0,883±0,035) и NEN (Q3/3 = 0,885±0,031, Q4/4 = 0,813±0,038), при этом следует отметить, что в популяции TUV выделялась одна (Q1/3 = 0,912, Q1/4 + Q2/4 = 0,914), а в популяции NEN — три особи (Q1/3 = 0,624-0,870, Q1/4 + Q2/4 = 0,838-0,935), имеющие генетически иное происхождение, близкое к группам EVN и EVK (кластеры Q1/3 и Q1/4 + Q2/4). Одна особь в популяции TUV была генетически близка к NEN (Q3/3 = 0,676 и Q4/4 = 0,485).
Популяции пород EVN и EVK оказались менее дифференцированными, и четкой кластеризации между ними обнаружить не удалось. Среднее значение коэффициента подобия составило Q1/3 = 0,768 ± 0,030 и Q1/4 + Q2/4 = 0,874±0,021. При этом две особи в группе EVN генетически были отнесены к группам TUV (Q2/3 = 0,666 и Q3/4 = 0,690) и NEN (Q3/3 = 0,903 и Q4/4 = 0,912), а в группе EVK двух особей отнесли к TUV (Q2/3 = 0,905-0,950 и Q3/4 = 0,808-0,927).
Результаты кластерного анализа подтвердили данные расчета генетических дистанций по M. Nei (рис. 3). Близкая генетическая связь EVN и EVK, с одной стороны, и неоднородность их генетической структуры — с 760
другой проявлялись в формировании на генеалогическом дереве общих кластеров. Олени TUV (за исключением трех особей) объединялись в отдельный кластер, в который также входили 4 особи трех других популяций. В NEN выделялась большая группа особей (32 из 45 исследованных), формировавших отдельный кластер, в который также вошли 4 особи EVN и 1 EVK. Животные EVN и EVK формировали несколько смешанных кластеров, и в некоторых из них локализовались животные TUV (3 особи) и NEN (13 особей).
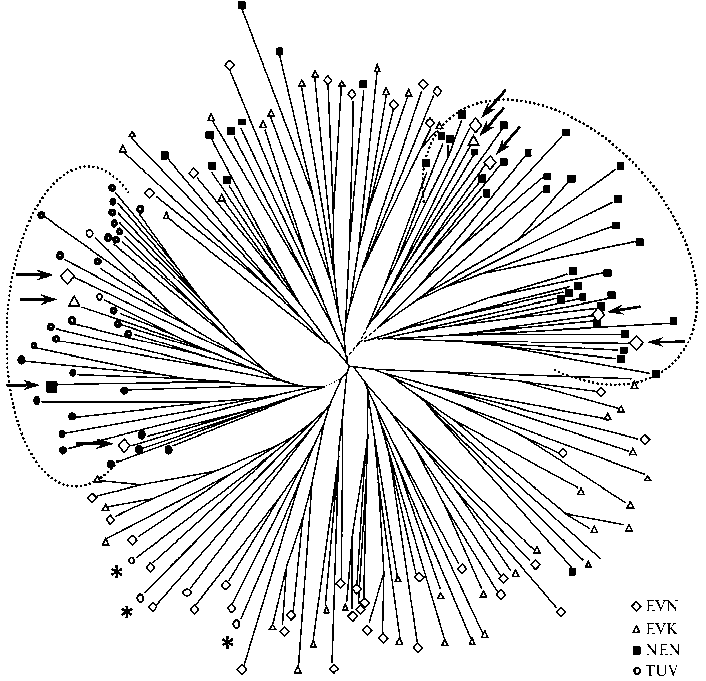
Рис. 3 . Дендрограмма генетических дистанций для особей изучаемых популяций северного оленя Rangifer tarandus , построенная по M. Nei (41) методом UPGMA: EVN, EVK, NEN, TUV — соответственно эвенская, эвенкийская, ненецкая породы и тувинская популяция (описание см. в разделе «Методика»). Звездочкой отмечены особи TUV, попавшие в «чужой» кластер, стрелками — особи других популяций, находящиеся в кластерах TUV и NEN.
2. Генетическая дифференциация изучаемых популяций северного оленя Rangifer tarandus
|
Порода/географическая группа |
EVN |
EVK |
NEN |
TUV |
|
EVN |
0,068 |
0,259 |
0,283 |
|
|
EVK |
0,032 |
0,273 |
0,356 |
|
|
NEN |
0,251 |
0,251 |
0,502 |
|
|
TUV |
0,299 |
0,347 |
0,452 |
П р и м еч ани е. EVN, EVK, NEN, TUV — соответственно эвенская, эвенкийская, ненецкая породы и тувинская популяция (описание см. в разделе «Методика»). Над диагональю — генетические дистанции по M. Nei (41); под диагональю — значения Fst при парном сравнении.
Расчет значения Rst (AMOVA) показал, что общая молекулярная изменчивость на 11,4 % была обусловлена различиями между популяциями и на 88,6 % — индивидуальными различиями между животными (p < 0,01). При оценке степени генетической дифференциации популяций с использованием в качестве критериев значений генетических дистанций по M. Nei и индекса Fst при парном сравнении были обнаружены сходные тенденции. Популяция TUV характеризовалась наибольшей удаленностью от остальных, при этом она была наиболее дифференцирована от NEN и близка к EVN. Популяция TUV оказалась практически равноудалена от EVN и EVK. Минимальные генетические различия отмечались между EVN и EVK (табл. 2).
Для оценки биоразнообразия северных оленей находят применение
STR панели, различающиеся как количеством, так и спектром используемых маркеров. K. R 0 ed и L. Midthjell (18) выполняли исследование двух полуодомашненных популяций северного оленя в Норвегии с помощью 23 STR R. tarandus , 6 из которых оказались мономорфны, а 17 характеризовались средним числом аллелей на локус от 3 до 10. A. McDevitt с соавт. описали генетическую структуру популяций R. t. groenlandicus и R. t. caribou в Северной Америке с использованием 11 STR (25). G. Wilson с соавт. (33) показали, что из 13 STR, разработанных для оленей карибу, 12 полиморфны в канадской популяции северных оленей с числом аллелей на локус от 4 до 8. S. C o t e с соавт. (44) использовали 14 STR для сравнительного изучения разнообразия северного оленя в двух популяциях Шпицбергена (число аллелей на локус — от 1 до 5). R. Courtois и L. Bernatchez (45) применяли 8 STR, включая 7 локусов, разработанных для оленей карибу (33), и один STR крупного рогатого скота для оценки биоразнообразия семи популяций оленя карибу Северной Америки (число аллелей на локус — от 3 до 20).
Нами предложена мультилокусная панель, позволяющая одновременно анализировать девять STR. Все они оказались полиморфными в исследованных российских популяциях северного оленя: число аллелей на локус варьировало от 4 в локусах NVHRT21 и NVHRT24 в популяции TUV до 12 в локусе RT1 в популяции EVN и в локусе RT7 в популяции NEN. Российские популяции R. tarandus характеризовались более высоким генетическим разнообразием в отношении числа аллелей на локус по сравнению с другими рассмотренными популяциями (табл. 3).
3. Сравнительный анализ биоразнообразия девяти STR в популяциях северных оленей Rangifer tarandus
|
STR |
Число аллелей в локусе STR в популяциях |
||||||
|
EVN |
EVK |
NEN |
TUV |
А |
Б |
В |
|
|
Число голов, n |
44 |
44 |
45 |
35 |
23 182 |
561 |
|
|
NVHRT21 |
5 |
8 |
6 |
4 |
5 |
- |
- |
|
NVHRT24 |
6 |
6 |
5 |
4 |
5 |
- |
- |
|
NVHRT76 |
6 |
7 |
6 |
6 |
6 |
- |
- |
|
RT1 |
12 |
11 |
9 |
6 |
- |
2 |
2 |
|
RT6 |
7 |
8 |
9 |
5 |
- |
2 |
2 |
|
RT7 |
11 |
12 |
7 |
9 |
- |
4 |
3 |
|
RT9 |
8 |
8 |
7 |
7 |
- |
1 |
1 |
|
RT27 |
8 |
10 |
6 |
6 |
- |
- |
- |
|
RT30 |
7 |
10 |
5 |
8 |
- |
2 |
2 |
П р и м еч а ни е. EVN, EVK, NEN, TUV — соответственно эвенская, эвенкийская, ненецкая породы и тувинская популяция (описание см. в разделе «Методика»); А — два стада полуодомашненных северных оленей центральной Норвегии (18), Б — полуодомашненные северные олени, Сассендален, Шпицберген (44), В — полуодомашненные северные олени, Колесдален/Рейндален, Шпицберген (44). Прочерк означает, что локусы не исследовались.
В работах некоторых авторов (44, 45) установлено меньшее генетическое разнообразие в изолированных популяциях северных оленей Европы и Северной Америки, что нашло подтверждение в полученных нами результатах для российских популяций R. tarandus . Наименьшим генетическим разнообразием, оцененным по среднему числу аллелей на локус, степени наблюдаемой гетерозиготности и информационному индексу Шеннона, характеризовалась популяция TUV, которая географически изолирована от других исследованных популяций, наибольшим — EVN и EVK с перекрывающимися ареалами. Одной из причин высокой гетерогенности популяции EVK и ее сходства с EVN может быть прилитие крови эвенской породы из Оймяконского района Республики Саха (Якутия). Поэтому для дальнейшей оценки генофонда российских пород северных оленей необходимо расширять число и географическое разнообразие исследуемых популяций, а также использовать генетически чистые популяции (длительно разводимые без прилития крови и географически изолированные).
Таким образом, показана неоднородность генетической структуры всех изученных популяций северного оленя, при этом наибольшей степенью генетической чистоты и обособленности характеризовалась тувинская, наименьшей — эвенская и эвенкийская. Полученные результаты демонстрируют возможность использования разработанной мультиплексной панели микросателлитов в качестве одного из приемов выявления генетических различий между популяциями. Это поможет выяснить историю происхождения, границы и пути распространения отдельных популяций и послужит основой для дальнейшего изучения разнообразия одного из самых уникальных представителей семейства Cervidae. Кроме того, предложенная нами мультилокусная панель служит инструментом в оценке достоверности происхождения индивидуумов. Ее применение позволит кардинально усовершенствовать селекционно-племенную работу с породами и разработать систему геномной оценки в северном оленеводстве.
Список литературы Разработка мультиплексной панели микросателлитов для оценки достоверности происхождения и степени дифференциации популяций северного оленя Rangifer tarandus
- Столповский Ю.А. Популяционно-генетические основы сохранения генофондов доместицированных видов животных. Вавиловский журнал генетики и селекции, 2013, 17(4/2): 900-902.
- Давыдов A.B. Морфологическая и генетическая дифференциация популяций северного оленя Евразии. Автореф. канд. дис. М., 1997.
- Флеров К.К. Фауна СССР. Млекопитающие. Кабарги и олени. М., 1952.
- Гептнер В.Г., Насимович А.А., Банников А.Г. Млекопитающие Советского Союза. М., 1961.
- Данилкин А.А. Оленьи (Cervidae). М., 1999.
- Banfield A.W.F. A revision of the reindeer and caribou genus, Rangifer. Bulletin № 177. National Museum of Canada, Ottawa, 1961.
- Куприянов А.Г. Дикий северный олень на Европейском Севере России. Мат. 2-го Межд. симп. «Динамика популяций охотничьих животных Северной Европы». Петрозаводск, 1998: 25-26.
- Данилов П.И. Охотничьи животные Карелии: экология, ресурсы, управление, охрана. М., 2005.
- Данилов П.И., Панченко Д.В. Изменение распространения и численности копытных зверей на европейском севере России. Современные проблемы природопользования, охотоведения и звероводства, 2012, 1: 88-92.
- Южаков А.А., Мухачев А.Д. Социальное значение северного оленеводства. Аграрная наука, 2002, 10: 7-8.
- Эрнст Л.К., Дмитриев Н.Г., Паронян И.А. Генетические ресурсы сельскохозяйственных животных в России и сопредельных странах. СПб, 1994.
- Помишин С.Б. Проблемы породы и ее совершенствование в оленеводстве. Якутск, 1981.
- Storset A., Olaisen B., Wika M., Bjarghov R. Genetic markers in the Spitzbergen reindeer. Hereditas, 1978, 88: 113-115.
- Baccus R., Ryman N., Smith M.H., Reuterwall C., Cameron D. Genetic variability and differentiation of large grazing mammals. J. Mammalogy, 1983, 64: 109-120 ( ) DOI: 10.2307/1380756
- Roed K.H. Genetic differences at the transferrin locus in Norwegian semidomestic and wild reindeer (Rangifer tarandus L.). Hereditas, 1985, 102: 199-206.
- Cronin M.A., Renecker L., Pierson B.J., Patton J.C. Genetic variation in domestic reindeer and wild caribou in Alaska. Anim. Genet., 1995, 26: 427-434.
- Романенко Т.М., Калашникова Л.А., Филиппова Г.И., Лайшев К.А. Генетическая структура популяции северных оленей О. Колгуев ненецкого автономного округа. Достижения науки и техники АПК, 2014, 4: 68-71.
- Røed K.H., Midthjell L. Microsatellites in reindeer, Rangifer tarandus, and their use in other cervids. Mol. Ecol., 1998, 7: 1773-1776 ( ) DOI: 10.1046/j.1365-294x.1998.00514.x
- Зиновьева Н.А., Харзинова В.Р., Логвинова Т.И., Гладырь Е.А., Сизарева Е.И., Чинаров Ю.И. Микросателлитные профили как критерии определения чистопородности и оценки степени гетерогенности подборов родительских пар в свиноводстве. Сельскохозяйственная биология, 2011, 6: 47-53.
- Weber J.L., May P.E. Abundant class of human DNA polymorphisms which can be typed using the polymerase chain reaction. Am. J. Hum. Genet., 1989, 44(3): 388-396.
- Tautz D. Hypervariability of simple sequences as a general source for polymorphic DNA markers. Nuc. Acids Res., 1989, 17: 6463-6471.
- DeWoody J., Avise J.C. Microsatellite variation in marine, freshwater and anadromous fishes compared with other animals. J. Fish Biology, 2000, 56: 461-473 ( ) DOI: 10.1111/j.1095-8649.2000.tb00748.x
- Primmer C.R., Ellegren H., Saino N., Møller A.P. Directional evolution in germline microsatellite mutations. Nat. Genet., 1996, 13: 391-393.
- Ball M.C., Finnegan L., Manseau M., Wilson P. Integrating multiple analytical approaches to spatially delineate and characterize genetic population structure: an application to boreal caribou (Rangifer tarandus caribou) in central Canada. Conserv. Genet., 2010, 11: 2131-2143 ( ) DOI: 10.1007/s10592-010-0099-3
- McDevitt A.D., Mariani S., Hebblewhite M., DeCesare N.J., Morgantini L., Seip D., Weckworth B.V., Musiani M. Survival in the Rockies of an endangered hybrid swarm from diverged caribou (Rangifer tarandus) lineages. Mol. Ecol., 2009, 18: 665-679 ( ) DOI: 10.1111/j.1365-294X.2008.04050.x
- Zittlau K.A., Coffin J., Farnell R.S., Kuzyk G.W., Strobeck C. Genetic relationships of three Yukon caribou herds determined by DNA typing. Rangifer, 2000, 12: 59-62 ( ) DOI: 10.7557/2.20.5.1625
- Кол Н.В., Лазебный О.Е. Полиморфизм ISSR-PCR маркеров в тувинской популяции северного оленя (Rangifer tarandus L.). Генетика, 2006, 42(12): 1731-1734.
- Cronin M.A. Intraspecific variation in mitochondrial DNA of North American cervids. J. Mammalogy, 1992, 73(1): 70-82 ( ) DOI: 10.2307/1381867
- Cronin M.A., MacNeil M.D., Patton J.C. Variation in mitochondrial DNA and microsatellite DNA in caribou (Rangifer tarandus) in North America. J. Mammalogy, 2005, 86: 495-505 (doi: 10.1644/1545-1542(2005)86%5B495:VIMDAM%5D2.0.CO;2).
- Engel S.R., Linn R.A., Taylor J.F., Davis S.K. Conservation of microsatellite loci across species of artiodactyls: implications for population studies. J. Mammalogy, 1996, 77: 504-518 ( ) DOI: 10.2307/1382825
- Zittlau K.A. Population genetic analyses of North American caribou (Rangifer tarandus). PhD Thesis. University of Alberta, Canada, 2004.
- Tyler S.K., McFarlane K.A., Pamela Groves P., Mooers A.Ø., Shapiro B. Modern and ancient DNA reveal recent partial replacement of caribou in the southwest Yukon. Mol. Ecol., 2010, 19: 1312-1323 ( ) DOI: 10.1111/j.1365-294X.2010.04565.x
- Wilson G.A., Strobeck C., Wu L., Coffin J.W. Characterization of microsatellite loci in caribou Rangifer tarandus, and their use in other artiodactyls. Mol. Ecol., 1997, 65: 697-699 ( ) DOI: 10.1046/j.1365-294X.1997.00237.x
- Hartl D.L., Clark A.G. Principles of population genetics. Massachusetts, 1997.
- Waits L.P., Luikart G., Taberlet P. Estimating the probability of identity among genotypes in natural populations: cautions and guidelines. Mol. Ecol., 2001, 10: 249-256 ( ) DOI: 10.1046/j.1365-294X.2001.01185.x
- Jamieson A. The effectiveness of using co-dominant polymorphic allelic series for (1) checking pedigrees and (2) distinguishing full-sib pair members. Anim. Genet., 1994, 25(1): 37-44 ( ) DOI: 10.1111/j.1365-2052.1994.tb00401.x
- Jamieson A., Taylor S.C.S. Comparisons of three probability formulae for parentage exclusion. Anim. Genet., 1997, 28: 397-400 ( ) DOI: 10.1111/j.1365-2052.1997.00186.x
- Pritchard J.K., Stephens M., Donnelly P. Inference of population structure using multilocus genotype data. Genetics, 2000, 155: 945-959.
- Slatkin M.A. A measure of population subdivision based on microsatellite allele frequencies. Genetics, 1995, 139: 457-462.
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38: 1358-1370.
- Nei M. F-statistics and analysis of gene diversity in subdivided populations. Ann. Hum. Genet., 1977, 41: 225-233.
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research-an update. Bioinformatics, 2012, 28: 2537-2539 ( ) DOI: 10.1093/bioinformatics/bts460
- Felsenstein J. Phylip (Phylogeny Inference Package) version 3.5c. Seattle, 1993.
- Côté S.D., Dallas J.F., Marshall F., Irvine R.J., Langvatn R., Albon S.D. Microsatellite DNA evidence for genetic drift and philopatry in Svalbard reindeer. Mol. Ecol., 2002, 11: 1923-1930 ( ) DOI: 10.1046/j.1365-294X.2002.01582.x
- Courtois R., Bernatchez L., Ouellet J.-P., Breton L. Significance of caribou (Rangifer tarandus) ecotypes from a molecular genetics viewpoint. Conservation Genetics, 2003, 4: 393-404.


