Разработка ПЦР-тест системы для выявления вируса картофеля Y
Автор: Бессолицына Е.А., Тулинов А.Г., Новоселова Н.В., Харина А.В.
Журнал: Известия Коми научного центра УрО РАН @izvestia-komisc
Рубрика: Растениеводство
Статья в выпуске: 7 (65), 2023 года.
Бесплатный доступ
Вирус картофеля Y поражает растения картофеля, нанося серьезный урон сельскому хозяйству за счет снижения урожайности данной культуры. Поэтому существует необходимость выявления данного возбудителя. Вирусы картофеля РНК-содержащие, поэтому могут быть использованы иммунологические методы или ПЦР, совмещенный с обратной транскрипцией. В ходе данной работы была разработана основа для ПЦР-тест системы для выявления вируса картофеля Y. Подобраны праймеры для реакции обратной транскрипции и последующей ПЦР, рассчитаны температуры отжига и размер амплифицируемого фрагмента. Проверены праймеры и условия реакции на растительном материале. Получены ПЦР-продукты рассчитанного размера. Определение их нуклеотидной последовательности подтвердило выявление генетического материала вируса картофеля Y. Таким образом, данная ПЦР-тест система может быть использована для выявления вируса картофеля Y.
Картофель, диагностика заболеваний, вирус y, праймеры, пцр-диагностика, пцр-тест система, обратная транскрипция
Короткий адрес: https://sciup.org/149144400
IDR: 149144400 | УДК: 578.85/.86 | DOI: 10.19110/1994-5655-2023-7-5-11
Текст научной статьи Разработка ПЦР-тест системы для выявления вируса картофеля Y
В настоящее время известно 52 вида вирусов, поражающих картофель [1]. В Российской Федерации (далее – РФ) пять наиболее опасных видов: вирус картофеля Y (PVY), вирус картофеля X (PVX), вирус скручивания листьев (PLRV) и вироид веретеновидности клубней (PSTVd). Менее опасными, но при этом увеличивающими потери урожая при совместном заражении с другими вирусами, являются вирусы картофеля М и S (PVM и PVS соответственно) [2].
Вирус картофеля Y вызывает серьезные потери урожая картофеля (до 30 % и более в зависимости от времени воздействия, типа вируса, устойчивости сорта растения-хозя- ина), причем он поражает не только картофель, но и другие сельскохозяйственные культуры как в РФ, так и в других регионах планеты. Все эти факторы делают вирус картофеля Y экономически важным фитопатогеном [3, 4].
Вирус картофеля Y относится к роду Potyvirus, семейству Potiviridae. Вирусные частицы нитевидные, размер составляет 730×11 нм, осаждаются как единый компонент с коэффициентом седиментации около 150S [5]. Геном представлен одноцепочечной позитивной молекулой РНК, длина которой составляет 9.7 тыс. нуклеотидов, 5’-конец РНК ковалентно соединен 5’-концевым терминальным белком (Vpg), на 3’-конце располагается полиА-тракт [6]. Молекула РНК содержит 10 открытых рамок считывания, транслируемых одним полибелком [7].
Вирус картофеля Y передается при контакте поврежденных клубней, а также через семена. Другим агентом распространения вируса являются тли [8].
Симптомы заражения вирусом картофеля Y вариабельные и зависят от таких факторов, как штамм вируса, сорт и условия выращивания растения-хозяина. Может развиваться резкий некроз вдоль жилок, вплоть до полного отмирания листьев. Некроз может быть мозаичным, и сопровождаться морщинистостью листьев, которая может проявляться и без некротических изменений [9].
Множественность путей распространения вируса, вариабельность симптомов выявляют необходимость разработки достаточно быстрого, точного, чувствительного метода для выявления вируса как в организме растения-хозяина, так и в организмах переносчиков.
В настоящее время обычно используется метод имму-ноферментного анализа (далее – ИФА) – это высокоточный и высокочувствительный метод, но он достаточно дорог, и для него может быть использован только свежий материал [10]. Второй метод, активно используемый для выявления возбудителей заболеваний, – метод ПЦР, но его классический вариант нельзя применять, так как матрицей для ПЦР является молекула ДНК, а вирус картофеля Y (PVY) РНК-содержащий. Но существует модификация метода ПЦР – обратная транскрипция ПЦР (далее – ОТ-ПЦР), которая позволяет выявлять РНК различных возбудителей [там же]. Метод ОТ-ПЦР по себестоимости сопоставим с иммуноферментным анализом, но позволяет работать не только со свежим, но и фиксированным материалом.
Цель данной работы – разработка ПЦР-тест системы для выявления вируса картофеля Y в растительном ма- териале. Разработка включает в себя подбор структуры праймеров, условий реакции ОТ-ПЦР, анализ размера амплифицируе-
мого фрагмента, воспроизведение ОТ-ПЦР на растительном материале, определение нуклеотидной последовательности полу- ченного фрагмента, что является подтверждением того факта, что данная ПЦР-тест система выявляет именно наличие РНК вируса картофеля Y.
Материалы и методы
Последовательности геномов вируса картофеля Y (PVY) были скопированы из базы данных Genome, Nucleotide (www. . Названия последовательностей и их паспортные номера представлены в табл. 1.
Данные последовательности были проанализированы в программе AliBee - Multiple alignment Release 3.0 на сайте В выявленных консервативных участках отби- рали фрагменты 20–25 нуклеотидов, начинающиеся и заканчивающиеся на гуанин или цитозин (предполагаемые праймеры, представленные в табл. 2). Далее для праймеров проводился расчет температуры отжига по формуле:
Тотжига = (2×(количество аденина + тимина) + (1) 4×(количество гуанина + циозина)) – 5 .
Также в программе Nucleotide blast ( gov) проверялась уникальность праймеров и присваивался номер от 0 – последовательность не выявляется в нужных видах и генах, до 10 – последовательность выявляется только в геноме вируса и больше нигде.
Из табл. 1 были выбраны два уникальных праймера (уровень уникальности – от 8 и выше) с одинаковыми температурами отжига (допускаются различия температуры отжига до 2 оС), которые были синтезированы в фирме Синтол.
В качестве источника нуклеиновых кислот использовались пазушные почки клубня картофеля и покровная ткань вокруг них. Суммарные нуклеиновые кислоты выделялись с помощью модификации гуанидин-изотиоцианатного метода [11].
Реакцию обратной транскрипции проводили следующим образом: 50 нг РНК растворяли в 4 мкл воды, добавляли по 1 мкл обратного праймера в концентрации 40 рМ/мкл, прогревали при 95 оС 5 мин, затем добавляли 1 мкл 10Х буфера для обратной транскрипции, 1 мкл смеси dNTPs (концентрация 4 мкМ), 1 мкл обратной транскриптазы MMLV (15 е.а./мкл) (Сибэнзим) и деионизированную воду до 10 мкл. Полученную смесь инкубировали при 37 оС 1 ч 10 мин. Затем переосаждали и вносили на реакцию ПЦР: 2 мкл раствора матрицы после обратной транскрипции, 1 мкл буфера для ПЦР с 1.5 мМ MgCl2, 0.5 мкл смеси dNTPs
Таблица 1
Названия изолятов вируса картофеля Y (PVY) и их идентификационные номера в базе данных Genome, Nucleotide
Table 1
Names of Potato virus Y (PVY) isolates and their identification numbers in the database Genome, Nucleotide
|
№ |
Название |
Идентификационный номер |
|
1 |
Potato virus Y isolate May4B, complete genome |
MK563993 |
|
2 |
Potato virus Y isolate Egypt35 polyprotein gene, complete |
KY863551 |
|
3 |
Potato virus Y isolate Egypt7 polyprotein gene, complete cds |
KY863549 |
|
4 |
Potato virus Y isolate Egypt11 polyprotein gene, complete cds |
KY863548 |
|
5 |
Potato virus Y isolate VarB, complete genome |
KT290512 |
|
6 |
Potato virus Y isolate VarA, complete genome |
KT290511 |
|
7 |
Potato virus Y isolate GF_YL20, complete genome |
KJ634023 |
|
8 |
Potato virus Y genomic RNA, complete genome, isolate: T13 |
AB714135 |
|
9 |
Potato virus Y isolate WWA150131_G4_8, complete genome |
MT200673 |
|
10 |
Potato virus Y isolate MT12_Oth288, complete genome |
MT200671 |
|
11 |
Potato virus Y isolate BL2, complete genome |
MT200669 |
|
12 |
Potato virus Y strain N, complete genome |
MT350290 |
|
13 |
Potato virus Y strain O, complete genome |
MT350288 |
|
14 |
Potato virus Y isolate P097, complete genome |
MT264732 |
|
15 |
Potato virus Y isolate PVYO_R3S16L8, complete genome |
MK572844 |
|
16 |
Potato virus Y isolate PVYN_O_R3S2L8, complete genome |
MK572835 |
(концентрация 4 мкМ), по 1 мкл праймеров, концентрация каждого 10 рМ/мкл, 0.75 мкл Taq-полимеразы (5 ед.а./мкл) (Сибэнзим) и деионизированную воду до 10 мкл. Реакция ПЦР состоит из трех этапов 1-й и 3-й состоят из одного цикла, так как не требуется циклическое изменение температуры. Продукты реакции амплификации разделяли в 6 %-ной нативном полиакриламидном геле, который окрашивался бромистым этидием [там же].
ПЦР-продукты экстрагировали из геля [там же] и определяли нуклеотидную последовательность в фирме Евроген. Полученные результаты сравнивали в программе Nucleotide blast ( с уже имеющимися
Последовательности, использованные для праймеров, их положение в геноме, длина, температура отжига и степень уникальности
Table 2
Sequences used for primers, their position in the genome, length, annealing temperature and degree of uniqueness
|
№ |
Вирус |
Положение праймера |
Последовательность |
T, отжига , °С |
Уникальность 0–10 |
|
1 |
PVY |
2130 – 2157 |
GACATGTGTGTGCCAAAGCTTGGAACC |
59 |
8 |
|
2 |
PVY |
2140 – 2166 |
GTGCCAAAGCTTGGAACCTGGCCAAC |
60 |
7 |
|
3 |
PVY |
2220 – 2244 |
CATGATGCAGAACTGCCTAGAATA |
52 |
7 |
|
4 |
PVY |
2434 – 2457 |
ATATCACCTTTCAGAGAAGGAGG |
52 |
9 |
|
5 |
PVY |
2811 – 2835 |
GATGGTGTTGCAAGTTGTTAAGAA |
50 |
7 |
|
6 |
PVY |
2962 – 2983 |
GAAAAATTATCCGCAACATGG |
47 |
7 |
|
7 |
PVY |
3163 – 3189 |
AATATTTCATCCTTTTTCATTCGTAG |
48 |
7 |
|
8 |
PVY |
3683 – 3705 |
AGGGGAGCTGTTGGGTCTGGAA |
57 |
7 |
|
9 |
PVY |
4480 – 4503 |
GTTGATGGCAGAACAATGAAGCA |
52 |
7 |
|
10 |
PVY |
4948 – 4976 |
GATGGATCAATGCATCCTGTCATACATG |
56 |
7 |
|
11 |
PVY |
4959 – 4986 |
GCATCCTGTCATACATGACATTCTTAA |
53 |
7 |
|
12 |
PVY |
5323 – 5346 |
CAGAGCCAATTCAGAAGTCTCAT |
52 |
7 |
|
13 |
PVY |
5349 – 5377 |
GAAGGATGCTCAAGCATGTTTTCAATTG |
55 |
7 |
|
14 |
PVY |
5806 – 5833 |
GAATTCTTTGGATCTGCATACAGGAA |
53 |
7 |
|
15 |
PVY |
5857 – 5884 |
GTTGGTATGGGCAAGTCAAGCAGGAGG |
60 |
7 |
|
16 |
PVY |
6085 – 6111 |
TACTTCAGGAAAGATTGGTCTGACAA |
53 |
8 |
|
17 |
PVY |
7152 – 7175 |
TTCTTCAGGCCTTTGATGGATGC |
53 |
7 |
|
18 |
PVY |
7562 – 7586 |
GGACATTCACTGCTGCACCTTTAG |
55 |
7 |
|
19 |
PVY |
7804 – 7829 |
AGAAGCACATACATGGAAGATTGGG |
54 |
7 |
|
20 |
PVY |
7832 – 7857 |
TGGGGTTGCAAATGTTGCGCAATTT |
54 |
7 |
|
21 |
PVY |
7885 – 7908 |
TCAACTCCAGATGGAACAATTGT |
50 |
7 |
|
22 |
PVY |
8346 – 8374 |
CACCAAATCAGGAGATTCTACTCATGGT |
56 |
7 |
|
23 |
PVY |
8567 – 8591 |
ACACAATCGATGCAGGAGGAAGCA |
55 |
8 |
|
24 |
PVY |
8703 – 8730 |
GCTATCACGTCCAAAATGAGAATGCCC |
57 |
8 |
|
25 |
PVY |
8812 – 8839 |
GCAACTCAATCACAGTTTGATACGTGG |
56 |
7 |
|
26 |
PVY |
8900 – 8927 |
GGCTTATGGTTTGGTGCATTGAAAATG |
54 |
7 |
|
27 |
PVY |
8907 – 8935 |
GGTTTGGTGCATTGAAAATGGAACCTCG |
58 |
8 |
|
28 |
PVY |
8942 – 8969 |
CAACGGAGTTTGGGTTATGATGGATGG |
57 |
9 |
|
29 |
PVY |
8992 – 9019 |
TGAAACCAATCGTTGAGAATGCAAAAC |
53 |
8 |
|
30 |
PVY |
9025 – 9051 |
CTTAGGCAAATCATGGCACATTTCTC |
54 |
8 |
|
31 |
PVY |
9038 – 9065 |
TGGCACATTTCTCAGATGTTGCAGAAG |
56 |
8,5 |
|
32 |
PVY |
9051 – 9079 |
GATGTTGCAGAAGCGTATATAGAAATGC |
55 |
8 |
|
33 |
PVY |
9080 – 9108 |
GCAACAAAAAGGAACCATATATGCCACG |
56 |
8 |
|
34 |
PVY |
9094 – 9118 |
CCATATATGCCACGATATGGTTTA |
50 |
8 |
|
35 |
PVY |
9208 – 9231 |
CACATTCAAATGAAGGCCGCAGC |
55 |
8 |
|
36 |
PVY |
9272 – 9300 |
AACACAGAGAGGCACACCACCGAGGATG |
62 |
8 |
|
37 |
PVY |
9322 – 9351 |
TCTCCAAGTATGCATACTCTACTTGGAGT |
56 |
8,5 |
в них последовательностями генома различных изолятов вируса картофеля Y.
Результаты и их обсуждение
Вирус картофеля Y имеет белковый капсид, протеоме-ры которого могут быть использованы для детекции вируса в растительных тканях методом иммуноферментного анализа, однако этот метод достаточно дорог. Метод ПЦР дешевле, при этом имеет сопоставимые чувствительность и точность. Но геном вируса представлен молекулой РНК. Данная молекула не может быть матрицей для стандартного метода ПЦР. Поэтому был выбран метод обратной Таблица 2 транскрипции с последующей полимеразной цепной реакцией.
Для выявления РНК вируса PVY необходимо разработать праймеры. Для этого в базе данных ( gov) было выбрано 16 последовательностей полных геномов различных изолятов вируса картофеля Y. Последовательности полных геномов использовали для большей простоты выравнивания и получения максимального количества фрагментов последовательности РНК вируса, которые можно было бы использовать в качестве праймеров.
Названия изолятов и их номера в базе данных представлены в табл. 1.
Так как геном вируса представляет собой мРНК (+ цепь РНК), кодирующую один полибелок, который транслируется, а затем разрезается на отдельные субъединицы, выбор открытых рамок считывания, кодирующих отдельный белок, не производился, а анализу подвергались нуклеотидные последовательности всего генома.
Далее последовательности были проанализированы в программе AliBee - Multiple alignment Release 3.0 на сайте В результате сравнения последовательностей выявлены идентичные у всех исследованных изолятов участки геномов, в которых выбирали последовательности для будущих праймеров. Последовательности для праймеров должны быть длиной 20–25 нуклеотидов, начинаться и заканчиваться на гуанин или цитозин.
Затем с помощью формулы (1) были рассчитаны температуры отжига, а также посчитано количество нуклеотидов во всех фрагментах. Далее в программе Nucleotide blast ( проверяли уникальность праймеров. Для этого сравнивали исследуемую последовательность с уже имеющимися в базе. В зависимости от уникальности исследу- емым последовательностям присваивалось значение уникальности от 10 (обнаруживается только в геномах различных изолятов вируса картофеля Y) до 0 (не обнаруживается в геномах PVY, но выявляется в геномах других организмов).
Последовательности и их положение в геноме, температура отжига, количество нуклеотидов и его уникальность представлены в табл. 2.
Наибольшей степенью уникальности обладают фрагменты под №№ 28 (уникальность – 9), 31 и 37 (уникальность – 8.5), но у фрагмента № 28 температура отжига составила 57 °С. Фрагменты №№ 31 и 37 имеют одинаковую температуру отжига (56 °С). Поскольку в температурах отжига допускается различие до 2 °С, все три фрагмента могут быть использованы в качестве праймеров, но также необходимо учитывать размер ПЦР-продукта. Оптимальным для разделения с помощью гель-электрофореза в полиакриламидном геле являются фрагменты ДНК длиной от 150 до 300 пар нуклеотидов. Фрагменты №№ 28 и 31 будут образовывать ПЦР-продукт длиной 158 пар нуклеотидов, такая длина ближе к нижней границе оптимума. Пары фрагментов №№ 28/37 и №№ 31/37 будут образовывать фрагменты длиной 451 и 313 пар нуклеотидов. Соответственно, такие фрагменты длиннее оптимальных.
Поэтому были рассмотрены фрагменты с №№ 32 по 36, их уникальность составила 8. По температуре отжига совпал фрагмент № 33, у остальных температура отжига не совпадает больше допустимых различий. Определение длины ПЦР-продукта показало, что для пары №№ 33 и 37 она будет составлять 271 пару нуклеотидов, которая попадает в пределы оптимальных длин фрагмента.
Следует отметить, что фрагмент № 4 (уникальность – 9) не рассматривался в связи с далеким расположением от фрагмента № 28, с вероятностью не получить ампликон обычными методами и низкой уникальностью соседнерасположенных фрагментов.
Таким образом, последовательности №№ 33 и 37 были использованы для создания праймеров.
Как видно из результатов гель-электрофореза, положительный результат выявлен в образце № 1. В дорожке 1 видна единственная четкая полоса чуть ниже полосы маркера длиной 300 пар нуклеотидов. Также слабо светящаяся полоса на том же уровне присутствует в дорожке 6 (образец картофеля № 6). Наличие полос означает, что праймеры провзаимодействовали с матрицей, а размер полученного фрагмента ДНК соответствует ожидаемому. Эти факты позволяют предполагать, что в образцах картофеля №№ 1 и 6 присутствует РНК вируса PVY. Тогда как в других образцах вирус отсутствует. Отсутствие дополнительных полос на дорожках 1 и 6 указывает на то, что праймеры отжигаются только на одном участке в образце нуклеиновых кислот, что подтверждает высокую степень уникальности, которая была определена теоретически. Отсутствие одинаковых полос во всех образцах подтверждает отсутствие реакции праймеров с РНК хозяина, а также их уникальность.
Кроме того, задачей являлась оценка возможности использовать данные праймеры в существующих условиях реакции ПЦР для выявления вируса картофеля Y.
Так как положительный контроль – образец ткани картофеля, зараженного вирусом Y, отсутствовал, проверку того, что данный амплифицированный фрагмент и есть ДНК-копия генома вируса картофеля Y, проводили, определяя нуклеотидную последовательность полученного ПЦР-продукта.
Образец РНК № 1 использовали для получения ПЦР-про-дукта, который был очищен и отправлен на определение нуклеотидной последовательности в фирму Евроген.
Таблица 3
Последовательность праймеров для выявления вируса картофеля Y (PVY)
Table 3
Potato virus Y (PVY) primer sequence
|
Название праймера |
Последовательность праймера |
T, отжига , °С |
Размер ПЦР-продукта |
|
PVY 1 F |
5’ - GCAACAAAAAGGAACCATATATGCCACG - 3’ |
56 |
271 п.н. |
|
PVY 1 R |
5’ - ACTCCAAGTAGAGTATGCATACTTGGAGA - 3’ |
56 |
Название и последовательность, температура отжига праймеров представлены в табл. 3, а также предполагаемый размер ПЦР-продукта, рассчитанный по последовательности генома вируса.
Праймеры после обработки были заказаны в фирме Синтол. Далее проводилась проверка работы праймеров в реальных условиях. Для анализа использованы образцы пазушных почек клубней картофеля, так как растущие ткани более пригодны для выделения нуклеиновых кислот как ткани с меньшим размером клеточных стенок и большим объемом генетического материала. Клубни были предоставлены с фермерского поля в Куменском районе Кировской области.
Выбрано восемь клубней с прорастающими пазушными почками, из которых брался материал для обратной транскрипции и ПЦР. Продукты реакции визуализировали методом электрофореза в нативном полиакриламидном геле. Полученный результат представлен на рис. 1.
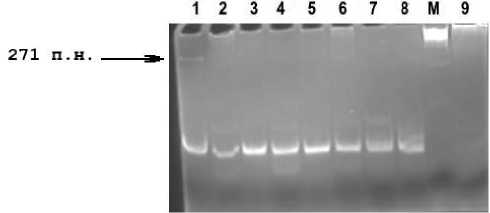
Рисунок 1. Гель-электрофорез в 6 %-ном нативном полиакриламидном геле, продуктов реакции ОТ-ПЦР на наличие вируса PVY. Дорожки 1–8 – реакции с образцами картофеля №№ 1–8; № 9 – отрицательный контроль; М – маркер длин фрагментов ДНК (Сибэнзим). Ожидаемый размер ПЦР-продукта PVY – 271 п.н.
Figure 1. Gel electrophoresis in 6 % native polyacrylamide gel of the RT-PCR reaction products for the presence of PVY. Lanes 1–8 – reactions with potato samples № 1 – № 8; № 9 – negative control; M – marker of DNA fragment lengths (Sibenzyme). The expected size of the PVY PCR product is 271 bp.
Получена следующая последовательность нуклеотидов: TGGCATTCTCATTTTGGACGTGATAGCTAAGCCTAAATATGCAA TGCAGGGATATTTACAATTTTTTATAAAAACACCCAGACCAAAACA AGTATCAACAGTCTGCTTCCTCCTGCATCGATTGTGTCA.
Она короче, чем размер ПЦР-продукта, так как секвенирование ПЦР-продуктов затруднено, поэтому в текстовый редактор переведен только тот участок, в котором были наиболее точные результаты капиллярного гель-электро-фореза.
Полученная последовательность проанализирована в Nucleotide blast (. Результаты представлены на рис. 2.
|
□ Potato virus Y strain 0 g ene for coat protein , partial cds |
PotatovirusYstralnO |
60,2 |
60.2 |
23% |
6e-05 |
100.00% |
799 |
10340445.1 |
|
Q Potato virus Y isolate PVY-CJ ooly protein ч епе , complete cds |
Potato virus Y |
60.2 |
60,2 |
23% |
6e-05 |
100.00% |
9614 |
KY6Q0342 1 |
|
Q Potato virus y isolate WG015 2Ю polv protein g ene , complete cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6^05 |
100.00% |
9653 |
MF624291 1 |
|
Q Potato virus Y Is ola te WQQ15 204 RBteoteln gens. ЕОПЩе cd s |
Potato Y lrys Y |
Ы) 7 |
fin 2 |
23% |
Re-QR |
100,00% |
U64II |
MF624290 I |
|
Q Potato virus Y isolate WGO1S 201 p olv protein g ene, complete cds |
Potato virus Y |
fifi 9 |
60 2 |
93% |
Кя-Пб |
100,00% |
9633 |
MF624289.1 |
|
Q Potato virus Y Isolate Qth2016 153 polv protein g ene , complete cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
9655 |
MF624268.1 |
|
Q Potato virus Y isolate Yunnan 1 coat protein g ene , partial cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
801 |
KJ513434.1 |
|
Q Potato virus Y Isolate Guizhou 2 coat protein g ene , partial cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
801 |
KJ513425.1 |
|
Q Potato virus Y Isolate Gulzhou_1 coat protein g ene , partial cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
801 |
KJ513423.1 |
|
Q Potato virus Y isolate WI120018. complete g enome |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
9609 |
KY848028.1 |
|
Q Potato virus Y isolate WA316. complete g enome |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
9571 |
KY848025.1 |
|
Q Potato virus Y isolate SU2 . ,complete g enome |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
9655 |
К Y848024.1 |
|
Pvlutu Virus V isulsle ND 100040 uvmuleto ueriuiue |
Potato virus Y |
100.00% |
К Y847992.1 |
|||||
|
0 Potato virus Y isolate ID26 . complete genome |
Potato virus Y |
en 9 |
60 7 |
93% |
Ra-ПП |
100.00% |
germ |
KYM7955.1 |
|
Q Potato virus Y isolate 1021 . complete g enome |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
9676 |
К Y64 7954,1 |
|
Q Potato virus Y isolate ID13_6l0Brw . complete genome Q Potato virus Y Isolate ID1280, complete genome |
Potato virus Y Potato virus Y |
60.2 |
бе-05 |
100.00% 100.00% |
9605 |
KY847953.1 KY847951.1 |
||
|
Q Potato virus Y Isolate Xch-Wh nonfuncbOnal PIPO and nonfunctional polyprotein genes, complete segue.. |
. Potato virus Y |
tie-OD |
100.00% |
I ( Л.1- J ] |
||||
|
Q Potato virus Y Isolate ZLT-2012H Y coat protein mRNA , partial cds |
Potato virus Y |
60.2 |
602 |
23% |
6e-05 |
100.00% |
801 |
KU893142.1 |
|
Я Potato virus Y strain PVY-NE11 polvprotein gene, complete cds |
Potato virus Y |
fin 9 |
fifl 7 |
93% |
Rn.nh |
100.00% |
дЛЗЙ |
KP793716.1 |
|
Q Potato virus Y Isolate 1021 coat protein g ene , partial cds |
Potato virus Y |
60.2 |
60.2 |
23% |
6e-05 |
100.00% |
801 |
KJ741190.1 |
|
Я Potato virus Y Isolate CN:XT27:16 coat protein gene, partial cds |
Potato virus Y |
60.2 |
60 2 |
23% |
6e-05 |
100.00% |
801 |
MH054516.1 |
Рисунок 2. Результаты проверки нуклеотидной последовательности в Nucleotide blast ( gov).
Figure 2. Results of verification of the nucleotide sequence in Nucleotide blast (. 1
Как видно из результатов сравнения последовательности с последовательностями из базы данных генов в Nucleotide blast (, исследуемая последовательность на 100 % совпадает с последовательностью генома вируса картофеля Y (PVY). Соответственно, данная пара праймеров в выбранных условиях обеспечивает амплификацию с матрицы РНК PVY и может быть использована как пара праймеров для ПЦР-тест системы для выявления вируса картофеля Y.
Данная система апробирована с использованием материалов, предоставленных Институтом агробиотехнологий ФИЦ Коми НЦ УрО РАН (Республика Коми, г. Сыктывкар). Проанализировано 48 клубней картофеля по окончании периода зимней лежкости в хранилище Института. Клубни отобраны от 16 селекционных сортообразцов питомников предварительного, основного и конкурсного испытаний по три клубня от каждого номера. В данном случае повторность не учитывалась, так как клубни были выкопаны с разных участков полей и не могли быть перезаражены через насекомых или какие-либо микромеханические повреждения. Поэтому можно было анализировать клубни как отдельных представителей, возможно, зараженные патогеном. Из 48 образцов зараженными оказались восемь, т.е. процент общей зараженности составил 16.7 %. Только в одном сортообразце отмечено заражение двух клубней из трех, в остальных же случаях заражен был только один клубень из трех. Можно предположить, что, с одной стороны, это показывает, что механические микроповреждения, получаемые клубнями в ходе уборки копателем и контактом с его рабочим органом, обеспечивают непосредственно перенос патогена, так как сами клубни не контактировали и находились на разных участках полей, а с другой – заражение произошло в ходе контакта уже непосредственно во время закладки в лечебный период и при последующем зимнем хранении. Таким образом, данная аналитическая система требует дальнейшей проверки с подбором материала.
Заключение
Разработана ПЦР-тест система для выявления генетического материала вируса картофеля Y. По результату анализа генома подобраны праймеры, апробированные в условиях реакции. Таким образом, праймеры PVY 1 F и PVY 1 R готовы для разработки тест-системы для выявления материала вируса PVY. Так же были подобраны условия реакции, и за счет определения нуклеотидной последовательности подтверждена достоверность реакции.
Список литературы Разработка ПЦР-тест системы для выявления вируса картофеля Y
- Kreuze, J. F. Viral diseases in potato / J. F. Kreuze, J. A. C. Souza-Dias, A. Jeevalatha, A. R. Figueira, J. P. T. Valkonen, R. A. C. Jones // The Potato Crop. – Cham: Springer, 2020. – P. 389–429.
- Рогозина, Е. В. Широко распространенные и потенциально опасные для Российского агропроизводства возбудители вирусных болезней картофеля / Е. В. Рогозина, Н. В. Мироненко, О. С. Афанасенко, Ю. Мацухито // Вестник защиты растений. – 2016. – № 4 (90). – С. 24–33.
- Kerlan, C. Potato virus Y. – URL: https://dpvweb.net/dpv/showdpv/?dpvno=414.
- Lacomme, C. General characteristics of potato virus Y (PVY) and its impact on potato production: an overview / C. Lacomme, L. Glais, D. Bellstedt, B. Dupuis, A. Karasev, E. Jacquot // Potato virus Y: biodiversity, pathogenicity, epidemiology and management. – Cham: Springer, 2017. – P. 1–19.
- Baunoch, D.A. Potato virus Y helper component is associated with amorphous inclusions / D.A. Baunoch, P. Das, V. Hari // Journal of General Virology. – 1990. – Vol. 71(10). – P. 2479–2482. doi:10.1099/0022-1317-71-10-2479.
- Chung, Betty Y.-W. An overlapping essential gene in the Potyviridae / Betty Y.-W. Chung, W. Allen Miller, John F. Atkins, Andrew E. Firth // PNAS. – 2008. – Vol. 105(15). – P. 5897–5902. doi:10.1073/pnas.0800468105.
- Kneller, E.L. Cap-independent translation of plant viral RNAs / E.L. Kneller, A.M. Rakotondrafara, W.A. Miller // Virus Research. – 2006. – Vol. 119(1). – P. 63–75. doi: 10.1016/j.virusres.2005.10.010.
- Сухорученко, Г. И. Система интегрированной защиты репродукционного семенного картофеля от комплекса вредных организмов в Северо-Западном регионе Российской Федерации / Г. И. Сухорученко, Г. П. Иванова, С. А. Волгарев. – Санкт-Петербург: Пушкин, 2016. – 64 с.
- Galvino-Costa, S. B. F. Molecular and serological typing of potato virus Y isolates from Brazil reveals a diverse set of recombinant strains / S. B. F. Galvino-Costa, A. R. Figueira, F. A. C. Rabelo-Filho, F. H. R. Moraes, O. V. Nikolaeva, A. V. Karasev // Plant Disease. – 2012. – Vol. 96(10). – P. 1451–1458. doi: 10.1094/PDIS-02-12-0163-RE.
- Onozuka, N. One-step real-time multiplex reverse transcription- polymerase chain reaction assay with melt curve analysis for detection of potato leafroll virus, potato virus S, potato virus X, and potato virus Y / N. Onozuka, T. Ohki, N. Oka, T. Maoka // Virology Journal. – 2021. – Vol. 18. – № 131. doi: 10.1186/s12985-021-01591-3.
- Sambrook, J. Molecular cloning: a laboratory manual / J. Sambrook, T. Fritch, T. Maniatis. – New York: Cold Spring Harbor Laboratory Press, 1989. – 1659 p.


