Роль генов общего стрессорного ответа в формировании бактериальной персистенции
Автор: Хаова Елена Александровна, Кашеварова Наталья Михайловна, Шумков Михаил Сергеевич, Сидоров Роман Юрьевич, Ткаченко Александр Георгиевич
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 4, 2018 года.
Бесплатный доступ
Изучена функциональная активность генов общего стрессорного ответа rpos, rmf, relA и spoT в формировании бактериальной персистенции в условиях периодического культивирования Escherichia coli. Генная экспрессия исследована на транскрипционном уровне с помощью двух методов: репортерных генных слияний и ПЦР в реальном времени с предварительным проведением обратной транскрипции. Полученные результаты представлены в сопоставлении с данными о частоте персистенции родительского штамма и делеционных мутантов по исследованным генам. На основании комплексной оценки показано участие генов rpoS, rmf, relA и spoТ в персистенции E. coli при переходе в стационарную фазу роста и в поздней стационарной фазе.
Бактериальная персистенция, экспрессия генов, стресс, стационарная фаза
Короткий адрес: https://sciup.org/147227047
IDR: 147227047 | УДК: 579.22:579.25 | DOI: 10.17072/1994-9952-2018-4-393-401
Текст научной статьи Роль генов общего стрессорного ответа в формировании бактериальной персистенции
Ключевым свойством любой популяции является её генотипическая и фенотипическая гетерогенность. Это свойство обеспечивает выживание популяции при воздействии многочисленных стрессов в меняющихся условиях среды [Тимофеев-Ресовский, Воронцов, Яблоков, 1977; Олескин, 2001; Миркин, Наумова, 2005]. Одним из примеров фенотипической гетерогенности бактериальных популяций являются опубликованные в последнее время данные об их субпопуляционной структуре по признаку персистенции [Rotem et al., 2010; Jong, Haccou, Kuipers, 2011; Maisonneuve, Gerdes, 2014; Bergh, Fauvart, Michiels, 2017].
Персисторы – это бактериальные клетки, которые в относительно небольшом количестве присутствуют в любой популяции и за счет значительного замедления скорости метаболических процессов (дормантное состояние) характеризуются пониженной чувствительностью к стрессорному воздей- ствию различных внешних физико-химических факторов [Lewis, 2010; Maisonneuve, Gerdes, 2014]. При этом сама субпопуляция персисторов неоднородна и представляет собой континуум микросубпопуляций, качественно и количественно различающихся по отношению к различным типам стрессоров [Tkachenko et al., 2014; Zhang, 2014; Bergh, Fauvart, Michiels, 2017]. Таким образом, персистенция представляет собой инструмент биологической «страховки» (bet hedging) бактериальной популяции от вымирания в условиях изменчивости среды [Carey, Goulian, 2018].
Переход бактериальной клетки из «нормального» состояния в дормантное обеспечивается за счет случайных флуктуаций около некоторого порогового уровня содержания в ней регуляторных молекул, относящихся к механизмам общего стрессор-ного ответа [Rotem et al., 2010]. Другой механизм представляет собой собственно стрессорный ответ
в виде индукции упомянутых клеточных систем, что приводит к возрастанию количества персисто-ров и способствует выживанию популяции в целом [Lewis, 2010; Maisonneuve, Gerdes, 2014]. Поскольку в естественных условиях бактерии часто испытывают стрессорные воздействия, можно говорить о значительной роли механизмов общего стрессорного ответа в персистообразовании. Наибольшее количество персисторов при периодическом культивировании образуется в стационарной фазе роста [Lewis, 2010; Bergh, Fauvart, Michiels, 2017], наиболее близкой к естественным условиям [Ленгелер, Древс, Шлегель, 2005]. Исходя из этого, переход периодической культуры бактерий в стационарную фазу часто используется в качестве экспериментальной модели персистообразования в природной популяции.
С использованием этого подхода нами была изучена роль в бактериальной персистенции генов rpoS, rmf, relA и spoT, относящихся к механизмам общего стрессорного ответа. Известно, что ген rpoS кодирует σS-субъединиицу РНК-полимеразы. Гены, промоторы которых имеют сродство к данной σ-субъединиице, объединены в структуру rpoS-регулона и участвуют в адаптации к стрессам преимущественно при переходе в стационарную фазу роста [Hengge-Aronis, 2002; Ткаченко, 2012]. Гены relA и spoT кодируют (p)ppGpp-синтетазу и (p)ppGpp-синтетазу/гидролазу соответственно. При этом для SpoT сигналом для синтеза (p)ppGpp является отклонение скорости трансляции от максимальной, тогда как RelA реагирует на истощение в клетках хотя бы одной из аминокислот. Незаряженные тРНК, связываясь с аминоацильным центром рибосом, служат для RelA в качестве сигна- ла, индуцирующего синтез (p)ppGpp. Первоначально (p)ppGpp был описан как сигнал стресса голодания и отнесен к категории алармонов, участвующих в адаптации к аминокислотному голоданию. Однако в настоящее время показано его участие в регуляции множества процессов в бактериальной клетке, связанных с ее ростом, адаптацией к стрессам, вторичным метаболизмом, персистенцией, клеточным делением, подвижностью, биопленкообразованием и вирулентностью. Под регуляторным контролем (p)ppGpp находятся около 500 генов, и этот список постоянно пополняется [Hauryliuk et al., 2015; Ткаченко, 2018]. Одним из них является ген стационарной фазы rmf [Izutsu, Wada, Wada, 2001]. Продукт его экспрессии, белок RMF, ингибирует трансляцию, превращая рибосомы в неактивную димерную форму [Wada, 1998].
В связи со своей практической значимостью, наиболее полно изученной является персистенция к антибактериальным препаратам. Находясь в дормантном состоянии, персисторы обладают множественной антибиотикотолерантностью, благодаря чему способны возобновлять рост после прекращения воздействия антибиотиков. Это дает основание рассматривать их в качестве основной причины рецидивов инфекционных заболеваний [Lewis, 2010; Maisonneuve, Gerdes, 2014]. Таким образом, изучение персистенции имеет важное значение для разработки новых антибактериальных средств.
Материалы и методы
В качестве объектов исследования использовали штаммы Escherichia coli (табл. 1) .
Таблица 1
Штаммы, использованные в работе
|
Штамм |
Генотип |
Источник |
|
BW25141 |
F-, Δ(araD-araB)567, ΔlacZ4787(::rrnB-3), Δ(phoB-phoR)580, λ-galU95, ΔuidA3::pir+, recA1, endA9(del-ins)::FRT, rph-1, Δ(rhaD-rhaB)568, hsdR514 |
Северинов К.В. [Datsenko, Wanner, 2000] |
|
EAT01 |
как BW25141, но ∆rmf |
Tkachenko et. al., 2017 |
|
EAT03 |
как BW25141, но ∆rpoS |
|
|
SRY1 |
как BW25141, но ∆relA |
Коллекция лаборатории адаптации микроорганизмов ИЭГМ УрО РАН |
|
SRY2 |
как BW25141, но ∆relA ∆spoT |
|
|
EAT16 |
как BW25141, но λRZ5rpoS742::lacZ |
|
|
EAT17 |
как BW25141, но λRS45rmf39::lacZ |
Штаммы высевали на 5 мл питательной среды LB («Sigma», США) с добавлением соответствующих антибиотиков: ампициллин 25 мкг/мл («MP Biomedicals», Франция), канамицин 25 мкг/мл («Синтез», Россия). После 7 ч. культивирования в термостате при 37ºС бактериальную культуру переносили в соотношении 1:1000 (по объему) на 50 мл LB в колбу Эрленмейера (250 мл) с добавлением антибиотиков. Культивировали 16 ч. в термо- статируемом шейкере («GFL 1092», Германия) при 37ºС, 120 об/мин. Далее, для проведения эксперимента культуру переносили в 50 мл свежей среды LB в колбу Эрленмейера (250 мл), доводя оптическую плотность (OD600) культуры до 0.1 (соответствует 0 ч. культивирования). Инкубировали в термостатируемом шейкере при 37ºС, 120 об/мин. Биомассу клеток оценивали по оптической плотно- сти на спектрофотометре UV1280 («Shimadzu», Япония).
Экспрессию генов определяли на транскрипционном уровне с помощью двух методов: полимеразной цепной реакции (ПЦР) в реальном времени с предварительным проведением обратной транскрипции и репортерных генных слияний. Репор-терное слияние rpos::lacZ трансдуцировано из штамма RO200 [Muffler et al., 1997]. Репортерное слияние rmf::lacZ сконструировано в соответствии с протоколом, предложенным R.W. Simons, F. Houman, N. Kleckner [1987]. Уровень экспрессии определяли методом Миллера [Миллер, 1976].
РНК выделяли из бактериальной культуры родительского штамма BW25141 (табл. 1) с помощью коммерческого набора «GeneJET Purification Kit» («Thermo Fisher Scientific», США) согласно протоколу. Концентрацию РНК в полученных пробах измеряли при OD260, качество проб оценивали по OD260/OD280 и OD260/OD230 при помощи спектрофотометра NanoDrop 2000 («Thermo Fisher Scientific», США) и методом электрофореза в 1%-ном агарозном геле с окрашиванием бромистым этидием [Кузнецов, Кузнецов, Романов, 2012; Великов, 2013]. Пробы обрабатывали ДНКазой, входящей в состав коммерческого набора «TurboTM DNase» (2U/мкл) («Thermo Fisher Scientific»,США). В качестве матрицы для обратной транскрипции использовали 100 нг суммарной РНК. Также в состав реакционной смеси входили: праймеры Random 9 («Евроген», Россия) 5мкМ, смесь dNTPs («Thermo Fisher Scientific», США) 1 мМ, ревертаза 200U «Thermo Scientific RevertAid Reverse Transcriptase» с буфером («Thermo Fisher Scientific», США). Реакцию проводили при 42°С 1 ч. с предварительной инкубацией при комнатной температуре 10 мин. и с последующей инактивацией при 70°С 10 мин. Синтезированную кДНК использовали в качестве матрицы для ПЦР в реальном времени. Праймеры подобраны с помощью программы PrimerSelect версия 7.1.0 (DNASTAR, Inc.) и представлены в табл. 2. Реакцию проводили при помощи коммерческого набора «qPCRmix-HS SYBR» («Евроген», Россия) на амплификато-ре «CFX96 RT Systems C1000 Thermal Cycler» («BioRad», США). Процедура ПЦР включала начальную денатурацию 95°С 5 мин., далее следовали 40 циклов из последовательно повторяющихся шагов: денатурация – 95°С 15 сек., отжиг праймеров – 60°С 15 сек., элонгация – 72°С 15 сек. После завершения ПЦР строили кривую диссоциации для оценки наличия димеров праймеров и других артефактов. Эффективность реакций и пороговые циклы устанавливали при помощи программы LinRegPCR (2014.x) [Ruijter et al., 2009]. Полученные данные нормализованы с использованием геномной ДНК [Demidenok, Kaprelyants, Gon- charenko, 2014].
Статистическую обработку полученных результатов проводили с использованием пакета стандартных программ STATISTICA 5.0 (StatSoft, Inc.). Повторность экспериментов 3-кратная.
Таблица 2
Праймеры, использованные для ПЦР в реальном времени
|
ген |
Последовательность праймеров |
|
rpoS |
5’ – TGAAGATGCGGAATTTGATGAGAA – 3’ 5’ – TCGGCCGTTAACAGTGGTGAA – 3’ |
|
rmf |
5’ – CGCGCACATCAACGTGGTTATCA – 3’ 5’ – GCCAGCCTCCCAGCCATTGT – 3’ |
|
relA |
5’ – GCTGAAGGCGTTAAAGCGGAAGTGT – 3’ 5’ – CGGCAGGTGGCGATAGTGAGTGT – 3’ |
|
spoT |
5’ – GCGCACGCTGGGCTCACTT – 3’ 5’ – GCGCGGCTTTCACCACTTCTTT – 3’ |
Результаты
Для выяснения роли генов rpoS, rmf, relA и spoT в бактериальной персистенции нами определена экспрессия каждого из них на транскрипционном уровне. Полученные результаты представлены в сопоставлении с данными о частоте персистенции родительского штамма и делеционных мутантов по исследуемым генам. Нокаутные штаммы в целом демонстрируют более низкие значения частоты персистенции по сравнению с контролем при переходе бактериальной культуры в стационарную фазу роста и собственно в стационарной фазе.
Полученные результаты свидетельствуют о том, что экспрессия гена rpoS на транскрипционном уровне достигает максимума при переходе в стационарную фазу на 8 ч. культивирования, а также в глубоком стационарном состоянии на 72 ч. Эти результаты согласуются с данными о частоте персистенции нокаутного штамма ∆ rpoS , демонстрирующего наибольшее снижение частоты персистенции по сравнению с родительским штаммом также при переходе культуры в стационарную фазу роста (8–24 ч.). После 24 ч. различие в значениях частоты персистенции между мутантом и родителем снижается, однако уровень персисторов в культуре нокуатного штамма ∆ rpoS остается сравнительно низким (рис. 1).
Наиболее высокие значения экспрессии генов relA и spoT наблюдаются в экспоненциальной фазе и при переходе в стационарную фазу (рис. 3). Ген rmf демонстрирует максимум экспрессии также при переходе в стационарную фазу (8–24 ч.) (рис. 2). Однако мутанты ∆rmf, ∆relA и ∆relA∆spoT показывают максимальное снижение частоты персистенции в поздней стационарной фазе (48–72 ч.). Наиболее вероятно, что активация экспрессии генов relA и spoT в стационарной фазе происходит преимущественно на трансляционном уровне, как ранее нами показано в отношении rmf [Tkachenko et al., 2017].
Полученные нами результаты показали, что исследуемые гены rpoS, rmf, relA и spoT вовлечены в частота
BW25141°D»-600 пеР™стенЧии
ArpoS ■-
-е- экспрессия rpoSJacZ
-■■ уровень rpoS мРНК
ODeoo чаСТ0ТЭ персистенции
-•- экспрессия rmf::lacZ
-■-уровень rmf мРНК
BW25141 -•- &rmf ■»
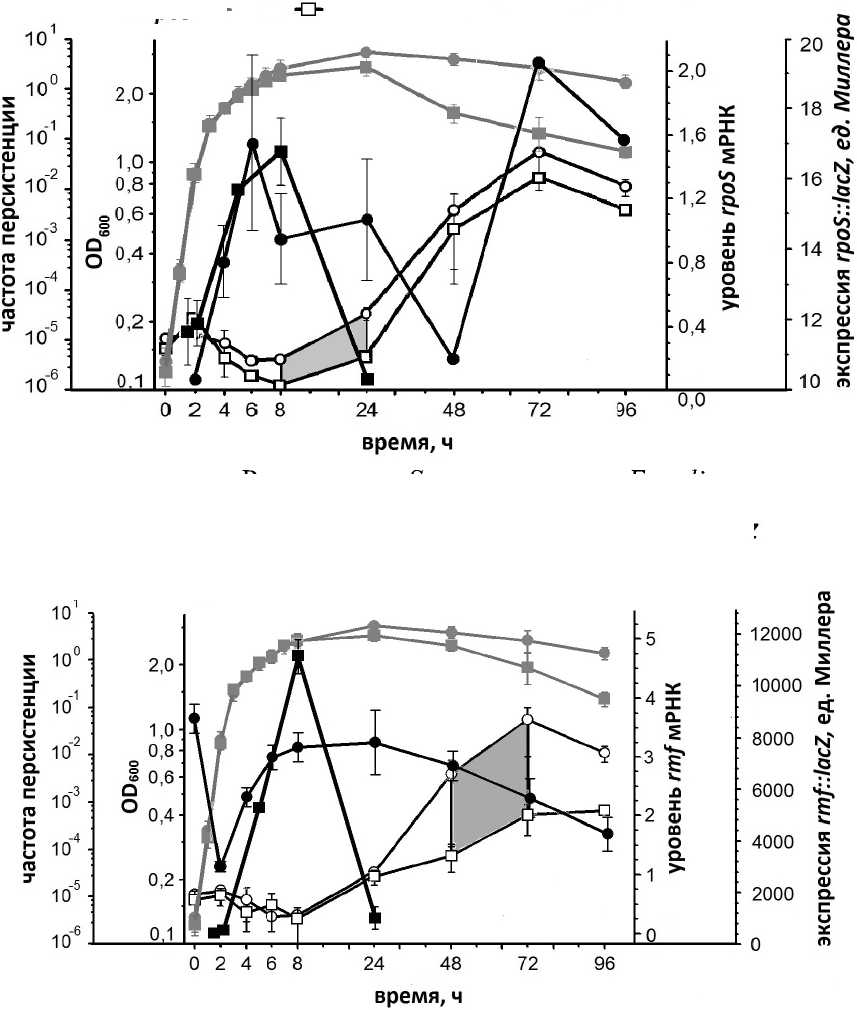
Рис. 2 . Роль гена rmf в персистенции E. coli
Рис. 1 . Роль гена rpoS в персистенции E. coli
частота уровень персистенции уровень spoT мРНК
ODeoo
BW25141 ♦
Are/Д *
ODeoo ЧаСТ0Та
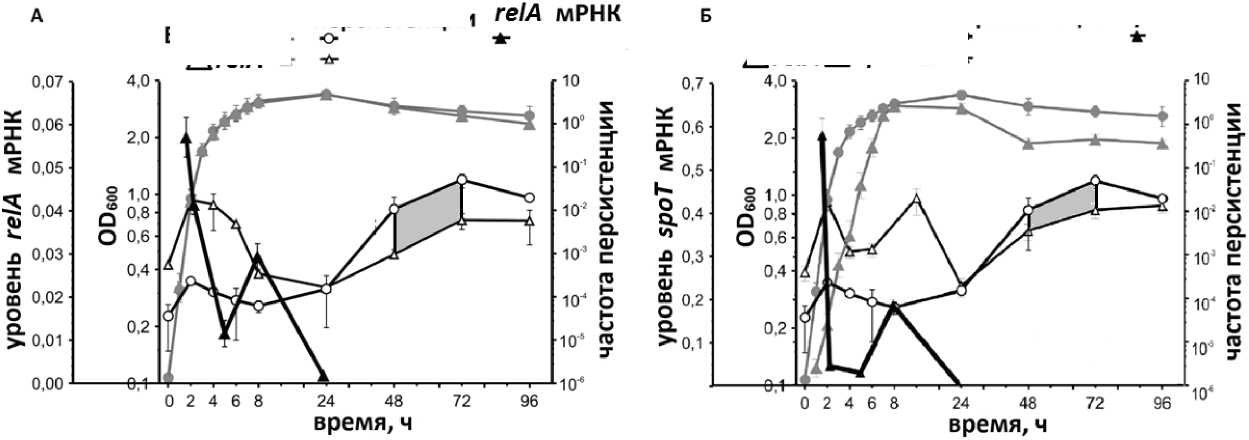
Рис. 3. Роль генов relA (А) и spoT (Б) в персистенции E. coli
Обсуждение
Участие генов rpoS, rmf, relA и spoT в регуляции бактериальной персистенции связано с ком- регуляцию формирования персисторных клеток E.coli при переходе в стационарную фазу и собственно в стационарной фазе.
BW25141. ^РСИСТеНЧИИ Дге/ДДброГ* *
плексным воздействием на клетки в этот период различных стрессоров, включая голодание, накопление метаболитов, закисление среды, недостаток кислорода и др. В этом отношении стационарная фаза сходна с естественными условиями обитания
[Ленгелер, Древс, Шлегель, 2005], активирующими механизмы общего стрессорного ответа [Ткаченко, 2012].
Результатом действия этих механизмов, к которым относятся действия изученных нами генов, может быть формирование состояния замедления скорости метаболических процессов (дормантного состояния), способствующего развитию персистенции [Lewis, 2010; Maisonneuve, Gerdes, 2014]. В частности, это может происходить под регуляторным контролем (p)ppGpp, который, согласно литературным данным, ингибирует транскрипцию генов rrn и тем самым снижает количество рибосом в клетке. Это, в свою очередь, приводит к уменьшению интенсивности процесса трансляции [Ткаченко, 2012]. Помимо этого, (p)ppGpp, опосредованно активируя Lon-протеазы, высвобождает токсины, входящие в состав токсин-антитоксиновых систем [Maisonneuve, Castro-Camargo, Gerdes, 2013]. Токсины ингибируют различные клеточные процессы и способствуют формированию дор-мантного состояния клеток [Wen, Behiels, Devreese, 2013; Hall, Gollan, Helaine, 2017]. Ток-син-антитоксиновые системы впервые были описаны как факторы персистенции [Maisonneuve, Gerdes, 2014], в регуляции которых принимает участие (p)ppGpp. Его регуляторные функции распространяются также на rpoS -регулон, играющий значительную роль в адаптации к стрессам, формируемым при переходе в стационарную фазу роста. При этом (p)ppGpp индуцирует транскрипцию гена rpoS и повышает стабильность продукта его экспрессии [Ткаченко, 2012].
Полученные нами данные свидетельствуют о том, что максимальный уровень транскрипции гена rpoS совпадает по стадии культивирования с наибольшим снижением частоты персистенции мутанта ∆rpoS по сравнению с контролем и наблюдается при переходе в стационарную фазу. Таким образом, ген rpoS участвует в регуляции персистенции преимущественно при переходе в стационарную фазу роста.
Под регуляторным контролем (p)ppGpp находится также исследованный нами ген rmf. Основываясь на полученных результатах и ранее опубликованных данных [Tkachenko et al., 2017], можно прийти к заключению, что ген rmf вовлечен в регуляцию персистенции в поздней стационарной фазе, когда происходит активация его экспрессии преимущественно на трансляционном уровне. Участие rmf в персистенции согласуется с его функциональной активностью, направленной на ингибирование трансляции. При этом, продукт данного гена – белок RMF – способствует остановке роста клеток, блокируя функции рибНосаомос.новании описанных выше данных можно говорить о значительной роли (p)ppGpp в форми- ровании персисторных клеток, что объясняет значительное снижение частоты персистенции нока-утных штаммов ∆relA и ∆relA∆spoT в поздней стационарной фазе. Вместе с тем в экспоненциальной фазе мутанты демонстрируют более высокий уровень персистенции по сравнению с родительским штаммом. Это связано с мнимым завышением частоты персистенции нокаутных штаммов в этот период. При помощи фазово-контрастной микроскопии и LIVE/DEAD-окрашивания обнаружены значительные изменения морфологических свойств клеток мутантов в виде формирования нитей, состоящих из нескольких не разделившихся клеток (рис. 4). Это приводит к занижению данных при подсчете числа живых клеток методом высева и, следовательно, мнимому завышению значений частоты персистенции, определяемой методом I. Keren et al. [2004]. Последующее разделение нитей на отдельные клетки при переходе в стационарную фазу дает возможность объективно выявить снижение частоты персисторных клеток в нокаутных штаммах по сравнению с контрольным. Согласно ранее опубликованным данным [Shan et al., 2015], низкая внутриклеточная концентрация (р)ppGpp приводит к повышению активности полифосфатазы Ppx. В результате снижения концентрации полифосфатов ингибируется активность Lon-протеазы, для которой эти соединения являются коферментами. Это приводит к недостаточному гидролизу ингибитора клеточного деления SulA и проявляется в фенотипе в виде не до конца разделившихся клеток. Исходя из этого, гены relA и spoT, согласно результатам мутационного анализа, осуществляют регуляцию персистообразования преимущественно в поздней стационарной фазе, хотя этот вывод не нашел прямого подтверждения при анализе экспрессии relA и spoT на транскрипционном уровне, что требует проведения дополнительных исследований.
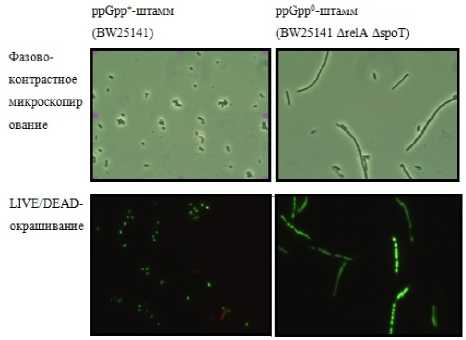
Рис. 4. Фенотип клеток штаммов BW25141и BW25141 ∆relA ∆spoT в экспоненциальной фазе роста (6 ч. культивирования)
Таким образом, на основании комплексной оценки данных определения уровня генной экспрессии методами репортерных генных слияний и ПЦР в реальном времени, сопряженной с обратной транскрипцией, в сопоставлении с результатами мутационного анализа показано участие генов стационарной фазы rpoS, rmf, relA и spoТ в формировании персисторного состояния клеток E. coli.
Работа выполнена в рамках государственного задания (номер госрегистрации темы: 01201353249, а также при финансовой поддержке РФФИ и Администрации Пермского края (проект №16-44-590279 р_а).
Список литературы Роль генов общего стрессорного ответа в формировании бактериальной персистенции
- Великов В.А. Молекулярная биология: практ. руководство. Саратов: Саратовский источник, 2013. 84 с.
- Кузнецов Вл.В., Кузнецов В.В., Романов Г.А. Молекулярно-генетические и биохимические методы в современной биологии растений. М.: БИНОМ. Лаборатория знаний, 2012. 487 с.
- Ленгелер Й., Древс Г., Шлегель Г. Современная микробиология. Прокариоты. М.: Мир, 2005. Т. 2. 656 с.
- Миллер Дж. Эксперименты в молекулярной генетике. М.: Мир, 1976. 437 с.
- Миркин Б.М., Наумова Л.Г. Основы общей экологии. М.: Университетская книга, 2005. 240 с.
- Олескин А.В. Экологически важные свойства популяций микроорганизмов // Соросовский образовательный журнал. 2001. Т. 7, № 8. С. 712.
- Тимофеев-Ресовский Н.В., Воронцов Н.Н., Яблоков А.В. Краткий очерк теории эволюции. М.: Наука, 1977. 303 с.
- Ткаченко А.Г. Молекулярные механизмы стрессорных ответов у микроорганизмов. Екатеринбург, 2012. 268 с.
- Ткаченко А.Г. Стрессорные ответы бактериальных клеток как механизм развития толерантности к антибиотикам // Прикладная биохимия и микробиология. 2018. Т. 54. С. 110-133
- Bergh B., Fauvart M., Michiels J. Formation, physiology, ecology, evolution and clinical importance of bacterial persistence // FEMS Microbiology Reviews. 2017. Vol. 41, №. 3. P. 219-251.
- Carey J.N., Goulian M. A bacterial signaling system regulates noise to enable bet hedging // Current Genetics. 2018. P. 1-6.
- Datsenko K.A., Wanner B.L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products // PNAS. 2000. Vol. 97, № 12. Р. 6640-6645.
- Demidenok O.I., Kaprelyants A.S., Goncharenko A.V. Toxin-antitoxin vapBC locus participates in formation of the dormant state in Mycobacterium smegmatis // FEMS Microbiology Letters. 2014. Vol. 352. P. 69-77.
- Hall M.J., Gollan B., Helaine S. Toxin-antitoxin systems: reversible toxicity // Current Opinion in Microbiology. 2017. Vol. 36. P. 102-110.
- Hauryliuk V. et al. Recent functional insights into the role of (p)ppGpp in bacterial physiology // Nature Reviews Microbiology. 2015. Vol. 13, № 5. P. 112.
- Hengge-Aronis R. Signal transduction and regulatory mechanisms involved in control of the sigma (S) (RpoS) subunit of RNA polymerase // Microbiology and Molecular Biology Reviews. 2002. Vol. 66, № 3. P. 373-395.
- Izutsu K., Wada A., Wada C. Expression of ribosome modulation factor (RMF) in Escherichia coli requires ppGpp // Genes Cells. 2001. Vol. 6, № 8. P. 665-676.
- Jong I., Haccou P., Kuipers O. Bet hedging or not? A guide to proper classification of microbial survival strategies // BioEssays. 2011. Vol. 33, № 3. P. 215-223.
- Keren I. et al. Persister cells and tolerance to antimicrobials // FEMS Microbiology Letter. 2004. Vol. 230, № 1. P. 13-18.
- Lewis K. Persister cells // The Annual Review of Microbiology. 2010. P. 357-372.
- Maisonneuve E., Castro-Camargo M., Gerdes K. (p)ppGpp controls bacterial persistence by stochastic induction by toxin - antitoxin activity // Cell. 2013. Vol. 154, № 5. P. 1140-1150.
- Maisonneuve E., Gerdes K. Molecular mechanisms underlying bacterial persisters // Cell. 2014. Vol. 157, № 3. P. 539-548.
- Muffler A. et al. Heat shock regulation of cS turnover: a role of DnaK and relationship between stress responses mediated by cS and c32 in Escherichia coli // Journal of Bacteriology. 1997. Vol. 179, № 2. P. 445-452.
- Rotem E. et al. Regulation of phenotypic variability by a threshold-based mechanism underlies bacterial persistence // Proceedings of the National Academy of Sciences. 2010. Vol. 107, № 28. P. 12541-12546.
- Ruijter J.M. et al. Amplification efficiency: linking baseline and bias in the analysis of quantitative PCR data // Nucleic Acids Research. 2009. Vol. 37, № 6. P. 1-12.
- Shan Y. et al. Genetic basis of persister tolerance to aminoglycosides in Escherichia coli // mBio. 2015. Vol. 6, № 2. P. 1-10.
- Simons R. W., Houman F., Kleckner N. Improved single and multicopy lac-based cloning vectors for protein and operon fusions // Gene. 1987. Vol. 53, № 1. P. 85-96.
- Tkachenko A.G. et al. Putrescine controls the formation of Escherichia coli persister cells tolerant to aminoglycoside netilmicin // FEMS Microbiology Letters. 2014. Vol. 361, № 1. Р. 1-9.
- Tkachenko A.G. et al. Stationary-phase genes upregu-lated by polyamines are responsible for formation of Escherichia coli persister cells tolerant to netilmicin // FEMS Microbiology Letters. 2017. Vol. 364, № 9. P. 1-9.
- Wada A. Growth phase coupled modulation of Escherichia coli ribosomes // Genes Cells. 1998. Vol. 3, № 4. P. 203-208.
- Wen Y., Behiels E., Devreese B. Toxin - antitoxin systems: their role in persistence, biofilm formation, and pathogenicity // Pathogens and disease. 2013. Vol. 70, № 3. P. 240-249.
- Zhang Y. Persisters, persistent infections and the Yin-Yang model // Emerging microbes and infections. 2014. Vol. 3, № 1. P. 1-10.


