Скрининг гена альфа-субъединицы бензоат диоксигеназы в бактериальных ассоциациях, полученных в результате селекции на (хлор)ароматических соединениях
Автор: Егорова Дарья Олеговна, Пьянкова Анна Александровна
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Генетика
Статья в выпуске: 4, 2019 года.
Бесплатный доступ
Показано, что в тотальной ДНК шести бактериальных ассоциаций, полученных в результате накопительного культивирования в присутствии бифенила/хлорированных бифенилов, обнаружены фрагменты гена benA, кодирующего альфа-субъединицу бензоат диоксигеназы. В результате филогенетического анализа нуклеотидных последовательностей амплифицированных фрагментов гена benA установлено, что большинство изученных фрагментов формируют кластеры с гомологичными генами представителей отдела/филума Proteobacteria, уровень сходства при этом составил 100%. Уровень сходства с benA-генами известных грамотрицательных штаммов-деструкторов составил 79.5-89.7%. В ассоциации CHN4 выявлен ген benA, формирующий единый кластер с геном benA штаммов рода Rhodococcus, и на 96% схожий с гомологичным геном штамма-деструктора R. jostii RHA1.
Полихлорированные бифенилы, бактериальные ассоциации, бензоат диоксигеназа, ген
Короткий адрес: https://sciup.org/147227113
IDR: 147227113 | УДК: 579.222 | DOI: 10.17072/1994-9952-2019-4-464-470
Текст научной статьи Скрининг гена альфа-субъединицы бензоат диоксигеназы в бактериальных ассоциациях, полученных в результате селекции на (хлор)ароматических соединениях
Бензойная кислота по химической структуре принадлежит к классу ароматических соединений, имеющих в своем составе одно ароматическое кольцо. В окружающей среде широко встречаются как замещенные бензойные кислоты, содержащие в молекуле в качестве заместителей гидрокси-, метокси- группы, атомы хлора, так и незамещенная бензойная кислота. Данные соединения являются компонентами метаболических путей растений и бактерий, в результате чего они попадают в почву, воду и донные отложения [Abd El-Mawla, Beerhues, 2002; Field, Sierra-Alvarez, 2008a; Soly-anikova et al., 2015].
Значительное количество бензойной кислоты, выявленной в объектах окружающей среды, имеет антропогенное происхождение. Бензойная кислота используется в качестве консерванта при производстве широкого круга продуктов питания, а также в составе препаратов противомикробного и фунгицидного действия. Кроме этого, бензойная кислота входит в состав лекарственных средств, направленных против кожных заболеваний грибкового и клещевого происхождения. В промышленности бензойная кислота используется как исходное сырье для синтеза широкого спектра химических соединений []. Стоит отметить, что при биотрансформации соединений группы стойких органических загрязнителей, та-
ких как полихлорированные бифенилы (ПХБ), также возможно образование бензойной/хлор-бензойной кислоты [Pieper, 2005; Field, Sierra-Alvarez, 2008b].
Основными биологическими агентами, осуществляющими разложение бензойной кислоты в природе, являются аэробные бактерии. Способность к трансформации замещенных и незамещенных бензойных кислот описана для представителей филумов Proteobacteria, Actinobacteria, Fermicutes [Pieper, 2005; Field, Sierra-Alvarez, 2008a, b]. Биоразложение бензойной кислоты начинается с окисления молекулы под действием фермента бензоат 1,2-диоксигеназы (БДО) [Parales, Resnick, 2006]. БДО (КФ 1.14.12.10) состоит из двух α- и двух β-субъединиц []. Показано, что субстратная специфичность обусловливается α-субъединицей БДО [Parales, Resnick, 2006; Solyanikova et al., 2015]. Анализ нуклеотидной последовательности гена benA, кодирующего α-субъединицу БДО, выявил существенные различия данного гена у грамположительных и грамотрицательных бактерий [Haddad, Eby, Neidle, 2001; Field, Sierra-Alvarez, 2008a; Solyanikova et al., 2015]. Высказано предположение, что эволюция гена benA у данных групп бактерий протекала независимо друг от друга, однако они имеют общего предка [Haddad, Eby, Neidle, 2001; Field, Sierra-Alvarez, 2008a].
Цель настоящей работы – изучить разнообразие гена benA , кодирующего α-субъединицу бензоат 1,2-диоксигеназы, в тотальной ДНК бактериальных ассоциаций, полученных в результате селекции почвенных микробиоценозов в присутствии бифенила/полихлорированных бифенилов.
Материалы и методы исследования
Бактериальные ассоциации
Бактериальные ассоциации CHN1, CHN2, CHN3, CHN4, CHN5, CHN6 были получены в результате накопительного культивирования в присутствии бифенила/ПХБ из почв, отобранных на территории ОАО «СВЗХ» (г. Чапаевск, Самарская обл., Россия), а PN1 и PN2 – в результате селекции бактериоценоза почв, отобранных на территории ОАО «Пермский завод смазок и СОЖ» (г. Пермь, Пермский край, Россия) с применением в качестве селективного фактора бифенила/ПХБ.
Культивирование бактериальных ассоциаций
Все бактериальные ассоциации, использованные в настоящем исследовании, поддерживаются в метаболически активном состоянии методом периодического культивирования в минеральной среде К1 состава (г/л): K 2 HPO 4 ×3H 2 O – 3.2, NaH 2 PO 4 ×2H 2 O – 0.4, (NH 4 ) 2 SO 4 – 0.5,
MgSO 4 ×7H 2 O – 0.15, Ca(NO 3 ) 2 – 0.01 с внесением в качестве селективного фактора бифенила в концентрации 1 г/л.
Тотальная ДНК
Тотальную ДНК из бактериальных ассоциаций выделяли с использованием коммерческого набора реактивов FastDNA Spin Kit for Soil (MP Biomedicals, USA). Концентрацию ДНК измеряли на приборе QubitTM Fluorometer (Invitrogen, США) с приминением реактивов производителя.
Амплификация гена benA, кордирующего α-субъединицу бензоат диоксигеназы
Амплификацию гена benA на матрице тотальной ДНК, выделенной из бактериальных ассоциаций, проводили с использованием бактериальных праймеров прямого benA-F [5’-GCCCACGAGAGCCAGATTCCC-3’] и обратного benA-R [5’-GGTGGCGGCGTAGTTCCAGTG-3’] [Baggi et al., 2008]. Праймеры подобраны к консервативному участку гена benA штамма Acineto-bacter baylyi ADP1, амплифицируемая область со 175 нуклеотида до 712 нуклеотида (размер фрагмента 521 пн) [Baggi et al., 2008]. ПЦР проводили в 25 мкл смеси, содержащей 1х буфер для Taq -полимеразы с MgCl 2 (Синтол, Россия), 0.25 мМ дНТФ, 0.5 мкМ каждого праймера, 2 ед. акт. Taq -полимеразы (Синтол, Россия) и 2 мкл ДНК матрицы. Амплификацию осуществляли на приборе С1000 Touch («Bio-Rad Laboratories», США) в режиме: начальный денатурирующий шаг при 95°С в течение 5 мин., далее 30 циклов: 40 сек. при 94°С, 50 сек. при 60°С с понижением при каждом шаге на 0.4°С, 1 мин. при 72°С, завершающий шаг 7 мин. при 72°.
Визуализация амплифицированных фрагментов ДНК
Продукты ПЦР разделяли электрофорезом в агарозном геле (концентрация агарозы 0.8%) в 1х Трис-боратном буфере (Thermo scientific, Литва) при напряжении 10V/см и визуализировали в проходящем УФ-свете с использованием системы Gel Doc XRtm («Bio-Rad Laboratories», США) после окрашивания в растворе бромистого этидия.
Определение нуклеотидных последовательностей амплифицированных фрагментов ДНК
Определение нуклеотидных последовательностей гена benA осуществляли на автоматическом секвенаторе Genetic Analyzer 3500xl (Applied Biosystems, США), с применением реактивов Big Dye Terminator Ready Reaction Kit v 3.1 (Applied Biosystems, США), согласно рекомендациям производителя, в молекулярно-генетической лабора- тории кафедры ботаники и генетики растений ПГНИУ.
Анализ нуклеотидных последовательностей амплифицированных фрагментов гена benA
Поиск гомологичных последовательностей был произведен по базе данных GenBank . С применением пакета программ Mega версия 7.0, выявленные по базе данных и секвенированные в настоящем исследовании нуклеотидные последовательности гена benA были выровнены с последующим расчетом их сходства, которое было отображено в виде графической модели эволюционного дерева.
Размещение нуклеотидных последовательностей в международных базах данных
Нуклеотидные последовательности фрагмента гена benA , полученные в настоящем исследовании, были депонированы в международной базе данных GenBank. Номера, присвоенные данным последовательностям, представлены в разделе «Результаты и обсуждение».
Результаты и их обсуждение
Бактериальные ассоциации, полученные в результате селекции на минеральной среде в условиях, когда источником углерода является труднодоступное ароматическое вещество (бифенил/поли-хлорированные бифенилы), характеризуются наличием определенного набора генов, обусловливающих способность членов ассоциации использовать данный источник углерода в качестве ростового субстрата. Одним из таких генов является ген benA, кодирующий α-субъединицу бензоат диоксигеназы.
На ДНК-матрице бактериальных ассоциаций CHN1, CHN2, CHN3, CHN4, CHN5, CНN6, PN1 и PN2 с помощью праймеров benA-F и benA-R были амплифицированы фрагменты гена benA . Со всех образцов тотальной ДНК были получены ПЦР-продукты ожидаемого размера – 500 п.н. (рис. 1).
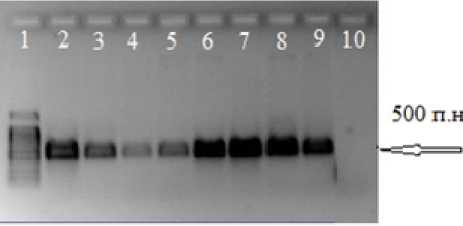
Рис. 1 . Электрофореграмма продуктов амплификации гена benA , кодирующего α-субъеди-ницу бензоат диоксигеназы:
1 – молекулярный маркер O'GeneRulerTM 100bp Plus DNA Ladder (Fermentas, Lithuania), 2 – CHN1, 3 – CHN2, 4 – CHN3, 5 – CHN4, 6 – CHN5, 7 – CHN6, 8 – PN1, 9 – PN2, 10 – отрицательный контроль
В результате проведения секвенирующей реакции и последующего секвенирования по Сэнгеру ампли-фицированных фрагментов, были получены хроматограммы, позволяющие осуществлять дальнейший анализ нуклеотидных последовательностей.
Проведены филогенетический и кластерный анализы полученных фрагментов гена benA с гомологичными последовательностями из базы данных GenBank (таблица, рис. 2).
Филогенетический анализ амплифицированных фрагментов гена benA из тотальной ДНК бактериальных ассоциаций
|
Ассоциация / номер в GenBank |
Штамм с наиболее близким геном benA , номер GenBank Штамм-деструктор с наиболее близким геном benA , номер GenBank |
Сходство, % / Перекрывание, % |
Количество проанализированных нуклеотидов |
|
CHN1 / MN396893 |
Pseudomonas putida JBC17 (CP029693.1) Pseudomonas putida KT2440 (LT799039.1) |
100 / 100 89.7 / 96 |
540 |
|
CHN2 / MN396894 |
Ralstonia euthropha JMP134 (CP000091.1) |
82.3 / 100 |
485 |
|
CHN3 / MN396895 |
Methylobacterium currus PR1016A (CP028843.1) Ralstonia euthropha JMP134 (CP000091.1) |
100 / 100 83.3 / 99 |
517 |
|
CHN4 / MN396896 |
Rhodococcus sp. S2-17 (CP021354.1) Rhodococcus jostii RHA1 (CP000431.1) |
100 / 100 96 / 100 |
476 |
|
CHN5 / MN396897 |
Ralstonia solanacearum SL3022 (CP023016.1) Ralstonia euthropha JMP134 (CP000091.1) |
100 / 99 88.8 / 99 |
493 |
|
CHN6 / MN396898 |
Burkholderia gladioli Co14 (CP033431.1) Ralstonia euthropha JMP134 (CP000091.1) |
100 / 100 79.5 / 98 |
498 |
|
PN1 / MN396900 |
Pseudomonas stutzeri CCUG 29243 (CP003677.1) Pseudomonas putida KF715 (AP015029.1) |
100 / 100 82.5 / 100 |
491 |
|
PN2 / MN396899 |
Pseudomonas putida JBC17 (CP029693.1) Pseudomonas knackmussii B13 (HG322950.1) |
100 / 100 89.5 / 100 |
524 |
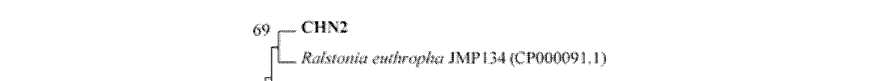

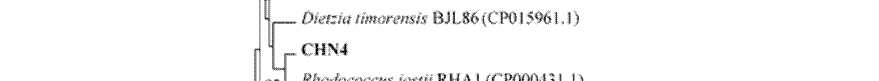
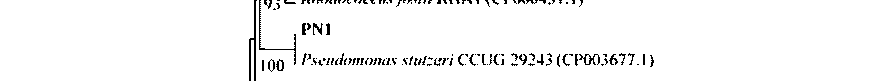


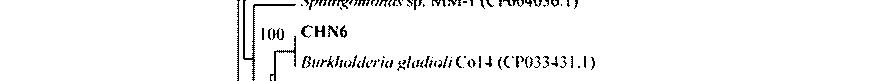

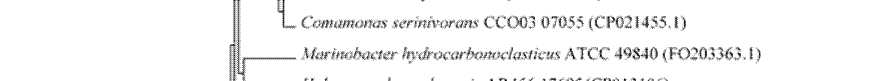
Сот.ишпшх чтатгар ( ( 003 (17055 ICP021455 I)
Marinч:кп ter iiydnivarbonoclnxtivus Al ((' 40X40 (H >20 3363. !)
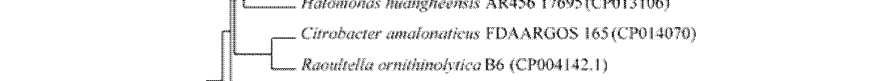

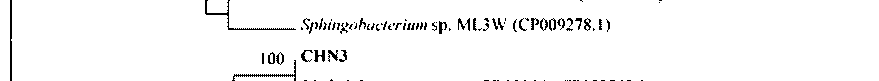

L PN2
—t-----------1-----------1-----------1-----------1-----------1-----------(
0 6 0,5 0.4 0.5 0.2 ill 0 0
Рис. 2. Дерево сходства выявленных генов с известными генами α-субъединицы бензоат 1,2-ди-оксигеназы, построенное методом UPGMA.
Масштаб соответствует 10 нуклеотидным заменам на каждые 100 нуклеотидов. «Bootstrap»-анализ проведен на 1000 повторностях. Значения рядом с «ветвями» показывают вероятность расположения последовательностей в данных группах. Жирным шрифтом выделены нуклеотидные последовательности, исследуемые в настоящей работе
Выявлен высокий уровень сходства амплифи-цированных участков гена benA исследуемых ассоциаций (100%) с генами подсемейства бензоат диоксигеназ бактерий различных таксономических групп, осуществляющих деструкцию ароматических соединений (таблица). Установлено, что в
ДНК исследуемых ассоциаций преимущественно амплифицировались гены бензоат диоксигеназ, характерных для бактерий отдела/филума Proteobacteria . Наиболее филогенетически близким среди генов бензоат диоксигеназы активных штаммов-деструкторов ароматических соединений для фрагментов гена benA из ассоциаций CHN2, CHN3, CHN5 и CHN6 является соответствующий ген штамма Ralstonia euthropha JMP134 (уровень сходства 79.5–88.8%). Известно, что штамм R. euthropha JMP134 осуществляет разложение широкого спектра ароматических соединений, в том числе бензойной и хлорбензойных кислот [Field, Sierra-Alvarez, 2008а]. Стоит отметить, что для фрагмента гена benA ассоциации CHN1 наиболее близким является ген бензоат диоксигеназы другого известного штамма-деструктора Pseudomonas putida KT2440 (таблица). Штамм P. putida KT2440 осуществляет разложение ароматических соединений и является одним из модельных штаммов, при анализе генетических систем [Kahlon R.S., 2016].
Фрагмент гена benA ассоциации CHN4, формирует единый кластер с соответствующим геном штаммов рода Rhodococcus класса Actinobacteria (таблица, рис. 2). Стоит отметить, что benA CHN4 имеет высокий уровень сходства (96%) с соответствующим геном известного штамма-деструктора бифенила/ПХБ Rhodococcus jostii RHA1 [Kitagawa et al., 2001].
Таким образом, в ассоциациях, селектированных из почв, отобранных на территории ОАО «СВЗХ» (г. Чапаевск, Самарская обл., Россия), выявлены фрагменты гена бензоат диоксигеназы, обладающие высоким уровнем сходства с benA -генами штаммов разных таксономических групп.
Иная картина получена при анализе нуклеотидных последовательностей фрагментов гена бензоат диоксигеназы, амплифицированных с тотальной ДНК ассоциаций PN1 и PN2, селектированных из почв ОАО “Пермский завод смазок и СОЖ” (г. Пермь, Пермский край, Россия). Наиболее близкими в данном случае по benA гену являются штаммы рода Pseudomonas, в том числе деструкторы Pseudomonas putida KF715 и Pseudomonas knackmussii B13 (уровни сходства 82.5 и 89.5%, соответственно). Штамм P. putida KF715 является высокоактивным деструктором хлорированных бифенилов, содержащих различное количества атомов хлора в молекуле бифенила, а также смесей ПХБ и обладает ферментативными путями, обусловливающими разложение бифенила до соединений основного обмена клетки [Suenaga et al., 2017; Kimura et al., 2018]. Штамм P. knackmussii B13 активно разлагает хлорбензойные кислоты, а в результате переноса ряда его генов в реципиент-ные штаммы получены трансконъюганты, способные использовать широкий спектр моно- и ди- хлорбензойных кислот [Kahlon, 2016]. Ранее было показано, что при культивировании почвенных микробиоценозов в присутствии бензола и 4-хлорбензойной кислоты в качестве селективных факторов, так же формируются сообщества, в которых присутствует бензоат диоксигеназа бактерий рода Pseudomonas [Назарова, Кирьянова, Егорова, 2019]. Стоит отметить, что кластерный анализ показал наличие существенных различий в нуклеотидных последовательностях выявленных генов benA ассоциаций PN1 и PN2 (рис. 2). Вероятно, ген бензоат диоксигеназы имеет значительную вариабельность в пределах рода Pseudomonas, что подтверждается формированием нескольких кластеров при графической визуализации результата анализа (рис. 2). Такое разнообразие может быть обусловлено как длительными эволюционными изменениями, так и адаптационными процессами, вызванными присутствием в окружающей среде химических поллютантов.
Заключение
В результате проведенных исследований изучены фрагменты гена benA , кодирующего α-субъ-единицу бензоат 1,2-диоксигеназы, присутствующие в тотальной ДНК восьми ассоциаций аэробных бактерий, селектированных на бифениле/по-лихлорированных бифенилах.
Показано, что амплифицированные участки гена benA имеют высокий уровень сходства (до 100%) с генами бензоат диоксигеназ штаммов, принадлежащих родам Burkholderia , Methylobacte-rium, Pseudomonas, Ralstonia и Rhodococcus . Установлено, что выявленные нуклеотидные последовательности были сходны на 79.5–96.0% с ben A-генами известных штаммов-деструкторов бензой-ных/хлорбензойных кислот и бифенила/поли-хлорированных бифенилов. Полученный результат позволяет высказать предположение, что в результате селекции в присутствии бифенила/ПХБ формируются бактериальные сообщества, обладающие набором генетических систем, обусловливающих биотрансформацию ароматических соединений.
Таким образом, в результате проведенных исследований изучено разнообразие гена benA , представленного в аэробных бактериальных ассоциациях, селектированных из почвенных микробиоценозов под воздействием бифенила/полихло-рированных бифенилов.
Работа выполнена при поддержке Российского фонда фундаментальных исследований, грант №18-29-05016мк.
Список литературы Скрининг гена альфа-субъединицы бензоат диоксигеназы в бактериальных ассоциациях, полученных в результате селекции на (хлор)ароматических соединениях
- Назарова Э.А., Кирьянова Т.Д., Егорова Д.О. Разнообразие гена бензоат диоксигеназы в бактериальных ассоциациях, сформировавшихся под давлением хлорорганического загрязнения // Экологическая генетика. 2019. Т. 17, № 3. С. 13-22. DOI: 10.17816/ecogen17313-22
- Abd El-Mawla A.M., Beerhues L. Benzoic acid biosynthesis in cell cultures of Hypericum androsaemum // Planta. 2002. Vol. 214. P.727. DOI: 10.1007/s004250100657
- Baggi G. et al. Co-metabolism of di- and trichlorobenzoates in a 2-chlorobenzoate-degrading bacterial culture: Effect of the position and number of halo-substituents // International Biodeterioration and Biodegradation. 2008. Vol. 62, № 1. P. 57-64. DOI: 10.1016/j.ibiod.2007.12.002
- Field J.A., Sierra-Alvarez R. Microbial transformation of chlorinated benzoates // Reviews in Environmental Science and BioTechnology. 2008a. Vol. 7. P. 191-210. DOI: 10.1007/s11157-0089133-z
- Field J.A., Sierra-Alvarez R. Microbial transformation and degradation of polychlorinated biphenyls // Environmental Pollution. 2008b. Vol. 155, № 1. P. 1-12.
- Haddad S., Eby D.M., Neidle E.L. Cloning and expression of the benzoate dioxygenase genes from Rhodococcus sp. strain 19070. // Applied Environmental Microbiology. 2001. Vol. 67. P. 2507-2514.
- Kahlon R.S. Pseudomonas: Molekular and Applied Biology. Springer International Publishing Switzerland, 2016. 519 p.
- DOI: 10.1007/978-3-319-31198-2_4
- Kimura N. et al. Pseudomonas furukawaii sp. nov., a polychlorinated biphenyl-degrading bacterium isolated from biphenyl-contaminated soil in Japan // International Journal Systematic Evolution Microbiology. 2018. Vol. 68, № 5. P. 1429-1435
- Kitagawa W. et al. Cloning and characterization of benzoate catabolic genes in the gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1 // Journal of Bacteriology. 2001. Vol. 183. P. 6598-6606.
- Parales R.E., Resnick S.M. Aromatic Ring Hydroxylating Dioxygenases // Pseudomonas / J.L. Ramos, R.C. Levesque, eds. Boston: Springer, 2006. P. 287-340.
- DOI: 10.1007/2F0-387-28881-3_9
- Pieper D.H. Aerobic degradation of polychlorinated biphenyls. // Applied Microbiology Biotechnology. 2005. Vol. 67, № 2. P. 170-191.
- DOI: 10.1007/s00253-004-1810-4
- Solyanikova I.P. et al. Pecularities of the degradation of benzoate and its chloro- and hydraxysubstituted analogs by Actinobacteria. // International Biodeterioration and Biodegradation. 2015. Vol. 100. P. 155-164.
- DOI: 10.1016/j.ibiod.2015.02.028
- Suenaga H. et al. Insights into the genomic plasticity of Pseudomonas putida KF715, a strain with unique biphenyl-utilizing activity and genome instability properties // Environmenmental Microbiology Reports. 2017. Vol. 9, № 5. P. 589-598.


