Скрининг и изучение ключевых генов катаболизма бифенила и полихлорированных бифенилов у аэробных бактерий
Автор: Шумковa Е.С., Ананьина Л.Н., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 9, 2008 года.
Бесплатный доступ
Методом полимеразной цепной реакции проведен скрининг бактерий-деструкторов бифенила на наличие гена bphA1, кодирующего a-субъединицу бифенил диоксигеназы, ключевого фермента разложения бифенила и полихлорированных бифенилов (ПХБ). Анализ нуклеотидных последовательностей показал высокий уровень сходства (98%) участка гена bphA1 длиной около 500 п.н. исследуемых штаммов рода Rhodococcus и известных бактерийдеструкторов того же рода. Показан высокий уровень сходства гена bphA1 грамотрицательных штаммов родов Pseudomonas и Comamonas и активных деструкторов ПХБ рода Pseudomonas (99-100%). Выявлены бактерии, у которых нуклеотидные последовательности, кодирующие a-субъединицу бифенил диоксигеназы, существенно отличаются от описанных в литературе.
Короткий адрес: https://sciup.org/147204436
IDR: 147204436 | УДК: 579.25
Текст научной статьи Скрининг и изучение ключевых генов катаболизма бифенила и полихлорированных бифенилов у аэробных бактерий
Е. С. Шумковaа, Л. Н. Ананьинаа, Е. Г. Плотниковаа,b a aИнститут экологии и генетики микроорганизмов, 614081, Пермь, ул. Голева, 13 b Пермский государственный университет, 614990, Пермь, ул. Букирева, 15
Методом полимеразной цепной реакции проведен скрининг бактерий-деструкторов бифенила на наличие гена bphA1 , кодирующего α-субъединицу бифенил диоксигеназы, ключевого фермента разложения бифенила и полихлорированных бифенилов (ПХБ). Анализ нуклеотидных последовательностей показал высокий уровень сходства (98%) участка гена bphA1 длиной около 500 п.н. исследуемых штаммов рода Rhodococcus и известных бактерий-деструкторов того же рода. Показан высокий уровень сходства гена bphA1 грамотрицатель-ных штаммов родов Pseudomonas и Comamonas и активных деструкторов ПХБ рода Pseudomonas (99–100%). Выявлены бактерии, у которых нуклеотидные последовательности, кодирующие α-субъединицу бифенил диоксигеназы, существенно отличаются от описанных в литературе.
Полихлорированные бифенилы (ПХБ) – синтетические соединения, широко использующиеся в электрохимической и электротехнической промышленности. Благодаря исключительной устойчивости к химическим, физическим воздействиям и к биодеструкции они входят в число наиболее распространенных и опасных загрязнителей окружающей среды. Известно, что, несмотря на низкую биодоступность ПХБ, основная роль их трансформации в природной среде принадлежит микроорганизмам (Dua et al., 2002; Hickey et al., 1993; Abraham et al, 2002). Многочисленные грамположительные и грамотрицатель-ные бактерии-деструкторы ПХБ, принадлежащие к различным систематическим группам, были выделены из почв и донных осадков (Furukava et al., 1986; Bedard et al., 1986; Seto et al., 1995; Sakai et al., 2002).
Изучение организации биохимических путей деструкции этих соединений и генетических систем, кодирующих ферменты деградации ПХБ, приобретает все большую актуальность. Работы в этом направлении важны как в теоретическом плане, так и для решения практических задач, таких как разработка методов биоремедиации загрязненных почв и водных объектов (Suenaga et al., 2002).
Происхождение генов, позволяющих бактериям использовать ПХБ в качестве субстрата, до сих пор остается неясным и является источником дискуссий (Abraham et al., 2002). Знания о путях их эволюции, в свою очередь, позволят более эффективно решать прикладные проблемы, такие, как искусственное конструирование путей деструкции тех или иных ксенобиотиков и создание ферментов с заданными свойствами.
На первом этапе разложения бифенил и ПХБ окисляются до (хлор)бифенил-дигидродиола бифенил 2,3-диоксигеназой (БДО). Бифенил 2,3-диоксигеназа относится к семейству негеминовых бифенил/толуольных оксигеназ, содержащих железо-серный кластер Риске (Gibson, Parales, 2000). Это мультикомпонентный фермент, состоящий из большой (α) и малой (β) субъединиц терминальной диоксигеназы (кодируются генами bphA1 и bphA2 соответственно), ферредоксина ( bphA3 ) и ферре-доксин-редуктазы ( bphA4 ). BphA1 и BphA2 ассоциированы в гетерогексамер α3β3 и катализируют внедрение двух атомов кислорода в ароматическое кольцо (хлор)бифенила. В значительной степени спектр окисляемых бактерией ПХБ определяется субстратной специфичностью ее БДО. α-Субъединица отвечает за распознавание субстрата и связывание с ним (Pieper, 2005).
Ранее из почв, загрязненных отходами производства галогенированных соединений (г. Пермь, г. Березники, Пермский край), были выделены аэробные бактерии, способные осуществлять разложение бифенила и его хлорированных производных (Плотникова и др., 2006; Плотникова и др., 2007; Рыбкина, 2003).
Целью настоящей работы является поиск и изучение генов, кодирующих большую субъединицу БДО, у бактерий-деструкторов бифенила и ПХБ, выделенных из техногенных почв.
Материалы и методы исследования
Бактериальные штаммы. В работе использованы бактериальные штаммы, выделенные из образцов почв, отобранных на территории предприятия ОАО «Галоген» (г. Пермь) и из почв района солеразработок г. Березники (Пермский край): Cel-lulomonas (P27, P28), Rhodococcus (P1, P2, Р2(51), P12, P13, P20, P25, P9, G10), Microbacterium (P26), Pseudomonas (B7, B106а, G12, G13, P22, P23а, P24а, S9, S13), Comamonas (S210, S211, S212), Al-caligenes (P39), Flavimonas (S214) (Плотникова и др., 2006; Плотникова и др., 2007; Рыбкина, 2003).
Среды и условия культивирования. Культивирование бактерий проводили в колбах объемом 250 мл в 100 мл минеральной среды K1 (Zaitsev et al., 1991) при 28°С с аэрацией на шейкере (100 об/мин). В качестве ростового субстрата использовали бифенил в концентрации 1 г/л.
Молекулярно-генетические методы. Выделение тотальной ДНК исследуемых штаммов проводили по стандартной методике (Ausbel et al., 1995).
ПЦР осуществляли на приборе MyCycler (BioRad, США) с вырожденными праймерами, подобранными к консервативным участкам гена bphA1 при условиях, предложенных R. Witzig (2006).
Анализ полиморфизма длин рестрикционных фрагментов (ПДРФ) амплифицированных участков генов bphА1 осуществляли с использованием эндонуклеаз рестрикции Hha I , Hae III (Sigma, Германия). Реакцию проводили при 37°С в течение 2 часов в объеме реакционной смеси 20 мкл. Построение карт сайтов рестрикции для участков исследуемых генов bphA1 осуществляли с помощью программы Vector NTI 9.1.0 на основе гомологичных нуклеотидных последовательностей известных деструкторов ПХБ Burkholderia xenovorans LB400 (GenBank 86348) и Rhodococcus globerulus P6 (GenBank Х80041).
Секвенирование продуктов амплификации проводили с помощью набора реактивов DYEnam-ic ET Dye Terminator Cycle sequencing Kit на автоматическом секвенаторе MegaBASE 1000 (JSC GE Healthcare, США) согласно рекомендациям производителя. Полученные нуклеотидные последовательности анализировали с использованием программ CLUSTAL W (Thompson et al., 1994), TREECON (van de Peer, DeWachter, 1994), BLAST .
Результаты исследования и их обсуждение
Скрининг штаммов на наличие гена bphA1
Методом полимеразной цепной реакции (ПЦР) был проведен скрининг 26 штаммов на наличие в геноме нуклеотидных последовательностей, гомо- логичных участку гена bphA1. С матриц ДНК четырнадцати штаммов (P1, P9, P12, P13, P20, P24а, P27, P28, S9, S13, S210, S211, S212, G10) прошла специфичная амплификация. Размер ПЦР-продук-та составлял около 500 п.н. Протяженность гена bphA1 у Rhodococcus globerulus P6 и Burkholderia xenovorans LB400 составляет 1375 и 1378 п.н. соответственно. Амплифицируемая область гена bphA1, располагающаяся в районе 668 – 1152 п.н., охватывает область около 500 п.н. Таким образом, длина амплифицированных фрагментов bphA1-гена с матриц ДНК исследуемых штаммов совпадала с ожидаемой.
Полученные данные позволили нам предположить наличие bphA1 -гена, кодирующего большую субъединицу БДО, у скринируемых бактерий в тех случаях, когда прошла специфичная амплификация.
ПДРФ-анализ амплифицированных участков гена bphA1
Для выявления сходства и различий между ам-плифицированными участками гена bphA1 был проведен анализ полиморфизма длин рестрикционных фрагментов (ПДРФ-анализ) полученных ампликонов.
По результатам гидролиза ДНК эндонуклеазами рестрикции Hha I и Hae III исследуемые штаммы можно подразделить на три группы. К первой группе относятся штаммы рода Rhodococcus (P1, P12, P13, P20), набор рестрикционных фрагментов амплифицированного участка гена bphA1 которых был сходен с таковым известных бактерий-деструкторов ПХБ рода Rhodococcus . При гидролизе ДНК грамотрицательных штаммов родов Comamonas (S210, S211, S212) и Pseudomonas (S9, S13) эндонуклеазой рестрикции Hae III были получены фрагменты других размеров. Расположение сайтов рестрикции на участке гена bphA1 было сходно с таковым известных бактерий рода Pseudomonas и Burkholderia . Эти штаммы составляют вторую группу. В третью группу по результатам гидролиза амплифицированной ДНК эндонуклеазами Hha I и Hae III можно включить штаммы Pseudomonas sp. Р24а, Rhodococcus sp. P9 и Cellu-lomonas spp. Р27, P28.
Анализ нуклеотидных последовательностей гена bphА1
Для определения нуклеотидных последовательностей амплифицированных фрагментов bphA1 представителей каждой из трех групп, выявленных в результате ПДРФ-анализа, были выбраны штаммы P12, P13, G10, S13, S212, S9, S210, P27, P24а, P9.
С помощью программ BLAST и CLUSTALW нуклеотидные последовательности фрагментов гена bphA1 сравнивали с аналогичными последовательностями, имеющимися в международных базах данных GenBank/EMBL/DDBJ. Идентичность нуклеотидных последовательностей bphA1 штаммов Rhodococcus spp. P12 и P13 с Rhodococcus opacus BIE-20 (GenBank AJ544524) составляет 98%. Уровень гомологии гена bphA1 грамотрицательных штаммов Pseudomonas spp. S9 и S13, Comamonas spp. S210, S212 с Pseudomonas sp. B2A (GenBank AJ544518) составляет 99–100%, с Pseudomonas sp. B4 (GenBank PSU95054) – 99%; с Ralstonia eutropha H850 (GenBank AJ544525) и c Burkholderia xenovorans LB400 (GenBank M86348) – 97%. Участок гена bphA1 штамма Rhodococcus sp. G10 на 98% сходен с гомологичной нуклеотидной последовательностю штамма Arthrobacter sp. 3YC3 (GenBank DQ336942).
Построена дендрограмма сходства нуклеотидных последовательностей, кодирующих α-субъединицы бифенил диоксигеназ, известных штаммов-деструкторов ПХБ и исследуемых бактерий (рисунок). Кластерный анализ показал, что грамотрицательные и грамположительные штаммы образуют две разные группы. Штаммы Rhodococcus spp. P12 и P13 группируются с представителями рода Rhodococcus, а штаммы Pseudomonas spp. S9, S13 и Comamonas spp. S210, S212 попадают в один кластер с грамотрица-тельными деструкторами ПХБ, принадлежащими к родам Pseudomonas, Burkholderia и Ralstonia (рисунок).
Нуклеотидные последовательности фрагментов гена bphA1 штаммов Rhodococcus sp. P9, Pseudomonas sp. P24 и Cellulomonas sp. P27 значительно отличаются от имеющихся в базах данных. Перекрывание их с гомологичными последовательностями известных деструкторов ПХБ составляет всего 20–30% от длины полученных ампликонов. Штаммы P9, P24 и P27 образуют самостоятельную группу на дендрограмме.
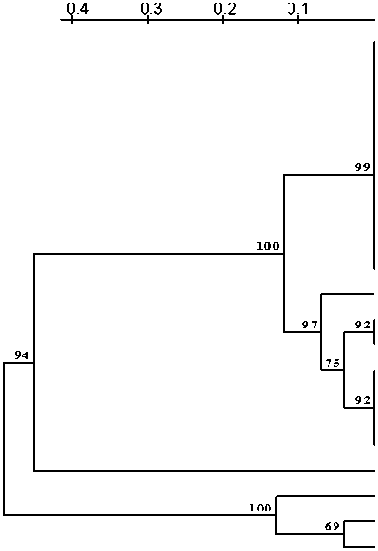
Comamonas sp. 8210
Pseudomonas sp. В2А (AJ544518)
Pseudomon as sp 89
Pseudom onas sp. 81 3
Pseudomonas sp. Cam-2 (A1792 7652)
P. pseudoalcaligenes (M83673)
Comamonas sp. 8212
Burkholderiaxenov orans (M86384)
Ralstonia eut-opha H850 (AJ544525)
Pseudomonas sp. B4 (U95054)
Rhodococcus erythropolis (D88020)
Rhodococcus sp. GIO
Arthrobactersp. 3YC3 (DQ336942)
Rhodococcus opacus (AJ544524)
Rhodococcus sp. RHA1 (D32142)
RhadQCQCsus sp. P12
Rhodococcus sp. P13
Rhodococcus sp. Ml (U27o91)
Rhodococcus sp. P9
Pseudomonas sp. P24
Cellulomonas sp. P27
Древо сходства нуклеотидных последовательностей, гомологичных исследуемым участкам генов bphА1 , построенное методом UPGMA. Масштаб соответствует 10 нуклеотидным заменам на каждые 100 нуклеотидов. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью «bootstrap»-анализа 100 альтернативных деревьев. В скобках указаны номера последовательностей в GenBank.
Заключение
Двадцать шесть штаммов бактерий-деструкторов бифенила были исследованы на наличие гена большой субъединицы БДО, фермента, осуществляющего первый этап разложения бифенила и ПХБ – внедрение двух атомов кислорода в ароматическое кольцо. Скрининг исследуемых бактериальных культур методом полимеразной цепной реакции позволил предположить функционирование у ряда исследованных штаммов бифенил диоксигеназ, сходных с описанными в научной литературе (Erickson., Mondello, 1992; Kah., Hofer, 2003; Masai et al., 1995; Master., Mohn, 2001; Taira et al.,
1992). В то же время отрицательный результат ПЦР-скрининга у других двенадцати штаммов-деструкторов бифенила может свидетельствовать о существенных отличиях нуклеотидных последовательностей их гена bphA1 от аналогичных последовательностей известных бактерий-деструкторов.
На основании ПДРФ-анализа амплифици-рованных фрагментов гена bphA1 можно говорить о значительном сходстве амплифицированных участков bphA1 у исследуемых штаммов рода Rhodococcus и их отличии от грамотрицательных бактерий.
Анализ нуклеотидных последовательностей участка гена bphA1 показал высокий уровень сходства (98%) исследуемых штаммов рода Rhodococcus с известными деструкторами ПХБ того же рода. Выявлен высокий уровень сходства грамотрица-тельных штаммов родов Pseudomonas и Comamonas с представителями рода Pseudomonas (99–100%). В то же время в результате ПДРФ-анализа и анализа нуклеотидных последовательностей у нескольких исследуемых штаммов (Р9, Р24а, Р27) были установлены существенные различия в структуре ключевого гена катаболизма бифенила и ПХБ (bphA1), что позволяет предположить наличие у данных штаммов бифенил диоксигеназ, отличающихся от описанных в литературе.
Работа поддержана грантом РФФИ-Урал_офи 07-04-97625.
Список литературы Скрининг и изучение ключевых генов катаболизма бифенила и полихлорированных бифенилов у аэробных бактерий
- Плотникова, Е.Г. Характеристика микроорганизмов, выделенных из техногенных почв Прикамья/Е.Г. Плотникова, Д.О. Рыбкина, Л.Н. Ананьина, О.В. Ястребова, В.А. Демаков//Экология. 2006. №. 4. С. 261-268.
- Плотникова, Е.Г. Бактерии-деструкторы полихлорированных бифенилов, перспективные для биоремедиации загрязненных почв/Е.Г. Плотникова, Д.О. Рыбкина//Биотехнология: состояние и перспективы развития: материалы конгресса. Ч. 2. М. 2005. С. 135.
- Рыбкина, Д.О. Исследование аэробных бактерий, разлагающих полихлорированные бифенилы и хлорбензойные кислоты: дис. канд. биол. наук/Д.О. Рыбкина. Пермь, 2003. 181 с.
- Abraham, W.R. Polychlorinated biphenyl-degrading microbial communities in soils and sediments/W.R.Abraham, B. Nogales, P.N. Golyshin, D.H. Pieper, K.N. Timmis//Curr. Opin. Microbiol. 2002. Vol. 5. P. 246-253.
- Bedard, D.L. Rapid assay for screening and characterizing microoranisms for the ability to degrade polychlorinated biphenyls/D.L. Bedard, R. Unterman, N. Ropp, M.I. Brennan, M.L. Haber, C. Jonson//Appl. Environ. Microbiol. 1986. Vol. 51. P. 761-768.
- Dua,M. Biotechnology and bioremediation: successes and limitations/M. Dua, A. Singh, N. Ssethunathan, A.K. Johri//Appl. Microbiol. Biothechnol. 2002. Vol.59. P. 143-152.
- Erickson, B.D. Nucleotide sequencing and transcriptional mapping of the genes encoding biphenyl dioxygenase, a multicomponent polychlorinatedbiphenyl-degrading enzyme in Pseudomonas strain LB400/B.D. Erickson, F.J. Mondello//J. Bacteriol. 1992. Vol. 174 (9). P. 2903-2912.
- Furukava, K. Oxidation of polychlorinated biphenyls by Pseudomonas sp. Strain LB400 and Pseudomonas pseudoalcaligenes KF707/K. Furukava, N. Tomizuka, A. Kamibayashi//J. Bacteriol. 1979. Vol. 175. Р. 4561-4564.
- Gibson, D.T. Aromatic hydrocarbon dioxygenases in environmenyal biotechnology/D.T. Gibson, R.E. Parales//Curr. Opin. Biotechnol. 2000. Vol. 11. P. 236-243.
- Hickey, W.J. Enhanced mineralization of polychlorinated biphenyls in soil inoculated with chlorobenzoate-degradin bacteria/W.J. Hickey, D.B. Searles, D.D. Focht//Appl. Environ. Microbiol. 1993. Vol. 59. P. 1194-1200.
- Kah, S. A genetic system for the rapid isolation of aromatic-ring-hydroxylating dioxygenase activities/S. Kah, B. Hofer//Microbiology. 2003. Vol. 149 (PT 6). P. 1475-1481.
- Masai, E. Characterization of biphenyl catabolic genes of gram-positive polychlorinated biphenyl degrader Rhodococcus sp. strain RHA1/E. Masai, A. Yamada, J.M. Healy, T. Hatta, K. Kimbara, M. Fukuda, K. Yano//Appl. Environ. Microbiol. 1995. Vol. 61 (6). P. 2079-2085.
- Master, E.R. Induction of bphA, encoding biphenyl dioxygenase, in two polychlorinated biphenyldegrading bacteria, psychrotolerant Pseudomonas strain Cam-1 and mesophilic Burkholderia strain LB400/E.R. Master, W.W. Mohn//Appl. Environ. Microbiol. 2001. Vol. 67 (6). P. 2669-2676.
- Pieper D.H. Aerobic degradation of polychlorinated biphenyls//Appl. Microbiol. Biotechnol. 2005. Vol. 67. P. 170-191.
- Sakai, M. Diversity of 2,3-dihydroxybiophenyl dioxygenase genes in a strong PCB degrader, Rhodococcus sp. strain RHA1/M. Sakai, E. Masai, H. Asami, K. Sugiyama, K. Kimbara, M. Fukuda//Journal of Bioscience and Bioengineering. 2002. Vol. 93. P. 421-427.
- Seto, M. A novel transformation of polychlorinated biphenyls by Rhodococcus sp. strain RHA1/M. Seto, K. Kimbara, M. Shimura, T. Hatta, M. Fukuda//Appl. Environ. Microbiol. 1995. Vol. 61. P.3353-3358.
- Ausubel. Short protocols in molecular biology/Ausubel et al.. Third edition. 1995. 450 p.
- Suenaga, H. Alteration of regiospecificity in biphenyl dioxygenase by active-site engeneering/H. Suenaga, T. Watanabe, M. Sato, Ngadiman, K. Furukawa//J. Bacteriol. 2002. Vol. 184. P. 3682-3688.
- Taira, K. Analysis of bph operon from the polychlorinated biphenyl-degrading strain of Pseudomonas pseudoalcaligenes KF707/K. Taira, J. Hirose, S. Hayashida, K. Furukawa//J. Biol. Chem. 1992. Vol. 267 (7). P. 4844-4853.
- Thompson, J.D. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice/J.D. Thompson, D.G. Higgins, T.J. Gibson//Nucleic. Acids. Res. 1994. Vol. 22. P. 4673-4680.
- van de Peer Y. TREECON for Windows a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment/Y. van de Peer, R. DeWachter//Comput. Appl. Biosci. 1994. Vol. 10. P. 569-570.
- Witzig, R. Bensene-Polluted Soils: Lnks between Benzene Biodegradation and enes Similar to Those Encoding Isopropilbenzene Dioxygenases/R. Witzig, J. Howard, H-J. Heht, D.H. Pieper//Appl.Environ.Microbiol. 2006. Vol. 72. No. 5. P. 3504-3514.
- Zaitsev, G.M. Genetic control of degradation of chlorinated benzoic acids in Arthrobacter globiformis, Corynebacterium sepedonicum and Pseudomonas cepacia strains/G.M. Zaitsev, T.V. Tsoi, V.G. Grischenkov, E.G. Plotnikova, A.M. Boronin//FEMS Microbiol. Lett. 1991. Vol. 81. P. 171-176.


