Сравнение методов выделения метагеномной ДНК из образцов почвы
Автор: Галиев Вадим Васильевич, Цырульников А.О.
Журнал: Science for Education Today @sciforedu
Рубрика: Биологические, химические, медицинские науки
Статья в выпуске: 1 (1), 2011 года.
Бесплатный доступ
В данной статье проведен обзор и практическое сравнение различных методов извлечения метагеномной ДНК из почвенных микроорганизмов. Выбор метода извлечения ДНК является важным звеном в проведении научной работы с метагеномными образцами, так как именно от качества выделенной ДНК зависит вся дальнейшая исследовательская деятельность. В работе впервые были сравнены представленные методы извлечения применительно к серым лесным почвам окрестностей г. Новосибирска.
Днк, микроорганизмы, метагеномные образцы, почва
Короткий адрес: https://sciup.org/147137342
IDR: 147137342 | УДК: 577.1
Текст научной статьи Сравнение методов выделения метагеномной ДНК из образцов почвы
В XX веке микроорганизмы позволили производить огромное количество химических соединений, разработанные методики культивирования микроорганизмов подтолкнули к развитию нового сектора экономики – биотехнологической промышленности.
Однако общеизвестно, что культивировать возможно лишь незначительную часть известных науке видов микроорганизмов. Следовательно, еще более значительное количество разнообразных продуктов, синтезируемых бактериями и имеющих коммерческий интерес, предстоит еще ввести в производство.
Такое положение вещей ставит исследователей перед необходимостью поиска методики работы с большим количеством видов микроорганизмов без их культивирования. В последние годы были хорошо отработаны технологии создания рекомбинантных генов, позволяющие, в том числе, встраивать интересующие исследователей нуклеотидные последовательности из некультивируемых микроорганизмов в культивируемые (E. coli, B. subtilis).
Целью данного исследования являлся подбор максимально эффективного метода извлечения суммарной ДНК микроорганизмов из почвенных образцов, пригодной для дальнейшего исследования с помощью ПЦР амплификации.
Материалы и методы исследования
Образцы почвы были собраны под кроной Betula Pendula в лесу смешанного типа в окрестностях НГУ леса Новосибирского Академгородка и хранятся в замороженном состоянии (-20 0С) при естественной влажности.
В работе проведено сравнение методов, ранее успешно примененных различными группами исследователей.
Экстракция почвенной ДНК
Метод1(с использованием протеиназы К и SDS [1])
Почва (0.5 г) была суспендирована в 1.5 мл литического буфера (100 мМ трис-HCl (pH 8.0), 100 мМ Na 3 PO 4 (pH 8.0), 100 мМ Na-EDTA1 (pH 8.0), 1% CTAB2, 1.5 М. NaCl) и 150 мкл раствора протеиназы K (10 мкг/мл).
Суспензию интенсивно перемешивали на горизонтальном шейкере на частоте 225 колебаний в минуту в течение 30 минут при комнатной температуре.
Затем добавили 160 мкл 20%-го SDS3, и пробу выдержали 2 часа в жидкостном термостате при температуре 65оС с легким покачиванием каждые 15 – 20 минут.
Осадок отделяли центрифугированием (10 минут, 6 000 об/мин при комнатной температуре в 1.5 мл пробирке Eppendorf на центрифуге Eppendorf 5415C).
К супернатанту добавляли равный объем смеси фенол/хлоро-форм/изоамиловый спирт (25:24:1). Водную фазу собрали после центрифугирования при 6000 оборотах в течение 10 минут, после чего осаждали изопропанолом (0.6 объема) при 4оC в течение ночи. Осадок собирали центрифугированием при 6000 об/мин в течение 15 минут, осадок промывали холодным 70% этанолом, высушивали и разбавляли водой до объема 50 мкл.
Метод 2 (CTAB метод [1])
0.5 г почвы суспендировали в буфере (100 мМ трис-HCl (pH 8.0), 100 мМ Na 3 PO 4 (pH 8.0), 100 мМ Na-EDTA (pH 8.0), 2% [в/о] CTAB, 1.5 М NaCl), затем приливали 160 мкл 20% SDS. Образцы выдерживали при 65оС на водяной бане в течение 2-х часов с аккуратным покачиванием каждые 15 – 20 мин.
Остальные процедуры проводили аналогично первому методу.
Метод 3 (замораживания – оттаивания [1])
0.5 гр. почвы смешивали с 1.5 мл буфера (100 мМ трис-HCl (pH 8.0), 100 мМ Na 3 PO 4 (pH 8.0), 100 мМ Nа-EDTA (pH 8.0), 1% [в/о] CTAB, 1.5 М NaCl), затем пробу замораживали в жидком азоте (10 мин) и разогревали до 65оС (10 мин), операцию повторяли три раза. Остальные процедуры проводили аналогично первому методу.
Данный метод был дополнен стадией выдерживания при 65оС в течение 2 часов с добавлением 160 мкл SDS ( Метод 4 ). Таким образом, этот метод представлен в исследовании в 2 вариациях, одна из которых дополнена выдерживанием с SDS ( Метод 4 ), а другая без таковой ( Метод 3 ).
Метод 5 (с использованием TENC буфера [2])
Почвенную навеску (0.5 г) смешивали с 1.5 мл TENС буфера (100 мМ трис-HCl, 10 мМ EDTA, 100 мМ NaCl, 1% [в/о] CTAB, 1 мл 50 мкг/мл протеиназы K, pH 8.0), затем смесь замораживали в жидком азоте и нагревали до 56оС на 10 мин, после чего к смеси добавляли 100 мкл 20% SDS и смесь нагревали до 65оС на 10 минут. Далее – как в первом методе.
Метод 6 (измельчения в жидком азоте [3])
Почвенную навеску (0.5 г) замораживали в жидком азоте и мелко измельчали в ступке пестиком, пыль суспендировали в 1 мл буфера (0.3% SDS, 0.14 M NaCl, 50 мМ ацетат натрия [pH 5.1]). Суспензию смешивали с равным объемом насыщенного водного раствора фенола и перемешивали на микрошейкере в течение 2 мин.
Очистка центрифугированием при 12 000 об/мин проводилась в течение 10 мин. Супернатант смешивали с 2.5 объемами этанола и осаждали при – 20оС в течение 16 часов.
После центрифугирования при 14 000 об/мин в течение 6 мин, выделенную ДНК дважды промывали этанолом, а затем высушили. Сухие гранулы растворяли в 50 мкл воды.
Хранение всей извлеченной ДНК при – 20оС.
Сравнение методов экстракции ДНК
Для сравнения методов между собой принимали во внимание 3 критерия:
-
1. Извлечение ДНК максимальной длины, чтобы работать с наименее поврежденными генами.
-
2. Извлечение максимального количества молекул ДНК.
-
3. Выделение наименьшего количества сопутствующих веществ, ингибирующих амплификацию (в частности гуминовых кислот), для облегчения дальнейших процедур очистки.
В большинстве случаев концентрацию ДНК в растворе определяют с помощью спектрофотомерии . В нашем случае это невозможно , поскольку помимо ДНК дополнительно была выделена масса других органических и неорганических соединений (в большинстве представленных гуминовыми кислотами). Помимо прочего для выделения ДНК использовался фенол, от которого трудно отмыть пробу полностью, не потеряв значительного количества ДНК. Фенол даже в следовых количествах интенсивно поглощает свет в той же области спектра, что и нуклеиновые кислоты. Поэтому в нашем случае количество выделенной ДНК с помощью разных методов сопоставили между собой по интенсивности свечения дорожек в УФ свете окрашенного бромистым этидием геля агарозы. Сравнение проводили с помощью прибора Gel Doc Imager System производства компании Bio-Rad (США).
Размер выделенных молекул был оценен с помощью горизонтального электрофореза в геле агарозы.
Параметры проведения электрофореза
Для проведения электрофореза были использованы комплекты для горизонтального электрофореза производства компании Bio-Rad (США).
При сопоставлении количества выделенной ДНК использовался 1%-й агарозный гель, на дорожку наносилось по 1 мкл экстракта, смешанного с 2 мкл красителя (5% SDS, 25% глицерин, 0.025% бромфеноловый синий). Электрофорез проводился 40 минут при напряжении электрического тока 80 Вольт. Силу тока прибор выставлял автоматически, согласно заложенной производителем программе.
При определении размера выделенных молекул с целью увеличить расстояние между молекулами разного размера использовался 0.3%-й агарозный гель, нанесенный на основание из 1%-го агарозного геля (для стабилизации формы). Прочие параметры – как в первом случае.
В качестве маркера был использован Hind III – гидролизат ДНК фага лямбда производства компании «СибЭнзайм».
Сравнение по коэкстракции сопутствующих веществ
При выборе метода извлечения ДНК из почвы и прочих объектов исследований важным критерием является коэкстракция побочных продуктов, способных ингибировать амплификацию, так как экстракт, выделенный с наименьшим количеством сопутствующих веществ, проще очистить для дальнейших исследований.
Для сравнения методов на предмет коэкстракции сопутствующих продуктов был применен метод спектрофотометрии, в диапазоне длин волн от 200 до 600 нм.
В кювету (3 мл) налили 2 мл деионизированной воды и добавили 10 мкл экстракта, выделенного из почвы. Замеры производились в сравнении с кюветой, содержащей чистую деионизированную воду. Таким образом были поочередно произведены замеры всех 6 экстрактов, после чего графики были наложены друг на друга с помощью специальной программы, поставляемой производителем с прибором.
Результаты и обсуждения
Сравнение по объему экстрагирования ДНК из почвенной навески
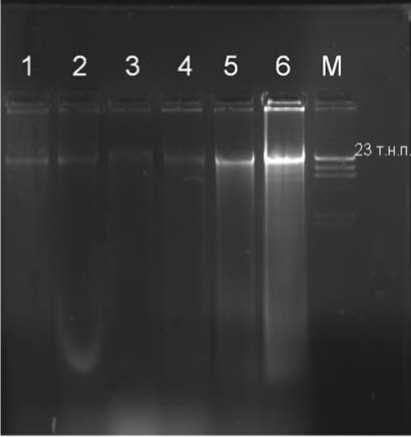
Рис. 1. Электрофорез экстрактов почвы в 1%-м агарозном геле. 1. Метод № 1. 2.Метод № 2. 3.Метод № 3. 4.Метод № 4. 5.Метод № 5. 6.Метод № 6. М. Маркер – Hind III – гидролизат ДНК фага лямбда (λ)
Как видно на рис. 1, наиболее яркое свечение наблюдается на дорожке под номером 6, куда наносили 1 мкл экстракта ДНК, полученного методом измельчения в жидком азоте. По всей видимости, интенсивное растирание при низких температурах наиболее полно разрушило клетки в исследуемом образце. Чуть менее значительное свечение наблюдается при экстрагировании ДНК с помощью TENС буфера (дорожка №5). Причиной, вероятно, является более высокая концентрация протеиназы К, чем в первом методе, а также комплексное использование термошока и химического разрушения клеток с помощью SDS. Прочие методы показали примерно равный массовый выход ДНК из навески 0.5 г.
Сравнение по размеру молекул ДНК
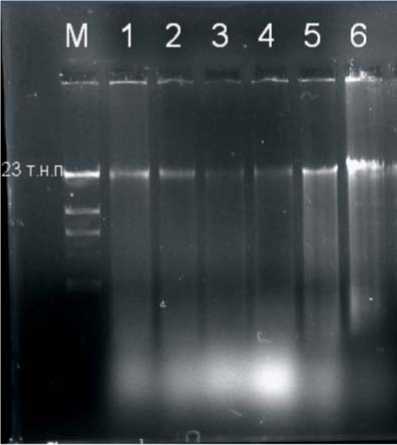
Рис.2. Электрофорез экстрактов почвы на 0.3%-м агарозном геле.
1.Метод № 1. 2.Метод № 2. 3.Метод № 3. 4.Метод № 4. 5.Метод № 5.
6.Метод № 7. М. Маркер – Hind III – гидролизат ДНК фага лямбда (λ)
Как показал электрофорез, в 0,3%-м геле агарозы (рис. 2), существенной разницы в размере экстрагированных молекул ДНК не выявлено. Все пробы показали наличие фрагментов размером около 23 тысяч пар нуклеотидов и некоторого количества высокомолекулярной ДНК, незначительно вошедшей в гель.
Сравнение по экстракции сопутствующих веществ
Как видно из спектров поглощения света в диапазоне от 200 до 600 нм (рис. 3), наибольшую оптическую плотность, а следовательно, наибольшее количество коэкстрагированных веществ имеет экстракт ДНК, выделенный с помощью метода заморозки – оттаивания, дополненного инкубированием с SDS (метод № 4). Следующий (в порядке уменьшения оптической плотности) — метод заморозки и оттаивания без инкубирования с SDS (метод № 3). Вероятно, резкая смена температур ведет к интенсивному разрушению комплексов, образованных органическими соединениями и почвенными частицами, а SDS, являясь сильным ПАВ , усиливает этот эффект. Относительно средние объемы коэкстракции показали методы CTAB (метод № 2) и с использованием TENC буфера (метод № 5).
Наиболее низкие значения коэкстракции показали методы с использованием протеиназы К (метод № 1) и метод измельчения в жидком азоте (метод № 6). В случае с методом выделения протеиназой К относительно низкая коэкстракция объясняется, вероятно, отсутствием стадии резкой смены температур; в случае с растиранием в жидком азоте (самый низкий показатель коэкстракции) можем предположить, что водный раствор фенола лучше осаждает примеси органических веществ из почвы, чем смесь фенол/холоформ/изоамилового спирта.
После сопоставления методов между собой была проведена повторная экстракция почвенной ДНК методами измельчения в жидком азоте (метод № 6) и методом с использованием TENC буфера (метод № 5) в трех повторах для сопоставления между собой в целях установить статистическую достоверность эффективности выделения ДНК (рис. 4).
Кривая спектрального сканирования
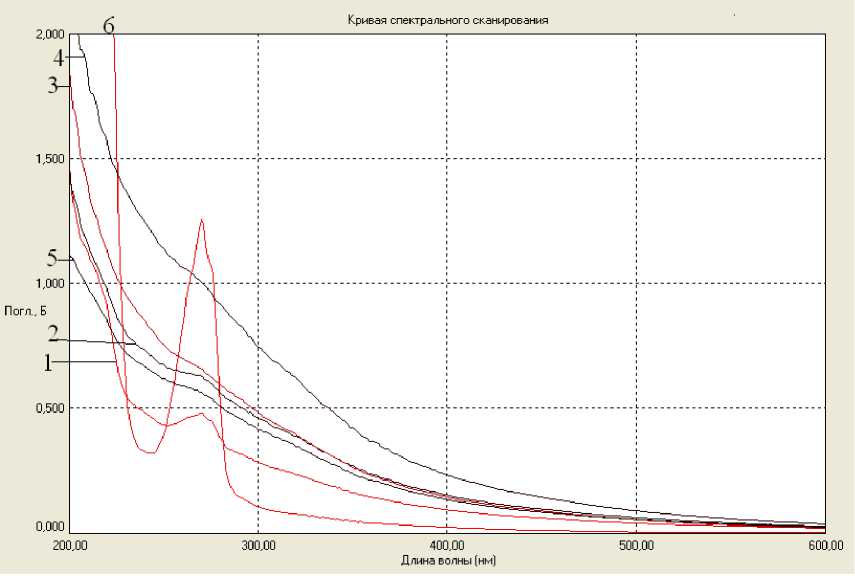
Рис.3. Сравнительные кривые спектрального сканирования экстрактов почвы.
1.Метод № 1. 2.Метод № 2. 3.Метод № 3. 4.Метод № 4. 5.Метод № 5. 6.Метод № 6
Как показало повторное исследование, в трех случаях из трех объем выделенной ДНК с размером фрагментов около 23 тысяч нуклеотидных пар, был существенно выше при использовании метода №6, чем в случае метода с использованием TENC буфера (метод №5). Однако ДНК большего размера в случае метода №6 была выявлена только на дорожке 2, а при выделении с помощью метода №5 ДНК размером существенно большим, чем 23 тысячи пар нуклеотидов, наблюдаются во всех трех случаях (дорожки 4,5 и 6).
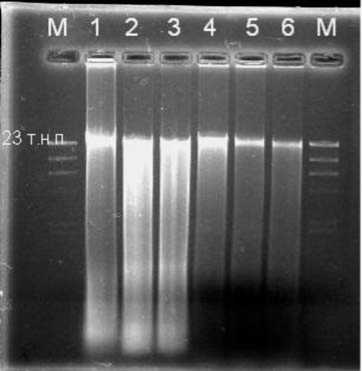
Рис. 4. Сравнение результатов выделения ДНК почвенных организмов методом измельчения в жидком азоте (метод № 5) и методом с использованием TENC буфера (метод № 6)
1,2,3 – экстракт ДНК, выделенной измельчением в жидком азоте; 4,5,6 – экстракт ДНК, выделенной с использованием TENC буфера; М. Маркер – Hind III – гидролизат ДНК фага лямбда (λ)
Выводы
Согласно полученным данным, наиболее значительное количество ДНК выделяется методом измельчения в жидком азоте, он же является и наиболее чистым методом относительно других, экстрагируя наименьшее количество сопутствующих веществ, что в дальнейшем упростит очистку экстракта для последующей амплификации. По всей видимости, тщательное растирание глубокозамороженной почвы позволяет разрушить максимальное количество содержащихся в ней клеток. Однако, как показало последующее сравнение, данный метод не всегда способен выделить фрагменты размером более 23 тысяч нуклеотидных пар; разрушение ДНК, очевидно, происходит ввиду грубых механических воздействий, что в некоторых случаях является недопустимым.
Метод № 6 показал средние значения с точки зрения чистоты от сопутствующих веществ и оказался вторым с точки зрения объема выделенной ДНК. Однако этот метод продемонстрировал стабильный выход ДНК с большим размером фрагментов и рекомендуется для выделения нуклеотидных цепочек значительного размера.
Метод № 1 (с использованием протеиназы К) – единственный, в котором не используется жидкий азот, - показал приемлемый выход ДНК при экстракции из почвенных образцов. В то же время является относительно чистым методом и может быть применен при отсутствии возможности использования жидкого азота в работе.
Список литературы Сравнение методов выделения метагеномной ДНК из образцов почвы
- Jia Xia, Han Shi-jie, Zhao Yong-hua, Zhou Yu-mei. Comparisons of extraction and purification methods of soil microorganism DNA from rhizosphere soil//Journal of Forestry Research 2006 -№ 17(1). -P. 31-34.
- Roh, C. H., Villatte, F., Kim, B. G., and Schmid, R. D.//Electrophoresis. -2005. -V. 26. -P. 3055-3061.
- Tatiana Volossiouk, E. Jane Robb, and Ross N. Nazar. Direct DNA Extraction for PCR-Mediated Assays of Soil Organisms//Applied and environmental microbiology. -1995. -P. 3972 -3976.


