Сравнительный анализ длинных некодирующих РНК NEAT1, MALAT1, GAS5, ROR, HOTAIR в крови больных раком простаты до и после лучевой терапии
Автор: Шуленина Л.В., Михайлов В.Ф., Незнанова М.В., Салеева Д.В., Засухина Г.Д.
Журнал: Саратовский научно-медицинский журнал @ssmj
Рубрика: Научные школы и памятные даты
Статья в выпуске: 4 т.15, 2019 года.
Бесплатный доступ
Цель: исследовать содержание в периферической крови пациентов с раком простаты длинных некодирующих РНК, отрицательно регулирующих активность онкосупрессора Р53. Материал и методы. Объектом исследования явилась венозная кровь пациентов с диагнозом «рак простаты» (от Т1N0M0 до T3N0M0), собранная до и после проведения полного курса лучевой терапии. С помощью метода ПЦР в реальном времени, совмещенной с обратной транскрипцией и специфическими праймерами, определялось содержание длинных некодирующих РНК NEAT1, MALAT1, GAS5, RoR, HOTAIR в крови онкобольных. Данные представлены как медиана и квартили относительно медианы показателей контрольной группы здоровых доноров, условно принятой за 1. Статистическая обработка результатов проводилась с использованием непараметрического критерия Манна - Уитни. Результаты. Обнаружено увеличение содержания NEAT1, MALAT1, GAS5, HOTAIR в крови у больных раком простаты по сравнению с группой «Доноры». Выявлено, что проведение курса лучевой терапии не влияло на величину исследуемых показателей. Заключение. Анализ полученных данных и литературного материала позволяет полагать, что в крови у больных раком простаты уровни NEAT1, MALAT1 и HOTAIR отражают их количество в клетках опухоли. NEAT1, MALAT1, GAS5 и HOTAIR могут быть потенциальными маркерами для диагностики и лечения рака.
Днрнк, кровь, рак простаты
Короткий адрес: https://sciup.org/149135473
IDR: 149135473 | УДК: 577.218
Текст научной статьи Сравнительный анализ длинных некодирующих РНК NEAT1, MALAT1, GAS5, ROR, HOTAIR в крови больных раком простаты до и после лучевой терапии
отвечающих за сомообновление и дифференцировку эмбриональных стволовых клеток. HOTAIR, связываясь своим 5’ — концом с PRC2 и 3’ — концом с LSD1 и образуя мостик, координирует их работу и направляет к генам-мишеням для последующего метилирования гистона H3 по остатку лизина в 27-м положении и деметилирования гистона H3 по остатку лизина в 4-м положении соответственно. Показано, что днРНК MALAT1 косвенно контролирует альтернативный сплайсинг, изменяя распределение регуляторов сплайсинга в ядерных спеклах, может функционировать как молекулярный каркас для взаимодействия между неметилированным Pc2, фактором транскрипции E2F, гистонами и комплексом транскрипционных коактиваторов [7], а кроме этого, может снимать ингибирующее действие микроРНК на мРНК, вступая с микроРНК в реципрокные взаимодействия. Гиперэкспрессия перечисленных днРНК приводит к ингибированию активности онкосупрессора Р53, инактивация которого является важной для сохранения жизнедеятельности раковых клеток.
В настоящее время интенсивно исследуются длинные некодирующие РНК в образцах тканей больных РПЖ, играющие существенную роль в прогрессии и метастазировании рака, с целью выявления новых терапевтических мишеней, а также изучается содержание днРНК в биологических жидкостях больного (крови, слюне и моче). Однако пока остается непонятным, на каком аналитическом и клиническом уровне находятся биомаркеры днРНК при РПЖ. В связи с этим целью нашей работы было исследование методом ПЦР в реальном времени содержания в периферической крови пациентов с РПЖ днРНК, отрицательно регулирующих активность онкосупрессора Р53.
Цель: исследовать содержание в периферической крови пациентов с раком простаты длинных некодирующих РНК, отрицательно регулирующих активность онкосупрессора Р53.
Материал и методы. Объектом исследования явились образцы венозной крови 32 мужчин, имею- щих в анамнезе РПЖ с классификацией от T1N0M0 до T3N0M0, которым была оказана дистанционная γ-терапия (источник радионуклид 60Со, аппарат «РОКУС»).
В группы сравнения включены образцы крови пациентов с диагнозом РПЖ до и после курса лучевой терапии (ЛТ), а также крови здоровых доноров (группа «Доноры», согласно данным медицинского обследования рассматриваемая нами как контрольная), в количестве 19 человек.
Исследование выполнено в соответствии со стандартами надлежащей клинической практики (Good Clinical Practice) и принципами Хельсинкской Декларации.
Выделение общей РНК, включая длинные некодирующие РНК, проводили с использованием набора Trizol RNA Prep 100 (ООО «Лаборатория Изоген», Россия) по протоколу фирмы-производителя. Полученный осадок РНК был растворен в 50 мкл Экстрагена Е и заморожен при –70°С до последующего использования. Для выражения количества выделенной РНК использованы единицы массовой концентрации.
Обратную транскрипцию проводили на матрице РНК в объеме 20 мкл с использованием набора GenePak RT Corе (ООО «Лаборатория Изоген», Россия) по протоколу фирмы-производителя со cлучайными гекса- и нанонуклеотидными праймерами, позволяющими синтезировать большие количества одноцепочечной кДНК, в том числе и на матрице неполиаденилированной РНК. Полученную кДНК хранили при –70°С до последующего использования.
ПЦР в реальном времени кДНК проводили на приборе DT prime 5М3 (НПО «ДНК-Технология», Россия) в присутствии интеркалирующего красителя SYBR Green I (Thermo Scientific Maxima sYbR Green/ROX qPCR Master Mix (2X) (ООО «ДНК-синтез», Россия) или TaqMan-зонда (ООО «ДНК-синтез») со специфическим праймерами для днРНК NEAT1, MALAT1,
GAS5, RoR, HOTAIR (ООО «ДНК-Синтез»), олиго-нуклетидная последовательность которых была заимствована из оригинальных работ и представлена в таблице. Данные праймеры имеют все необходимые характеристики для эффективной ПЦР, в том числе и длину 20–25 пар нуклеотидов, за исключением праймеров к днРНК NEAT1 (длина прямого F- и обратного R-праймеров составляет 27 пар нуклеотидов). Эта особенность в дальнейшем учтена при отработке концентрационных и температурных условий амплификации днРНК NEAT1 (таблица).
Отсутствие ингибирования ПЦР продуктами выделения РНК, а также выбор вносимого в пробирку объема матрицы для ПЦР контролировались методом последовательных разведений матриц (нераз-веденная, разведенная в 5 раз, в 25 раз, в 125 раз, в 625 раз).
Для оценки контаминации исследуемых проб, а также наличия димеров праймеров с помощью программного обеспечения DT prime 5М3 построены кривые плавления кДНК с инкорпорированным красителем SYBR Green I после окончания ПЦР.
Полученные данные ПЦР анализировали с использованием метода определения порогового цикла амплификации ΔΔCt, где Ct — пороговый цикл флуоресценции. В качестве нормировочного гена использован ген ghr (ген рецептора гормона роста человека для нормировки по ДНК). Каждая ПЦР проводилась не менее двух раз.
Статистическую обработку результатов проводили с использованием программы Statistica 7.0 методом непараметрической статистики (критерий Манна-Уитни), где количественные показатели в исследуемых группах выражались в виде медианы и квартилей (25%-75%). Значения медианы в контрольной группе «Доноры» условно приняты за 1.
Результаты. Методом ПЦР в реальном времени в крови больных РПЖ до и после лучевой терапии (ЛТ) обнаружены изменения содержания днРНК MALAT1, GAS5, HOTAIR, RоR и NEAT1 (рисунок).
Олигонуклеотидная последовательность праймеров и зондов, а также условия ПЦР в реальном времени
|
Название |
Праймеры (прямой F и обратный R) и зонды для ПЦР и их конечная концентрация (нМ) |
Условия ПЦР в реальном времени |
|
кДНК NEAT1 |
F-5/-CTTCCTCCCTTTAACTTATCCATTCAC-3/(100) R-5/-CTCTTCCTCCACCATTACCAACAATAC-3/(100) |
Активация UDG (0,02 ед/мкл): 50°С/2мин; предварительная денатурация: 95°С/10 мин; далее 40 циклов: 95°С/15 с, 60°С/20с |
|
кДНК MALAT1 |
F-5/-GCAGTATTGCATGTTAGG-3/(500) R-5/-CCTCTGAGTGAAGTGTACTATC-3/(500) |
Предварительная денатурация: 95°С/10 мин; далее 45 циклов: 94°С/10 с, 65°С/20 с (с температурным инкрементом до 55°С) и 72°С/30с |
|
кДНК RoR |
F-5/-CTCAGTGGGGAAGACTCCAG-3/(200) R-5/-AGGAAGCCTGAGAGTTGGC-3/(200) |
Предварительная денатурация: 94°С/10 мин; далее 45 циклов: 95°С/15 с, 60°С/15 с |
|
кДНК GAS5 |
F-5/-TGAAGTCCTAAAGAGCAAGCC-3/(200) R-5/-ACCAGGAGCAGAACCATTAAG-3/(200) |
Предварительная денатурация: 94°С/10 мин; далее 45 циклов: 95°С/15 с, 60°С/15 с |
|
кДНК HOTAIR |
F-5/-GGCGGATGCAAGTTAATAAAAC-3/(500) R-5/-TACGCCTGAGTGTTCACGGAG-3/(500) |
Предварительная денатурация: 95°С/10 мин; далее 45 циклов: 94°С/10 с, 65°С/20 с (с температурным инкрементом до 55°С) и 72°С/30с |
|
ДНК ghr |
F-5/-CATTCCCATCATTGAGTGTGGAGTGAG-3/(350) R-5/-CTGGGGATCAGGTGTTTATGGACCA-3/(350) Зонд TaqMan- (BHQ1) — 5’ — CCTTCTGCCTGGCTTGCTTTCCC-3’ — (FAM) (100) |
Предварительная денатурация: 80°С/30 с, 94°С/1 мин; далее 50 циклов: 94°С/10 с, 64°С/20 с |
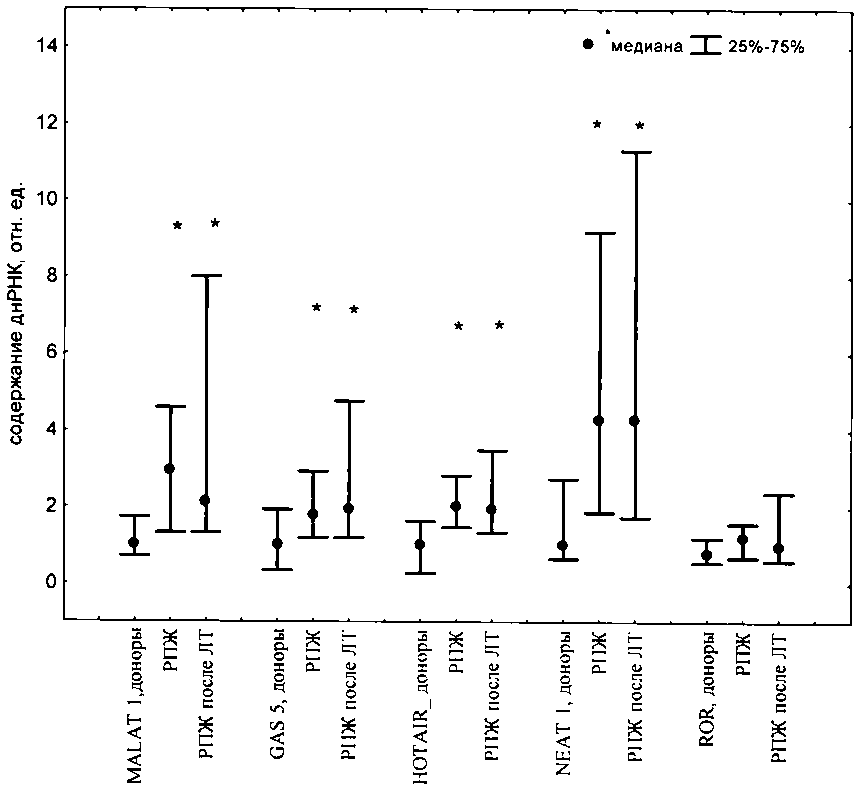
Содержание днРНК NEAT1, MALAT1, GAS5, RoR, HOTAIR в периферической крови пациентов с РПЖ до и после проведения курса лучевой терапии:
* — отличия статистически значимы по сравнению с группой «Доноры» (р<0,05)
У пациентов с РПЖ наблюдается статистически значимое увеличение содержания днРНК MALAT1, GAS5, HOTAIR и NEAT1 по сравнению с контрольной группой «Доноры», условно принятой за 1 (р<0.05). Медиана содержания днРНК MALAT1 до ЛТ cоставляет 2,93 (1,32–4,59) отн. ед., GAS5–1,8 (1,19– 2,83) отн. ед., HOTAIR — 2,0 (1,46–2,83) отн. ед., NEAT1–4,29 (1,87–9,19) отн. ед. После проведения курса ЛТ значение этих показателей остается на более высоком уровне, чем в группе «Доноры», однако статистически значимых отличий как между группой «РПЖ» и «Доноры», так и между «РПЖ» и «РПЖ после ЛТ» не наблюдается.
Медиана содержания днРНК RоR в группе «РПЖ» и «РПЖ после ЛТ» не отличается от группы «Доноры» и составляет 1,19 (0,68–1,57) и 0,97 (0,59–2,38) отн. ед. соответственно.
Обсуждение. Для успешного лечения онкологических заболеваний важны профилактические мероприятия и как можно более ранняя диагностика опухолей. В связи с этим поиск биомаркеров рака в биологических жидкостях организма привлекателен, поскольку он является минимально инвазивным, а кроме того, отражает системный ответ организма на развитие опухоли. В последние годы пристальное внимание уделяется РНК-соединениям. Этот интерес обусловлен открытием регуляторных РНК, которые модифицируют экспрессию белоккоди-рующих генов, как на транскрипционном, так и посттранскрипционном уровне. Интенсивно изучались микроРНК, которые, включаясь в РНК-индуцируемый комплекс, взаимодействуют с микроРНК-респонсив-ным элементом, находящимся на мРНК в области З/UTR-peruoHa, предотвращают рибосомальный синтез и таким образом модулируют экспрессию генов-мишеней. Серьезным препятствием для применения этих соединений в медицинской практике явилось, как показывает биоинформационный анализ, наличие многочисленных потенциальных генов-мишеней практически для большинства микроРНК. При этом выбор активных для каждой микроРНК генов-мишеней зависел от типа клеток, их функционального состояния и т. д., что затрудняло оценку очагов поражения в организме по изучению содержания этих веществ в биологических жидкостях.
Установлено, что изучение днРНК имеет большие перспективы их использования в качестве маркеров онкологических заболеваний. Эти РНК-соединения привлекли значительное внимание исследователей из-за их роли в генетической регуляции (включая эпигенетическую, транскрипционную и посттранскрипционную регуляцию) участия в онкогенезе [8].
Обнаруживаемые в периферической крови днРНК могут находиться как в циркулирующих клетках крови, так и в экзосомах — наноразмерных мембранных везикулах, секретируемых различными клетками, включая опухолевые. Таким образом, изменения содержания днРНК в крови могут быть не только связаны с изменением их экспрессии в клетках крови, но и иметь экзосомальное происхождение. В настоящее время формируется база данных по содержанию РНК-продуктов в этих экзосомах [9]. В ней собраны сведения по 15501 днРНК. Из обследованных нами днРНК в базу данных входит лишь днРНК RоR. По данным базы установлено, что содержание днРНК RоR в экзосомах здоровых доноров колеблется от 0 до 8,4 отн. ед., а у больных раком поджелудочной железы от 0 до 19,3 отн. ед., причем частота обнаружения этой днРНК составляет 0,47. Таким образом, примерно у каждого второго обследованного в крови не обнаруживается экзосом, содержащих днРНК RоR. В нашем эксперименте данные по содержанию
RоR в крови у больных РПЖ не отличались от величин, наблюдаемых в группе «Доноры». Лучевая терапия также не оказывала эффект на величины этой днРНК.
Известно, что одним из мишеней транскрипционного фактора Р53 является ген для днРНК RоR, однако увеличение содержания RoR, вызываемое действием Р53 на промотор гена, синтезирующего эту днРНК, ингибирует экспрессию Р53. Таким образом, Р53 и днРНК RоR образуют между собой отрицательную обратную петлю, подобную петли между белком Mdm2 и P53, днРНК NEAT1 и P53, днРНК MALAT1 и P53, когда увеличение содержания транскрипционного фактора Р53 приводит к усилению экспрессии его ингибиторов, что снижает активность Р53. Представленные нами данные показывают, что изменений функционирования отрицательной обратной петли между Р53 и днРНК RоR в крови у пациентов с РПЖ не выявлено. В то же время содержание днРНК MALAT1 и днРНК NEAT1 увеличивается. Высокий уровень содержания онкогенных днРНК MALAT1 и NEAT1 может быть связан как с усилением их экспрессии в клетках крови, так и с выбросом опухолевыми клетками экзосом, содержащих эти днРНК, поскольку установлено, что в трансформированных клетках простаты наблюдается высокий уровень этих регуляторных РНК. Так, у 82,5% пациентов с РПЖ обнаруживается гиперэкспрессия днРНК MALAT1 в опухолевой ткани [10]. Такой же высокий уровень этой днРНК отмечен в плазе крови и моче пациентов с РПЖ. Курс ЛТ также не снижал уровень днРНК MALAT1 и NEAT1 в периферической крови.
В настоящее время показано участие днРНК GAS5 в регуляции программ пролиферации и остановки клеточного цикла. Во многих злокачественных опухолях наблюдается снижение уровня днРНК GAS5, который проявляет функции онкосупрессора [11]. Для раскрытия механизмов выявленного нами увеличения содержания днРНК GAS5 в периферической крови пациентов с РПЖ необходимо проведение дальнейших исследований. Наконец, обнаруженный нами высокий уровень содержания днРНК HOTAIR в крови у пациентов с РПЖ был ожидаем, поскольку высокий уровень этого показателя в сыворотке крови больных плоскоклеточным раком гортани уже предлагался другими авторами в качестве прогностического маркера [12]. Установлено, что во многих опухолях наблюдается увеличение экспрессии днРНК HOTAIR. Показано, что возрастание содержания этой днРНК приводит к ингибированию Р53, а высокая активность Р53 снижает уровень HOTAIR [13].
В работах, посвященных исследованиям экзо-сом, сообщается, что выбрасываемые в биологические жидкости раковыми клетками экзосомы содержат днРНК. В связи с этим мы оценили содержание днРНК, ингибирующих активность Р53 в периферической крови пациентов с РПЖ. Установлено увеличение днРНК, подавляющих функциональную активность Р53, в периферической крови пациентов с РПЖ, что свидетельствует о необходимости дальнейших исследований в этом направлении.
С нашей точки зрения, необходимо выяснить: каков точный источник появления в периферической крови высоких значений содержания днРНК у пациентов с РПЖ; каков вклад экзосом, выделяемых опу- холями в этот эффект; какова роль системы крови, отвечающей за иммунный ответ?
Заключение. Выявлено увеличение содержания днРНК NEAT1, MALAT1 и HOTAIR в периферической крови пациентов с РПЖ. Анализ полученных данных и литературного материала позволяет заключить следующее:
-
1. У больных РПЖ уровень днРНК NEAT1, MALAT1 и HOTAIR в периферической крови отражает их количество в клетках опухоли.
-
2. В клетках опухоли высокое содержание этих днРНК может вызывать снижение уровня Р53 с помощью петли обратной связи между днРНК NEAT1, MALAT1 и HOTAIR и онкосупрессором Р53, что приводит к защите онкотрансформированных клеток с поврежденным геномом от гибели и сохранению высокого пролиферативного потенциала.
-
3. Длинные некодирующие РНК NEAT1, MALAT1, GAS5 и HOTAIR могут быть потенциальными мишенями для диагностики и лечения рака.
Список литературы Сравнительный анализ длинных некодирующих РНК NEAT1, MALAT1, GAS5, ROR, HOTAIR в крови больных раком простаты до и после лучевой терапии
- Cancer Stat Facts: Prostate Cancer. URL: https:// seer.cancer.gov/statfacts/html/prost. html, 2017.
- Volders P-J, Helsens K, Wang X, et al. LNCipedia: a database for annotated human lncRNA transcript sequences and structures. Nucleic Acids Res 2013; 41: D246-51.
- Schmitt AM, Chang HY. Long noncoding RNAs in cancer pathways. Canc Cell 2016; 29: 452-63.
- Iyer MK, Niknafs YS, Malik R, et al. The landscape of long noncoding RNAs in the human transcriptome. Nat. Genet 2015; 47: 199-208.
- Mercer TR, Mattick JS. Structure and function of long noncoding RNAs in epigenetic regulation. Nat Struct Mol Biol 2013; 20: 300-7.
- Jalali S, Bhartiya D, Lalwani MK, et al. Systematic transcriptome wide analysis of lncRNA-miRNA interactions. PLoS One 2013; 8: e53823.
- Yang L, Lin C, Liu W, et al. NcRNA- and Pc2 methylation-dependent gene relocation between nuclear structures mediates gene activation programs. Cell 2011; 147 (4): 773-88.
- Oliveira JC de, Oliveira LC, Mathias C, et al. LncRNAs in Cancer: another layer of complexity. J Gene Med 2018; 14: e3065.
- Shengli Li, Yuchen Li, Bing Chen, et al. ExoRBase: a database of circRNA, lncRNA and mRNA in human blood exosomes. Nucleic Acids Research 2018; 46: D106-12.
- Ren S, Peng Z, Mao J-H, et al. RNA-seq analysis of prostate cancer in the Chinese population identifies recurrent gene fusions, cancer-associated long noncoding RNAs and aberrant alternative splicings. Cell Res 2012; 22: 806-21.
- Liu Y, Zhao J, Zhang W, Gan J, et al. lncRNA GAS5 enhances G1 cell cycle arrest via binding to YBX1 to regulate p21 expression in stomach cancer. Sci Rep 2015; 5: 10159.
- Wang J, Zhou Y, Lu J, et al. Combined detection of serum exosomal miR-21 and HOTAIR as diagnostic and prognostic biomarkers for laryngeal squamous cell carcinoma. Med Oncol 2014; 31 (9):148.
- Zhai N, Xia Y, Yin R. et al. A negative regulation loop of long noncoding RNA HOTAIR and p53 in non-small-cell lung cancer. Onco Targets Ther 2016; 16; 9: 5713-20.


