Сравнительный анализ филогений симбиотических генов клубеньковых бактерий с использованием метадеревьев
Автор: Карасев Е.С., Чижевская Е.П., Симаров Б.В., Проворов Н.А., Андронов Е.Е.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Симбиогенетика
Статья в выпуске: 5 т.52, 2017 года.
Бесплатный доступ
Для изучения закономерностей эволюции различных групп «симбиотических» генов ( nod -генов, контролирующих образование клубеньков, nif / fix -генов, контролирующих симбиотическую азотфиксацию) был применен модифицированный нами метод филогенетического анализа - построение метадеревьев. Суть метода заключается в попарном сравнении топологий дендрограмм и построении комбинированных дендрограмм (метадеревьев), на которых относительное положение двух деревьев является мерой конгруэнтности филогений соответствующих генов. Для реализации данного метода были выбраны 18 симбиотических генов ( nodABCDIJN, nifABDEHKN, fixABC, fdxB ), гомологи которых присутствуют у каждого из исследуемых организмов (9 штаммов, относящихся к родам Bradyrhizobium, Mesorhizobium, Rhizobium, Sinorhizobium и Neorhizobium ), а также ген 16S рРНК - традиционный хромосомный таксономический маркер. Для каждого из этих генов были построены и сопоставлены филогенетические деревья, затем были рассчитаны коэффициенты попарного сходства их топологий. По полученным данным было построено «метадерево», в пределах которого были выявлены два статистически различающихся кластера генов. В кластер 1 вошли преимущественно nif- и fix- гены, а в кластер 2 - преимущественно nod- гены, что согласуется с данными о раздельной локализации этих групп генов в геномах ризобий. Исключением было расположение генов nifB и fixC в кластере 2 вместе с геном nodA, а также локализация гена nodI в кластере 1 вместе с геном nifD. При анализе структуры выявленных кластеров не было обнаружено строгой зависимости между относительным положением изучаемых генов и особенностями их локализации в геномах клубеньковых бактерий. Важно отметить, что различия между кластерами 1 и 2 выражены не менее четко, чем различия между группами nod - и nif / fix -генов. Очевидно, что кластеры 1 и 2 на построенном нами метадереве отражают в первую очередь различие механизмов эволюции процессов образования клубеньков и симбиотической азотфиксации, связанное с независимым происхождением соответствующих групп генов, а возможно, и с их раздельным горизонтальным переносом между разными группами ризобий. Дальнейшее изучение эволюции симбиотических генов клубеньковых бактерий требует усовершенствования использованной методики филогенетического анализа, включая раздельный анализ метадеревьев для ризобий, представляющих разные этапы эволюции симбиоза.
Филогенетический анализ, метадеревья, клубеньковые бактерии, симбиотические гены
Короткий адрес: https://sciup.org/142214089
IDR: 142214089 | УДК: 631.461.52:575.1:577.257.065 | DOI: 10.15389/agrobiology.2017.5.995rus
Текст научной статьи Сравнительный анализ филогений симбиотических генов клубеньковых бактерий с использованием метадеревьев
Работа выполнена в рамках проекта РНФ ¹ 14-26-00094П и проводилась с использованием оборудования ЦКП «Геномные технологии, протеомика и клеточная биология» ФГБНУ ВНИИСХМ.
сферного азота, — nif -гены (контролируют синтез и регуляцию активности фермента нитрогеназы) и fix -гены (разнородная группа генов, вовлеченных в работу нитрогеназного комплекса, в первую очередь в его снабжение электронами и энергией) (3).
У большинства клубеньковых бактерий nod- и nif / fix -гены собраны в группы, расположенные в геноме в виде отдельных кластеров, структура которых варьирует у разных видов. Например, у клубеньковых бактерий люцерны ( Sinorhizobium meliloti и S . medicae ), гороха ( Rhizobium leguminosarum bv. viciae ), клевера ( R. leguminosarum bv. trifolii ), козлятника ( Neorhizobium galegae ) и сои ( Bradyrhizobium japonicum ) основные nod -гены тесно сцеплены, при этом гены nodABC организованы в один оперон (рис. 1). У многих ризобий структурные гены нитрогеназы nifHDK представляют собой один оперон и тесно сцеплены с генами nifEN . Такая организация генов азот-фиксации характерна для представителей Sinorhizobium , R. leguminosarum , N. galegae и Mesorhizobium loti . Однако у B. japonicum дополнительная копия гена nifH расположена вне nifHDK -оперона, а ген nifA — за пределами nif / fix -кластера совместно с nod -генами.
Рис. 1. Расположение симбиотических генов у различных видов клубеньковых бактерий: • — nod -гены, D — nif -гены, D — fix -гены, 1 — fdx -гены, ^ — несимбиотические гены; ген nodN в геноме R . elti (отмечен звездочкой) имеет хромосомную локализацию. Схемы построены по данным секвенирования геномов изучаемых ризобиальных штаммов (4-11).
У эволюционно продвинутых видов клубеньковых бактерий (например, R. leguminosarum ) симбиотические гены расположены на высокомолекулярных плазмидах (12). В то же время их хромосомная локализация характерна для анцестральной формы — B. japonicum, а также для M. loti (13, 14). Есть ризобии, у которых на хромосоме могут располагаться только некоторые симбиотические гены. Это свойственно, например, для R. etli : большинство симбиотических генов находятся на плазмиде, а ген nodN — на хромосоме (15) (см. рис. 1).
Долгое время молекулярные механизмы эволюции бобово-ризоби-ального симбиоза, насчитывающей 60-70 млн лет (16), оставались неясными. Лишь благодаря методам высокопроизводительного секвенирования, когда началось быстрое накопление данных о составе и строении геномов различных видов бактерий, пангеномному и метагеномному анализу удалось реконструировать тот длинный путь, который прошли микро- и макросимбионты от рекрутирования в бактериальные клетки генов азотфиксации ( nif- генов) до развития современных высокоспециализированных бобово-996
ризобиальных симбиотических систем. Этот процесс, по-видимому, происходил в несколько этапов, на которых рассматриваемые группы генов были приобретены из разных источников: nif -гены и большинство fix -генов — получены анцестральными медленнорастущими ризобиями ( Bradyrhizobium ) от своих свободноживущих предков ( Rhodopseudomonas ) посредством вертикального наследования, тогда как гены синтеза липо-хито-олигосахаридных Nod-факторов, отвечающих за образование клубеньков ( nod -гены), — приобретены от грибов либо от грамположительных бактерий при горизонтальном переносе (17). Возникшие позднее быстрорастущие ризобии ( Rhizobium, Sinorhizobium, Neorhizobium ) получили nod - и nif / fix -гены от анцестральных ризобий при горизонтальном переносе генов (ГПГ), что привело к образованию особых плазмид (p Sym ), содержащих обе группы sym -генов.
Связь филогении sym -генов с механизмами их происхождения и эволюции, а также с локализацией в геномах ризобий исследована недостаточно. Для ее изучения мы использовали новый метод филогенетического анализа с построением «метадеревьев» — комбинированных дендрограмм, на которых относительное положение двух деревьев служит мерой конгруэнтности филогений индивидуальных генов. Ранее этот метод был разработан и использован для анализа альтернативных филогений, полученных при изучении функционально разнородных генов в различных группах эукариот (дрожжи, рыбы), в которых ГПГ ограничен (18).
В нашем исследовании названный метод был применен для филогенетического анализа двух функционально различающихся групп генов ( nod и nif / fix ), которые имеют неодинаковое происхождение и распространялись среди таксономически неродственных групп ризобий посредством как вертикального наследования, так и ГПГ. Выявленные нами особенности топологий филогений, построенных для генов образования клубеньков и симбиотической азотфиксации, позволяют существенно дополнить возникшие ранее представления о направлениях и механизмах эволюции генных систем симбиоза у клубеньковых бактерий.
Целью работы было использование метадеревьев для анализа участия горизонтального переноса в процессе эволюционной сборки кластера симбиотических генов у ризобий.
Методика . Для анализа использовали нуклеотидные последовательности симбиотических генов из полногеномных сиквенсов 9 представителей клубеньковых бактерий: R. leguminosarum bv. viciae 3841 (GenBank GI:115259115) (4), R. leguminosarum bv. trifolii WSM2304 (GI:209537694) (5), S. meliloti 1021 (GI:25168258) (6), S. medicae WSM419 (GI:150031715) (7), B. ja-ponicum USDA110 (GI:47118316) (8), M. loti MAFF303099 (GI:47118328) (9), R. etli CFN42 (GI:89213252) (10), а также N. galegae bv. officinalis HA-MBI1141 (GI:659665307) и N. galegae bv. orientalis HAMBI540 (GI:659657635) (11).
Кластерный анализ выполняли в компьютерной программе MEGA 5.1 . Выравнивание нуклеотидных последовательностей проводили с помощью алгоритма ClustalW; математическая модель при составлении древ для каждого исследуемого гена — p-distance, механизм кластеризации — Neighbor-Joining. Численное выражение сходств топологий филогенетических деревьев рассчитывали с применением статистических подходов (реализованы в программе, представленной на сайте (19). Для построения метадерева и анализа достоверности кластеризации использовали коэффициенты различия, которые вычисляли как разность единицы (100 %, уменьшаемое) и соответствующего коэффициента сходства (вычитаемое).
Результаты. На начальном этапе работы были построены филоге- нетические деревья для каждого из следующих 18 генов: генов вирулентности nodA, nodB, nodC (отвечают за синтез коровой части Nod-фактора) (20-22), nodD (при наличии нескольких копий nodD1 это конститутивно экспрессируемый ген флавоноид-чувствительного активатора транскрипции nod-генов) (23), nodI, nodJ (гены мембранных транспортеров Nod-фактора) (24), nodN (кодирует фермент дегидратазу) (25); генов симбиотической азотфиксации fixA, fixB, fixC (компоненты электронно-транспортной цепи нитрогеназы) (26), nifA (ген транскрипционного регулятора nif-генов), nifB, nifN, nifE (отвечают за синтез Fe-Mo кофактора нитрогеназы), nifD, nifH, nifK (структурные компоненты нитрогеназы) (27), fdxN (ферредоксин). Выбор был обусловлен тем, что гомологи указанных генов присутствуют у всех представителей клубеньковых бактерий, отобранных для анализа. Кроме того, в исследование включили традиционный хромосомный таксономический маркер — ген 16S рРНК в связи с консервативностью его нуклеотидной последовательности и низкой частотой горизонтального переноса.
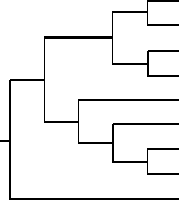
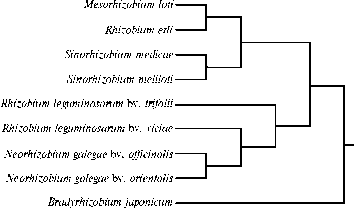
Методология сравнения топологий основана на представлении о том, что горизонтальный перенос генов от одного вида ризобий к другому в ходе эволюции вносит существенные различия в топологии филогений этих генов. В том случае, когда топология деревьев двух генов не различается, предполагают, что они либо не участвуют в горизонтальном переносе совсем, либо переносятся совместно (рис. 2, А).
A
Rhizobium etli
Sinorhizobium medicae
Sinorhizobium meliloti
Rhizobiutn leguminosarum bv. viciae
Neorhizobium galegae bv. officinalis
Neorhizobium galegae bv. orientalis
Mesorhizobium loti
Bradyrhizobium japonicum
Rhizobium leguminosarum bv. trifolii
Б
Mesorhizobium loti
Rhizobium etli
Sinorhizobium medicae
Sinorhizobium meliloti
Rhizobium leguminosarum bv. viciae
Neorhizobium galegae bv. officinalis
Rhizobium leguminosarum bv. trifolii
Bradyrhizobium japonicum
Neorhizobium galegae bv. orientalis
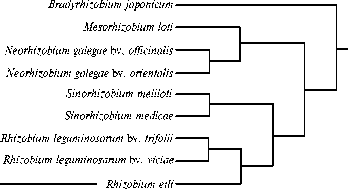
Рис. 2. Сравнения топологии филогенетических деревьев для генов nifH (слева) и nifN (справа) со степенью сходства 100 % (А) и для генов fdxN (слева) и 16S рРНК (справа) со степенью сходства 50,5 % (Б) . Стрелкой отмечено наиболее заметное различие в топологии: перемещение Rhizobium etli из кластера R. leguminosarum на древе гена 16S рРНК в кластер M. loti на древе fdxN .
В то же время несоответствие в топологии деревьев, построенных на основании анализа сиквенсов одного и того же набора штаммов, представляет собой следствие горизонтального переноса симбиотических генов (см. рис. 2, Б). По этим различиям в топологии мы можем предполагать, как происходил ГПГ в ходе эволюции. Например, видно, что в древе по гену 16S рРНК в одном кластере находятся два биовара R. leguminosarum и R. etli . Так как ген 16S рРНК — хромосомный маркер, то допустимо утвер-998
ждать, что кластеризующиеся вместе представители эволюционно близки. В то же время на древе плазмидного маркера fdxN ризобии R. etli группировались в одном кластере с M. loti, хотя R. etli и M. loti относятся к разным семействам — Rhizobiaceae и Phyllobacteriaceae. Этот факт позволяет предположить, что имел место горизонтальный перенос гена fdxN между клубеньковыми бактериями M. loti и R. etli либо при горизонтальном переносе указанные виды получили этот ген из одного источника.
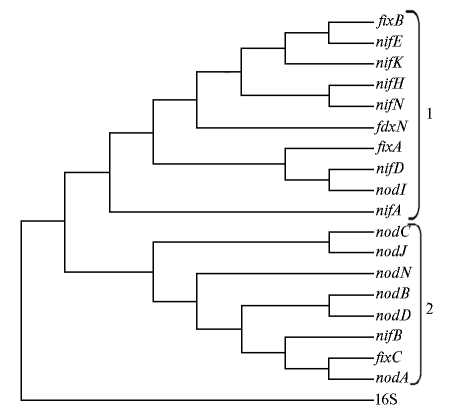
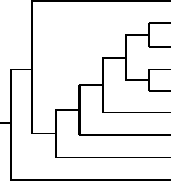
Рис. 3. Основные кластеры метадерева по матрице сходств топологий в филогенетических деревьях, построенных по данным о нуклеотидном полиморфизме симбиотических генов: 1 — nif/fix- кластер, 2 — nod- кластер.
Затем мы сравнили все полученные 19 филогенетических деревьев для выявления различий в их топологиях. Би-оинформатический анализ полученных данных позволил рассчитать коэффициенты попарного сходства топологий для всех деревьев, варьирующие от 1 (полная конгруэнтность двух филогений) до 0 (полное отсутствие конгруэнтности). Затем построенная матрица сходств была преобразована в матрицу различий вычитанием соответствующих коэффициентов сходства из единицы. По данным этой матрицы было построено метадерево (рис. 3).
Видно, что в метадере- ве достоверно выделились два кластера: группа nif/fix-генов оказалась пре- имущественно сосредоточена в кластере 1, nod-генов — в кластере 2, тогда как хромосомный маркер (ген 16S рРНК) не вошел ни в один из кластеров (табл.).
Оценка достоверности кластеризации метадерева в соответствии со средним расстоянием внутри и между кластерами / группами генов с использованием t -критерия Стьюдента
|
Группа сравнения |
TD |
SD |
SE |
Достоверность различий по t -критерию Стьюдента при уровне значимости p |
|
Внутри кластера 1 |
0,197 |
0,094 |
0,013 |
< 0,001 |
|
Внутри кластера 2 |
0,216 |
0,104 |
0,020 |
< 0,01 |
|
Между кластерами |
0,269 |
0,107 |
0,012 |
|
|
Внутри группы nif-fix |
0,194 |
0,084 |
0,011 |
< 0,001 |
|
Внутри группы nod |
0,201 |
0,082 |
0,018 |
< 0,01 |
|
Между группами симбиотических генов |
0,270 |
0,099 |
0,011 |
П р и м е ч а н и е. TD — среднее различие топологий, SD — стандартное отклонение, SE — стандартная ошибка. Проводили сравнение среднего расстояния между топологиями деревьев внутри одного класте-ра/группы, а также между деревьями одного кластера/группы и каждым деревом другого кластера/группы. Структура кластеров приведена на рисунке 3.
Такая кластеризация может указывать на то, что в эволюционной истории группы генов nod и nif/fix не связаны между собой и с хромосомными генами. Она определяется их самостоятельным происхождением и раздельным горизонтальным переносом. Действительно, различие групп nod- и nif/fix-генов выражено не менее четко, чем несходство между кластерами 1 и 2. Однако были выявлены и исключения: ген nodI, который находится в nif/fix кластере 1 и переносится вместе с геном nifD, а также гены nifB и fixC, попадающие в nod-кластер 2 вместе с nodA.
Как отмечалось выше, в геномах многих клубеньковых бактерий симбиотические гены объединены в кластеры. В связи с этим совместный горизонтальный перенос групп nod - и nif / fix генов может быть обусловлен особенностями их кластерного расположения в геноме (см. рис. 1). Тем не менее, объяснить полученную картину только спецификой расположения генов на генетических картах не представляется возможным. Так, в кластере 1 были обнаружены все структурные гены нитрогеназы, причем гены nifH , nifK , nifE и nifN группировались друг с другом. Подобная кластеризация генов соответствует их расположению в геномах, поскольку у всех изучаемых организмов гены nifHDK составляют один оперон, к которому прилегают гены nifE и nifN . Единственное исключение — локализация гена nifN за пределами nif / fix -региона у клубеньковых бактерий люцерны ( Sinorhizobium meliloti ). Вместе с теми же nod -генами в кластере 2 был обнаружен nodN , однако внутри кластера он располагался отдельно. Следует отметить, что локализация nodN в геноме может быть различной. У некоторых представителей он находится в одном кластере с генами nodABC ( S. meliloti , S. medicae , R. leguminosarum ), у других — расположен отдельно от общих nod -генов. Например, у R. galegae nodN находится в другом nod -кластере, а у B. japonicum, M. loti и R. etli — вообще вне кластера симбиотических генов. Кроме того, у R. etli ген nodN имеет хромосомную локализацию.
Интересным результатом анализа стало попадание генов nifB и fixC в кластер 2 и их группировка вместе с геном nodA . В настоящее время известно, что гены, отвечающие за синтез корового Nod-фактора ( nodABC ), имеют неризобиальное происхождение. Так, A. Hirch с соавт. (28) проанализировали последовательности бактериальных и грибных генов, кодирующих ферменты с каталитическими центрами, схожими с таковыми у продуктов генов nodABC . Было сделано предположение, что гены nodB и nodC могли быть получены ризобиями в процессе горизонтального переноса от грамположительных бактерий, у которых они участвуют в биогенезе муреиновой клеточной стенки. Кроме этого указанные гены могли быть получены клубеньковыми бактериями от грибов, активно синтезирующих хитиноподобные вещества. Известно, что многие грибы вступают с бактериями в тесные симбиотические отношения, при которых вполне возможен горизонтальный перенос генов. Однако для гена nodA гомологичных последовательностей в других организмах не обнаружили. Таким образом, ген nodA имеет иное происхождение, нежели nodB и nodC (28). В связи с этим его кластеризация вместе с генами nifB и fixC может свидетельствовать об их изначально совместном горизонтальном переносе из не идентифицированных пока организмов.
Важно отметить, что на специфику кластеризации генов на метадереве, несомненно, должно влиять не только их расположение у современных форм ризобий, но и происходившие ранее эволюционные события, включая независимые перестройки геномов (см. рис. 1), причем свою локализацию могли изменять как отдельные гены, так и содержащие их участки генома. В качестве примера таких перестроек можно привести инверсию nif / fix региона у N. galegae bv. orientalis (18) и транслокацию генов nifHDKEN у R. leguminosarum bv. viciae (11). Несомненно, что геномные перестройки оказывают существенное воздействие на порядок кластеризации генов, однако степень их влияния в рамках примененной модели недостаточно ясна. Существенную роль в возникновении различий в топологиях двух генов, вероятно, играет и неодинаковая скорость их эволюции.
Полученные в нашем исследовании результаты не согласуются с данными, полученными методами молекулярной филогении. Ранее было показано, что полиморфизм nod-генов сформировался независимо от дивергенции коровых элементов бактериального генома, однако коррелирует с таксономией хозяев, тогда как полиморфизм nif-генов коррелирует с дивергенцией коровой части генома бактерий, но не связан с дивергенцией хозяев (29). В нашем исследовании метод метадеревьев показал независимость эволюционной истории как nod-, так и nif/fix-генов от хромосомного маркера (ген 16S рРНК). Возможно, использованный нами метод чувствительнее к влиянию эволюционных факторов, не связанных с ГПГ и не учитываемых в традиционных филогенетических подходах.
Таким образом, результаты филогенетического анализа nod- и nif / fix -генов, который мы провели с помощью метода построения и анализа метадеревьев, подтверждают выявленные ранее различия в эволюционных историях этих генов. Обнаружение двух кластеров, один из которых состоит преимущественно из nif / fix -генов (кластер 1), другой — из nod- генов (кластер 2), может отражать тот факт, что сопоставляемые группы генов возникли на разных этапах эволюции ризобий (в связи с чем они занимают различные участки на хромосомах или плазмидах ризобий), а также экспрессию этих генов на разных стадиях развития симбиоза (инфицирование корневых волосков и освобождение в растительную цитоплазму для nod- генов, симбиотическая фиксация азота — для nif / fix -генов). Присутствие некоторых nod- генов в кластере 1 и некоторых nif / fix -генов — в кластере 2, возможно, обусловлено тем, что на поздних этапах эволюции ризобий весь комплекс sym -генов переносился как единое целое в составе Sym -плазмид или геномных островов. Нельзя также исключить, что отсутствие корреляции между структурой кластеров, выявленных при анализе метадерева, и расположением изучаемых генов на генетических картах — следствие интенсивных внутригеномных перестроек, характерных для многих ризобий, и разной скорости эволюции первичной структуры у изучаемых генов.
Дальнейший анализ роли указанных эволюционных механизмов требует модификации использованного нами подхода, в частности раздельного анализа структур метадеревьев, построенных с использованием выборок штаммов, возникших на ранних стадиях эволюции ризобий (когда ГПГ был ограничен в связи с хромосомной локализацией sym -генов у Bradyrhizobium ) либо на поздних стадиях (когда ГПГ был наиболее интенсивен и скорее всего захватывал весь комплекс sym -генов, локализованных на плазмидах Rhizobium и Sinorhizobium ). Важная задача усовершенствования использованного метода состоит также в разработке статистических и биоинформационных критериев поддержки кластеров, выявляемых на метадеревьях, поскольку на первом этапе работы мы применили для решения этой задачи лишь стандартные биометрические подходы.
Итак, филогенетический анализ nod- и nif/fix-генов, проведенный нами с помощью построения и сопоставления метадеревьев, подтвердил выявленные ранее различия в эволюционных историях этих генов. Наличие двух кластеров в метадереве, один из которых объединяет преимущественно nif/fix-, другой — nod-гены, может быть связано с тем, что этапы эволюции ризобий, на которых возникли сопоставляемые группы, не совпадали, а также с экспрессией этих генов на разных стадиях развития симбиоза. Отсутствие корреляции между структурой кластеров в метадереве и результатами генетического картирования, возможно, обусловлено внутригеномными перестройками и неодинаковой скоростью эволюции нуклеотидных последовательностей генов. Для усовершенствования примененного метода необходимо разработать статистические и биоинформа- ционные критерии поддержки кластеров, выявляемых на метадеревьях.
Список литературы Сравнительный анализ филогений симбиотических генов клубеньковых бактерий с использованием метадеревьев
- Проворов Н., Тихонович И., Андронов Е., Белимов А., Борисов А., Воробьёв Н., Долгих Е., Жернаков А., Жуков В., Кимеклис А., Копать В., Курчак О., Онищук О., Сафронова В., Сулима А., Чижевская Е., Чирак Е., Штарк О. Генетические основы эволюции бактерий -симбионтов растений. СПб, 2016.
- Доуни Дж. Функции ризобиальных генов клубенькообразования. В кн.: Rhizobiaceae. СПб, 2002: 417-434.
- Камински П., Батут Ж., Боистард П. Контроль симбиотической фиксации азота ризобиями. В кн.: Rhizobiaceae. СПб, 2002: 465-492.
- Young J., Crossman L., Johnston A. The genome of Rhizobium leguminosarum has recognizable core and accessory components. Genome Biol., 2006, 7(4): R34 ( ) DOI: 10.1186/gb-2006-7-4-r34
- Reeve W., O'Hara G., Chain P. Complete genome sequence of Rhizobium leguminous-arum bv. trifolii strain WSM2304, an effective microsymbiont of the South American clover Trifolium polymorphum. Stand Genomic Sci., 2010, 2(1): 66-76 ( ) DOI: 10.4056/sigs.44642
- Barnett M., Fisher R., Jones T. Nucleotide sequence and predicted functions of the entire Sinorhizobium meliloti pSymA megaplasmid. PNAS USA, 2001, 98(17): 9883-9888 ( ) DOI: 10.1073/pnas.161294798
- Reeve W., Chain P., O'Hara G. Complete genome sequence of the Medicago microsymbiont Ensifer (Sinorhizobium) medicae strain WSM419. Stand Genomic Sci., 2010, 2(1): 77-86 ( ) DOI: 10.4056/siqs.43526
- Kaneko T., Nakamura Y., Sato S. Complete genomic sequence of nitrogen-fixing symbiotic bacterium Bradyrhizobium japonicum USDA110. DNA Res., 2002, 9(6): 189-197 ( ) DOI: 10.1093/dnares./9.6.189
- Kaneko T., Nakamura Y., Sato S. Complete genome structure of the nitrogen-fixing symbiotic bacterium Mesorhizobium loti. DNA Res., 2000, 7(6): 331-338 ( ) DOI: 10.1093/dnares./7.6.331
- Girard M., Flores M., Brom S. Structural complexity of the symbiotic plasmid of Rhizobium leguminosarum bv. phaseoli. J. Bacteriol., 1991, 173(8): 2411-2419.
- Österman J., March J., Laine P.K. Genome sequencing of two Neorhizobium galegae strains reveals a noeT gene responsible for the unusual acetylation of the nodulation factors. BMC Genomics, 2014, 15: 500 (doi: 10.1186/1471-2164-15-500).
- Хайнес М., Финан Т. Общая генетика. В кн.: Rhizobiaceae. СПб, 2002: 41-62.
- Göttfert M., Röthlisberger S., Kündig C. Potential symbiosis-specific genes uncovered by sequencing a 410-kilobase DNA region of the Bradyrhizobium japonicum chromosome. J. Bacteriol., 2001, 183(4): 1405-1412 ( ) DOI: 10.1128/JB.183.4.1405-1412.2001
- Sullivan J., Ronson C. Evolution of rhizobia by acquisition of a 500-kb symbiosis island that integrates into a phe-tRNA gene. PNAS USA, 1998, 95(9): 5145-5149.
- González V., Santamaría R., Bustos P. The partitioned Rhizobium etli genome: Genetic and metabolic redundancy in seven interacting replicons. PNAS USA, 2006, 103(10): 3834-3839 ( ) DOI: 10.1073/pnas.0508502103
- Воробьёв Н., Проворов Н. Моделирование эволюции бобово-ризобиального симби-оза в условиях экологической нестабильности. Экологическая генетика, 2013, 11: 73-85.
- Provorov N., Andronov E. Evolution of root nodule bacteria: reconstruction of the speciation processes resulting from genomic rearrangements in a symbiotic system. Microbiology, 2016, 83(2): 131-139 ( ) DOI: 10.1134/S0026261716020156
- Nye T. Trees of trees: an approach to comparing multiple alternative phylogenies. Syst. Biol., 2008, 57(5): 785-794 ( ) DOI: 10.1080/10635150802424072
- Nye T., Lio P., Gilks W. A nowel algorithm and web-based tool for comparing two alternative phylogenetic trees. Bioinformatics, 2005, 22(1): 117-119.
- Atkinson E., Palcic M., Hindsgaul O., Long S. Biosynthesis of Rhizobium meliloti lipooligosaccharide Nod-factors: NodA is required for an N-acyltransferase activity. PNAS USA, 1994, 91(18): 8418-8422.
- John M., Rohrig H., Schmidt J. Rhizobium NodB protein involved in nodulation signal synthesis is chitooligosaccharide deacetylase. PNAS USA, 1993, 90: 625-629.
- Debelle F., Rosenberg C., Denarie J. The Rhizobium, Bradyrhizobium and Azorhizobium NodC proteins are homologous to yeast chitin synthases. Mol. Plant-Microbe Interact., 1992, 5: 443-446.
- Peck M., Fisher R., Long S. Diverse flavonoids stimulate NodD1 binding to nod gene promoters in Sinorhizobium meliloti. J. Bacteriol., 2006, 188(15): 5417-5427 ( ) DOI: 10.1128/JB.00376-06
- Evans I., Downie J. The NodI product of Rhizobium leguminosarum is closely related to ATP-binding bacterial transport proteins: nucleotide sequence of nodI and nodJ Genes. Gene, 1986, 43: 95-101.
- Baev N., Shultze M., Barlier I. Rhizobium nodM and nodN genes are common nod genes: nodM encodes functions for efficiency of nod signal production and bacteroid maturation. J. Bactreriol., 1992, 174(23): 7555-7565.
- Putnoky P., Grosskopf E., Cam D. Rhizobium fix-genes mediate at least two communication steps in symbiotic nodule development. J. Cell Biol., 1988, 106: 597-607.
- Arnold W., Rump A., Klipp W. Nucleotide sequence of 24,206-base-pair DNA fragment carrying the entire nitrogen fixation gene cluster of Klebsiella pneumonia. J. Mol. Biol., 1988, 203: 715-738.
- Hirch A., Lum M., Downie J. What makes the rhizobia-legume symbiosis so special? Plant Physiol., 2001, 127: 1484-1492 ( ) DOI: 10.1104/pp.010866
- Проворов Н., Воробьёв Н. Генетические основы эволюции растительно-микроб-ного симбиоза. СПб, 2012.


