Сравнительный анализ профилей экспрессии генов опухолевого контроля и микроРНК в опухолевой и перифокальной ткани у пациентов с колоректальным раком
Автор: Служев М.И., Зарайский М.И., Семиглазов В.В., Семиглазова Т.Ю., Ткаченко Е.В., Кондратьев С.В., Бриш Н.А., Алексеева Ю.В., Петрик Ю.В., Сидорова А.Н.
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Лабораторные и экспериментальные исследования
Статья в выпуске: 2 т.21, 2022 года.
Бесплатный доступ
Введение. В развитии и прогрессировании колоректального рака (КРР) важную роль играют различные гены опухолевого контроля и микроРНК. Экспрессия этих генов может значительно отличаться в опухолевой и перифокальной здоровой ткани. Нет точных данных, на каком расстоянии от опухоли находится перифокальная здоровая ткань с точки зрения экспрессии генов. Цель исследования -изучить профили экспрессии генов опухолевого контроля ( E2F3, TGFB, NFKB, KLF-12, EGFR и MMP9 ), а также экспрессии генов микроРНК ( микроРНК-15, -16, -21 и -210 ) в опухолевой и перифокальной здоровой тканях при КРР. Материал и методы. В исследование были включены 19 пациентов с диагнозом КРР. В рамках постановки диагноза при колоноскопии забирался материал опухолевой и рядом расположенной перифокальной визуально здоровой ткани на расстоянии 1-2 см от края опухоли. В полученном материале исследовали уровни экспрессии генов опухолевого контроля ( E2F3, TGFB, NFKB, KLF-12, EGFR и MMP9 ), а также уровни экспрессии генов микроРНК ( микроРНК-15, -16, -21 и -210 ). Результаты. Было обнаружено снижение уровня экспрессии E2F3 (Медиана 3,73, Q1-Q3 2,64 УЕ против Медиана 6,5, Q1-Q3 6,39 УЕ, p=0,01) и микроРНК-16 (Медиана 2,83, Q1-Q3 4,74 УЕ против Медиана 4,29, Q1-Q3 3,73 УЕ, p=0,027) и повышение уровня экспрессии микроРНК-21 (Медиана 2,64, Q1-Q3 1,38 УЕ против Медиана 1,41, Q1-Q3 1,21 УЕ, p function show_abstract() { $('#abstract1').hide(); $('#abstract2').show(); $('#abstract_expand').hide(); }
Микрорнк, колоректальный рак, экспрессия генов, e2f3, микрорнк-16, микрорнк-21, перифокальная ткань
Короткий адрес: https://sciup.org/140293899
IDR: 140293899 | УДК: 616.34-006.6:575.113.1
Текст научной статьи Сравнительный анализ профилей экспрессии генов опухолевого контроля и микроРНК в опухолевой и перифокальной ткани у пациентов с колоректальным раком
Колоректальный рак (КРР) занимает лидирующие позиции в структуре заболеваемости и смертности среди всех злокачественных новообразований. Хирургическое удаление опухоли с последующей химиотерапией является стандартом лечения КРР. Общепризнанным подходом к выбору края резекции опухоли при КРР является иссечение кишки в пределах не менее 10 см от опухоли при операциях по поводу рака ободочной кишки, не менее 5 см – при операциях по поводу рака дистальной трети сигмовидной кишки, ректосигмоидного отдела толстой кишки и рака прямой кишки [1, 2]. Однако по данным анализа 17 исследований ширина резекции 1 см и более может быть достаточной в отдельных случаях при раке прямой кишки, и это не увеличивает риск рецидива [3]. Аналогичные данные в обзоре представил C.-G. Ker, где безопасное расстояние при хирургическом лечении рака прямой кишки составило от 1 до 3 см [4].
Отсутствие опухолевой ткани в конкретной области может быть подтверждено гистологически. Однако существуют генетические особенности, которые могут быть определены только путем молекулярно-генетического исследования. Имеющиеся данные дают основания для оценки различий геномного профиля опухолевой и перифокальной тканей. На образцах опухолей и прилежащей нормальной ткани толстой кишки мышей показано, что профиль экспрессии генов значимо отличается в опухолевой и перифокальной нормальной ткани на расстоянии 1–2 см и 4 см от опухоли [5]. Однако до сих пор нет данных о качественных различиях опухолевой и перифокальной ткани на генном уровне у пациентов с КРР в зависимости от расстояния от опухоли.
В онкогенезе и метастазировании КРР участвует множество различных факторов: белков сигна-линга, межклеточного взаимодействия, факторов транскрипции, эпигенетических факторов. Одним из наиболее изученных сигнальных путей, повреж- дающихся при КРР, является путь фактора роста опухоли (TGF-B) [6]. M. Nakano et al. показали, что сигнальный путь TGF-B участвует в прогрессировании КРР, приводя к дедифференцировке неопухолевых стволовых клеток в опухолевые стволовые [7].
Другими важными факторами контроля онкогенеза КРР являются гены семейства E2F . Так, ген E2F3 обеспечивает точную передачу генетического материала каждой дочерней клетке во время клеточного цикла [8]. Однако он может участвовать и в развитии опухолевого процесса. В частности, показано, что уровни экспрессии E2F3 были выше в опухолевых тканях, что может быть причиной усиления пролиферации и роста клеток КРР [9]. Существует универсальный фактор – ген NFkB . Он является важным участником в передаче сигналов, способствующих развитию метастатического процесса при КРР [10]. Экспрессия рецептора эпидермального фактора роста (EGFR) наблюдается в большинстве случаев КРР, при этом гиперэкспрессия EGFR достоверно связана с плохим прогнозом и чаще наблюдается при распространенном опухолевом процессе [11]. Семейство Круппель-подобных транскрипционных факторов ( KLF ) также участвует в контроле роста, пролиферации, апоптоза и ангиогенеза. В частности, KLF8 способствует онкогенезу, инвазии и метастазированию клеток КРР [12], а потеря KLF3 коррелировала с агрессивными фенотипами и плохими результатами выживания [13]. Матриксная металлопротеиназа 9 (MMP9), известная также как «желатиназа В», участвует в ремоделировании структур внеклеточного матрикса. Изменение экспрессии MMP-9 может влиять на эффективность лечения злокачественных новообразований. Например, гиперэкспрессия MMP9 предсказывает плохую выживаемость и хороший ответ на химиотерапию у пациентов с КРР [14].
МикроРНК представляют собой большую группу некодирующих РНК, которые не могут быть транслированы в белок. МикроРНК регулируют экспрессию более 30 % генов человека [15]. Аномальная экспрессия микроРНК является значимым элементом канцерогенеза [16]. Низкая экспрессия микроРНК-15 коррелирует с плохим прогнозом пациента с КРР [17]. Гиперэкспрессия микроРНК-15 увеличивает чувствительность клеток рака толстой кишки к 5-фторурацилу и оксалиплатину [18]. Снижение экспрессии микроРНК-16 может быть маркером плохого прогноза [19]. W. Zhang et al. показали в метаанализе, что низкий уровень экспрессии микроРНК-16 был связан с плохим прогнозом при солидных опухолях [20]. Повышенная экспрессия микроРНК-21 и микроРНК-210 наблюдается при КРР [19, 21]. По данным метаанализа, гиперэкспрессия микроРНК-21 при колоректальном раке связана с плохой выживаемостью [22].
Перечисленные выше гены играют важную роль в онкогенезе КРР и могут служить отличительными характеристиками опухолевого процесса. Это дает возможность провести сравнительный анализ экспрессии генов для определения различия между опухолевой тканью и перифокальной тканью на расстоянии 1–2 см.
Цель исследования – изучить профили экспрессии генов опухолевого контроля ( E2F3, TGFB, NFKB, KLF-12, EGFR и MMP9 ), а также экспрессии генов микроРНК ( микроРНК-15, -16, -21 и -210 ) в опухолевой и перифокальной здоровой тканях при КРР.
Материал и методы
Исследование выполнено в соответствии с Хельсинкской декларацией [23]. С 02.2020 по 04.2021 в обсервационное исследование было включено 20 пациентов, из них 13 мужчин и 7 женщин (табл. 1), которые соответствовали критериям включения и подписали информированное согласие. Из анализа был исключен 1 пациент (мужчина) в связи с наличием значительного воспалительного компонента в биопсийных тканях, что могло исказить окончательные результаты. В исследование были включены мужчины и женщины в возрасте от 42 до 80 лет с подозрением на КРР, которым в плановом порядке производили верификацию опухоли. Все пациенты с верифицированным КРР получали стандартное лечение в соответствии с национальными и международными рекомендациями по клинической практике с учетом стадии заболевания.
У каждого пациента с помощью видеоколоноскопии была выполнена биопсия опухоли и нормальной здоровой эпителиальной ткани толстой кишки. Участок «здоровой» ткани выбирался визуально врачом-эндоскопистом на расстоянии 1–2 см от опухоли. Биоматериал (опухоль и здоровая ткань кишечника) исследовали на уровни экспрессии генов опухолевого контроля ( E2F3, TGFB, NFKB, KLF-12, EGFR и MMP9 ), относительно референц-гена GAPDH , а также на уровни экспрессии генов микроРНК ( микроРНК-15, -16, -21 и -210 ), используя в качестве референц-гена малую ядерную РНК U6 .
Исследование относительной экспрессии генов опухолевого контроля, а также генов микроРНК проводили по полуколичественному протоколу [24]. Тотальную РНК выделяли из биоматериала (биопсийный материал) с помощью реактива ExtractRNA (Евроген, Москва) в соответствии с инструкцией.
Для приготовления кДНК генов опухолевого контроля использовали набор для обратной транскрипции «ОТ-1» (Синтол, Москва) в модификации для гексануклеотидных праймеров, согласно инструкции. Приготовление кДНК генов микроРНК проводили технологией StemLoop со специфическими праймерами, раздельно для
Таблица 1/table 1
|
Характеристика/Characteristics |
Значение/Values |
|
Возраст, лет/Age, years |
63,2 ± 11 |
|
Мужчины/Males |
12 (63 %) |
|
Женщины/Females |
7 (37 %) |
|
N0 |
10 (53 %) |
|
N+ |
9 (47 %) |
|
Стадия I/Stage I |
5 (26 %) |
|
Стадия II/Stage II |
4 (21 %) |
|
Стадия III/Stage III |
4 (21 %) |
|
Стадия IV/Stage IV |
6 (32 %) |
|
Low-grade High-grade |
16 (84 %) 3 (16 %) |
|
M1 HEP |
5 (26 %) |
|
M1 PUL/PLE |
3 (16 %) |
|
Левая половина ободочной кишки, рак прямой кишки/Left side of the colon, rectal cancer |
15 (80 %) |
|
Правая половина ободочной кишки/Right half of the colon |
2 (10 %) |
|
Первично-невыясненная локализация/Primary unexplained localization |
2 (10 %) |
Примечание: Low-grade – низкая степень злокачественности; High-grade – высокая степень злокачественности; M1 HEP – наличие метастазов в печени; M1 PUL/PLE – наличие метастазов в легких и/или плевре.
Характеристика пациентов characteristics of patients
Note: Low-grade – low degree of malignancy; High-grade – high degree of malignancy; M1 HEP – the presence of metastases in the liver; M1 PUL/ PLE – the presence of metastases in the lungs and / or pleura.
Таблица 2/table 2
Последовательности праймеров для обратной транскрипции генов микроРНК primer sequences for reverse transcription of miRNa genes
|
МикроРНК/MicroRNA |
Последовательности праймеров/Primer sequences |
|
МикроРНК-15 |
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACCACAAA |
|
МикроРНК-16 |
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACCGCCAA |
|
МикроРНК-21 |
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTCAAC |
|
МикроРНК-210 |
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACTCAGCC |
U6 GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACAAAAATATG каждой микроРНК и гена сравнения U6 (табл. 2) с использованием набора для обратной транскрипции «ОТ-1» (Синтол, Москва). Температурный профиль реакции был следующий: 16 ºС – 30 мин, 42 ºС – 30 мин, 85 ºС – 5 мин.
В качестве полимеразной цепной реакции (ПЦР) смеси была использована 2,5× реакционная смесь с интекалирующим красителем EVA Green (Синтол, Москва). Амплификацию проводили в режиме RealTime на приборе DTLite-4 (ДНК-Технология, Москва) по стандартной двухпраймерной схеме по полуколичественному протоколу. Праймеры для ПЦР представлены в табл. 3. Температурный профиль амплификации генов опухолевого контроля: 95 ºС – 7 мин, затем 40 циклов (95 ºС – 15 сек и 56 ºС – 1 мин), для микроРНК: 95 ºС – 5 мин, затем 45 циклов (95 ºС – 15 сек и 60 ºС – 1 мин). Оценку относительного уровня экспрессии генов проводили по протоколу ΔСt и рассчитывали по формуле 2(-(А-В)), где А – Ct гена микроРНК, а В – Ct гена U6 [25]. Полученный результат выражали в условных единицах экспрессии (УЕ).
Статистический анализ проводили с использованием непараметрических методов. Тип распределения был протестирован с использованием гистограмм распределения и критериев Шапиро– Уилка и Колмогорова–Смирнова. Категориальные данные представлены как частота и процент. Непрерывные переменные представлены как среднее ± стандартное отклонение. Количественные параметры когорт сравнивали с использованием критерия Манна–Уитни. Корреляции между стадией, степенью злокачественности, локализацией первичной опухоли, поражением лимфатических узлов и печени, с одной стороны, и всеми факторами – с другой анализировали с использованием ранговой корреляции Спирмена. Уровень p<0,05 считался статистически значимым. Для статистических тестов использовали программный пакет SPSS 23.0 (IBM, Чикаго, Иллинойс).
Результаты
Оценка уровней экспрессии генов опухолевого контроля проводилась путем сравнения экс-
Таблица 3/table 3
|
Ген/Gene |
Последовательности праймеров/Primer sequences |
|
E2F3 (прямой)/(forward) |
GCACTACGAAGTCCAGATAGTCC |
|
E2F3 (обратный)/(reverse) |
AGACTGCAGCCCATCCATTG |
|
TGFB (прямой)/(forward) |
GCCCTGGACACCAACTATTG |
|
TGFB (обратный)/(reverse) |
CGTGTCCAGGCTCCAAATG |
|
NFKB (прямой)/(forward) |
CCTCTCTCTAATCAGCCCTCTG |
|
NFKB (обратный)(reverse) |
GAGGACCTGGGAGTAGATGAG |
|
EFFR (прямой)/(forward) |
GGGTTCAGAGGCTGATTGTGATAGA |
|
EGFR (обратный)(reverse) |
CGCCTTGACTGAGGACAGCATA |
|
KLF-12 (прямой)/(forward) |
CACCTGGAAATGTGAACAACA |
|
KLF-12 (обратный)/(reverse) |
TTTTACTTTGTCTGGGAGATAGGC |
|
MMP9 (прямой)/(forward) |
CGCAGACATCGTCATCCAGT |
|
MMP9 (обратный)/(reverse) |
GGATTGGCCTTGGAAGATGA |
|
GAPDH (прямой)/(forward) |
AGAAGGCTGGGGCTCATTTG |
|
GAPDH (обратный)/(reverse) |
AGGGGCCATCCACAGTCTTC |
|
МикроРНК-15/MicroRNA-15 |
GACGCCTAGCAGCACATA |
|
МикроРНК-16/MicroRNA-16 |
CCGGAGTAGCAGCACGTAAAT |
|
МикроРНК-21/MicroRNA-21 |
GCCCGCTAGCTTATCAGACTGATG |
|
МикроРНК-210/MicroRNA-210 |
CAGAGTGCTGTGCGTGTGA |
|
U6 |
GCGCGTCGTGAAGCGTTC |
|
Общий праймер для микроРНК/Total primer for microRNA |
GTGCAGGGTCCGAGGT |
Последовательности праймеров для ПЦР pcR primer sequences
прессии в двух группах: 1) опухолевый биоптат и 2) визуально здоровая перифокальная область. Относительные уровни экспрессии генов E2F3, TGFB, NFKB, KLF-12, EGFR и MMP9 представлены на рис. 1. Уровень экспрессии гена E2F3 был статистически значимо выше в нормальной ткани по сравнению с опухолевой (Медиана 6,5, Q1-Q3 6,39 УЕ vs Медиана 3,73, Q1-Q3 2,64 УЕ, p=0,01).
Уровни экспрессии остальных генов не показали статистически значимых отличий в исследуемых группах.
Оценка уровней экспрессии генов опухолевого контроля проводилась также путем сравнения экспрессии в группах материала кишки из фокуса опухоли и здоровой перифокальной области. Относительные уровни экспрессии генов микроРНК-15,
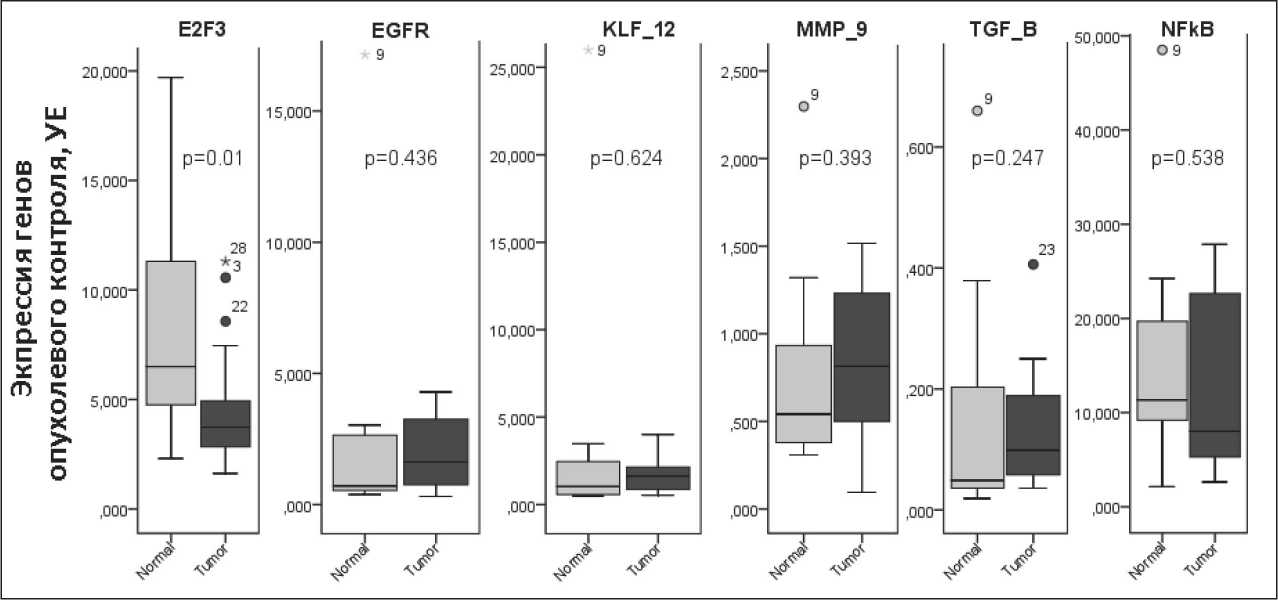
Рис.1. Экспрессия генов опухолевого контроля в нормальной и опухолевой тканях.
Примечание: Normal – визуально здоровая перифокальная ткань; Tumor – опухолевая ткань; УЕ – условные единицы Fig. 1. Expression of tumor control genes in normal and tumor tissues.
Note: Normal – visually healthy perifocal tissue; Tumor – tumor tissue; УЕ (REU) – relative units of expression
-
-16, -21 и -210 представлены на рис. 2. Уровень экспрессии генов микроРНК-16 был статистически значимо выше в нормальной ткани (Медиана 4,29, Q1-Q3 3,73 УЕ vs Медиана 2,83, Q1-Q3 4,74 УЕ, p=0,027), а уровень экспрессии генов микроРНК-21 , наоборот, статистически значимо оказался выше в опухолевой ткани (Медиана 2,64, Q1-Q3 1,38 УЕ vs Медиана 1,41, Q1-Q3 1,21 УЕ, p<0,001).
При многофакторном анализе корреляций уровней экспрессии исследуемых генов в ткани опухоли между собой и клиническими данными были получены следующие результаты. Существует положительная корреляция экспрессии генов E2F3 с TGFB и NFkB (r=0,67, p=0,035 и r=0,73, p<0,001 соответственно). Также выявлена положительная корреляция между экспрессией генов TGFB и NFkB (r=0.84, p=0,002). Найдена сильная положительная корреляция экспрессии генов EGFR с KLF и MMP-9 (r=0,81, p=0,004 и r=0,84, p=0,003 соответственно). Кроме того, зарегистрирована корреляция между экспрессиями генов KLF-12 и MMP-9 (r=0,66, p=0,038).
При оценке связи экспрессии исследуемых генов с клиническими данными выявлена отрицательная корреляция уровня экспрессии генов микроРНК-210 с опухолевым поражением лимфатических узлов, метастатическим поражением печени и стадией опухолевого процесса (r=-0,59, p=0,008; r=-0,54, p=0,018; r=-0,60, p=0,007 соответственно). Также обнаружена корреляция экспрессии генов микроРНК-210 с микроРНК-16 и микроРНК-15 (r=0,55, p=0,014 и r=0.70, p=0,025 соответственно). Отдельно хотелось бы подчеркнуть отсутствие корреляций микроРНК-21 со всеми исследуемыми генами.
При многофакторном анализе корреляций уровней экспрессии исследуемых генов между собой в перифокальной области были получены следующие результаты. Обнаружено наличие высокодостоверной статистической положительной корреляции экспрессии гена E2F3 и генов KLF-12 и NFkB (r=0,49, p=0,035 и r=0,72, p=0,001 соот- ветственно). Схожие экспрессионные характеристики имел и ген EGFR с генами KLF-12 и NFkB (r=0,85, p=0,002 и r=0,65, p=0,042 соответственно). Дополнительно к указанным ген KLF-12 имел положительную корреляцию с экспрессией генов TGFB и NFkB (r=0,69, p=0,029 и r=0,62, p=0,004 соответственно) и отрицательную корреляцию с экспрессией микроРНК-15 (r=-0,65, p=0,043), -16 (r=-0,63, p=0,004) и -210 (r=-0,47, p=0,040). Экспрессия гена MMP-9 коррелировала с таковой гена TGFB (r=0,78, p=0,008). Также выявлена положительная корреляция экспрессий генов TGFB и NFkB (r=0,65, p=0,042).
При оценке экспрессионных взаимодействий генов микроРНК показано, что микроРНК-15 имела отрицательную корреляцию с генами TGFB (r=-0,67, p=0,033) и NFkB (r=-0,71, p=0,023) и положительную с микроРНК-16 (r=0,67, p=0,035), -21 (r=0,90, p<0,001) и 210 (r=0,66, p=0,038). Дополнительно, экспрессия микроРНК-210 высоко коррелировала с микроРНК-16 (r=0,59, p=0,008).
Обсуждение
Настоящая работа посвящена изучению экс-прессионного профиля некоторых генов, в том числе и регуляторных, включенных в онкологический процесс, в опухолевой и перифокальной тканях при КРР. В качестве исследуемого материала использовались биоптаты, полученные при колоноскопии. Перифокальная область рассматривалась как ткань толстой кишки в 1–2 см от видимого опухолевого очага. Были найдены значимые отличия в экспрессии генов E2F3 , микроРНК-16 и микроРНК-21 в опухолевой и перифокальной тканях на расстоянии 1–2 см от опухоли. В нашей работе не обнаружено достоверных различий в экспрессии генов опухолевого контроля TGFB , NFKB , KLF-12 , EGFR и MMP9 , а также экспрессии генов микроРНК-15 и микроРНК-210 .
Согласно полученным данным, уровень экспрессии E2F3 в нормальной ткани оказался выше, чем в опухолевой ткани. Повышение уровня экспрессии E2F3 наблюдается при усилении пролифе-
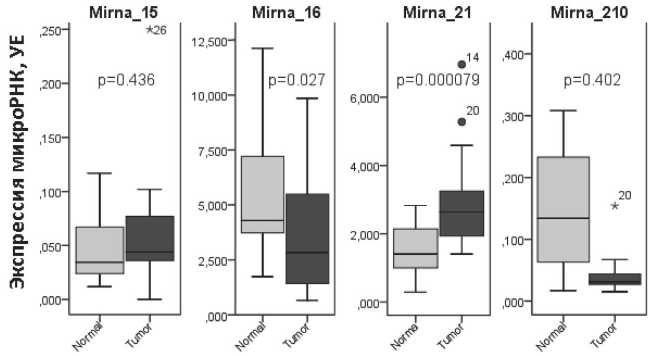
Рис. 2. Экспрессия генов микроРНК в нормальной и опухолевой тканях. Примечание: Normal – визуально здоровая перифокальная ткань;
Tumor – опухолевая ткань;
УЕ – условные единицы
Fig. 2. Expression of microRNA genes in normal and tumor tissues Note: Normal – visually healthy perifocal tissue; Tumor – tumor tissue; УЕ (REU) – relative units of expression
рации и роста клеток [9]. Повышенный рост клеток нормальной ткани несет риски злокачественного перерождения и прогрессирования. С другой стороны, сниженная экспрессия E2F3 в опухолевой ткани также является неблагоприятным признаком, так как может служить причиной генетической нестабильности и тем самым увеличивать метастатический потенциал опухолевых клеток [8].
Выявлено, что у пациентов с повышенной экспрессией E2F3 повышены также экспрессии TGF-B и NFkB . Белок NFkB является частью сигнального пути AKT/NFkB/MDR1, ответственного за развитие резистентности к лекарственной терапии при колоректальном раке [26]. Учитывая наличие корреляции с NFkB , высокая экспрессия E2F3 потенциально является прогностическим фактором развития лекарственной устойчивости. Данное наблюдение может быть подтверждено только в рамках последующих клинических исследований.
Нарушение регуляции экспрессии микроРНК связано с инициацией и прогрессированием злокачественного новообразования. Негативные последствия в отношении прогноза при КРР имеет изменение экспрессии нескольких микроРНК .
Выявлена низкая экспрессия микроРНК-16 в опухолевой ткани по сравнению с перифокальной. Известно, что микроРНК-16 снижает пролиферацию и индуцирует апоптоз опухолевых клеток при колоректальном раке [27]. C. Farace et al. сообщили, что низкая экспрессия микроРНК-16 при колоректальном раке является неблагоприятным прогностическим фактором [19]. Сниженная экспрессия микроРНК-16 в опухолевой ткани в нашей работе может являться фактором негативного прогноза.
Уровень экспрессии микроРНК-21 оказался выше в опухолевой ткани. Как показали Y. Wu et al., микроРНК-21 при колоректальном раке способствует росту и инвазии опухолевых клеток с помощью подавления активности опухолевого супрессора PTEN [28]. Y. Yu et al. показали в метаанализе, что повышенная экспрессия микроРНК-21 в тканях предсказывала снижение общей выживаемости и выживаемости без прогрессирования у пациентов с КРР [22]. Повышенная экспрессия микроРНК-21 в опухолевой ткани в нашей работе может являться негативным прогностическим фактором рецидива и прогрессирования колоректального рака.
Уровень экспрессии микроРНК-210 в перифокальной ткани оказался выше, чем в опухолевой, хотя разница и не была статистически значима. Как показали K.E. Tagscherer et al., гиперэкспрессия микроРНК-210 может индуцировать апоптоз опухолевых клеток через ROS механизм [29]. Однако в работах других авторов было показано, что экспрессия микроРНК-210 повышена при опухолевом процессе [19, 21], в особенности в условиях гипоксии [30]. Учитывая эти данные, не исключено, что высокая экспрессия микроРНК-210 в нормальной перифокальной ткани может быть фактором повышенного риска рецидива, развития опухоли de novo и неблагоприятного прогноза. Обнаружено, что чем ниже экспрессия генов микроРНК-210 в опухолевой ткани, тем более вероятно метастатическое поражение лимфатических узлов, печени, а также выше стадия опухолевого процесса.
Кроме того, выявлена корреляция экспрессии EGFR с KLF и MMP-9 , а также корреляция экспрессии KLF и MMP-9 между собой. EGFR и KLF являются важными участниками роста опухолевых клеток, в то время как MMP-9 участвует в ремоделировании структур внеклеточного матрикса и может обеспечивать необходимые изменения в ткани, способствуя развитию опухолевого процесса. Гиперэкспрессия этих факторов связана с плохим прогнозом при КРР [11, 12, 14].
Важно отметить, что при перекрестном анализе экспрессионных ландшафтов нормальной и опухолевой ткани нами был выявлен ряд идентичных взаимосвязей. Так, экспрессия микроРНК-210 имела одинаково достоверную корреляцию с микроРНК-15 и -16 , ген NFkB – с генами E2F3 и TGFB , а ген KLF-12 – с геном EGFR .
Таким образом, опухолевая и перифокальная ткани при КРР имеют значимые различия в уровнях экспрессии генов E2F3 , микроРНК-16 и микроРНК-21 , определяющих регуляцию опухолевого процесса.
Заключение
Обнаружено снижение уровня экспрессии E2F3 и микроРНК-16 и повышение уровня экспрессии микроРНК-21 в опухолевой ткани по сравнению с нормальной тканью у пациентов с КРР. Это дает возможность дифференцировать опухолевую и перифокальную ткань на расстоянии 1–2 см от опухоли не только гистологическим, но и молекулярно-генетическим методом исследования. Повышенный уровень экспрессии E2F3 и микроРНК-16 на расстоянии 1–2 см от опухоли может быть предиктором злокачественного перерождения, рецидива и прогрессирования опухоли. Экспрессия гена E2F3 коррелирует с уровнем экспрессии гена NFkB , ответственного за развитие резистентности к лекарственной терапии, и потенциально может служить неблагоприятным прогностическим фактором лекарственной устойчивости. Повышенная экспрессия микроРНК-21 в опухолевой ткани может являться негативным прогностическим фактором.
Существует возможная диагностическая ценность таких факторов, как микроРНК-210 , KLF и MMP-9 . Повышенная экспрессия микроРНК-210 в перифокальной ткани, а KLF и MMP-9 в опухолевой ткани может быть фактором рецидива и неблагоприятного прогноза.
Совместная оценка экспрессионных характеристик генов контроля опухолевого роста и генов-регуляторов – микроРНК – в опухолевой и нормальной ткани на до- или интраоперационном этапах, безусловно, может рассматриваться как потенциальный прогностический критерий. Так, возможно, чем более различным является характер генетической дисрегуляции в ткани опухоли и перифокальной ткани, остающейся после резекции,
Список литературы Сравнительный анализ профилей экспрессии генов опухолевого контроля и микроРНК в опухолевой и перифокальной ткани у пациентов с колоректальным раком
- Каприн А.Д. Клинические рекомендации. Злокачественные новообразования ободочной кишки и ректосигмоидного отдела. Общероссийский национальный союз Ассоциация онкологов России; 2020. [KaprinA.D. Clinical guidelines. Malignant neoplasms of the colon and rectosigmoid region. All-Russian National Union. Association of Oncologists of Russia; 2020. (in Russian)].
- Каприн А.Д. Клинические рекомендации. Рак прямой кишки. Общероссийский национальный союз Ассоциация онкологов России; 2020. [Kaprin A.D. Clinical guidelines. Rectal cancer. All-Russian National Union Association of Oncologists of Russia; 2020. (in Russian)].
- Bujko K., Rutkowski A., Chang G.J., Michalski W., Chmielik E., Kusnierz J. Is the 1-cm rule of distal bowel resection margin in rectal cancer based on clinical evidence? A systematic review. Ann Surg Oncol. 2012; 19(3): 801-8. doi: 10.1245/s10434-011-2035-2.
- Ker C.-G. Surgical safety margin of gastroenterological cancer surgery: A truth or a dream? Formosan J Sur. 2014; 47(3): 83-9.
- AranD., CamardaR., OdegaardJ., PaikH., OskotskyB., Krings G., Goga A., Sirota M., Butte A.J. Comprehensive analysis of normal adjacent to tumor transcriptomes. Nat Commun. 2017; 8(1): 1077. doi: 10.1038/ s41467-017-01027-z.
- Cai J., Xia L., Li J., Ni S., Song H., Wu X. Tumor-Associated Macrophages Derived TGF-p-Induced Epithelial to Mesenchymal Transition in Colorectal Cancer Cells through Smad2,3-4/Snail Signaling Pathway. Cancer Res Treat. 2019; 51(1): 252-66. doi: 10.4143/crt.2017.613.
- Nakano M., Kikushige Y., Miyawaki K., Kunisaki Y., Mizuno S., Takenaka K., Tamura S., Okumura Y., Ito M., Ariyama H., Kusaba H., Nakamura M., Maeda T., Baba E., Akashi K. Dedifferentiation process driven by TGF-beta signaling enhances stem cell properties in human colorectal cancer. Oncogene. 2019; 38(6): 780-93. doi: 10.1038/s41388-018-0480-0.
- SaavedraH.I., Maiti B., Timmers C., AlturaR., Tokuyama Y., Fuka-sawaK., Leone G. Inactivation of E2F3 results in centrosome amplification. Cancer Cell. 2003; 3(4): 333-46. doi: 10.1016/s1535-6108(03)00083-7.
- Yao H., Lu F., Shao Y. The E2F family as potential biomarkers and therapeutic targets in colon cancer. PeerJ. 2020; 8. doi: 10.7717/ peerj.8562.
- Jana A., Krett N.L., Guzman G., Khalid A., Ozden O., Staudacher J.J., Bauer J., Baik S.H., Carroll T., Yazici C., Jung B. NFkB is essential for activin-induced colorectal cancer migration via upregulation of PI3K-MDM2 pathway. Oncotarget. 2017; 8(23): 37377-93.
- Del Carmen S., CorcheteL.A., GervasR., Rodriguez A., GarciaM., Alcazar J.A., Garcia J., Bengoechea O., Munoz-Bellvis L., Sayagues J.M., AbadM. Prognostic implications of EGFR protein expression in sporadic colorectal tumors: Correlation with copy number status, mRNA levels and miRNA regulation. Sci Rep. 2020; 10(1): 4662. doi: 10.1038/s41598-020-61688-7.
- Yan Q., Zhang W., Wu Y., WuM., ZhangM., Shi X., Zhao J., Nan Q., Chen Y., Wang L., Cheng T., Li J., Bai Y., Liu S., Wang J. KLF8 promotes tumorigenesis, invasion and metastasis of colorectal cancer cells by transcriptional activation of FHL2. Oncotarget. 2015; 6(28): 25402-17.
- Wang X., Jiang Z., Zhang Y., Wang X., Liu L., Fan Z. RNA sequencing analysis reveals protective role of kruppel-like factor 3 in colorectal cancer. Oncotarget. 2017; 8(13): 21984-93. doi: 10.18632/ oncotarget.15766.
- Yang X.Z., Cui S.Z., Zeng L.S., Cheng T.T., Li X.X., Chi J., Wang R., Zheng X.F., Wang H.Y. Overexpression of Rab1B and MMP9 predicts poor survival and good response to chemotherapy in patients with col-orectal cancer. Aging (Albany NY). 2017; 9(3): 914-31. doi: 10.18632/ aging.101200.
- Lewis B.P., Burge C.B., Bartel D.P. Conserved seed pairing, often flanked by adenosines, indicates that thousands of human genes are microR-NA targets. Cell. 2005; 120(1): 15-20. doi: 10.1016/j.cell.2004.12.035.
- TruongA.D., RengarajD., Hong Y., Hoang C.T., Hong Y.H., Lille-hojH.S. Differentially expressed JAK-STAT signaling pathway genes and target microRNAs in the spleen of necrotic enteritis-afflicted chicken lines. Res Vet Sci. 2017; 115: 235-43. doi: 10.1016/j.rvsc.2017.05.018.
- Fesler A., Liu H., Ju J. Modified miR-15a has therapeutic potential for improving treatment of advanced stage colorectal cancer through inhibition of BCL2, BMI1, YAP1 and DCLK1. Oncotarget. 2017; 9(2): 2367-83. doi: 10.18632/oncotarget.23414.
- Liu L., Wang D., Qiu Y., Dong H., Zhan X. Overexpression of microRNA-15 increases the chemosensitivity of colon cancer cells to 5-fluorouracil and oxaliplatin by inhibiting the nuclear factor-кВ signalling pathway and inducing apoptosis. Exp Ther Med. 2017; 2655-60. doi: 10.3892/etm.2017.5675.
- Farace C., Pisano A., Grinan-Lison C., Solinas G., Jiménez G., Serra M., Carrillo E., Scognamillo F., Attene F., Montella A., Marchal J.A., Madeddu R. Deregulation of cancer-stem-cell-associated miRNAs in tissues and sera of colorectal cancer patients. Oncotarget. 2020; 11(2): 116-30. doi: 10.18632/oncotarget.27411.
- Zhang W., Zhou F., Jiang D., Mao Y., Ye D. Association of the Expression Level of miR-16 with Prognosis of Solid Cancer Patients: A Meta-Analysis and Bioinformatic Analysis. Disease Markers. 2020; 1-9.
- Sabry D., El-Deek S.E.M., Maher M., El-Baz M.A.H., ElBader H.M., Amer E., Hassan E.A., Fathy W., El-Deek H.E.M. Role of miRNA-210, miRNA-21 and miRNA-126 as diagnostic biomarkers in colorectal carcinoma: impact of HIF-1a-VEGF signaling pathway. Mol Cell Biochem. 2019; 454(1-2): 177-89. doi: 10.1007/s11010-018-3462-1.
- Yu Y., Chen Z., Liu H., Jin W., Ding Z., Zheng S. Tissue microR-NA-21 expression predicted recurrence and poor survival in patients with colorectal cancer – a meta-analysis. OTT. 2016; 2615.
- WorldMedical Association. World Medical Association Declaration of Helsinki: ethical principles for medical research involving human subjects. JAMA. 2013; 310(20): 2191-4. doi: 10.1001/jama.2013.281053.
- Sazanov A.A., Kiselyova E.V., Zakharenko A.A., Romanov M.N., Zaraysky M.I. Plasma and saliva miR-21 expression in colorectal cancer patients. J Appl Genet. 2017; 58(2): 231-7. doi: 10.1007/s13353-016-0379-9.
- Seliverstov R.Yu., Zaraiskiy M.I., Tyurin R.V., Naryshkin A.G., Valerko V.G., Semiglazov V.V., Takahachi Ch. Microrna in monitoring of the evolution of glial cerebral tumors. Sib J Onkol. 2020; 19(3): 47-53. doi: 10.21294/1814-4861-2020-19-3-47-53.
- Yuan Z., Liang X., Zhan Y., Wang Z., Xu J., Qiu Y., Wang J., Cao Y., Le V.M., Ly H.T., Xu J., Li W., Yin P., Xu K. Targeting CD133 reverses drug-resistance via the AKT/NF-kB/MDR1 pathway in colorectal cancer. Br J Cancer. 2020; 122(9): 1342-53. doi: 10.1038/s41416-020-0783-0.
- Ma Q., Wang X., Li Z., Li B., Ma F., Peng L., Zhang Y., Xu A., Jiang B. microRNA-16 represses colorectal cancer cell growth in vitro by regulating the p53/survivin signaling pathway. Oncol Rep. 2013; 29(4): 1652-8. doi: 10.3892/or.2013.2262.
- Wu Y., Song Y., Xiong Y., Wang X., Xu K., Han B., Bai Y., Li L., Zhang Y., Zhou L. MicroRNA-21 (Mir-21) Promotes Cell Growth and Invasion by Repressing Tumor Suppressor PTEN in Colorectal Cancer. Cell Physiol Biochem. 2017; 43(3): 945-58. doi: 10.1159/000481648.
- Tagscherer K.E., Fassl A., Sinkovic T., Richter J., Schecher S., Macher-Goeppinger S., Roth W. MicroRNA-210 induces apoptosis in colorectal cancer via induction of reactive oxygen. Cancer Cell Int. 2016; 16: 42. doi: 10.1186/s12935-016-0321-6.
- NijhuisA., Thompson H., Adam J., ParkerA., Gammon L., LewisA., Bundy J.G., Soga T., Jalaly A., Propper D., Jeffery R., Suraweera N., McDonald S., Thaha M.A., Feakins R., Lowe R., Bishop C.L., Silver A. Remodelling of microRNAs in colorectal cancer by hypoxia alters metabolism profiles and 5-fluorouracil resistance. Hum Mol Genet. 2017; 26(8): 1552-64. doi: 10.1093/hmg/ddx059.


