Связь микро-РНК кластера MIR-143/145 с онкогенезом: тканевой и клеточный контекст
Автор: Воропаева Е.Н., Поспелова Т.И., Нестерец А.М., Максимов В.Н.
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Обзоры
Статья в выпуске: 3 т.22, 2023 года.
Бесплатный доступ
Цель исследования - представить современные данные о регуляции экспрессии, функции в нормальных тканях и разнонаправленной активности в онкогенезе микроРНК кластера miR-143/145, а также оценить возможности и ограничения терапевтического использования микроРНК данного кластера при злокачественных новообразованиях. Материал и методы. Проведен поиск доступных отечественных и зарубежных литературных источников, опубликованных в базах данных PubMed и РИНЦ за последние 10 лет. Найдено 427 статей, из которых 41 была включена в данный обзор. Результаты. Консерва-тивный кластер miR-143/145 является одним из наиболее интенсивно изучаемых при опухолях. На основании результатов анализа дифференциальной экспрессии микроРНК, экспериментов in vitro в раковых клеточных линиях и in vivo в мышиных моделях опухолей было показано снижение уровня miR-143 и miR-145 при злокачественных новообразованиях эпителиального происхождения. До недавнего времени данные микроРНК считались классическими онкосупрессорами. Приведенные в обзоре данные демонстрируют, что результаты целого ряда работ, учитывающих клеточные аспекты экспрессии микроРНК, противоречат этой концепции. Для микроРНК miR-143, например, известно участие в метаболической перестройке опухоли и активации неоангиогенеза. Показано, что онкосупрессорная или проонкогенная активность miR-143 и miR-145 зависят от тканевого и клеточного контекста и могут объясняться наличием у них нескольких регулируемых мишеней, оказывающих противоположные эффекты на онкогенез. В совокупности полученные данные говорят о необходимости проявлять осторожность при выборе микроРНК описываемого кластера для экзогенной терапевтической доставки. Заключение. Дальнейшая детальная расшифровка механизмов функционирования miR-143 и miR-145 в различных типах тканей и клеток, а также идентификация новых мРНК-мишеней необходимы для лучшего понимания участия данных молекул в онкогенезе.
Онкогенез, микрорнк, mir-143, mir-145, экспрессия
Короткий адрес: https://sciup.org/140300174
IDR: 140300174 | УДК: 616-006+575.853+577.216 | DOI: 10.21294/1814-4861-2023-22-3-134-143
Текст научной статьи Связь микро-РНК кластера MIR-143/145 с онкогенезом: тканевой и клеточный контекст
МикроРНК представляют собой небольшие некодирующие РНК, которые регулируют экспрессию генов на посттранскрипционном уровне. Многие из них играют решающую роль в канцерогенезе и могут действовать как онкогены или супрессоры опухолевого роста [1]. У человека идентифицированы тысячи микроРНК, которые, как предполагается, регулируют, по меньшей мере, одну треть всех транскриптов мРНК. Каждая микроРНК нацеливается на более чем 200 транскриптов, в то же время каждую из мРНК-мишеней регулируют более одной микроРНК [2]. Таким образом, потенциальные регуляторные сети с участием микроРНК крайне сложны.
Общие данные о механизмах экспрессии микроРНК бицистронного кластера miR-143/145
Одним из наиболее интенсивно изучаемых при опухолях является кластер микроРНК miR-143/145. Он расположен в регионе q33 5-й хромосомы и образован двумя высококонсервативными, находящимися друг от друга на расстоянии 1,3-килобазы генами MIR-143 и MIR-145 , кодирующими различные по спектру регулируемых мишеней микроРНК [3].
В ходе биогенеза микроРНК МIR-143 и MIR-145 совместно транскрибируются в единый первичный транскрипт (рис. 1). Далее он подвергается процессингу DGCR8 и Drosha с образованием соответствующих пре-микроРНК. Экспортином пре-микроРНК доставляются в цитозоль клетки и расщепляются Dicer с образованием зрелых молекул miR-143 и miR-145. Последние включаются в РНК-индуцированный комплекс сайленсинга (RISC) и действуют каждая на свои мРНК-мишени.
Механизмы, участвующие в стадии транскрипции miR-143 и miR-145, в целом идентичны, поскольку их геномные локусы расположены на небольшом расстоянии друг от друга. Экспрессия всего кластера угнетается Ras-чувствительным элементсвязывающим белком 1 (RREB1) и EGFR, а активируется ТР53. Однако для гена MIR-145 описана отдельная промоторная область, регулирующаяся метилированием и репрессором транскрипции SNAI1 [4]. Это может быть возможной причиной несоответствия в характере экспрессии miR-145 и miR-143 в некоторых типах клеток [5].
Экспрессия miR-143 и miR-145 в норме
В настоящее время четко показано, что экспрессия большинства микроРНК тканеспецифична [7]. В норме уровень miR-143 колеблется от самой высокой в толстой кишке до самой низкой в головном мозге и печени [8]. Экспрессия miR-145 высокая в подавляющем большинстве изученных тканей [9]. Гемопоэтическая ткань обладает особенностями. Так, в костномозговых кроветворных клетках различных стадий дифференцировки miR-143 экспрессируется на высоком уровне, тогда как miR-145 обнаруживается лишь в небольших количествах [10].
Во всех ранних работах, изучающих роль кластера miR-143/145 в онкогенезе, для определения экспрессии miR-143 и miR-145 РНК выделяли из гетерогенных образцов опухоли [2]. В настоящее время стало понятно, что определяемая на тканевом уровне экспрессия зависит от уровня экспрессии микроРНК в составляющих ее клетках конкретного типа и их обилия в образце. На примере толстой кишки показаны значимые различия в уровне miR-143 в клетках различного типа, а именно: низкая экспрессия в эритроцитах и лимфоцитах,
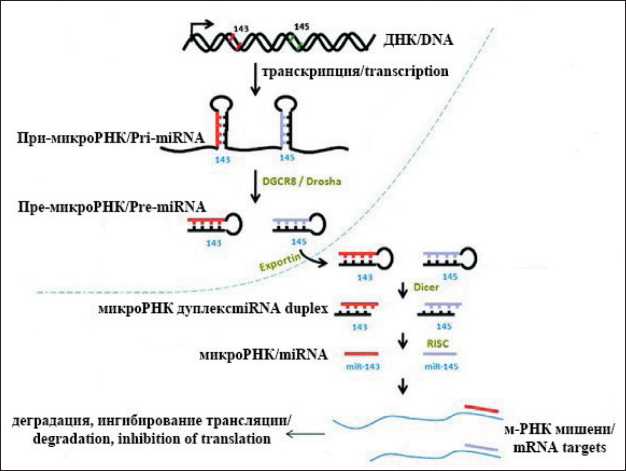
Рис. 1. Биогенез микроРНК кластера miR-143/145 (источник [6] с изменениями) Fig. 1. Biogenesis of the microRNA cluster miR-143/145 (Ref. [6] with changes)

лимфоцит/lymphocyte эндотелий/endothelium гладкая мускулатура/smooth muscle фибробласт/fibroblast эпителий/epithelial эритроцит/erythrocyte

очень высокая/ very high
отсутствует/ none
Рис. 2. Уровень экспрессии miR-143 и miR-145 в клетках различного типа (источник [2] c изменениями)
Fig. 2. Expression level of miR-143 and miR-145 in different cell types (Ref. [2] with changes)
умеренная в эндотелиальных и эпителиальных элементах и высокая в гладкомышечных клетках и фибробластах (рис. 2). Экспрессия miR-145 была низкой во всех перечисленных типах клеток, кроме фибробластов, где она имела умеренный уровень [1]. Авторы пришли к логичному выводу, что гомогенизированная ткань толстой кишки будет содержать все эти типы клеток в соотношениях, количество которых может сильно варьировать в образцах, что отразится на экспрессионных профилях.
Недавние же наблюдения свидетельствуют, что на уровень miR-143 и miR-145 влияет не только клеточный состав ткани, но даже степень дифференцировки клеточных элементов [1]. Предпринимались попытки идентифицировать микроРНК, которые важны для нормального протекания дифференцировки лимфоцитов. По меньшей мере, 4,5-кратную разницу в экспрессии между
В-клетками зародышевого центра и лимфоцитами с более поздним фенотипом показала miR-145. Ее уровень увеличивался в лимфоцитах от стадии наивных В-клеток герминального центра до активированных клеток и далее В-клеток памяти [11]. Повышение экспрессии miR-143 наблюдали при дифференцировке CD8+ Т-клеток, что сопровождалось также снижением их чувствительности к апоптозу и увеличением секреции провоспали-тельных цитокинов [12].
Из сказанного выше следует, что для адекватной оценки уровня экспрессии данных микроРНК необходимо не только учитывать клеточный состав анализируемой ткани, но и проводить сортировку клеток [13] или использовать методы микродиссекции, которые позволяют выделять отдельные клетки на основе морфологии или наличия/отсут-ствия специфических маркеров [14].
Разнонаправленные изменения экспрессии и эффекты miR-143 и miR-145 при опухолях
Выводы недавней работы на мышиной модели аденокарциномы легкого полностью противоречат данным, описанным выше [13]. Авторы наблюдали отсутствие предрасположенности мышей с нокаутом miR-143/145 к развитию опухоли, не отмечали влияния потери miR-143/145 на скорость пролиферации аденокарциномы, а также подавляющих опухоль эффектов сверхэкспрессии miR-143 и miR-145 в злокачественных клетках in vitro и in vivo . В другом исследовании с применением опухолевых клеточных линий, образцов пациентов и мышиных моделей изучалась роль miR-143 в развитии глиобластом. Показано, что относительная экспрессия данной микроРНК была выше у пациентов с глиобластомой по сравнению с контрольными образцами, а транзиторная трансфекция клеток ингибитором miR-143 приводила к снижению клеточной пролиферации, усилению апоптоза и остановке клеточного цикла. Более того, многократные инъекции мышам, несущим опухоль, липосомальной композиции ингибитора miR-143 значительно снижали рост новообразования по сравнению с контрольными животными. Повышенный же уровень miR-143 увеличивал инвазивную способность клеток глиобластомы [8]. В совокупности эти данные свидетельствуют о том, что онкосупрессорная или проонкогенная активности miR-143 и miR-145 зависят от тканевого контекста.
Насколько трудной может быть интерпретация роли микроРНК данного кластера в онкогенезе, свидетельствуют результаты работы C. Johannessen et al., в которой in vitro изучали три клеточные линии: (ER+), трижды-негативного (TN) и (HER2+) рака молочной железы. Интересно, что трансфекция miR-143 привела к усилению пролиферации клеточных линий ER+ и TN рака, но не вызывала каких-либо существенных изменений в пролиферации клеточной линии HER2+. В то же время все три изученные клеточные линии рака молочной железы продемонстрировали резкое снижение пролиферации при трансфекции miR-145. Пролиферативная активность клеток, ко-трансфицированных miR-143 и miR-145, была резко снижена во всех трех клеточных линиях. Вместе с тем, miR-143 и miR-145 ингибировали инвазию in vitro всех клеточных линий как при изолированной, так и при сочетанной трансфекции [3].
В последние два десятилетия проведено небольшое число исследований, целью которых было профилирование экспрессии микроРНК miR-143 и miR-145 при гемобластозах. Их результаты наиболее постоянны лишь для работ с использованием клеточных линий. Так, все изученные четыре клеточные линии лимфомы Беркитта (ЛБ) (Raji, Daudi, P32 и KHM-10B) [15], девять клеточных линий Т-ОЛЛ (ATN-1, HUT-102, MJ, TL-Om1, ATL-102, MT-1, MT-2, MT-4 и TL-SU) [16] и четыре клеточные линии ММ (U266, NCI-H929, RPMI-8226 и LP-1) [17] демонстрировали снижение экспрессии микроРНК описываемого кластера по сравнению с нормальными клетками. В экспериментах с клеточными линиями ЛБ и лейкемии [18] микроРНК данного кластера проявляли онкосупрессорные свойства.
Результаты же работ с использованием первичных образцов опухолей больных гемобластозами в меньшей степени согласованы (табл. 1). В отношении miR-143 описано снижение экспрессии при ЛБ [19] и острых лейкозах миелоидного [20, 21] и лимфоидного происхождения [22], а также отсутствие изменений при синдроме Сезари [23]. В отношении miR-145 описано снижение экспрессии при ЛБ [19], остром миелоидном лейкозе (ОМЛ) [20, 21] и множественной миеломе [17], тогда как при синдроме Сезари – повышение [23]. При Т-ОЛЛ снижение уровня miR-143 и miR-145 наблюдалось лишь у четверти пациентов и было тесно связано с ухудшением клинического течения заболевания [16].
Наибольшее внимание привлекают противоречивые результаты по изменению экспрессии микроРНК описываемого кластера при хроническом лимфолейкозе [15, 24], фолликулярной лимфоме (ФЛ) [25, 26] и диффузной В-крупноклеточной лимфоме (ДВККЛ) [15, 25–27], что может быть связано с небольшим числом исследованных образцов, различными методическими подходами и неоднородностью как контрольных образцов, выбираемых авторами для расчета дифференциальной экспрессии miR-143 и miR-145, так и самих опухолевых образцов в пределах одной нозологической формы.
Например, ДВККЛ является крайне гетерогенным заболеванием. Профилирование экспрессии генов в злокачественных лимфоцитах позволяет
Таблица 1/table 1
Дифференциальная экспрессия miR-143 и miR-145 в образцах гемопоэтических опухолей и клетках/тканях сравнения
differential expression of miR-143 and miR-145 in hematopoietic tumor samples and referencecells/tissues
|
Исследуемые образцы/ Studied samples |
Образцы сравнения/ Comparison samples |
Метод/ Method |
Изменение экспрессии/ |
Источник/ Reference |
||
|
Опухоль/ Tumor |
Кол-во образцов/ Number of samples |
Expression < miR-143 |
changes miR-145 |
|||
|
ЛУ ХЛЛ/ LN CLL |
13 |
CD19+ B-клетки миндалин, ПК/ CD19+ tonsil B-cells, PB |
Количественная ПЦР/ Q-PCR |
Экстремально низкая/ Extremely low |
Экстремально низкая/ Extremely low |
[15] |
|
ЛУ ХЛЛ/ LN CLL |
41 |
ЛУ, миндалины, ПК/ LN, tonsils, PB |
Микрочипы/ Microarray |
Не изменена/ Not changed |
Не изменена/ Not changed |
[24] |
|
ЛУ ЛБ/ LN BL |
46 |
Реактивные ЛУ/ Reactive LN |
Количественная ПЦР/ Q-PCR |
Экстремально низкая/ Extremely low |
Экстремально низкая/ Extremely low |
[19] |
|
КМ, ПК ОЛЛ/ BM, PB ALL |
11 |
ПК, КМ/ PB, BM |
Количественная ПЦР/ Q-PCR |
Значительно ниже/ Significantly lower |
– |
[22] |
|
КМ, ПК ОMЛ/ BM, PB AML |
– |
ПК, КМ/ PB, BM |
Количественная ПЦР/ Q-PCR |
Значительно снижена/ Significantly decreased |
Значительно снижена/ Significantly decreased |
[20, 21] |
|
Кожа синдром Сезари/ Skin Cesari syndrome |
11 |
Кожа/ Skin |
Количественная ПЦР/ Q-PCR |
Не изменена/ Not changed |
Повышена в 7,4 раза/ Increased by 7.4 times |
[23] |
|
ЛУ Т-ОЛЛ/ LN T-ALL |
40 |
CD4+ Т-клетки/ CD4+ Т-cells |
Количественная ПЦР/ Q-PCR |
– |
Снижена/ Decreased |
[16] |
|
ЛУ ФЛ/ LN FL |
18 |
Нормальные лимфоциты/ Normal lymphocytes |
Микрочипы/ Microarray |
Повышена в 14,2 раза/ Increased by 14.2 times |
Повышена в 11,4 раза/ Increased by 11.4 times |
[25] |
|
ЛУ ФЛ/ LN FL |
46 |
Неопухолевые ЛУ/ Non-tumor LN |
Количественная ПЦР/ Q-PCR |
– |
Не изменена/ Not changed |
[26] |
|
ММ/ MM |
30 |
Нормальные плазмоциты/ Normal plasmocytes |
Количественная ПЦР/ Q-PCR |
– |
Заметно ниже/ Markedly lower |
[17] |
|
ЛУ ДВККЛ/ DLBCL LN |
8 |
CD19+ B-клетки миндалин, ПК/ CD19+ tonsil B-cells, PB |
Количественная ПЦР/ Q-PCR |
Экстремально низкая/ Extremely low |
Экстремально низкая/ Extremely low |
[15] |
|
ЛУ ДВККЛ/ DLBCL LN |
98 |
В-клетки/ B-cells |
Микрочипы/ Microarray |
Повышена в 14,2 раза/ Increased by 14.2 times |
Повышена в 11,4 раза/ Increased by 11.4 times |
[25] |
|
ЛУ ДВККЛ/ DLBCL LN |
58 |
Неопухолевые ЛУ/ Non-tumor LN |
Количественная ПЦР/ Q-PCR |
– |
Значительно ниже/ Significantly lower |
[26] |
|
ПЛЦНС/ PCNSL |
11 |
ЛУ ДВККЛ/ DLBCL LN |
Количественная ПЦР/ Q-PCR |
– |
Снижена в 3,3 раза/ Decreased by 3.3 times |
[27] |
Примечание: ХЛЛ – хронический лимфоидный лейкоз; ЛБ – лимфома Беркитта; ОЛЛ – острый лимфобластный лейкоз; ОМЛ – острый миелобластный лейкоз; ФЛ – фолликулярная лимфома; ММ – множественная миелома; ДВККЛ – диффузная В-крупноклеточная лимфома; ПЛЦНС – первичная лимфома ЦНС; ЛУ – лимфоузлы; ПК – периферическая кровь; КМ – костный мозг.
Note: CLL – chronic lymphcytic leukemia; LB – Burkitt’s lymphoma; ALL – acute lymphoblastic leukemia; AML – acute myeloblastic leukemia; FL – follicular lymphoma; MM – multiple myeloma; DLBCL – diffuse large B-cell lymphoma; PLCNS – primary lymphoma of the central nervous system; LN – lymph nodes; PB – peripheral blood; BM – bone marrow; Q-PCR – quantitative PCR.
Таблица 2/table 2
Перечень избранных мишеней микроРНК кластера miR-143/145
list of selected miR-143/145 cluster microRNa targets
|
miR-143 |
miR-145 |
||
|
Oнкогены/ Oncogenes |
Онкосупрессоры/ Oncosuppressors |
Oнкогены/ Oncogenes |
Онкосупрессоры/ Oncosuppressors |
|
KLF5, KRAS, DNMT3A, AKT1, SERPINE1, BCL2 |
PUMA ITGA7 |
KRAS, KLF4/5, SOX2, CD-KN1A, STAT1, VEGFA, EGFR, |
|
|
MAPK7, BRAF, IGF1R, ABL2, CD44 |
ITM2B ELK-1 VASH1 |
MYC, CDK4/6, NRAS, SERPINE1, CD44, SOX9, SMAD3, |
|
TGFBR2
выделить, как минимум, два прогностически различных подтипа ДВККЛ: из клеток зародышевого центра и активированных В-клеток. К сожалению, в представленных в литературе работах при анализе результатов не выполнялась разбивка ДВККЛ на подтипы. Вместе с тем, описаны различия в экспрессии микроРНК при данных подтипах опухоли [28]. Более того, недавно высказано предположение, что описываемая ранее дифференциальная экспрессия miR-143 и miR-145 при солидных опухолях может быть не связана с трансформированными клетками как таковыми, а объясняется различиями в составе клеток микроокружения опухоли тестируемых образцов [13]. Важно отметить, что ДВККЛ характеризуется различным составом микроокружения опухоли, что также может вносить вклад в получаемые значения экспрессии микроРНК. Так, на основании профиля экспрессии генов клетками микроокружения ДВККЛ можно выделить подгруппы заболевания «стромальная-1» и «стромальная-2». Прогностически благоприятная подгруппа «стромальная-1» отражает отложение внеклеточного матрикса и гистиоцитарную инфильтрацию. Прогностически неблагоприятная «стромальная-2» – высокую плотность кровеносных сосудов опухоли [29].
Таким образом, можно говорить о разнонаправленных изменениях экспрессии miR-143 и miR-145 при злокачественных новообразованиях, в основе которой могут лежать не только различия в тканевом происхождении, но и неоднородность опухолей, включая состав микроокружения, в пределах одной нозологии.
Значимые для онкогенеза мишени микроРНК кластера miR-143/145
Проявление онкосупрессорных или проонко-генных активностей микроРНК кластера miR-143/145 при различных опухолях или их сочетание при одном и том же типе злокачественных новообразований может объясняться наличием у них нескольких регулируемых мишеней, которые оказывают противоположные эффекты на онкогенез [8]. Следует отметить, что данные микроРНК име- ют лишь несколько общих мишеней, в остальном они действуют независимо друг от друга [5].
В списке проверенных мишеней обеих микроРНК (табл. 2), по данным онлайн-базы данных miRTarBase, указаны проонкогены [30]. Однако, по меньшей мере, для miR-143 показано нацеливание на мРНК ряда онкосупрессоров, таких как PUMA (проапоптотический белок), ITM2B (р53-независимый активатор апоптоза), ELK-1 (транскрипционный фактор), ITGA7 (интегрин альфа 7, который приводит к подавлению роста опухоли и замедлению миграции), VASH1 (фактор, ингибирующий ангиогенез) [8].
В различных типах опухолей, включая рак молочной железы, толстой кишки и глиобластому, miR-143 была связана в опухолевых клетках с метаболизмом глюкозы и регуляцией гликолиза посредством нацеливания на гексокиназу-2 (НК2) [31, 32]. Как известно, метаболическая перестройка является отличительной чертой злокачественного роста.
В настоящее время известен вклад микросреды в стимулирование роста опухоли. Иммунные клетки, фибробласты, эндотелиальные клетки, мезенхимальные стволовые клетки, перициты и, в некоторых случаях, адипоциты относятся к числу клеток, которые окружают и взаимодействуют со злокачественными элементами, образуя поддерживающее микроокружение [33]. В этой связи имеются данные, что miR-143 и miR-145, экспрессирующиеся не в злокачественных клетках, а в стромальных элементах опухоли, играют роль в стимулировании ее развития посредством активации неоангиогенеза [31, 34]. Особенно ярко это показано на примере модельных животных с опухолями легких [13, 35]. Анализ гибридизации тканей с микроРНК in situ при немелкоклеточном раке легкого выявил значительно повышенную экспрессию miR-143 в опухолевых и прилегающих стромальных клетках по сравнению со здоровыми тканями [35].
Показано, что miR-143 нацеливается на мРНК вазогибина VASH1, который является биологически активным веществом, угнетающим ангиогенез путем подавления миграции и пролиферации эндотелиальных клеток и экспрессии ими факторов
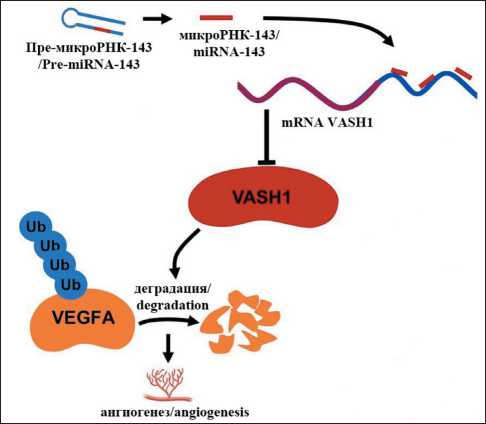
Рис. 3. Механизм активации miR-143 ангиогенеза (источник
[35] с изменениями)
Fig. 3. Mechanism of activation of miR-143 angiogenesis (Ref.
[35] with changes)
прорастания сосудов VEGFR-2 и VEGFA (рис. 3) [35]. Усложняют уже имеющуюся картину данные о том, что экзосомы, источником которых являются ассоциированные с раком фибробласты и в больших количествах содержащие miR-143, могут быть средством доставки данной микроРНК в клетки рака молочной железы и способствовать прогрессированию опухоли [36]. Наконец, недавние сообщения указывают на то, что кольцевые РНК действуют как губки микроРНК, которые изолируют miR-143 и miR-145, тем самым снижая их способность связываться с мРНК-мишенями [37, 38].
В заключение раздела следует сказать, что в настоящее время идентифицированы не все мишени микроРНК анализируемого кластера. Требуются дальнейшие усилия, направленные на предсказание in silico и проверку в эксперментах функционального значения нацеливания miR-143 и miR-145 на те или иные мРНК.
Ограничения терапевтического использования микроРНК кластера miR-143/miR-145
при злокачественных новообразованиях
Восстановление уровня подавленных или функционально дефицитных онкосупрессорных и/или ингибирование аберрантной продукции проонкогенных микроРНК могут способствовать восстановлению баланса экспрессии большого числа генов, вовлеченных в онкогенез. Одними из наиболее перспективных новых стратегий лечения злокачественных новообразований являются доставка в опухоль экзогенных микроРНК или их ингибиторов, что позволяет в первом случае функционально замещать эндогенные дерегулируемые микроРНК и восстанавливать функционирующие в норме сигнальные пути, во втором – понижать уровень эндогенной микроРНК и нарушать ее репрессирующие функции [1].
До недавнего времени считалось, что введение микроРНК кластера miR-143/145 в опухоль может оказаться эффективным терапевтическим подходом для лечения широкого спектра злокачественных новообразований. Так, данные функциональных исследований in vitro свидетельствуют о том, что эктопическая экспрессия miR-143 и miR-145 способствует апоптозу, ограничивает пролиферацию и миграционную способность клеток различных типов опухолей [5].
Терапевтический эффект доставки miR-143 или miR-145 был доказан в подкожных/ортото-пических моделях рака поджелудочной железы и ксенотрансплантатах колоректальной карциномы [39 ‒ 41]. В частности, эктопическая экспрессия miR-143 приводила к снижению пролиферации и усилению апоптоза опухолевых клеток колоректальной карциномы, а также снижению темпов роста опухолевого ксенотрансплантата [39].
Вместе с тем, в настоящее время считается, что перед терапевтическим применением экспрессия и эффекты микроРНК кластера miR-143/145 должны быть проанализированы при каждом типе злокачественных новообразований как в опухолевых клетках, так и в стромальных элементах. Следует также проверять потенциал коммуникации между опухолью и стромой через секрецию данных микроРНК в микроокружение [1].
Согласно современным представлениям, для клинического применения доставка miR-143 и miR-145 должна обладать высокой специфичностью путем прямого нацеливания на злокачественные клетки и избегания воздействия микроРНК на микроокружение опухоли [5]. Отметим, что такие подходы активно разрабатываются. В частности, тестируются способы адресной доставки искусственно синтезированных ингибиторов или аналогов микроРНК с помощью липосом, поверхность которых покрывают специфичными для тканей и клеток лигандами [42].
Таким образом, сведения об активации неоангиогенеза и метаболической перестройке опухоли под действием miR-143 говорят о необходимости проявлять осторожность при выборе микроРНК кластера miR-143/145 для экзогенной терапевтической доставки [13].
Заключение
Приведенные в обзоре данные демонстрируют, что результаты целого ряда исследований, учитывающих клеточный контекст изучаемых тканей, противоречат концепции, согласно которой микроРНК кластера miR-143/145 являются классическими супрессорами опухолей. Показано, что микроРНК miR-143 и miR-145 играют решающую роль в функционировании стромальных элементов органов и тканей, понимание которой имеет важное значение для разработки будущих методов лечения злокачественных новообразований, а эффекты микроРНК, установленные в экспериментах in vitro или в моделях с животными с нокаутом
Список литературы Связь микро-РНК кластера MIR-143/145 с онкогенезом: тканевой и клеточный контекст
- Almeida M.I., Calin G.A. The miR-143/miR-145 cluster and the tumor microenvironment: unexpected roles. Genome Med. 2016; 8(1): 29. https://doi.org/10.1186/s13073-016-0284-1.
- Kent O.A., McCall M.N., Cornish T.C., Halushka M.K. Lessons from miR-143/145: the importance of cell-type localization of miRNAs. Nucleic Acids Research. 2014; 42(12): 7528-38. https://doi.org/10.1093/nar/gku461.
- Johannessen C., Moi L., Kiselev Y., Pedersen M.I., Dalen S.M., Braaten T., Busund L.T. Expression and function of the miR-143/145 cluster in vitro and in vivo in human breast cancer. PLoS One. 2017; 12(10). https://doi.org/10.1371/journal.pone.0186658.
- PMikova P., Reis R., Herichova I. miRNA Clusters with Down-Regulated Expression in Human Colorectal Cancer and Their Regulation. Int J Mol Sci. 2020; 21(13): 4633. https://doi.org/10.3390/ijms21134633.
- Das A.V., Pillai R.M. Implications of miR cluster 143/145 as universal anti-oncomiRs and their dysregulation during tumorigenesis. Cancer Cell Int. 2015; 15: 92. https://doi.org/10.1186/s12935-015-0247-4.
- Rangrez A.Y., Massy Z.A., Metzinger-Le Meuth V., Metzinger L. miR-143 and miR-145: molecular keys to switch the phenotype of vascular smooth muscle cells. Circ Cardiovasc Genet. 2011; 4(2): 197-205. https://doi.org/10.1161/CIRCGENETICS.110.958702.
- Mishra S., Yadav T., Rani V. Exploring miRNA based approaches in cancer diagnostics and therapeutics. Crit Rev Oncol Hematol. 2016; 98: 12-23. https://doi.org/10.1016/j.critrevonc.2015.10.003.
- Lozada-Delgado E.L., Grafals-Ruiz N., Miranda-Roman M.A., Santana-Rivera Y., Valiyeva F, Rivera-Diaz M., Marcos-Martinez MJ., Rivas-Mejia P.E. Targeting MicroRNA-143 Leads to Inhibition of Glioblastoma Tumor Progression. Cancers (Basel). 2018; 10(10): 382. https://doi.org/10.3390/cancers10100382.
- MIR145 Gene - MicroRNA 145 [Internet]. [cited 2023 Apr 01]. URL: https://www.genecards.org/cgi-bin/carddisp.pl?gene=MIR145.
- Voropaeva E.N., Pospelova T.I., Berezina O.V., Churkina M.I., Gurazheva A.A., Maksimov V.N. Metilirovanie genov r53-responzivnykh onkosupressornykh mikroRNK pri gemoblastozakh. Sibirskii onkologicheskii zhurnal. 2022; 21(2): 130-42. https://doi.org/10.21294/1814-4861-2022-21-2-130-142.
- Tan L.P., Wang M., Robertus J.L., Schakel R.N., Gibcus J.H., Diepstra A., Harms G., Peh S.C., Reijmers R.M., Pals S.T., Kroesen B.J., Kluin P.M., Poppema S., van den Berg A. miRNA profiling of B-cell subsets: specific miRNA profile for germinal center B cells with variation between centroblasts and centrocytes. Lab Invest. 2009; 89(6): 708-16. https://doi.org/10.1038/labinvest.2009.26.
- Zhang T., Zhang Z., Li F., Ping Y., Qin G., Zhang C., Zhang Y. miR-143 Regulates Memory T Cell Differentiation by Reprogramming T Cell Metabolism. J Immunol. 2018; 201(7): 2165-75. https://doi.org/10.4049/jimmunol.1800230. Erratum in: J Immunol. 2022; 208(5): 1307-9.
- Dimitrova N., Gocheva V., Bhutkar A., Resnick R., Jong R.M., Miller K.M., Bendor J., Jacks T. Stromal Expression of miR-143/145 Promotes Neoangiogenesis in Lung Cancer Development. Cancer Discov. 2016; 6(2): 188-201. https://doi.org/10.1158/2159-8290.CD-15-0854.
- Lugli G., Kataria Y., Richards Z., Gann P., Zhou X., Nonn L. Lasercapture Microdissection of Human Prostatic Epithelium for RNA Analysis. J Vis Exp. 2015; (105): 53405. https://doi.org/10.3791/53405.
- Akao Y., Nakagawa Y., Kitade Y., Kinoshita T., Naoe T. Downregulation of microRNAs-143 and -145 in B-cell malignancies. Cancer Science. 2007; 98(12): 1914-20. https://doi.org/10.1111/j.1349-7006.2007.00618.x.
- Xia H., Yamada S., Aoyama M., Sato F., Masaki A., Ge Y., Ri M., Ishida T., Ueda R., Utsunomiya A., Asai K., Inagaki H. Prognostic impact of microRNA-145 down-regulation in adult T-cell leukemia/lymphoma. Hum Pathol. 2014; 45(6): 1192-8. https://doi.org/10.1016/j.humpath.2014.01.017.
- Wu H., Liu C., Yang Q., Xin C., Du J., Sun F., Zhou L. MIR145-3p promotes autophagy and enhances bortezomib sensitivity in multiple myeloma by targeting HDAC4. Autophagy. 2020; 16(4): 683-97. https://doi.org/10.1080/15548627.2019.1635380.
- Ferreira A.C., Robaina M.C., Rezende L.M., Severino P., Klumb C.E. Histone deacetylase inhibitor prevents cell growth in Burkitt's lymphoma by regulating PI3K/Akt pathways and leads to upregulation of miR-143, miR-145, and miR-101. Ann Hematol. 2014; 93(6): 983-93. https://doi.org/10.1007/s00277-014-2021-4.
- Ferreira A.C., Robaina M.C., Rezende L.M., Severino P., Klumb C.E. Histone deacetylase inhibitor prevents cell growth in Burkitt's lymphoma by regulating PI3K/Akt pathways and leads to upregulation of miR-143, miR-145, and miR-101. Ann Hematol. 2014; 93(6): 983-93. https://doi.org/10.1007/s00277-014-2021-4.
- Shi Q., Xing G., Qi M., Xing Y. Lower Serum miR-145 Predicts Poor Prognosis in Patients with Acute Myeloid Leukemia. Clin Lab. 2020; 66(6). https://doi.org/10.7754/Clin.Lab.2019.191143.
- Huang J.L., Fang J.P., Shen J.Z. [The Effect of MicroRNA-143 on the Proliferation and Apoptosis of Leukemia Cell Line U-937]. Zhong-guo Shi Yan Xue Ye Xue Za Zhi. 2021; 29(6): 1695-703. Chinese. https://doi.org/10.19746/j.cnki.issn.1009-2137.2021.06.001.
- Dou L., Zheng D., Li J., Li Y., Gao L., Wang L., Yu L. Methylation-mediated repression of microRNA-143 enhances MLL-AF4 oncogene expression. Oncogene. 2012; 31(4): 507-17. https://doi.org/10.1038/onc.2011.248.
- Ballabio E., Mitchell T., van Kester M.S., Taylor S., Dunlop H.M., Chi J., Tosi I., Vermeer M.H., Tramonti D., Saunders N.J., Boultwood J., Wainscoat J.S., Pezzella F., Whittaker S.J., Tensen C.P., Hatton C.S., Lawrie C.H. MicroRNA expression in Sezary syndrome: identification, function, and diagnostic potential. Blood. 2010; 116(7): 1105-13. https://doi.org/10.1182/blood-2009-12-256719.
- Calin G.A., Liu C.G., Sevignani C., Ferracin M., Felli N., Dumit-ru C.D., Shimizu M., Cimmino A., Zupo S., Dono M., Dell'Aquila M.L., Alder H., Rassenti L., Kipps T.J., Bullrich F., Negrini M., Croce C.M. MicroRNA profiling reveals distinct signatures in B cell chronic lymphocytic leukemias. Proc Natl Acad Sci USA. 2004; 101(32): 11755-60. https://doi.org/10.1073/pnas.0404432101.
- Lawrie C.H., Chi J., Taylor S., Tramonti D., Ballabio E., Palazzo S., Saunders N.J., Pezzella F., Boultwood J., Wainscoat J.S., Hatton C.S. Expression of microRNAs in diffuse large B cell lymphoma is associated with immunophenotype, survival and transformation from follicular lymphoma. J Cell Mol Med. 2009; 13(7): 1248-60. https://doi.org/10.1111/j.1582-4934.2008.00628.x.
- Roehle A., Hoefig K.P., Repsilber D., Thorns C., Ziepert M., Wesche K.O., Thiere M., Loeffler M., Klapper W., Pfreundschuh M., Matolcsy A., Bernd H.W., Reiniger L., Merz H., Feller A.C. MicroRNA signatures characterize diffuse large B-cell lymphomas and follicular lymphomas. Br J Haematol. 2008; 142(5): 732-44. https://doi.org/10.1111/j.1365-2141.2008.07237.x.
- Fischer L., Hummel M., Korfel A., Lenze D., Joehrens K., Thiel E. Differential micro-RNA expression in primary CNS and nodal diffuse large B-cell lymphomas. Neuro Oncol. 2011; 13(10): 1090-8. https://doi.org/10.1093/neuonc/nor107.
- Yang J.M., Jang J.Y., Jeon Y.K., Paik J.H. Clinicopathologic implication of microRNA-197 in diffuse large B cell lymphoma. J Transl Med. 2018; 16(1): 162. https://doi.org/10.1186/s12967-018-1537-0.
- Lenz G., Wright G., Dave S.S., Xiao W., Powell J., Zhao H., Xu W., Tan B., Goldschmidt N., Iqbal J., Vose J., Bast M., Fu K., Weisenburger D.D., Greiner T. C., Armitage J.O., Kyle A., May L., Gascoyne R.D., Connors JM., Troen G., Holte H., Kvaloy S., Dierickx D., Verhoef G., Delabie J., Sme-land E.B., Jares P., Martinez A., Lopez-Guillermo A., Montserrat E., Campo E., Braziel R.M., Miller T.P., Rimsza L.M., Cook J.R., Pohlman B., Sweetenham J., Tubbs R.R., Fisher R.I., Hartmann E., Rosenwald A., Ott G., Muller-Hermelink H.K., Wrench D., Lister T.A., Jaffe E.S., Wilson W.H., Chan W.C., Staudt L.M.; Lymphoma/Leukemia Molecular Profiling Project. Stromal gene signatures in large-B-cell lymphomas. N Engl J Med. 2008; 359(22): 2313-23. https://doi.org/10.1056/NEJMoa0802885.
- Chou C.H., Chang N.W., Shrestha S., Hsu S.D., Lin Y.L., Lee W.H., Yang C.D., Hong H.C., Wei T.Y., Tu S.J., Tsai T.R., Ho S.Y., Jian T.Y., Wu H.Y., Chen P.R., Lin N.C., Huang H.T., Yang T.L., Pai C.Y., Tai C.S., Chen W.L., Huang C.Y., Liu C.C., Weng S.L., Liao K.W., Hsu W.L., Huang H.D. miRTarBase 2016: updates to the experimentally validated miRNA-target interactions database. Nucleic Acids Res. 2016; 44(1): 239-47. https://doi.org/10.1093/nar/gkv1258.
- Peschiaroli A., Giacobbe A., Formosa A., Markert E.K., Bongiorno-Borbone L., Levine AJ., Candi E., D'Alessandro A., Zolla L., Finazzi Agro A., Melino G. miR-143 regulates hexokinase 2 expression in cancer cells. Oncogene. 2013; 32(6): 797-802. https://doi.org/10.1038/onc.2012.100.
- Zhao S., Liu H., Liu Y., Wu J., Wang C., Hou X., Chen X., Yang G., Zhao L., Che H., Bi Y., Wang H., Peng F., Ai J. miR-143 inhibits glycolysis and depletes stemness of glioblastoma stem-like cells. Cancer Lett. 2013; 333(2): 253-60. https://doi.org/10.1016/j.canlet.2013.01.039.
- Berindan-Neagoe I., Calin G.A. Molecular pathways: microRNAs, cancer cells, and microenvironment. Clin Cancer Res. 2014; 20(24): 6247-53. https://doi.org/10.1158/1078-0432.CCR-13-2500.
- Wang R., Zhang H., Ding W., Fan Z., Ji B., Ding C., Ji F., Tang H. MiR-143 promotes angiogenesis and osteoblast differentiation by targeting HDAC7. Cell Death Dis. 2020; 11: 179. https://doi.org/10.1038/s41419-020-2377-4.
- Wang H., Deng Q., Lv Z., Ling Y., Hou X., Chen Z., Dinglin X., Ma S., Li D., Wu Y., Peng Y., Huang H., Chen L. N6-methyladenosine induced miR-143-3p promotes the brain metastasis of lung cancer via regulation of VASH1. Mol Cancer. 2019; 18(1): 181. https://doi.org/10.1186/s12943-019-1108-x.
- Donnarumma E., Fiore D., Nappa M., Roscigno G., Adamo A., Iaboni M., Russo V., Affinito A., Puoti I., Quintavalle C., Rienzo A., Pis-cuoglio S., Thomas R., Condorelli G. Cancer-associated fibroblasts release exosomal microRNAs that dictate an aggressive phenotype in breast cancer. Oncotarget. 2017; 8(12): 19592-608. https://doi.org/10.18632/oncotarget.14752.
- Huang C.S., Tsai C.H., Yu C.P., Wu Y.S., Yee M.F., Ho J.Y., Yu D.S. Long Noncoding RNA LINC02470 Sponges MicroRNA-143-3p and Enhances SMAD3-Mediated Epithelial-to-Mesenchymal Transition to Promote the Aggressive Properties of Bladder Cancer. Cancers (Basel). 2022; 14(4): 968. https://doi.org/10.3390/cancers14040968.
- Wu Z., Huang W., Wang X., Wang T., Chen Y., Chen B., Liu R., Bai P., Xing J. Circular RNA CEP128 acts as a sponge of miR-145-5p in promoting the bladder cancer progression via regulating SOX11. Mol Med. 2018; 24(1): 40. https://doi.org/10.1186/s10020-018-0039-0.
- BorralhoP.M., SimdesA.E., Gomes S.E., LimaR.T., Carvalho T., Ferreira DM., Vasconcelos M.H., Castro R.E., Rodrigues CM. miR-143 overexpression impairs growth of human colon carcinoma xenografts in mice with induction of apoptosis and inhibition of proliferation. PLoS One. 2011; 6(8). https://doi.org/10.1371/journal.pone.0023787.
- Ibrahim A.F., Weirauch U., Thomas M., Grunweller A., Hartmann R.K., Aigner A. MicroRNA replacement therapy for miR-145 and miR-33a is efficacious in a model of colon carcinoma. Cancer Res. 2011; 71(15): 5214-24. https://doi.org/10.1158/0008-5472.CAN-10-4645.
- Pramanik D., Campbell N.R., Karikari C., Chivukula R., Kent O.A., Mendell J.T., Maitra A. Restitution of tumor suppressor microRNAs using a systemic nanovector inhibits pancreatic cancer growth in mice. Mol Cancer Ther. 2011; 10(8): 1470-80. https://doi.org/10.1158/1535-7163.MCT-11-0152.
- Ivkin D.Yu., Lisitskii D.S., Zakharov E.A., Lyubishin M.M., Karpov A.A., Burkova N.V., Okovityi S.V., Tyukavin A.I. MikroRNK kak perspektivnye diagnosticheskie i farmakologicheskie agenty. Astrakhanskii meditsinskii zhurnal. 2015; 4: 8-25.


