Таксономические и функциональные особенности микробиоты рубца у дойных коров с диагнозом кетоз
Автор: Лаптев Г.Ю., Йылдырым Е.А., Дуняшев Т.П., Ильина Л.А., Тюрина Д.Г., Филиппова В.А., Бражник Е.А., Тарлавин Н.В., Дубровин А.В., Новикова Н.И.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Молочная продуктивность
Статья в выпуске: 2 т.56, 2021 года.
Бесплатный доступ
В последние годы в животноводческих хозяйствах Российской Федерации резкое увеличения уровня молочной продуктивности у крупного рогатого скота за счет введения в рацион высоких количеств крахмала при одновременном снижении доли некрахмалистых полисахаридов отрицательно сказалось на многих функциях животных, с чем связан, в частности, рост распространенности метаболических заболеваний. Одно из подобных расстройств - кетоз, заболевание, которое отмечается у 23-80 % высокопродуктивных коров в послеотельный период. Отрицательный энергетический баланс у дойных коров в период лактации нарушает координацию липидного обмена между жировой тканью, печенью, кишечником и молочными железами, что приводит к кетозу. Микробное сообщество рубца играет значительную роль в энергетическом гомеостазе хозяина, его метаболической и физиологической адаптации к периодам недостатка энергии. Однако исследования, касающиеся состава микробного сообщества и реализуемых микроорганизмами метаболических путей при кетозе молочных коров, немногочисленны. В настоящем сообщении мы описали изменения микробного пейзажа и реализуемых микроорганизмами метаболических путей в рубце молочных коров при кетозе. Целью работы было сравнение состава и метаболического потенциала рубцовой микробиоты у двух дойных коров черно-пестрой голштинизированной породы 3-й лактации (клинически здоровой и кетозной) с применением NGS-секвенирования и количественной ПЦР с обратной транскрипцией. Была также проведена биоинформатическая реконструкция с прогнозированием функционального потенциала метагеномного сообщества. В микробиоте рубце коровы с кетозом повышалась доля таксонов, ассоциированных с нарушением энергетического баланса, в частности связанном с глюконеогенезом, повышением синтеза лактата, усилением патогенеза. Так, в 1,2 раза (при р £ 0,05) снижалась представленность бактерий филума Bacteroidetes , включавшего роды, связанные с образованием пропионата и сукцината - главных источников глюкозы для глюконеогенеза и высокоценных для коров янтарной и уксусной кислот. В рубце кетозного животного мы обнаружили повышение (р ≤ 0,05) доли родов Odoribacter и Frischella , среди которых встречаются патогены, а также представленности патогенного вида Bacteroides fragilis . У коровы, больной кетозом, произошло полное исчезновение из состава микробиоты рубца неатрибутируемых бактерий из рода Gp15. Выявлена достоверная разница (р £ 0,001) в экспрессии гена ldh-L L-лактатдегидрогеназы между животными: уровень экспрессии генов, ассоциированных с синтезом этого фермента, у коровы с кетозом был в 128±17,9 раза выше, чем у клинически здорового животного. С применением программного комплекса PICRUSt2 и MetaCyс было показано, что у коровы с диагнозом кетоз предсказанный потенциал функциональной активности микробиоты усилен в отношении 12 метаболических путей (р £ 0,05). В частности, повышен прогнозируемый метаболический потенциал, связанный с осуществлением гетероферментативного молочнокислого брожения, усилением деградации гекситолов, синтезом О-антигенов, используемых патогенными формами для избегания фагоцитоза и противостояния литическому действию системы комплемента.
Кетоз, микробиом рубца, жвачные, ngs-секвенирование, picrust2, metacyс, экспрессия генов, метаболические пути
Короткий адрес: https://sciup.org/142229482
IDR: 142229482 | УДК: 636.2:577.121:579.6:577.2 | DOI: 10.15389/agrobiology.2021.2.356rus
Текст научной статьи Таксономические и функциональные особенности микробиоты рубца у дойных коров с диагнозом кетоз
Объемистые корма в рационе крупного рогатого скота оказывают стимулирующее воздействие на работу рубца, улучшается состав рубцовой микрофлоры, процессы пищеварения, состояние здоровья животных. Тем не менее для таких кормов по сравнению с концентрированными характерно относительно низкое количество обменной энергии (ОЭ), имеющей принципиальное значение в кормлении животных. Потребление объемистых кормов ведет к снижению молочной продуктивности, раннему наступлению пика лактации и быстрому спаду лактационной кривой, что вынуждает компенсировать недостаток ОЭ в рационе с помощью дополнительного
∗ Исследование выполнено при поддержке гранта РФФИ ¹ 20-016-00168 «Исследование особенностей экспрессии генов метаболизма микробного сообщества рубца крупного рогатого скота под влиянием различных кормовых факторов».
количества концентратов. В последние годы в животноводческих хозяйствах Российской Федерации резкое увеличения уровня молочной продуктивности у крупного рогатого скота за счет использования рационов с высоким содержанием крахмала при одновременном снижении доли некрахмалистых полисахаридов отрицательно сказалось на многих физиологических функциях животных, что привело, в частности, к росту распространенности метаболических заболеваний (1, 2). Исследования ряда авторов (3-5) содержат информацию о том, что одно из подобных расстройств — кетоз, который отмечают у 23-80 % высокопродуктивных коров в послеотельный период. Кетоз характеризуется нарушением углеводно-жирового обмена и сопровождается расстройством пищеварения, гипогликемией, кетонемией, кетонурией, кетолактией и, как следствие, поражением гипофизарно-надпочечниковой системы, щитовидной, паращитовидной желез, печени, сердца, почек и других органов (6), что приводит с снижению молочной продуктивности в среднем на 10-15 % и сокращает продуктивное долголетие.
Обеспечение высокопродуктивных молочных коров питательными веществами, особенно в период лактации, требует специального внимания. Отрицательный энергетический баланс (превышение расхода энергии на выработку молока над ее поступлением с кормом) в этот физиологический период вызывает нарушение координации липидного обмена между жировой тканью, печенью, кишечником и молочными железами, что приводит к кетозу (7, 8). Таким образом, концентрация глюкозы в крови жвачных играет ключевую роль в поддержании энергетического метаболизма и профилактики болезней обмена веществ, прежде всего кетоза.
У жвачных животных приблизительно 90 % глюкозы образуется вследствие глюконеогенеза, который происходит главным образом в печени, в меньшей степени — в корковом слое почек и в эпителиальных клетках, выстилающих тонкую кишку. При этом важнейшие предшественники в глюконеогенезе — трехуглеродные соединения, в частности пропионат, пируват и лактат, которые образуются исключительно в результате деятельности рубцовой микрофлоры (9).
Некоторые исследования (10, 11) демонстрируют, что микробное сообщество пищеварительной системы играет значительную роль в энергетическом гомеостазе хозяина, его метаболической и физиологической адаптации к периодам недостатка энергии. Важно, что энергетический метаболизм жвачных отличается от такового у моногастрических животных: энергия поступает в основном за счет микробного брожения в рубце (12). У здоровых животных под действием ферментов микроорганизмов целлюлоза, которой богаты корма жвачных, гидролизуется в рубце до целлобиозы, затем до глюкозы и далее до лактата (13, 14). Наконец, в результате ряда ферментативных реакций, осуществляемых преимущественно бактериями порядка Selenomonadales , из лактата образуются летучие жирные кислоты (ЛЖК) (15). Количественное соотношение молочной, уксусной, пропионовой и масляной кислот, образующихся в рубце, зависит от состава микробиома рубца (16, 17). ЛЖК, поглощенные в рубце, транспортируются в печень и включаются в глюконеогенез, что обеспечивает животное глюкозой.
При филогенетическом анализе микробных сообществ посредством реконструкции функционального потенциала наблюдаемого микробиомного профиля на основе данных 16S-метагеномики могут использоваться программные комплексы для биоинформатических исследований (18). При этом функции генов достаточно точно воспроизводятся на основе данных метатаксономического изучения последовательностей ДНК (19).
У молочных коров в период стельности и ранней лактации, когда риск возникновения кетоза наиболее велик, выявлены значительные изменения в составе микробиоты рубца (20). Также показано, что численность ряда представителей микробного сообщества рубца коррелировала с концентрацией кетоновых тел в биологических жидкостях коров (21). В целом следует отметить, что состав микробиоты рубца изучается достаточно подробно. В то же время исследования, касающиеся структуры микробного сообщества и реализуемых микроорганизмами метаболических путей при кетозе молочных коров, немногочисленны (22).
В представляемой работы мы показали, что у молочных коров с диагнозом кетоз в рубце сдвиги в численности рубцовой микробиоты связаны с изменениями ее функциональной активности и экспрессии генов синтеза L-лактатдегидрогеназы.
Нашей целью было сравнение состава и метаболического потенциала микробиома рубца клинически здоровой коровы и животного с диагнозом кетоз.
Методика . Эксперимент проводили в зимний период в СПК «Коб-раловский» (Гатчинский р-н, Ленинградская обл., 2020 год) на двух дойных коровах (¹ 1 и ¹ 2) черно-пестрой голштинизированной породы 3-й лактации с молочной продуктивностью соответственно 11310 и 9800 л/год. На основании клинической картины (вялость, пониженная эластичность кожи, взъерошенность шерстного покрова, плотность фекалий, редкая дефекация) животному ¹ 2 был поставлен диагноз кетоз.
Животные находились в одинаковых условиях кормления на привязном содержании. Рацион коров включал 26 кг силоса, 1,3 кг сена, 2 кг жома свекловичного сухого, 2 кг дробины пивной сухой, 3,5 кг кукурузы дробленой, 1,5 кг кукурузы экструдированной, 3 кг ячменя плющеного, 0,8 кг отрубей пшеничных, 0,25 кг дрожжей кормовых, 5 кг шрота рапсового, 0,5 кг шрота соевого, 110 г мела, 250 г минеральных добавок, 100 г соли. Содержание в рационе энергетических кормовых единиц составляло 10,7 мДж в 1 кг сухого вещества.
Пробы содержимого рубца (10-50 г, в 3 повторностях от каждой коровы) отбирали с соблюдением требований асептики вручную с использованием стерильного зонда.
Содержание кетоновых тел в молоке определяли в соответствии с описанием (23).
Тотальную ДНК из исследуемых образцов выделяли с использованием набора Genomic DNA Purification Kit («Fermentas, Inc.», Литва) согласно прилагаемой инструкции.
Состав бактериального сообщества рубца анализировали методом NGS-секвенирования на платформе MiSeq («Illiumina, Inc.», США) с праймерами для V3-V4 региона гена 16S рРНК: прямой праймер — 5 ′ -TCGTC-GGCAGCGTCAGATGTGTATAAGAGACAGCCTACGGGNGGCWGCAG-3 ′ , обратный — 5 ′ -GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGGA-CTACHVGGGTATCTAATCC-3 ′ .
Секвенирование проводили с применением следующих реагентов: для приготовления библиотек — Nextera® XT Index Kit («Illiumina, Inc.», США), для очистки ПЦР-продуктов — Agencourt AMPure XP («Beckman Coulter, Inc.», США), для секвенирования — MiSeq® ReagentKit v2 (500 cycle) («Illiumina, Inc.», США). Максимальная длина полученных последовательностей составила 2½250 п.н.
Биоинформатический анализ данных выполняли в программном обеспечении Qiime2 version 2019.10 . После первоначального импорта последовательностей в формат Qiime2 выравнивали парные строки прочтений. Далее последовательности фильтровали по качеству с использованием параметров настроек по умолчанию. Фильтрацию шумовых последовательностей проводили с помощью метода Deblur, при этом использовали максимальную длину последовательности обрезки, равную 250 п.н. . В результате для образцов ¹ 1 и ¹ 2 получили соответственно 1527 и 5040 последовательностей. Для реконструкции филогении de novo применяли программный пакет MAFFT , далее проводили маскированное выравнивание последовательностей. Для анализа таксономии использовали справочную базу данных GreenGenes 13.5 99 % .
На основании полученной таблицы ОТЕ (оперативные таксономические единицы) с использованием плагинов программного пакета QIIME 1.9.1 рассчитали индексы альфа-разнообразия и построили график зависимости числа ОТЕ от числа прочтений. При статистическом анализе индексов разнообразия не проводили их дополнительного преобразования. Рассчитывали следующие индексы биоразнообразия микробиоты рубца: Сhao1, индекс Шеннона (Shannon index H) и индекс Сим-пона (Simpson index D) (24).
Реконструкцию и прогнозирование функциональной активности метагенома, семейств генов, ферментов проводили при помощи программного комплекса PICRUSt2 (v.2.3.0) (18). Работу с программой выполняли согласно рекомендованному сценарию, все настройки использовали по умолчанию. Индекс NSTI (nearest-sequenced taxon index) ближайшего по порядку таксона для обоих образцов составлял 0,3, что позволяет использовать эти данные для предсказания метаболических путей. Для анализа метаболических путей и ферментов пользовались базой данных MetaCyc . Оценку профилей метаболических путей MetaCyc проводили после нормализации обилия ASV (Amplicon Sequence Variants) логарифмом по основанию 2 (18).
Тотальную РНК из образцов рубцового содержимого выделяли с помощью набора Aurum Total RNA («Bio-Rad», США) согласно инструкции производителя.
При помощи набора iScript RT Supermix («Bio-Rad», США) осуществляли реакцию обратной транскрипции для получения кДНК на матрице РНК (25). Уровень относительной экспрессии генов ldb0101 и ldh-L у бактерий, способных осуществлять молочнокислое брожение, оценивали в количественной ПЦР (qPCR). Реакцию амплификации с праймерами для гена ldb0101 , связанного с синтезом D-лактатдегидрогеназы (F — 5 ′ -GCG-GGATCCGATGACTAAAATTTTTGCT-3 ′ и R — 5 ′ -GCGTGTCGACTTAGC-CAACCTTAACTGG-3 ′ ), и гена ldh-L L-лактатдегидрогеназы (F — 5 ′ -CAT-CAAAAAGTTGTGTTAGTCGGCG-3 ′ и R — 5 ′ -TCAGCTAAACCGTCGTTA-AGCACTT-3 ′ ) проводили с использованием набора SsoAdvanced™ Universal SYBR® Green Supermix («Bio-Rad», США) согласно протоколу производителя. Условия амплификации: 1 мин при 95 ° С (1 цикл); 15 с при 95 ° С, 1 мин при 50 ° С (45 циклов).
Относительную экспрессию рассчитывали методом 2 - AA Ct (26). В качестве референсного контроля использовали праймеры к универсальному гену, кодирующему рибосомную 16S-субъединицу прокариот (F — 5 ′ -AGG-CCTTCGGGTTGTAAAGT-3 ′ , R — 5 ′ -CGGGGATTTCACATCTCACT-3 ′ ).
Математическую и статистическую обработку результатов выполняли с применением программных пакетов Microsoft Office Excel 2003 и R-Studio (Version 1.1.453) . Различия считали статистически значимыми при p < 0,05 при использовании t-критерия Стьюдента.
Ðåçóëüòàòû . Проведенный анализ содержания кетоновых тел в молоке выявил значительное превышение рефересных значений и подтвердил диагноз кетоз у одной из дойных коров (¹ 2 — 0,9±0,04 г/л при референсных значениях 0,06-0,08 г/л), поставленный на основании наблюдаемой клинической картины. При этом в молоке клинически здоровой коровы мы не обнаружили кетоновых тел. Кетоз крупного рогатого скота характеризуется накоплением кетоновых тел, главным образом бета-гидроксибутирата, ацетоацетата и ацетона, которые могут быть измерены в жидкостях организма, включая кровь, мочу и молоко (27).
Анализ таксономического разнообразия на основе данных 16S-мета-геномного NGS-секвенирования ДНК из содержимого рубца обеих коров выявил 21 филум микроорганизмов, из которых доминировали Bacteroidetes , Firmicutes и Proteobacteria (рис. 1). Обилие представителей Bacteroidetes и Fir-micutes в рубце молочных коров подтверждается результатами других авторов, полученными ранее (28-30), однако доля представителей Bacteroidetes , детектированных в нашем исследовании, была значительно выше по сравнению с данными этих исследователей. Такие различия могут быть связаны с составами рационов, величиной удоя и другими факторами (31). Интересно, что у коровы с диагнозом кетоз по сравнению с клинически здоровым животным представленность бактерий филума Bacteroidetes снижалась в 1,2 раза (р ≤ 0,05) при увеличении доли филумов Firmicutes и Proteobacteria соответственно в 2,1 (р ≤ 0,001) и 3,8 (р ≤ 0,01) раза. Ранее G. Grum с соавт. (21) также продемонстрировали снижение численности представителей фи-лума Bacteroidetes — бактерий порядка Bacteroidales и семейства Prevotellaceae в сочетании с увеличением концентрации кетоновых тел в биологических жидкостях коров. Также сообщалось о других аналогичных результатах, касающихся семейства Prevotellaceae (32, 33).
А Б
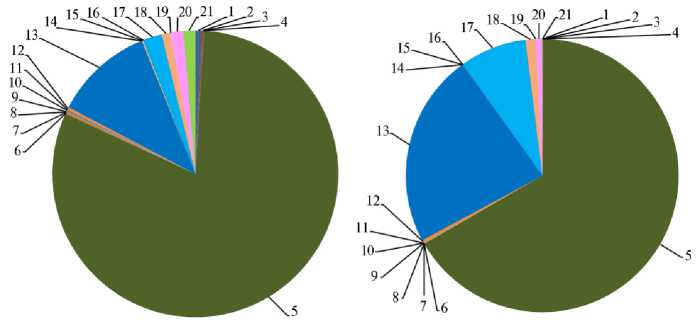
Рис. 1. Доля филумов бактерий в образцах содержимого рубца у коров черно-пестрой голштини-зированной породы 3-й лактации: А — клинически здоровая корова, Б — корова с диагнозом кетоз; 1 — Acidobacteria , 2 — Actinobacteria , 3 — Aquificae , 4 — Armatimonadetes , 5 — Bacteroidetes , 6 — Candidatus_Saccharibacteria , 7 — Chlamydiae , 8 — Chloroflexi , 9 – Cyanobacteria , 10 — Elusimicrobia , 11 — Euryarchaeota , 12 — Fibrobacteres , 13 — Firmicutes , 14 — Latescibacteria , 15 — Lentisphaerae , 16 — Planctomycetes , 17 — Proteobacteria , 18 — Spirochaetes , 19 — Synergistetes , 20 — Tenericutes , 21 — Verrucomicrobia (СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год).
Детальный анализ состава микробиома на уровне родов позволил выявить существенные различия между животными по 13 бактериальным семействам ( Acidaminococcaceae , Anaeroplasmataceae , Bacteroidaceae , Bifidobac-teriaceae , Enterococcaceae , Eubacteriaceae , Lachnospiraceae , Lactobacillaceae , Or-baceae , Porphyromonadaceae , Rikenellaceae , Ruminococcaceae , Succinivibrionaceae ) и 26 родам, в том числе Bacteroides , Bifidobacterium , Butyrivibrio , Coprococcus , Dysgonomonas , Enterococcus , Eubacterium , Frischella , Lachnospira .
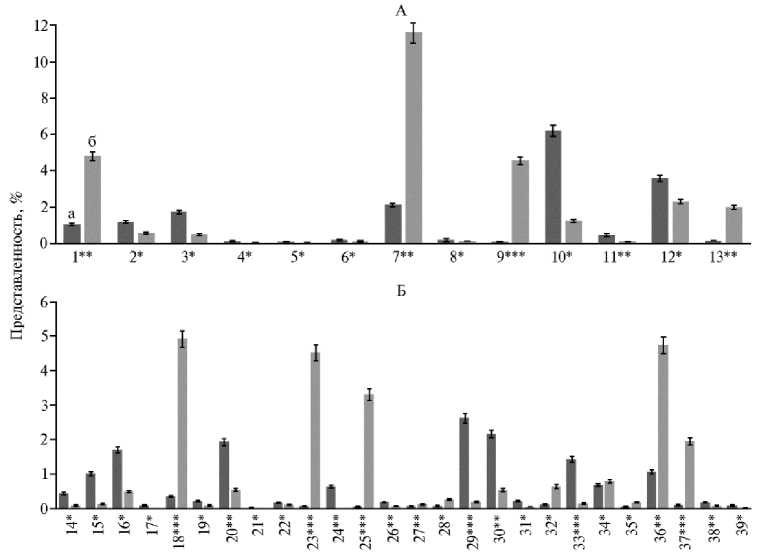
Рис. 2. Представленность бактериальных таксонов, доля которых имела достоверные различия между группами, в образцах содержимого рубца у коров черно-пестрой голштинизированной породы 3-й лактации: А — семейств, Б — роды; а — клинически здоровая корова, б — корова с диагнозом кетоз; 1 — Acidaminococcaceae , 2 — Anaeroplasmataceae , 3 — Bacteroidaceae , 4 — Bifidobacteriaceae , 5 — Enterococcaceae , 6 — Eubacteriaceae , 7 — Lachnospiraceae , 8 — Lactobacil-laceae , 9 — Orbaceae , 10 — Porphyromonadaceae , 11 — Rikenellaceae , 12 — Ruminococcaceae , 13 — Succinivibrionaceae ; 14 — Alistipes , 15 — Anaeroplasma , 16 — Bacteroides , 17 — Bifidobacterium , 18 — Butyrivibrio , 19 — Coprococcus, 20 — Dysgonomonas , 21 — Enterococcus , 22 — Eubacterium , 23 — Frischella , 24 — Gp15, 25 — Lachnospira , 26 — Lactobacillus , 27 — Odoribacter , 28 — Oribacterium , 29 — Parabacteroides , 30 — Paraprevotella , 31 — Porphyromonas , 32 — Pseudobutyrivibrio , 33 — Saccharofermentans , 34 — Selenomonas , 35 — Shuttleworthia , 36 — Succiniclasticum , 37 — Suc-cinivibrio , 38 — Lactobacillus , 39 — Bifidobacterium (СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год).
*, **, *** Различия при сравнении больного и здорового животного статистически значимы соответственно при p ≤ 0,05; p ≤ 0,01 и p ≤ 0,001.
В рубце коровы с диагнозом кетоз по сравнению с клинически здоровой особью мы детектировали более низкую долю представителей филума Bacteroidetes — бактерий семейств Bacteroidaceae (p ≤ 0,05) и Porphyromonad-aceae (p ≤ 0,05) (рис. 2, А). В частности, уменьшалась численность бактерий родов Bacteroides spp., принадлежащих к семейству Bacteroidaceae (в 2,1 раза ниже, p ≤ 0,05) и Dysgonomonas spр. из семейства Porphyromonadaceae (в 3,6 раза ниже, p ≤ 0,01). Например, такие виды рода Dysgonomonas, как D. gadei и D. hofstadii, полностью исчезали из рубца коровы с диагнозом кетоз. Среди бактерий рода Bacteroides в рубце коровы с метаболическими нарушениями не детектировались виды B. ñaecigallinarum и B. eggerthii. Известно, что многие представители родов Bacteroides и Dysgonomonas образуют в рубце жирные кислоты, в том числе значительные количества пропионата (34). Как отмечалось выше, ЛЖК, образуемые в результате рубцовой ферментации, используются как энергоресурсы для животных, более того, пропионат — это главный источник глюкозы для глюконеогенеза (8). Поэтому пропионат имеет важное физиологическое значение для жвачных животных, а снижение численности указанных микроорганизмов у одной из коров в нашем исследовании могло стать провоцирующим фактором при возникновении кетоза.
В рубце пропионат может образовываться через акрилат и сукцинат (35). Доказано, что представители рода Megasphaera выступают основными продуцентами пропионата через акрилатный путь (36, 37). В нашем исследовании содержание бактерий рода Megasphaera было следовым (от 0,006±0,0005 до 0,01±0,0007 %) (см. рис. 2, Б). Обнаруженные нами и имеющие достаточно высокую численность (по сравнению с бактериями рода Megasphaera ) представители Bacteroides spр. (от 0,47±0,03 до 1,7±0,2 %) и Dysgonomonas spр. (от 0,5±0,034 до 1,9±0,3 %) способны образовывать пропионат, используя не акрилат, а сукцинат (38-40). Подобный путь синтеза пропионата был ранее описан J.M. Macy с соавт. (41).
В то же время в рубце коровы с диагнозом кетоз численность бактерий семейства Porphyromonadaceae , относящегося к филуму Bacteroidetes , была в 5,1 раза ниже (p ≤ 0,05), чем у клинически здоровой особи. В частности, доля представителей этого семейства — бактерий рода Parabacteroides уменьшалась в 15,9 раза (р ≤ 0,001). Численность доминирующего представителя рода — P. chinchillae падала с 2,2±0,13 до 0,01±0,0004 %. Ранее K. Wang с соавт. (42) показали, что основным продуктом метаболизма бактерий рода Parabacteroides служит сукцинат, синтез которого был ассоциирован со снижением гипергликемии у мышей через активацию кишечного глюконеогенеза. Таким образом, представители семейства Porphyromona-daceae могут иметь связь с устойчивостью животных к кетозу.
Снижение численности бактерий рода Paraprevotella семейства Prevotellaceae , представленного преимущественно P. clara (р ≤ 0,01), в рубце коровы с диагнозом кетоз также могло обусловливать возникновение метаболических расстройств. На примере P. clara и P. xylaniphila , выделенных из кишечника человека, показано что эти микроорганизмы продуцируют высокоценные для коров янтарную и уксусную кислоты в качестве конечных продуктов метаболизма глюкозы (43).
В рубце кетозного животного мы обнаружили повышение (р ≤ 0,05) доли родов Odoribacter семейства Porphyromonoadaceae , Frischella семейства Orbaceae , среди которых встречаются патогены, а также представленности патогенного вида Bacteroides fragilis . Так, обнаруженный нами в рубце Odori-bacter denticanis является возбудителем брюшного абсцесса человека (44). Интересно, что бактерий рода Frischella ранее в рубце не выявляли. Обнаруженные нами микроорганизмы рода Frischella были представлены в рубце коров единственным видом — F. perrara . Известно, что бактерии F. perrara присутствуют в кишечнике медоносной пчелы, где индуцирует паршу — заболевание, которое проявляется в виде темной полосы, которая развивается на эпителиальной поверхности привратника (45). Bacteroides fragilis выделяли из тканей 105 абортированных плодов у коров. Гистопатологические поражения при этом включали плацентит и бронхопневмонию (46).
Увеличение обилия патогенных форм также могло повлиять на развитие кетоза, поскольку, как отмечалось выше, выделяют так называемые вторичные кетозы, которые сопутствуют инфекционным заболеваниям и болезням желудочно-кишечного тракта.
Возможно, более низкая численность патогенных форм в рубце клинически здоровой коровы было связано с вытеснением этих микроорганизмов представителями индигенной нормофлоры (родов Bifidobacterium , Alistipes и др.), обладающих антимикробной активностью. Например, у клинически здоровой коровы в рубце мы выявили представителей семейства Bifido-bacteriaceae (0,07±0,004 %), известных ярко выраженными антимикробными свойствами (47), тогда как у коровы с кетозом эти микроорганизмы отсутствовали. Повышенная численность (р ≤ 0,01) представителей семейства Rikenellaceae из филума Bacteroidetes в рубце коровы без нарушений метаболизма также могла положительно повлиять на состояние здоровья животного. Не так давно было проведено исследование, показавшее прямую связь между присутствием представителя этого семейства Alistipes finegoldii и снижением проявлений колита (48). Другие исследователи (49) выявили у бактерий Alistipes spр. способность продуцировать противовоспалительные сфинголипиды. Бактерии рода Anaeroplasma (филум Tenericutes ), численность которых в рубце клинически здоровой коровы тоже увеличивалась (р ≤ 0,05), известны свойством индуцировать противовоспалительный цитокин TGF- β , что усиливает кишечный барьер за счет повышения синтеза мукозального IgA (50). Бактерии рода Coprococcus (филум Firmicutes ), доля которых также оказалась выше (р ≤ 0,05) в рубце здоровой коровы, были связаны со снижением заболеваемости раком прямой кишки (51).
Полное вытеснение из состава микробиоты рубца неатрибутируемых бактерий из рода Gp15 у коровы, больной кетозом, указывает на позитивную роль этого рода бактерий в регуляции микробиологических и метаболических процессов пищеварительной системы. Такой вывод представляет фундаментальный интерес, поскольку в настоящее время функции представителей рода Gp15 практически не изучены, что связано с отсутствием возможности их лабораторного культивирования.
1. Индексы альфа-разнообразия микробного сообщества рубца у коров чернопестрой голштинизированной породы 3-й лактации по данным NGS-секвенирования ( M ±SEM, СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год)
|
Вариант |
1 OTE 1 |
Chao1 1 |
Shannon H 1 |
Simpson D |
|
Клинически здоровая 96,0±5,15 97,1±4,92 С диагнозом кетоз 125,0±6,40* 126,0±6,16* Примечание. OTE — оперативные таксономические единицы. * Различия между вариантами статистически значимы при р ≤ 0,05. |
6,1±0,54 5,8±0,41 |
0,98±0,06 0,97±0,05 |
||
В таблице 1 представлены индексы альфа-биоразнообразия микробного сообщества рубца у двух коров — клинически здоровой и кетозной. При сравнении по индексам видового разнообразия Shannon и Simpson статистически достоверных различий между животными мы не установили. Индекс Chao1, оценивающий предположительное реальное количество ОТЕ в сообществе, оказался выше в 1,3 раза (р ≤ 0,05) для микробиоты рубца коровы с диагнозом кетоз.
Сходные данные были получены и при расчете ОТЕ: как видно из рисунка 3, микробиом рубца у клинически здорового животного характеризовался более пологой кривой накопления ОТЕ, чем у коровы с диагнозом кетоз. Следовательно, можно предположить, что в микробиоте рубца при кетозе имеет место менее равномерное распределение видов по их обилию в сообществе на фоне большей численности представленных видов по сравнению с клинически здоровыми животными.
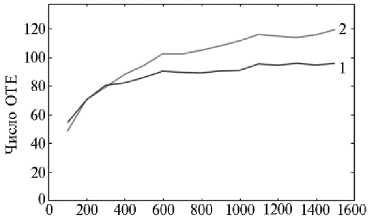
Число прочтений
Рис. 3. Зависимость числа оперативных таксономических единиц (ОТЕ) от числа прочтений по результатам анализа структуры микробного сообщества рубца у коров черно-пестрой голштинизи-рованной породы 3-й лактации (по данным NGS-секвенирования): 1 — клинически здоровая корова, 2 — корова с диагнозом кетоз (СПК «Ко-браловский», Гатчинский р-н, Ленинградская обл., 2020 год).
Образование лактата в рубце — это определяющий этап инициации метаболических расстройств у жвач- ных (52). С использованием количественной ПЦР с обратной транскрипцией мы изучили экспрессию бактериальных генов, связанных с продукцией лактатдегидрогеназ. Лактат в рубце может образовываться из пирувата под действием двух различных форм НАД-связанных лактатдегидрогеназ — ключевых бактериальных ферментов молочнокислого брожения: один фермент (ЕС 1.1.1.27) участвует в образовании L(+)-лактата (53, 54) (кодируется преимущественно геном ldh-L), другой (ЕС 1.1.1.28) — D(-)-лактата (55) (в синтез этого фермента вовлечены следующие известные на сегодняшний день гены: ldb0101, ldb0813, ldb1010, ldb1147 и ldb2021). Оценка экспрессии генов ldh-L, ldb0101, ldb0813, ldb1010, ldb1147 и ldb2021 должна дать адекватное представление о процессах синтеза лактата в рубце коров. Количественная ПЦР с обратной транскрипцией была выбрана для исследования экспрессии генов в связи с общепризнанной (56-58) воспроизводимостью, высокой чувствительностью и специфичностью этого метода для оценки экспрессии генов бактерий.
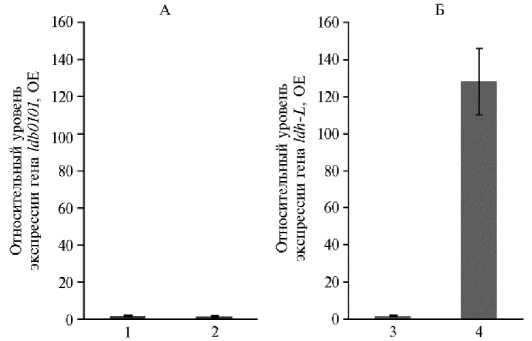
Рис. 4. Относительный уровень экспрессии генов, участвующих в синтезе лактатдегидрогеназ, в микробиоте рубца у коров черно-пестрой голштинизированной породы 3-й лактации: А — ген, вовлеченный в метаболический путь образования D( - )-лактата, Б — ген L-лактатдегидрогеназы; 1 — клинически здоровая корова, 2 — корова с диагнозом кетоз (СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год). ОЕ — относительные единицы экспрессии по сравнению с таковой референсного универсального гена 16S рРНК малой субъединицы рибосомы у прокариот.
В рубце обеих коров не было зафиксировано экспрессии генов ldb0813, ldb1010, ldb1147, ldb2021. По уровню экспрессии гена ldb0101 лактатдегидрогеназы ЕС 1.1.1.28 достоверных различий между животными мы не обнаружили (рис. 4, А). Тем не менее между животными была выявлена достоверная разница (р ≤ 0,001) в уровне экспрессии гена ldh-L лактатдегидрогеназы ЕС 1.1.1.27: экспрессия генов, ассоциированных с синтезом этого фермента, у коровы с кетозом была в 128±17,9 раза выше, чем у клинически здорового животного (см. рис. 4, Б). Полученные результаты закономерны, поскольку при несбалансированном кормлении у коров действительно могут возникнуть дисбиотические нарушения рубцовой микрофлоры (59), причем равновесие может сместиться в сторону образования лактата — промежуточного продукта ферментации в рубце. Избыток лактата коррелирует со снижением pH рубца и лактатным ацидозом (59). В условиях ацидоза в результате подавления чувствительных к снижению рН продуцентов ЛЖК, таких как Selenomonas ruminantium и Megasphaera elsdenii (8), скорость образования пропионата в рубце снижается, вследствие чего глюконеогенез у животных может замедляться, то есть увеличивается риск развития кетоза.
С использованием программного комплекса PICRUSt2 и MetaCyс была проведена реконструкция и прогнозирование функциональной активности сообщества рубца на основе метагеномного анализа нуклеотидных последовательностей генов 16S рРНК. В результате мы аннотировали различных метаболические пути (всего 232), из которых в 12 предсказанный функциональный потенциала был достоверно (р ≤ 0,05) усилен у коровы с диагнозом кетоз по сравнению с клинически здоровым животным (рис. 5).
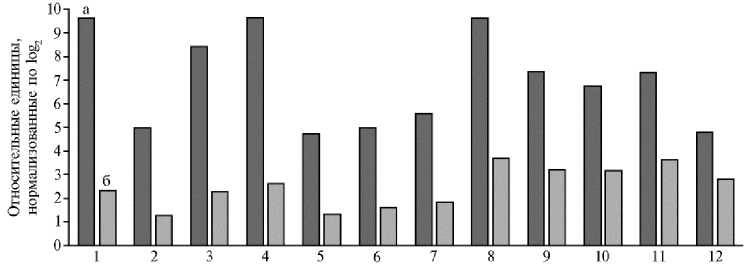
Рис. 5. Результаты (р ≤ 0,05) функциональной аннотации метаболических путей в метагеномном сообществе рубца у коров черно-пестрой голштинизированной породы 3-й лактации: а — корова с диагнозом кетоз, б — клинически здоровая корова; 1 — утилизация сахарозы, 2 — биосинтез метахинонов, 3 — утилизация фруктозы, 4 — биосинтез структурных блоков для синтеза О-антигена, 5 — деградация мио-, хиро- и сциллоинозита, 6 — биосинтез (R, R)-бутандиола, 7 — деградация 4-аминобутаноата, 8 — утилизация глюкозы и глюкозо-1-фосфата, 9 — деградация гекситолов, 10 — ферментация гекситолов до лактата, 11 — синтез витамина B 12 , 12 — гетероферментативное молочнокислое брожение (СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год).
В частности, по сравнению с клинически здоровой особью у коровы с диагнозом кетоз повышался прогнозируемый метаболический потенциал микробиоты, связанный с осуществлением гетероферментативного молочнокислого брожения (60), что согласуется с данными по экспрессии гена L-лактатдегидрогеназы ЕС 1.1.1.27, представленными выше (см. рис. 4, Б).
Более того, анализ показал присутствие у обеих коров в рубцовой микробиоте генов, кодирующих большинство критических ферментов пути молочнокислого брожения: фруктокиназы (EC 2.7.1.4), глюкозо-6-фос-фатизомеразы (EC 5.3.1.9), глюкозо-6-фосфатдегидрогеназы (EC 1.1.1.49), фосфокетолазы (EC 4.1.2.9), фосфат-ацетилтрансферазы (EC 2.3.1.8), D-лактатдегидрогеназы (ЕС 1.1.1.28), L-лактатдегидрогеназы (ЕС 1.1.1.27)
(см. рис. 5). При этом представленность генов, ассоциированных с синтезом D-лактатдегидрогеназы и L-лактатдегидрогеназы, у коровы с диагнозом кетоз была соответственно в 2,0 и 1,5 раза достоверно (р ≤ 0,05) выше, чем у клинически здоровой коровы (рис. 6).
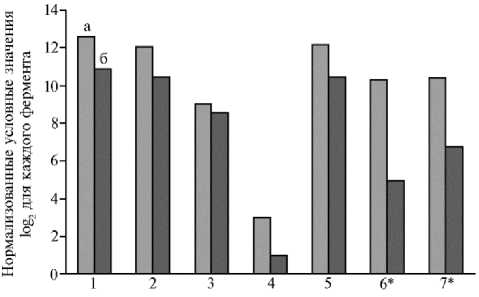
Рис. 6. Результаты (р ≤ 0,05) функциональной аннотации ферментов, прогнозируемых в метагеномном сообществе рубца у коров чернопестрой голштинизированной породы 3-й лактации: а — корова с диагнозом кетоз, б — клинически здоровая корова; 1 — фруктокиназа, 2 — глюкозо-6-фосфатизо-мераза, 3 — глюкозо-6-фосфат-дегидрогеназа, 4 — фосфокетолаза, 5 — фосфат-ацетилтрансфе-раза, 6 — D-лактатдегидрогеназа, 7 — L-лактатдегидрогеназа (СПК «Кобраловский», Гатчинский р-н, Ленинградская обл., 2020 год).
* Различия между больным и здоровым животным статистически значимы при р ≤ 0,05.
Интересно, что несмотря на выявленное повышение метаболического потенциала, связанного с продукцией лактата в рубце животного, страдающего кетозом, обилие молочнокислых бактерий — продуцентов лактата из семейства Lactobacillaceae было выше (р ≤ 0,05) в рубце клинически здорового животного (см. рис. 2, А). Это может быть обусловлено либо тем, что разные виды лактобактерий проявляют неодинаковую функциональную активность геномов и способность продуцировать лактат, либо этой способностью обладают микроорганизмы других таксонов. Например, как видно из рисунка 2, А, в рубце коровы, страдающей кетозом, по сравнению со здоровым животным доля бактерий семейства Lachnospiraceae увеличивалась в 5,6 раза (p ≤ 0,01) . Обилие бактерий указанного семейства — родов Oribacterium , Pseudobutyrivibrio и Shuttleworthia было повышено соответственно в 4,5; 6,3 и в 5,6 раза (р ≤ 0,05) в рубце коровы с кетозом. Нам удалось обнаружить, что вследствие заболевания животного в рубце появлялся новый вид рода Pseudobutyrivibrio — P. xylanivorans . Известно, что основные конечные продукты метаболического брожения у вышеназванных бактерий — органические кислоты, в том числе лактат (61-63). Аналогичным образом в рубце коровы с нарушением здоровья была также повышена (р ≤ 0,001) численность микроорганизмов рода Succinivibrio из семейства Succinivibrionaceae , способных производить лактат помимо ацетата, сукцината и формиата (64).
Возвращаясь к рисунку 5, необходимо подчеркнуть, что по сравнению с клинически здоровым животным у коровы с нарушением состояния здоровья повышались (в 2,6 раза, p ≤ 0,05) метаболические возможности микробиоты, связанные с утилизацией глюкозы и глюкозо-1-фосфата. Синтезируемые микрофлорой ЛЖК, всасываясь в рубце, транспортируются в печень и включаются в путь глюконеогенеза, в результате чего животное снабжается сахаром. Концентрация глюкозы в крови играет ключевую роль в поддержании энергетического метаболизма и профилактике болезней обмена веществ, прежде всего кетоза. Хорошо известно, что, помимо ЛЖК-синтезирующих бактерий, многие другие микроорганизмы, например представители семейства Enterobacteriaceae, активно используют глюкозу и глю-козо-1-фосфат как источники питательных веществ, образуя в качестве основного продукта спирты. Так, Escherichia coli проявляет способность к культивированию на экзогенно поставляемом глюкозо-1-фосфате как единственном источнике углерода (65). Это может привести к снижению количества питательного субстрата для бактерий порядка Selenomonadales и нарушению синтеза ЛЖК.
Кроме того, у коровы, страдающей кетозом, в 3,0 раза (p ≤ 0,05) усиливался функциональный потенциал рубцовой микробиоты, связанный с биосинтезом бутандиола (см. рис. 5). Как известно, бутандиол в рубце синтезируется с помощью альфа-ацетолактатдекарбоксилазы из пирувата, необходимого для синтеза глюкозы в процессе глюконеогенеза. В результате концентрация высокоценного для производства глюкозы пирувата у коровы с диагнозом кетоз могла быть снижена, что тоже может создавать предпосылки для развития метаболических нарушений. Метаболическая функция самого бутандиола не очень хорошо изучена, хотя есть указания на то, что он способен играть определенную роль в предотвращении внутриклеточного подкисления (66).
Интересно, что у коровы с диагнозом кетоз в содержимом рубца была также усилена (в 2,3 раза, p ≤ 0,05) возможность деградации гексито-лов. Увеличение в рационе таких гекситолов, как D-маннит, D-сорбит и галактит, было связано с накоплением гликогена в печени млекопитающих (67). Поэтому процесс деградации гекситолов мог провоцировать кетоз, который, как уже отмечалось, наступает при углеводном дефиците и связан с расходом запасов гликогена из печени.
Также важно, что по сравнению с клинически здоровой коровой у кетозного животного повышались метаболические возможности рубцовой микробиоты, ассоциированные с деградацией мио-, хиро- и сциллоинозита (в 3,6 раза, p ≤ 0,05), биосинтезом структурных блоков O-антигена (в 3,7 раза, p ≤ 0,05), деградацией 4-аминобутаноата (в 3,0 раза, p ≤ 0,05).
Метаболическая деградации миоинозита первоначально была охарактеризована для Klebsiella aerogenes (68). Миоинозит и другие стереоизомеры шестиатомного спирта инозитола в форме различных инозитолфосфатов и фосфатидилинозитоловых липидов выступают в качестве важных передатчиков сигнала во внутриклеточных сигнальных каскадах, участвуя в регуляции содержания внутриклеточного кальция, передаче сигнала от рецептора инсулина, расщеплении жиров и снижении концентрации холестерина в крови, модуляции активности нейротрансмиттеров. Снижение синтеза миоинозита в рубце связано с уменьшением образования печеночных липопротеинов. В период лактации молочных коров мобилизация липидов крайне интенсивна, а потребность в миоинозите может превышать его доступность. Таким образом, при повышении активности по деградации этого соединения рубцовая микрофлора могла участвовать в инициации кетоза как метаболического нарушения.
Структурные блоки полимеров О-антигена представляют собой олигосахаридные субъединицы, которые служат компонентом внешней мембраны некоторых грамотрицательных бактерий (69). Ряд облигатных и условно-патогенных микроорганизмов, таких как Salmonella enterica, Francisella tularensis и Burkholderia cepacia, используют О-антигены, чтобы избежать фагоцитоза и противостоять литическому действию системы комплемента (70-72). Способность синтезировать О-антигены, выявленная у рубцовой микробиоты при кетозе, имеет важное значение, поскольку различают не только первичные (метаболические) кетозы, возникающие вследствие нарушений в режимах кормления и содержании, но и вторичные, которые сопутствуют заболеваниям желудочно-кишечного тракта и некоторым инфекционным болезням (73). Это согласуется с приведенными выше данными о том, что в рубце кетозного животного мы обнаружили повышение (р < 0,05) доли родов Odoribacter семейства Porphyromonoadaceae и Frischella семейства Orbaceae, среди которых встречаются патогены, а также обилия патогенного вида Bacteroides fragilis.
Обнаруженное нами потенциальное усиление деградации 4-амино-бутаноата у коровы с диагнозом кетоз — еще один негативный фактор, поскольку 4-аминобутаноат служит главным ингибиторным нейромедиатором в головном мозге млекопитающих (74). Известно, что многие бактерии могут синтезировать 4-аминобутаноат в качестве промежуточного продукта в процессе деградации путресцина. Первое доказательство бактериальной деградации 4-аминобутаноата было найдено при изучении метаболизма бактерии Pseudomonas fluorescens (75), которая способна расти на 4-аминобута-ноате в качестве единственного источника углерода и азота (76).
Итак, применение совокупности молекулярно-биологических и био-информатических методов позволило нам не только подробно изучить структуру микробиома рубца у коров, но также описать и предсказать ее функциональный потенциал в зависимости от состояния здоровья. В рубце коровы с заболеванием кетоз повышалась доля таксонов, связанных с нарушением обеспечения энергетического баланса, в частности глюконеогенеза, повышением синтеза L-лактата, усилением патогенеза. Поэтому увеличение численности таких микроорганизмов могло вызвать и/или усугубить кетоз. У коровы с кетозом экспрессия генов, ассоциированных с синтезом фермента лактатдегидрогеназы (ЕС 1.1.1.27), усиливалась в 128±17,9 раза по сравнению с таковой у клинически здорового животного. Полученная оценка экспрессии генов согласуется с данными о метаболическом потенциале рубцовой микробиоты, предсказанном на основе результатов метагеном-ного анализа нуклеотидных последовательностей генов 16S рРНК. С использованием программного комплекса PICRUSt2 и MetaCyс аннотированы 232 метаболических пути, у 12 предсказанный функциональный потенциал усилен у коровы с диагнозом кетоз по сравнению с клинически здоровым животным. В частности, повышена представленность генов, ассоциированных с синтезом D-лактатдегидрогеназы и L-лактатдегидрогеназы, генов, связанных с утилизацией глюкозы и глюкозо-1-фосфата, биосинтезом бутандиола и возможностью деградации гекситолов. Результаты нашего исследования могут быть использованы для прогнозирования метаболических нарушений у коров, в частности кетоза. Однако для подтверждения того, действительно ли существуют причинно-следственные связи между возникновением кетоза, изменением численности специфических популяций микроорганизмов в рубце, их метаболическим потенциалом и экспрессией генов в микробном сообществе пищеварительной системы, необходимы дальнейшие исследования. Возможно, это позволит разработать приемы оптимизации состава микробного сообщества рубца с целью коррекции кетоза, в частности создать препараты на основе пробиотических штаммов бактерий для направленной регуляции численности у ряда представителей рубцовой микробиоты.
Список литературы Таксономические и функциональные особенности микробиоты рубца у дойных коров с диагнозом кетоз
- Калюжный И.И., Баринов Н.Д., Коробов А.В. Метаболические нарушения у высокопродуктивных коров. Саратов, 2010.
- Boichard D., Brochard M. New phenotypes for new breeding goals in dairy cattle. Animal, 2012, 6: 544-550 (doi: 10.1017/S1751731112000018).
- Koeck A., Jamrozik J., Schenkel F.S., Moore R.K., Lefebvre D.M., Kelton D.F., Miglior F. Genetic analysis of milk p-hydroxybutyrate and its association with fat-to-protein ratio, body condition score, clinical ketosis, and displaced abomasum in early first lactation of Canadian Holsteins. Journal of Dairy Science, 2014, 97: 7286-7292 (doi: 10.3168/jds.2014-8405).
- Gohary K., Overton M.W., Von Massow M., LeBlanc S.J., Lissemore K.D., Duffield T.F. The cost of a case of subclinical ketosis in Canadian dairy herds. Canadian Veterinary Journal, 2016, 57: 728-732.
- Pryce J.E., Parker Gaddis K.L., Koeck A., Bastin C., Abdelsayed M., Gengler N., Miglior F., Heringstad B., Egger-Danner C., Stock K.F., Bradley A.J., Cole J.B. Opportunities for genetic improvement of metabolic diseases. Journal of Dairy Science, 2016 99: 6855-6873 (doi: 10.3168/jds.2016-10854).
- Гавриш В.Г., Аганин А.В., Демкин Г.П. Справочник ветеринарного врача. Ростов-на Дону, 1996.
- Bell A.W. Regulation of organic nutrient metabolism during transition from late pregnancy to early lactation. Journal of Animal Science, 1995, 73: 2804-2819 (doi: 10.2527/1995.7392804x).
- Wang X., Li X., Zhao C., Hu P., Chen H., Liu Z., Liu G., Wang Z. Correlation between composition of the bacterial community and concentration of volatile fatty acids in the rumen during the transition period and ketosis in dairy cows. Applied and Environmental Microbiology, 2012, 78(7): 2386-2392 (doi: 10.1128/AEM.07545-11).
- Reynolds C.K., Huntington G.B., Tyrrell H.F., Reynolds P.J. Net metabolism of volatile fatty acids, d-p-hydroxybutyrate, nonesterified fatty acids, and blood gases by portal-drained viscera and liver of lactating Holstein cows. Journal of Dairy Science, 1988, 7: 2395-2405 (doi: 10.3168/jds.S0022-0302(88)79824-0).
- Crawford P.A., Crowley J.R., Sambandam N., Muegge B.D., Costello E.K., Hamady M., Knight R., Gordon J.I. Regulation of myocardial ketone body metabolism by the gut microbiota during nutrient deprivation. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106: 11276-11281 (doi: 10.1073/pnas.0902366106).
- Cotter D.G., Schugar R.C., Crawford P.A. Ketone body metabolism and cardiovascular disease American Journal of Physiology: Heart and Circulatory Physiology, 2013, 304: H1060-H1076 (doi: 10.1152/ajpheart.00646.2012).
- Bergman E.N. Energy contributions of volatile fatty acids from the gastrointestinal tract in various species. Physiological Reviews, 1990, 70: 567-590 (doi: 10.1152/physrev.1990.70.2.567).
- Sutton J.D. The fermentation of soluble carbohydrates in rumen contents of cows fed diets containing a large proportion of hay. British Journal of Nutrition, 1968, 22: 689-712 (doi: 10.1079/bjn19680080).
- Negrao F.D.M., Zanine A.D.M., Cabral L.D.S., Souza A.L.D., Alves G.R., Ferreira D.D.J., Dantas C.C.O., Lehmkuhl A. Fractionation of carbohydrates and protein and rumen degradation kinetic parameters of Brachiaria grass silage enriched with rice bran. Revista Brasileira de Zootecnia, 2014, 43: 105-113 (doi: 10.1590/S1516-35982014000300001).
- McDonald P., Edwards R.A., Greenhalgh J.F.D., Morgan C.A., Sinclair L.A., Wilkinson R.G. Animal Nutrition, 7th edition. Pearson, Harlow, England, 2010.
- Stewart C.S., Bryant M.P. The rumen microbial ecosystem /P.N. Hobson, C.S. Stewart (eds.). Elsevier Applied Science, London, 1988: 21-76.
- Tajimaa K., Arai S., Koretsugu O., Takafumi N., Hiroki M., Mutsumi N., Rustam I., Benno A.Y. Rumen bacterial community transition during adaptation to high-grain diet. Anaerobe, 2000, 6: 273-284 (doi: 10.1006/anae.2000.0353).
- Douglas G.M., Maffei V.J., Zaneveld J.R., Yurgel S.N., Brown J.R., Taylor C.M., Hut-tenhower C., Langille M.G.I. PICRUSt2 for prediction of metagenome functions. Nature Biotechnology, 2020, 38(6): 685-688 (doi: 10.1038/s41587-020-0548-6 0).
- Wilkinson T.J., Huws S.A., Edwards J.E., Kingston-Smith A.H., Siu-Ting K., Hughes M., Ru-bino F., Friedersdorff M., Creevey C.J. CowPI: a rumen microbiome focussed version of the PICRUSt func-tional inference software. Frontiers in Microbiology, 2018, 9: 1095 (doi: 10.3389/fmicb.2018.01095).
- Zhu Z., Kristensen L., Difford G.F., Poulsen M., Noel S.J., Abu Al-Soud W., Serensen S.J., Lassen J., Lavendahl P., Hejberg O. Changes in rumen bacterial and archaeal communities over the transition period in primiparous Holstein dairy cows. Journal of Dairy Science, 2018, 101: 9847-9862 (doi: 10.3168/jds.2017-14366).
- Grum G., Difford G.F., Buitenhuis B., Lassen J., Noel S.J., Hqjberg O., Plichta D.R., Zhu Z., Poulsen N.A, Sundekilde U.K., Levendahl P., Sahana G. Predictive ability of host genetics and rumen microbiome for subclinical ketosis. Journal of Dairy Science, 2020, 103(5): 4557-4569 (doi: 10.3168/jds.2019-17824).
- Khafipour E., Li S., Plaizier J.C., Krause D.O. Rumen microbiome composition determined using two nutritional models of subacute luminal acidosis. Applied and Environmental Microbiology, 2009, 75: 7115-7124 (doi: 10.1128/AEM.00739-09).
- Кондрахин И.П., Левченко В.И. Диагностика и терапия внутренних болезней животных. М., 2005.
- Zeka F., Vanderheyden K., De Smet E., Cuvelier C.A., Mestdagh P., Vandesompele Jo. Straightforward and sensitive RT-qPCR based gene expression analysis of FFPE samples. Scientific Reports, 2016, 6: 21418 (doi: 10.1038/srep21418).
- Kim B.R., Shin J., Guevarra R., Lee J.H., Kim D.W., Seol K.-H., Lee J.-H., Kim H.B., Isaacson R. Deciphering Diversity indices for a better understanding of microbial communities. Applied Microbiology and Biotechnology, 2017, 27(12): 2089-2093 (doi: 10.4014/jmb.1709.09027).
- Livak K.J., Schmittgen T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta DeltaC(T)) method. Methods, 2001, 25(4): 402-408 (doi: 10.1006/meth.2001.1262).
- Geishauser T., Leslie K., Kelton D., Duffield T. Evaluation of five cowside tests for use with milk to detect subclinical ketosis in dairy cows. Journal of Dairy Science, 1998, 81: 438-443 (doi: 10.3168/jds.S0022-0302(98)75595-x).
- Jami E., White B.A., Mizrahi I. Potential role of the bovine rumen microbiome in modulating milk composition and feed efficiency. PLoS ONE, 2014, 9(1): e85423 (doi: 10.1371/jour-nal.pone.0085423).
- Jewell K.A., McCormick C.A., Odt C.L., Weimer P.J., Suen G. Ruminal bacterial community composition in dairy cows is dynamic over the course of two lactations and correlates with feed efficiency. Applied and Environmental Microbiology, 2015, 81(14): 4697-4710 (doi: 10.1128/AEM.00720-15).
- Lima F.S., Oikonomou G., Lima S.F., Bicalho M.L., Ganda E.K., de Oliveira Filho J.C., Lorenzo G., Trojacanec P., Bicalho R.C. Prepartum and postpartum rumen fluid microbiomes: characterization and correlation with production traits in dairy cows. Applied and Environmental Microbiology, 2015, 81(4): 1327-1337 (doi: 10.1128/AEM.03138-14).
- McCann J.C., Wickersham T.A., Loor J.J. High-throughput methods redefine the rumen microbiome and its relationship with nutrition and metabolism. Bioinformatics and Biology Insights, 2014, 8: 109-125, (doi: 10.4137/BBI.S15389).
- Schären M., Frahm J., Kersten S., Meyer U., Hummel J., Breves G., Dänicke S. Interrelations between the rumen microbiota and production, behavioral, rumen fermentation, metabolic, and immunological attributes of dairy cows. Journal of Dairy Science, 2018, 101: 4615-4637 (doi: 10.3168/jds.2017-13736).
- Wallace R.J., Sasson G., Garnsworthy P.C., Tapio I., Gregson E., Bani P., Huhtanen P., Bayat A.R., Strozzi F., Biscarini F., Snelling T.J., Saunders N., Potterton S.L., Craigon J., Minuti A., Trevisi E., Callegari M.L., Cappelli F.P., Cabezas-Garcia E.H., Vilkki J., Pinares-Patino C., Fliegerová K.O., Mrázek J., Sechovcová H., Kopecny J., Bonin A., Boyer F., Taber-let P., Kokou F., Halperin E., Williams J.L., Shingfield K.J., Mizrahi I. A heritable subset of the core rumen microbiome dictates dairy cow productivity and emissions. Science Advances, 2019, 5(7): eaav8391 (doi: 10.1126/sciadv.aav8391).
- Kita A., Miura T., Okamura Y., Aki T., Matsumura Y., Tajima T., Kato J., Nakashimada Y. Dysgonomonas alginatilytica sp. nov., an alginate-degrading bacterium isolated from a microbial consortium. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(10): 35703575 (doi:10.1099/ijsem.0.000459).
- Chiquette J., Allison M.J., Rasmussen M.A. Prevotella bryantii 25A used as a probiotic in early-lactation dairy cows: effect on ruminal fermentation characteristics, milk production, and milk composition. Journal of Dairy Science, 2008, 91(9): 3536-3543 (doi: 10.3168/jds.2007-0849).
- Hino T., Shimada K., Maruyama T. Substrate Preference in a strain of Megasphaera elsdenii, a ruminal bacterium, and its implications in propionate production and growth competition. Applied and Environmental Microbiology, 1994, 60(6): 1827-1831 (doi: 10.1128/AEM.60.6.1827-1831.1994).
- Counotte G.H., Prins R.A., Janssen R.H., deBie M.J. Role of Megasphaera elsdenii in the fermentation of DL-(2-C) lactate in the rumen of dairy cattle. Applied and Environmental Microbiology, 1981, 42: 649-655 (doi: 10.1128/AEM.42.4.649-655.1981).
- Mountfort D.O., Roberton A.M. Origins of fermentation products formed during growth of Bac-teroides ruminicola on glucose. The Journal of General Microbiology, 1978, 106(2): 353-360 (doi: 10.1099/00221287-106-2-353).
- Martin S.A. Factors affecting glucose uptake by the ruminal bacterium Bacteroides ruminicola. Applied Microbiology and Biotechnology, 1992, 37: 104-108 (doi: 10.1007/BF00174212).
- Rios-Covian D., Salazar N., Gueimonde M., de Los Reyes-Gavilan C.G. Shaping the metabolism of intestinal bacteroides population through diet to improve human health. Frontiers in Microbiology, 2017, 8: 376 (doi: 10.3389/fmicb.2017.00376).
- Macy J.M., Ljungdahl L.G., Gottschalk G. Pathway of succinate and propionate formation in Bacteroides fragilis. Journal of Bacteriology, 1978, 134(1): 84-91 (doi: 10.1128/JB.134.1.84-91.1978).
- Wang K., Liao M., Zhou N., Bao L., Ma K., Zheng Z., Wang Y., Liu C., Wang W., Wang J., Liu S., Liu H. Parabacteroides distasonis alleviates obesity and metabolic dysfunctions via production of succinate and secondary bile acids. Cell Reports, 2019, 26(1): 222-235 (doi: 10.1016/j.celrep.2018.12.028).
- Morotomi M., Nagai F., Sakon H., Tanaka R. Paraprevotella clara gen. nov., sp. nov. and Par-aprevotella xylaniphila sp. nov., members of the family 'Prevotellaceae'isolated from human faeces. International Journal of Systematic and Evolutionary Microbiology, 2009, 59(8): 1895-900 (doi: 10.1099/ijs.0.008169-0).
- Hardham J.M., King K.W., Dreier K., Wong J., Strietzel C., Eversole R.R., Sfintescu C., Evans R.T. Transfer of Bacteroides splanchnicus to Odoribacter gen. nov. as Odoribacter splanchnicus comb. nov., and description of Odoribacter denticanis sp. nov., isolated from the crevicular spaces of canine periodontitis patients. International Journal of Systematic and Evolutionary Microbiology, 2008, 58(1): 103-109 (doi: 10.1099/ijs.0.63458-0)
- Engel P., Bartlett K., Moran, N. The bacterium Frischella perrara causes scab formation in the gut of its honeybee host. mBio, 2015, 6(3): e00193-15 (doi: 10.1128/mBio.00193-15).
- Kraipowich N.R., Morris D.L., Thompson G.L., Mason G.L. Bovine abortions associated with Bacteroides fragilis fetal infection. Journal of Veterinary Diagnostic Investigation, 2000, 12(4): 369371 (doi: 10.1177/104063870001200413).
- Lee D.K., Park S.Y., An H.M., Kim J.R., Kim M.Ji., Lee S.W., Cha M.K., Kim S.A., Chung M.J., Lee K.O., Ha N.J. Antimicrobial activity of Bifidobacterium spp. isolated from healthy adult Koreans against cariogenic microflora. Archives of Oral Biology, 2011, 56(10): 10471054 (doi: 10.1016/j.archoralbio.2011.03.002).
- Dziarski R., Park S.Y., Kashyap D.R., Dowd S.E., Gupta D. Pglyrp-regulated gut microflora Prevotella falsenii, Parabacteroides distasonis and Bacteroides eggerthii enhance and Alistipes fi-negoldii attenuates colitis in mice. PLoS ONE, 2016, 11(1): e0146162 (doi: 10.1371/jour-nal.pone.0146162).
- Walker A., Pfitzner B., Harir M., Schaubeck M., Calasan J., Heinzmann S.S., Turaev D., Rat-tei T., Endesfelder D., zu Castell W., Haller D., Schmid M., Hartmann A., Schmitt-Kopplin P. Sulfonolipids as novel metabolite markers of Alistipes and Odoribacter affected by high-fat diets. Scientific Reports, 2017, 7(1): 11047 (doi: 10.1038/s41598-017-10369-z).
- Beller A., Kruglov A., Durek P., von Goetze1 V., Hoffmann U., Maier R., Heiking K., Siegmund B., Heinz G.A., Mashreghi M.-F., Radbruch A., Chang H.-D. P104 Anaeroplasma, a potential anti-inflammatory probiotic for the treatment of chronic intestinal inflammation. Annals of the Rheumatic Diseases, 2019, 78 (1): A45-A46 (doi: 10.1136/annrheumdis-2018-EWRR2019.92).
- Xia L.C., Liu G., Yingxin G., Li X., Pan H., Ai D. Identifying gut microbiota associated with colorectal cancer using a zero-inflated lognormal model. Frontiers in Microbiology, 2019, 10: 826 (doi: 10.3389/fmicb.2019.00826).
- Nocek J.E. Bovine acidosis: implications on laminitis. Journal of Dairy Science, 1997, 80(5): 10051028 (doi: 10.3168/jds.S0022-0302(97)76026-0).
- Zhang X., Zhang S., Shi Y., Shen F., Wang H. A new high phenyl lactic acid-yielding Lactobacillus plantarum IMAU10124 and a comparative analysis of lactate dehydrogenase gene. FEMS Microbiology Letters, 2014, 356(1): 89-96 (doi: 10.1111/1574-6968.12483).
- Yeswanth S., Nanda Kumar Y., Venkateswara Prasad U., Swarupa V., Koteswara rao V., Venkata Gurunadha Krishna Sarma P. Cloning and characterization of L-lactate dehydrogenase gene of Staphylococcus aureus. Anaerobe, 2013, 24: 43-48 (doi: 10.1016/j.anaerobe.2013.09.003).
- Huang Y., You C., Liu Z. Cloning of D-lactate dehydrogenase genes of Lactobacillus delbrueckii subsp. bulgaricus and their roles in D-lactic acid production. Biotechnology, 2017, 7(3): 194 (doi: 10.1007/s13205-017-0822-6).
- Goerke C., Bayer M.G., Wolz C. Quantification of bacterial transcripts during infection using competitive reverse transcription-PCR (RT-PCR) and LightCycler RT-PCR. Clinical and Vaccine Immunology, 2001, 8(2): 279-282 (doi: 10.1128/CDLI.8.2.279-282.2001).
- Bustin S.A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology, 2002, 29(1): 23-39 (doi: 10.1677/jme.0.0290023).
- Nolan T., Hands R.E., Bustin S.A. Quantification of mRNA using real-time RT-PCR. Nature Protocols, 2006, 1(3): 1559-1582 (doi: 10.1038/nprot.2006.236).
- Russell J.B., Sharp W.M., Baldwin R.L. The effect of pH on maximum bacterial growth rate and its possible role as a determinant of bacterial competition in the rumen. Journal of Animal Science, 1979, 48: 251-255 (doi: 10.2527/jas1979.482251x).
- Kang T.S., Korber D.R., Tanaka T. Regulation of dual glycolytic pathways for fructose metabolism in heterofermentative Lactobacilluspanis PM1. Applied and Environmental Microbiology, 2013, 79(24): 7818-7826 (doi: 10.1128/AEM.02377-13).
- Downes J., Munson M.A., Radford D.R., Spratt D.A., Wade W.G. Shuttleworthia satelles gen. nov., sp. nov., isolated from the human oral cavity. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(5): 1469-1475 (doi: 10.1099/00207713-52-5-1469).
- Cepeljnik T., Zorec M., Kostanjsek R., Nekrep F.V., Marinsek-Logar R. Is Pseudobutyrivibrio xylanivorans strain Mz5T suitable as a probiotic? An in vitro study. Folia Microbiologica (Praha), 2003, 48(3): 339-345 (doi: 10.1007/BF02931363).
- Sizova M.V., Muller P.A., Stancyk D., Panikov N.S., Mandalakis M., Hazen A., Hohmann T., Doerfert S.N., Fowle W., Earl A.M., Nelson K.E., Epstein S.S. Oribacterium parvum sp. nov. and Oribacterium asaccharolyticum sp. nov., obligately anaerobic bacteria from the human oral cavity, and emended description of the genus Oribacterium. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(8): 2642-2649 (doi: 10.1099/ijs.0.060988-0).
- O'Herrin S.M., Kenealy W.R. Glucose and carbon dioxide metabolism by Succinivibrio dextrinosol-vens. Applied and Environmental Microbiology, 1993, 59(3): 748-755 (doi: 10.1128/AEM.59.3.748-755.1993)
- Pradel E., Boquet P.L. Utilization of exogenous glucose-1-phosphate as a source of carbon or phosphate by Escherichia coli K12: respective roles of acid glucose-1-phosphatase, hexose-phos-phate permease, phosphoglucomutase and alkaline phosphatase. Research in Microbiology, 1991, 142(1): 37-45 (doi: 10.1016/0923-2508(91)90095-r).
- Booth I.R., Kroll R.G. Regulation of cytoplasmic pH (pH1) in bacteria and its relationship to metabolism. Biochemical Society Transactions, 1983, 11(1): 70-72 (doi: 10.1042/bst0110070).
- Johnston C., Deuel H.J. Jr. Studies on ketosis: XXI. The comparative metabolism of the hexitols. Journal of Biological Chemistry, 1943, 149: 117-124 (doi: 10.1016/S0021-9258(18)72221-7).
- Vidal-Leiria M., van Uden N. Inositol dehydrogenase from the yeast Cryptococcus melibiosum. Biochimica et Biophysica Acta, 293(2): 295-303 (doi: 10.1016/0005-2744(73)90337-9).
- Kalynych S., Morona R., Cygler M. Progress in understanding the assembly process of bacterial O-antigen. FEMS Microbiology Reviews, 38(5): 1048-1065 (doi: 10.1111/1574-6976.12070).
- Murray G.L., Attridge S.R., Morona R. Altering the length of the lipopolysaccharide O antigen has an impact on the interaction of Salmonella enterica serovar Typhimurium with macrophages and complement. Journal of Bacteriology, 2006, 188: 2735-2739 (doi: 10.1128/JB.188.7.2735-2739.2006).
- Duerr C.U., Zenk S.F., Chassin C., Pott J., Gutle D., Hensel M., Hornef M.W. O-antigen delays lipopolysaccharide recognition and impairs antibacterial host defense in murine intestinal epithelial cells. PLOS Pathogens, 2009, 5(9): e1000567 (doi: 10.1371/journal.ppat.1000567).
- Saldias M.S., Ortega X., Valvano M.A. Burkholderia cenocepacia O antigen lipopolysaccharide prevents phagocytosis by macrophages and adhesion to epithelial cells. Journal of Medical Microbiology, 2009, 58(12): 1542-1548 (doi: 10.1099/jmm.0.013235-0).
- Гавриш В.Г, Калюжный И.И. Лечебник домашних животных и птиц для фермеров и животноводов-любителей. Ростов-на Дону, 1999.
- Bouche N., Fromm H. GABA in plants: just a metabolite? Trends in Plant Science, 2004, 9(3): 110-115 (doi: 10.1016/j.tplants.2004.01.006).
- Scott E.M., Jakoby W.B. Soluble gamma-aminobutyric-glutamic transaminase from Pseudomonas fluorescens. Journal of Biological Chemistry, 1959, 234(4): 932-936.
- Belitsky B.R., Sonenshein A.L. GabR, a member of a novel protein family, regulates the utilization of gamma-aminobutyrate in Bacillus subtilis. Molecular Microbiology, 2002, 45(2): 569-583 (doi: 10.1046/j.1365-2958.2002.03036.x).


