Технологии разработки программы содействия принятию решения в диагностике заболеваний системы крови с использованием свёрточных искусственных нейронных сетей
Автор: Масликова Ульяна Владиславовна, Супильников Алексей Александрович
Журнал: Вестник медицинского института "РЕАВИЗ": реабилитация, врач и здоровье @vestnik-reaviz
Рубрика: Информационные технологии в медицине
Статья в выпуске: 5 (47), 2020 года.
Бесплатный доступ
В рамках исполнения работы исследованы технологии получения, обработки, сегментации и передачи микрофотографий по протоколу для последующего распознавания. Выполнено исследование технологий получения, обработки, сегментации и передачи микрофотографий для последующего распознавания. Отобраны наиболее перспективные алгоритмы машинного обучения, зарекомендовавшие себя в обработке медицинских изображений. Исследованы технологии анализа данных текстов медицинской документации. Изучены аспекты применения нейросети Watson для анализа семантики медицинских изображений. Изучены аспекты применения единого медицинского языка UMLS для нужд синдромальной диагностики по изучению медицинских текстов истории болезни на натуральном языке. Разработан интерфейс получения, обработки, сегментации и передачи микрофотографий на вход искусственной нейронной сети. Создан интерфейс первичного получения и обработки микрофотографий на базе платформы обработки медицинских изображений OMERO. Для отправки данных в режиме онлайн подготовлен демо-скрипт для jupiter. Разработан интерфейс передачи текстов медицинской документации системе распознавания семантики медицинского текста. Для анализа медицинских текстов в первом приближении использован сервис IBM Watson Annotator for Clinical Data. Создана база данных медицинских изображений микрофотограмм костного мозга для подготовки нейросети. Получены микрофотографии мазков костного мозга при разрешении ×600 в световой микроскопии (окраска гематоксилин-эозин) общим числом 3 500 цветных изображений 600×400 пикселей. Проведена разметка на 11 типов клеток костного мозга. Создана база данных медицинских текстов для подготовки нейросети. Подготовлена база данных медицинских текстов 167 пациентов для обучения нейросети в объеме 40000 слов. Проведена деперсонализация личных данных пациентов.
Микроскопия костного мозга, сапр, системы содействия принятию решения, анализ семантики текста, машинное зрение
Короткий адрес: https://sciup.org/143176768
IDR: 143176768 | УДК: 519.861-612.419 | DOI: 10.20340/vmi-rvz.2020.5.16
Текст научной статьи Технологии разработки программы содействия принятию решения в диагностике заболеваний системы крови с использованием свёрточных искусственных нейронных сетей
Микроскопическое исследование костного мозга играет важную роль в области диагностики и контроля основных заболеваний. Оно способно выявлять клинически значимые морфологические особенности кроветворных клеток, в том числе аномальные лейкоциты при лимфомах, лейкозах, дисплазиях и других заболеваниях [1– 2]. Однако до сих пор морфологическое профилирование клеток костного мозга в значительной степени опирается на методы ручной обработки мазка и визуального осмотра с ограничениями по контролю качества и экономической масштабируемости [3]. Считается, что на подготовку и интерпретацию мазка крови отрицательно влия- ют усталость и степень компетентности наблюдателя, ошибки распределения слайдов, ошибки статистической выборки, ошибки записи, а также трудоемкие процессы, требующие высококвалифицированных специалистов [4–5]. В связи с этим возник значительный интерес к разработке систем автоматизированной классификации цифровых изображений мазков периферической крови с высокой чувствительностью и специфичностью. Традиционно исследователи прилагают усилия к автоматизированному морфологическому дифференциальному учёту лейкоцитов. Они использовали неглубокие модели машинного обучения, которые полагаются на входные данные, полученные таким же образом, как и анализ морфологов.
Обычные подходы к машинному обучению, реализованные в классификации лейкоцитов, включают искусственные нейронные сети (ИНС) [6–7], поддерживаемые векторные машины (SVM) [6, 8–11], Naive Bayes Classifier [12, 13], линейный дискриминантный анализ (LDA) [14, 15] и многослойный перцептрон (MLP) [7, 16]. Для получения высокой эффективности классификации многие исследования используют технологии предварительной обработки изображений [6], сегментации объектов [17], а также извлечения и отбора признаков [18], что является предпосылками классификационных моделей. В жестко контролируемых условиях, например в [16], или при использовании небольших наборов данных, например в [7, 12], традиционные системы распознавания лейкоцитов часто достигают высокой классификационной точности.
Основная часть
Исследование технологий получения, обработки, сегментации и передачи микрофотографий костного мозга для последующего распознавания
В распознавании клеток костного мозга при помощи нейронной сети можно выделить три этапа: (I) сегментация объекта (объектов) от фона; (II) извлечение эффективных или отличительных признаков вручную; (III) классификационный дизайн. Эти неглубокие модели обучения, основанные на изучении небольших образцов, часто могут достичь хороших результатов, но в значительной степени зависят от точности сегментации и эффективности характеристик. Сама по себе надежная и устойчивая сегментация является нетривиальной проблемой. Любые ошибки превыше-ния/невыполнения сегментации отрицательно сказываются на общей производительности системы. Извлечение функции вручную также является очень важным шагом, и как количество, так и качество чувствительны к конечной производительности точности системы. Для изучения метода распознавания лейкоцитов, не содержащего сегментации изображений, Дан и др. [19] охарактеризовали лейкоциты с локальными дескрипторами и применили известный набор слов в качестве механизма объединения. В данном исследовании использовались детектор SIFT (Scale-Invariant Feature Transform [20]), детектор oFAST (Oriented Features from Accelerated Segment Text [21]) и детектор CenSurE (CENter SURround Extrema [22]) для получения дескрипторов ключевых точек или точек интереса, чтобы эти локальные дескрипторы могли быть представлены для 5 типов лейкоцитов (нейтрофил, эозинофил, базофил, моноцит и лимфоцит). Но точность классификации была неудовлетворительной, особенно для эозинофила и базофила. Все вышеперечисленные недостатки традиционных систем распознавания приводят к острой необходимости разработки более адаптивных и практичных высокоэффективных систем распознавания. Затем постепенно появляются исследования по распознаванию лейкоцитов, основанные на глубоком обучении.
Технологии глубокого обучения показали впечатляющие результаты при решении различных задач зрения, таких как классификация изображений, обнаружение объектов и семантическая сегментация. Суть технологии глубокого обучения заключается в том, что путь извлечения признаков не разрабатывается людьми, а изучается на основе данных с использованием процедуры обучения общего назначения. В области глубокого обучения, конволюцион-ные нейронные сети (CNN) достигли отличных результатов при анализе изображений. Относительно легко построить комплексную модель для использования конволю-ционных нейронных сетей на основе CNN. Более того, архитектура глубокого обучения на основе CNN позволяет избежать сложной ручной разработки функций и достичь желаемой производительности. Таким образом, подходы на основе CNN быстро развиваются в области распознавания лейкоцитов. Жао и др. [23] предложили систему автоматического обнаружения и классификации клеток костного мозга, где последние детектировались с точки зрения расположения ядра лейкоцита, а архитектура CNN (5 конволюционных слоев и 2 пуловых слоя) была спроектирована для извлечения признаков высокого уровня. Эта конструкция дала более ценную идею для решения проблемы распознавания лейкоцитов путем объединения детектирования и классификации их вместе, однако типы клеток, участвовавшие в исследованиях, были ограничены пятью общими типами, а точность для некоторых типов (эозинофил 70 % и лимфоцит 74,8 %) все еще нуждается в улучшении. Шахин и др. [24] предложили архитектуру глубокого обучения на основе CNN для распознавания 5 зрелых СБК (базофил, эозинофил, лимфоциты, моноциты и нейтрофил) и добились более высокой точности классификации, чем традиционные подходы к идентификации СБК. Кроме того, в снимках мазка костного мозга Чой и др. [25] использовали автоматизированную систему дифференциального подсчета лейкоцитов с использованием двухступенчатой конво-люционной нейронной сети. Двухступенчатая архитектура CNN разделила изображения на 10 типов серий созревания миелои-дов и эритроидов и добилась отличных результатов. Кроме того, основываясь на теории глубокого остаточного обучения и медицинских знаниях в области медицины, Цинь и др. [26, 27] представили метод мелкозернистой классификации лейкоцитов для микроскопических изображений. Предложенная нейронная сеть глубокого обучения была протестирована на наборе данных микроскопических изображений с 40 категориями лейкоцитов и достигла желаемых результатов. Из вышеперечисленных исследований можно заметить, что объект исследования варьировал от 5 типов периферической крови до 10 или 40 типов костного мозга, а объём тренировочного набора варьировался от 2174, 2551 до 92480 изображений. Хотя глубокий CNN и традиционные методы машинного обучения показали хорошие результаты в классификации изображений кровяных клеток, они не в состоянии в полной мере использовать долгосрочную зависимость между определенными ключевыми особенностями изображений и этикеток изображений. Для решения этой проблемы была разработана основа CNN-RNN (Recursive Neural Network – рекурсивная нейронная сеть) [28], предназначенная для углубления понимания содержания изображений и изучения структурированных особенностей изображений. Большинство методов, упомянутых выше, разработаны с точки зрения классификации изображений, т.е. рассматривают распознавание клеток как классификационную задачу [24–27, 29–30], которая должна обеспечить наличие во входном изображении кандидатов на объекты по сегментации, а количество объектов не превышает одного за счет обрезки изображения вручную или сложного шага сегментации. Эти классификационно-задачные методы, как правило, направлены на распознавание пяти типов зрелых лейкоцитов, обычно встречающихся в периферической крови, и начинают классификацию с обрезанных формами лейкоцитов, что приводит к неудобствам в реальных приложениях.
Общее обнаружение объектов, направленное на определение местонахождения экземпляров объектов из большого числа предопределенных категорий в изображениях, является одной из самых фундаментальных и сложных проблем компьютерного зрения [31–32]. Тем не менее, несмотря на свой потенциал, этот объектно-обнаруженный подход с ориентацией на задачи раньше не применялся для решения проблемы распознавания лейкоцитов. Поэтому мы пытаемся решать проблему распознавания лейкоцитов с точки зрения распознавания объектов, а не классификации изображений, в надежде правильно определить, какой тип и где находится лей- коцит в изображении ресурса, захваченном непосредственно с микроскопа. Существуют две устоявшиеся серии в качестве представителей методов глубокого изучения: двухступенчатая система обнаружения, которая включает в себя этап предварительной обработки регионального предложения, что делает общую систему двухступенчатой; и одноступенчатая система обнаружения, или свободная система регионального предложения, которая не разделяет предложения по обнаружению, что делает общую систему одноступенчатой с элегантной манерой работы от конца до конца. Типичными архитектурами двухступенчатого алгоритма являются регионально-адаптированные CNN (R-CNN) [33], пул пространственных пирамид в глубоких свёрточных сетях (SPP-сеть) [34], быстрая R-CNN [35], ускоренная R-CNN [36], региональные полностью свёрточные сети (R-FCN) [37] и маска R-CNN [38], в то время как DetectorNet [39], MultiBox [30], OverFeat [31], алгоритм «ты смотришь только раз» (YOLO) [22], YOLOv2 [33], YOLOv3 [34] и однокадровый многоблочный детектор (SSD) [25] используются для одноступенчатого алгоритма. Среди различных вышеперечисленных конвейеров обнаружения объектов, SSD [35] является относительно быстрым и надежным для масштабирования вариаций, потому что он использует несколько слоев свертки и сочетает в себе все предсказания из множества функциональных карт с разным разрешением для обнаружения объектов. YOLO [22] является унифицированным детекторным кастинго-вым детектором обнаружения объектов как регрессионная проблема от пикселей изображения до пространственно-разделенных ограничительных ящиков и связанных с ними классовых вероятностей. Как инкрементная версия улучшения YOLO, YOLOv3 [34] работает значительно быстрее, чем другие методы обнаружения с сопоставимой производительностью. А именно, до сих пор в YOLOv3 достигнут наилучший компромисс между точностью детектиро- вания и вычислительной скоростью. Лян и др. [14] рассматривали распознавание мочевых частиц как обнаружение объектов и использовали более быстрые методы RCNN [32] и SSD [34], наряду с их вариантами, для распознавания мочевых частиц. Более того, результат их исследования был обнадеживающим.
Мы используем механизм глубокого трансфертного обучения (тонкая настройка соответствующих моделей до обучения, а не с нуля), а также проводим экспериментальный анализ для демонстрации влияния различных факторов. Подробно, при использовании SSD или YOLOv3, мы настраиваем несколько параметров, в том числе масштабы коробок по умолчанию, размер входных изображений и опорной сети для повышения производительности распознавания клеток костного мозга.
База микроскопических изображений мазков костного мозга построена нами с использованием микрофотографий мазков костного мозга при разрешении х600 в световой микроскопии (окраска гематоксилин-эозин). Все 3 500 аннотированных цветных изображений имеют разрешение 600 x 400 пикселей, размечены и включают 11 типов клеток костного мозга – бласты (Blast), промиелоциты (PRO), миелоциты (MYE), метамиелоциты (MET), палочкоядерный нейтрофил (bNEU), сегментированный нейтрофил (sNEU), лимфоциты (LYM), моноциты (MO), эозинофил (EO), базофил (BA) и реактивные лимфоциты (rLYM), зародышевые эритроциты (NRBC), мегакариоциты (MGKR) и артефакт (Artefact). 7 % выборки были случайно отобраны для формирования тестового набора данных, а оставшиеся изображения – для тренировочного.
Исследование технологий анализа данных текстов медицинской документации
Электронные медицинские карты (ЭМК) широко распространены в здравоохранении. Объём хранимых в них данных возрастает экспоненциально. При этом типичная
ЭМК содержит несколько сотен неструктурированных простых текстовых клинических записей, а также большие объемы по-луструктурированных данных, таких как назначенные медикаменты, значения лабораторных тестов, процедур и жизненных показателей. Итак, сама технология позволяет записывать каждый аспект ухода за пациентом, что делает его (совершенно непреднамеренно) сложным для понимания. Так как суммирование вручную отнимает много времени и предрасположено к ошибкам, существует настоятельная необходимость в автоматических методах.
Задача состоит в том, чтобы максимизировать информационное покрытие при минимизации избыточности в ограниченном пространстве. Развивающиеся точные аннотации записей пациентов требуют сложного подхода. Первый успех реализации технологии искусственного интеллекта в медицине принадледжит команде IBM Watson [31].
Так как запись о пациенте содержит различные наборы данных о пациенте и его лечении, т.е. о проблемах, лекарствах, лабораториях, процедурах и операциях, аллергии и т.д., естественный способ достижения охват и краткость, необходимые для подведения итогов, должны быть начаты с агрегатами этих наборов, которые мы называем клиническими.
Элементы каждого из этих агрегатов сами по себе могут быть резюмированы до некоторого уровня абстракции, как это концептуально отражено в [23]. Например, результаты лабораторного теста могут быть организованы, преобразованы и истолкованы таким образом, что резюме показывает последнее значение и указание на то, является ли оно сейчас или имеет когда-либо был, вне нормального диапазона. Следующая ключевая часть обобщения – это клинические отношения, которые определяют семантические отношения между элементами агрегатов. Например, решается проблема одним или несколькими лекарствами. Ни данные по проблеме агрегат, ни агрегат данных по лекарствам не содержит этого важная семантическая ассоциация. Эти отношения не являются непосредственно присутствующими в EMR, но они являются результатом врачебного суждения. Следующим элементом модели является схожесть элементов в совокупности данных. Атрибут близости определяет, как тесно связан элемент с другими элементами совокупности. Например, для лекарственного агрегата – клинически значимое пространство для определения близости состоит из фармакологических механизмов лекарства и классов фармакологического воздействия на физиологию человека.
Центральным агрегатом обобщения является формализованный список выявленных синдромов, и поэтому мы ссылаемся на это обобщение как на резюме пациента, ориентированного на проблемы.
Обобщение ЭМК пациента в Watson состоит из следующих агрегатов данных:
-
- сгенерированный список синдромов;
-
- медикаменты;
-
- лабораторные исследования;
-
- процедуры;
-
- жизненные показатели;
-
- таймлайн (расписание) осмотров пациентов;
-
- социальный анамнез, аллергия и демография.
Суммаризация автоматически генерирует следующие клинические семантические данные:
-
- взаимосвязь между списками проблем и элементами других агрегатов клинических данных;
-
- клинически значимая группировка элементов в каждой из агрегированных данных;
-
- категоризация встреч с пациентами на основе специальности врача;
-
- отфильтрованные и/или приоритетные сводные данные на основе специальности врача по резюме.
В целях анализа семантики используется единая система медицинского языка
UMLS [25], определяющая концепцию уникальных идентификаторов (УИ). В настоящее время в медицинской текстовой аналитике широко используется программное обеспечение UMLS Metamap [16] для сопоставление простого текста с концепциями UMLS, высокоэффективным оказалось также использование Watson NLP.
Естественный язык компонентов обработки Ватсон включает в себя парсер языка, концепт-картограф, детектор отрицания и связанные с ним технологии. В дополнение к тексту клинических примечаний, необходим анализ UMLS-концепций для записи полуструктурированных данных EMR, таких как название лекарств. Важная часть подведения итогов состоит в том, чтобы установить клинически значимую взаимосвязь между возникающими медицинскими проблемами и элементами других агрегированных клинических данных. Подведение итогов необходимо для количественной оценки пары клинических связей между проблемами и лекарствами, лабораториями и процедурами. Кроме того, латентносемантический анализ [28] применялся к медицинскому набору, он может также предоставить ассоциативный балл между медицинскими концепциями. Еще более точный подход под названием «Распределительное Обнаружение Отношений», включающее в себя дистрибутивную семантику [26], разрабатываемую для подсчета очков ассоциации между медицинскими концепциями.
Клинический групповой анализ на лекарства начинается с неупорядоченного списка лекарств в ЭМК, и заканчивается списком клинически назначенных препаратов, в котором сопутствующие препараты приведены вместе. Сначала анализ сопоставляет каждое лекарство с набором препаратов государственного реестра, включая его ингредиенты, химическую структуру, форму дозы, механизм действия и фармакокинетику. Следующий шаг в анализе состоит в кластеризации лекарственных средств на базе сходства их классов. Кла- стеризация происходит снизу вверх иерархическим методом с использованием косинусного сходства их класса. Аналогичный групповой анализ проводится для медицинских проблемы с использованием дескрипторов MeSH [35]. Другой анализ, который перспективен в использовании, состоит в классификации клинических записей по следующим категориям типа практики, которая его создала, т.е. был ли он создан врачом первичной медикосанитарной помощи, специалистом, медсестрой или врачем отделения скорой помощи. Метаданные клинических записей (описание) в ЭМП не являются надежным средством идентификации его категории примечаний. Однако при представлении графика встречи пациента с врачами это полезно, чтобы правильно классифицировать встречи по практике, потому что такая сгруппированная временная шкала позволяет врачу просмотреть сводку.
Алгоритм машинного обучения используется для идентификации заметки. Функции машинного обучения, извлеченные из каждой заметки для этой цели включают медицинские концепции UMLS, встречающиеся в тексте примечания, существуют ли определенные неофициальные разделы (например, предыдущий медицинский анамнез, оценка и план) в записке, и любую информацию о врачах-специалистах в записке.
В настоящее время подготовлен интерфейс HL7 для передачи данных ЭМК 167 пациентов в сервис IBM Watson Knowledge Studio для анализа и смысловой группировки деперсонифицированных медицинских документов.
Разработка интерфейса получения, обработки, сегментации и передачи микрофотографий на вход искусственной нейронной сети
Для подготовки, обработки и сегментации микрофотографий на вход искусственной нейронной сети используется интеграция решений с открытым исходным кодом.
В качестве инструмента первичного получения и обработки микрофотографий использована платформа обработки медицинских изображений OMERO. При помощи OMERO Public Api планируется экспорт кадров с микрофотограммами клеток костного мозга в приложение Python, c пакетом scikit-image. После автоматической сегментации врач-оператор корректирует результат сегментации и аннотирует ее с учетом ранее созданной базы типов клеток костного мозга. Далее данные сохраняются и распределяются в обучающий или тестовый набор данных. Для отправки данных в режиме онлайн подготовлен демо-скрипт для jupiter.
Разработка интерфейса передачи текстов медицинской документации системе распознавания семантики медицинского текста
Для анализа медицинских текстов в первом приближении использован сервис IBM Watson Annotator for Clinical Data – это сервис IBM Cloud, работающий на базе искусственного интеллекта, который обеспечивает получение значимой информации из неструктурированных данных, созданных специально для сферы здравоохранения и медико-биологических наук. Annotator for Clinical Data извлекает ключевые клинические понятия из текста на естественном языке, такие как заболевания, лекарства, аллергии и процедуры. Эти особенности обогащены глубоким контекстуальным пониманием, а также значениями ключевых клинических признаков, чтобы обеспечить более полное представление о доступных данных. Потенциальные источники данных включают различные источники в области здравоохранения и наук о жизни, такие как клинические примечания, резюме выписок, протоколы клинических испытаний и литературные данные. Клинические замечания
Функция клинического понимания – это готовая к использованию возможность аннотации в программе Watson Annotator for Clinical Data, которая предоставляет важ- ную контекстуальную информацию о проблемах, процедурах и лекарствах, указанных в тексте. Определение и кодирование клинических концепций с поддержкой SNOMED CT, RxNorm, ICD-10-PCS, CPT, NCI, MESH и LOINC.
Сервис NLP в медицинской области включает в себя различные аннотаторы для обнаружения метаданных (таких как сущности, концепции, значения концепций, отрицательные диапазоны, гипотетические диапазоны) и набор аннотаторов, которые обнаруживают, нормализуют и кодируют медицинские и социальные данные, полученные из неструктурированных клинических данных. Для анализа неструктурированных данных из одного запроса можно использовать несколько аннотаторов.
Создание базы данных медицинских изображений микрофотограмм костного мозга для подготовки нейросети
Получены микрофотографии мазков костного мозга при разрешении х600 в световой микроскопии (окраска гематоксилин-эозин) общим числом 3 500 цветных изображений 600 x 400 пикселей. Проведена разметка на 11 типов клеток костного мозга – бласты (Blast), промиелоциты (PRO), миелоциты (MYE), метамиелоциты (MET), палочкоядерный нейтрофил (bNEU), сегментированный нейтрофил (sNEU), лимфоциты (LYM), моноциты (MO), эозинофил (EO), базофил (BA) и реактивные лимфоциты (rLYM), зародышевые эритроциты (NRBC), мегакариоциты (MGKR) и артефакт (Artefact). Произведено деление выборки: 7 % для формирования тестового набора данных, а оставшиеся изображения – для тренировочного.
На рис. 1 представлен внешний вид первичной микрофотограммы с разметкой клеток.
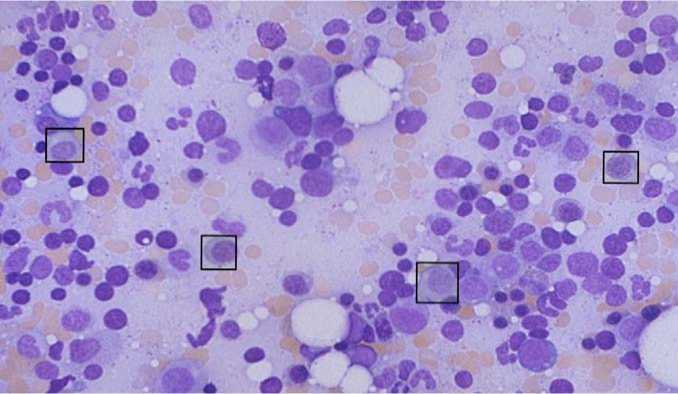
Рис. 1. Микрофотограмма мазка костного мозга, х600
На рис. 2 представлен скриншот папки с фотографиями костного мозга, использованными для обучения нейросети.
Создание базы данных медицинских текстов для подготовки нейросети.
Подготовлена база данных медицинских текстов 167 пациентов для обучения нейросети в объеме 40000 слов. Проведена деперсонализация личных данных пациентов. Проведено разделение текстовых данных на структурные компоненты, удаление повторяющихся фрагментов. Проведена проверка текстов на наличие синтаксических ошибок. Имена файлов приведены к ID пациентов.
На рис. 3 показан пример распознавания структуры текста медицинского документа в окне приложения Watson Annotator for Clinical Data.
Заключение
На первом этапе исполнения проекта исследованы технологии получения, обработки, сегментации и передачи микрофотографий по протоколу для последующего распознавания. Выполнено исследование технологий получения, обработки, сегментации и передачи микрофотографий для последующего распознавания. Проанализированы 123 источника научной литературы по данной тематике, проанализированы основные технологии обработки медицинских изображений. Отобраны наиболее перспективные алгоритмы машинного обучения, по данным литературы, зарекомендовавшие себя в обработке медицинских изображений. Исследованы технологии анализа данных текстов медицинской документации. Изучены аспекты применения нейросети Watson для анализа семантики медицинских изображений. Изучены аспекты применения единого медицинского языка UMLS для нужд синдромальной диагностики по изучению медицинских текстов истории болезни на натуральном языке. Проведена оценка используемых алгоритмов, их эффективности. Проанилизирова-ны перспективы использования IBM Watson Knowledge Studio для обработки текстов медицинских документов.
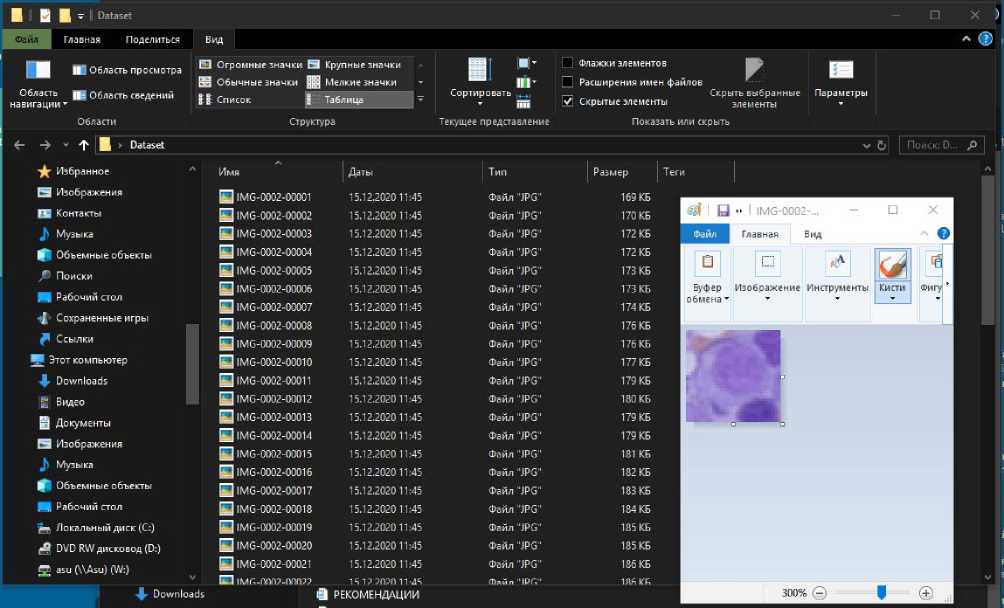
Рис. 2. Скриншот микрофотографий костного мозга – датасета, использующегося для обучения искусственной нейронной сети
IBM Watson Annotator for Clinical Data |
The Annotator for Clinical Data can be used to surface insights in a variety of healthcare scenarios. Select a use case to see relevant insights extracted from unstructured text.

COVID MrdKil I.itrritur
of twin June 17. 1996
modcai history ho was bom during the hrsl pregnancy. which had no particular symptoms I ixgont. indopondont belh with a long
waterless interval Weight of the body al twin was С55Э g, ri«ght » cm Dovetopment according to her age Pasi medical tvstory СТОЙ
on Л u’/JA adnwuslrabon
Pasi modcai history Side since November 5. when he body tornporaturo rose to <Э 2 о C and had । JIEJBiSHS took СЕИЯ1
♦2 on. spleen *8 cm), hard breathmg w Рис. 3. Аннотирование текста медицинского документа – автоматический перевод нейросетью deepl, аннотация сервисом IBM Watson Annotator for Clinical Data Разработан интерфейс получения, обработки, сегментации и передачи микрофотографий на вход искусственной нейронной сети. Создан интерфейс первичного получения и обработки микрофотографий на базе платформы обработки медицинских изображений OMERO. При помощи OMERO Public Api настроен экспорт кадров с микрофотограммами клеток костного мозга в приложение Python, c пакетом scikit-image. Подготовлен инструмент сохранения и распределения в обучающий или тестовый набор данных. Для отправки данных в режиме онлайн подготовлен демо-скрипт для jupiter. Разработан интерфейс передачи текстов медицинской документации системе распознавания семантики медицинского текста. Для анализа медицинских текстов в первом приближении использован сервис IBM Watson Annotator for Clinical Data. Проведена оценка определения и кодирования клинических концепций с поддержкой SNOMED CT, RxNorm, ICD-10-PCS, CPT, NCI, MESH и LOINC. Разработана технология перевода аннотаторов на английский язык сервисом deepl.com. Создана база данных медицинских изображений микрофотограмм костного мозга для подготовки нейросети. Получены микрофотографии мазков костного мозга при разрешении х600 в световой микроскопии (окраска ге-матоксилин-эозин) общим числом 3 500 цветных изображений 600x400 пикселей. Проведена разметка на 11 типов клеток костного мозга – бласты (Blast), промиелоциты (PRO), миелоциты (MYE), метамиелоциты (MET), палочкоядерный нейтрофил (bNEU), сегментированный нейтрофил (sNEU), лимфоциты (LYM), моноциты (MO), эозинофил (EO), базофил (BA) и реактивные лимфоциты (rLYM), зародышевые эритроциты (NRBC), мегакариоциты (MGKR) и артефакт (Artefact). Произведено деление выборки: 7 % для формирования тестового набора данных, а оставшиеся изображения – для тренировочного. Создана база данных медицинских текстов для подготовки нейросети. Подготовлена база данных медицинских текстов 167 пациентов для обучения нейросети в объеме 40000 слов. Проведена деперсонализация личных данных пациентов. Проведено разделение текстовых данных на структурные компоненты, удаление повторяющихся фрагментов. Проведена проверка текстов на наличие синтаксических ошибок. Имена файлов приведены к ID пациентов. Таким образом, существующие средства позволяют создать интегральный сервис распознавания изображений и текстов медицинских изображений с использованием технологий искусственного интеллекта. Дальнейшей задачей исполнения проекта будет интеграция данных сегментации обеих сетей и построение интерфейса выявления патологии костного мозга.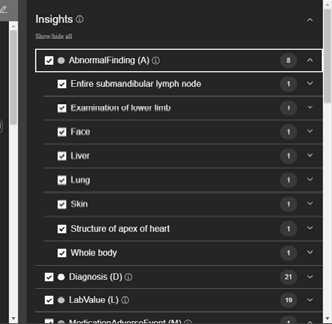
Список литературы Технологии разработки программы содействия принятию решения в диагностике заболеваний системы крови с использованием свёрточных искусственных нейронных сетей
- Bain, Barbara J. Diagnosis from the Blood Smear. New England Journal of Medicine. 2005;353(5):498-507. pmid:16079373
- Gallagher PG. Red Cell Membrane Disorders. Hematology. 2005;2005(1):13-18.
- Durant Thomas JS, Olson Eben M., Schulz Wade L, Torres R. Very Deep Convolutional Neural Networks for Morphologic Classification of Erythrocytes. Clinical Chemistry. 2017;63(12):1-9.
- Ford J. Red blood cell morphology. International Journal of Laboratory Hematology. 2013;35:351-357. pmid:23480230
- Ceelie H, Dinkelaar RB, van Gelder W. Examination of peripheral blood films using automated microscopy; evaluation of Diffmaster Octavia and Cellavision DM96. J Clin Pathol. 2007;60:72-79. pmid:16698955
- Seyed HR, Hamid SZ. Automatic recognition of five types of white blood cells in peripheral blood. Computerized Medical Imaging and Graphics. 2011;35:333-343. pmid:21300521
- Sedat N, Deniz K, Tuncay E, Murat HS, Osman K, Yavuz E. Automatic segmentation, counting, size determination and classification of white blood cells. Measurement. 2014;55:58-65.
- Lorenzo P, Giovanni C, Cecilia DR. Leucocyte classification for leukaemia detection using image processing techniques. Artificial Intelligence in Medicine. 2014;62:179-191. pmid:25241903
- Agaian S, Madhukar M, Chronopoulos AT. Automated Screening System for Acute Myelogenous Leukemia Detection in Blood Microscopic Images. IEEE SYSTEMS JOURNAL. 2014;8:995-1004.
- ALFEREZ S, MERINO A, BIGORRA L, RODELLAR J. Characterization and automatic screening of reactive and abnormal neoplastic B lymphoid cells from peripheral blood. INTERNATIONAL JOURNAL OF LABORATORY HEMATOLOGY. 2016;38:209-219. pmid:26995648
- Morteza M, Ahmad M, Nasser S, Saeed K, Ardeshir T. Computer aided detection and classification of acute lymphoblastic leukemia cell subtypes based on microscopic image analysis. Microscopy Research and Technique. 2016;79:908-916. pmid:27406956
- Mathur A, Tripathi AS, Kuse M. Scalable system for classification of white blood cells from Leishman stained blood stain images. Journal of pathology informatics. 2013;4:15. Available from: http://www.jpathinformatics.org/text.asp?2013/4/2/15/109883
- Jaroonrut P, Charnchai P. Segmentation of white blood cells and comparison of cell morphology by linear and naive Bayes classifiers. BioMed. Eng. OnLine. 2015; 14-63.
- Ramesh N, Dangott B, Salama ME, Tasdizen T. Isolation and two-step classification of normal white blood cells in peripheral blood smears. Journal of pathology informatics. 2012;3:3-13.
- Santiago A, Anna M, Laura B, Luis M, Magda R, Jose R. Automatic Recognition of Atypical Lymphoid Cells From Peripheral Blood by Digital Image Analysis. Am J Clin Pathol. 2015;143:168-176. pmid:25596242
- Su MC, Cheng CY, Wang PC. A neural-network-based approach to white blood cell classification. The Scientific World Journal. 2014;1-9.
- Tamalika C. Accurate segmentation of leukocyte in blood cell images using Atanassov's intuitionistic fuzzy and interval Type II fuzzy set theory. Micron. 2014;61:1-8. pmid:24792441
- Alferez S, Merino A, Bigorra L, Rodellar J. Characterization and automatic screening of reactive and abnormal neoplastic B lymphoid cells from peripheral blood. Jnl. Lab. Hem. 2016;38:209-219.
- Dan L-P, V. Javier T, Filiberto P. Recognizing white blood cells with local image descriptors. Expert Systems With Applications. 2019;115:695-708.
- Lowe DG. Distinctive image features from scale-invariant keypoints. International Journal of Computer Vision. 2004;60(2):91-110.
- Rublee E, Rabaud V, Konolige K, Bradski G. ORB: An efficient alternative to SIFT or SURF. In Proceedings of the international conference on computer vision. 2011;2564-2571.
- Agrawal M, Konolige K, Blas MR. CenSurE: Center surround extremas for realtime feature detection and matching. In Proceedings of the European conference on computer vision. 2008;102-115.
- Zhao JW, Zhang MS, Zhou ZH, Chu JJ, Cao FL. Automatic detection and classification of leukocytes using convolutional neural networks. Medical & Biological Engineering & Computing. 2016 Nov 07. https://doi.org/10.1007/s11517-016-1590-x
- Shahin AI, Guo YH, Amin KM, Sharawi AA. White Blood Cells Identification System Based on Convolutional Deep Neural Learning Networks. Computer Methods and Programs in Biomedicine. 2019;168:69-80. pmid:29173802
- Choi JW, Ku Y, Yoo BW, Kim J-A, Lee DS, Chai YJ, et al. White blood cell differential count of maturation stages in bone marrow smear using dual-stage convolutional neural networks. PLoS ONE 2017; 12(12):e0189259. pmid:29228051
- Jiang M, Cheng L, Qin FW, Du L, Zhang M. White Blood Cells Classification with Deep Convolutional Neural Networks. International Journal of Pattern Recognition and Artificial Intelligence. 2018;32(9):1857006.
- Qin FW, Gao NN, Peng Y, Wu ZZ, Shen SY, Artur G. Fine-grained leukocyte classification with deep residual learning for microscopic images. Computer Methods and Programs in Biomedicine. 2018;162:243-252. pmid:29903491
- Liang GB, Hong HC, Xie WF, Zheng LX. Combining convolutional neural network with recursive neural network for blood cell image classification. IEEE Access. 2018;6:36188-36197.
- Amjad R, Naveed A, Tanzila S, Syed IR, Zahid M, Hoshang K. Classification of acute lymphoblastic leukemia using deep learning. Microsc Res Tech. 2018;1-8.
- Tiwari P, Qian J, Li QC, Wang BY, Gupta D, Khanna A, et al. Detection of Subtype Blood Cells using Deep Learning, Cognitive Systems Research. 2018 August 25. pii: S1389-0417(18)30376-0. https://doi.org/10.1016/j.cogsys.2018.08.022
- Liu L, Ouyang WL, Wang XG, Paul F, Chen J, Liu XW, Matti P. Deep Learning for Generic Object Detection: A Survey. Preprint. Available from: arXiv: 1809.02165v1. Cited 6 Sep 2018.
- Zou ZX, Shi ZW Guo YH, and Ye JP. Object Detection in 20 Years: A Survey. Preprint. Available from: arXiv: 1905.05055v1. Cited 13 May 2019.
- Girshick R, Donahue J, Darrell T, Malik J. Rich feature hierarchies for accurate object detection and semantic segmentation. Preprint. Available from: arXiv:1311.2524v3 Cited 7 May 2014.
- He K, Zhang X, Ren S, Sun J. Spatial pyramid pooling in deep convolutional networks for visual recognition. In: European Conference on Computer Vision. 2014:346-361.
- Girshick R. Fast R-CNN. In: Proceedings of the IEEE International Conference on Computer Vision. 2015:1440-1448.


