Трансгенные растения томата (Solanum lycopersicum L.): прямые методы введения генов и факторы, влияющие на эффективность трансформации (обзор)
Автор: Михель И.М., Халилуев М.Р.
Журнал: Сельскохозяйственная биология @agrobiology
Статья в выпуске: 3 т.57, 2022 года.
Бесплатный доступ
Томат ( Solanum lycopersicum L.) - важнейшая продовольственная культура, которая также находит широкое применение в качестве модельного объекта в различных молекулярно-генетических исследованиях, затрагивающих вопросы вегетативного развития и репродуктивной биологии, механизмов устойчивости растений к абиотическим и биотическим стрессам, ассоциативного симбиоза с микроорганизмами и многие другие, имеющие как фундаментальное, так и прикладное значение. Получение трансгенных растений томата, экспрессирующих чужеродные гетерологичные гены, а также растений с индуцированным сайленсингом или нокаутом собственных генов, - важная составляющая исследований в современной физиологии растений. Существует два принципиально отличающихся друг от друга подхода для введения чужеродной ДНК в геном томата. Первый (метод агробактериальной, или Agrobacterim- опосредованной, трансформации) основан на естественном механизме заражения растений бактериальным патогеном рода Agrobacterim ( A. tumefaciens или A. rhizogenes ) и опосредованном им переносе чужеродной ДНК в растительный геном. Второй подход основан на непосредственной доставке чужеродной ДНК в растительную клетку сквозь плазмалемму с помощью химических веществ (Ca2+, полиэтиленгликоль - ПЭГ) или физических воздействий (электрический импульс или повышенное давление) (так называемые прямые методы генетической трансформации томата). При этом трансгенные растения томата можно получать как классическим способом с использованием метода культуры изолированных органов и тканей in vitro, так и без него (трансформация in planta). В представленном обзоре рассмотрены классические методы прямого введения чужеродной ДНК в геном томата (химически опосредованная трансфекция, электропорация протопластов, микроинъекция, биобаллистическая трансформация), а также методы трансформации томата in planta (метод пыльцевых трубок или pollen-tube pathway, электропорация зародышей зрелых семян) и проведен подробный анализ физических, генетических и физиологических факторов, влияющих на эффективность трансформации (ЭТ). Обсуждаются особенности получения растений томата как с транзиентной экспрессией трансгена, так и со стабильно наследуемой вставкой в ядерный или пластидный геном. Отдельно рассмотрено применение прямых методов генетической трансформации для доставки различных систем геномного редактирования (ZFNs, TALEN, CRISPR/Cas, редакторов оснований, prime editing), получивших широкое распространение в последние 5 лет.
Solanum lycopersicum l, электропорация, пэг-опосредованная трансформация, микроинъекция, биобаллистическая трансформация, трансформация in planta, геномное редактирование
Короткий адрес: https://sciup.org/142236340
IDR: 142236340 | УДК: 635.64:579.254.2:581.143.6 | DOI: 10.15389/agrobiology.2022.3.518rus
Текст обзорной статьи Трансгенные растения томата (Solanum lycopersicum L.): прямые методы введения генов и факторы, влияющие на эффективность трансформации (обзор)
Томат ( Solanum lycopersicum L.) — важнейшая продовольственная культура, занимающее второе место после картофеля среди сельскохозяйственных овощных растений по валовому сбору товарной продукции. Так, по данным Продовольственной и сельскохозяйственной организации Объединенных Наций (FAO), мировой валовый сбор плодов томата составил в 2020 году около 186,8 млн т при выращивании на площади 5,1 млн га, из этих показателей на долю России приходилось 2,9 млн т (около 1,6 %) при площади 80,7 тыс. га (1). В России томат выращивают в различных агроэкологических условиях как в открытом, так и защищенном грунте (соответственно около 60 и 40 % валового сбора) (2, 3). Основные зоны возделывания этой культуры сосредоточены преимущественно в южных регионах страны (Краснодарский и Ставропольский края, а также Поволжский и Центрально-Черноземный районы), территории которых в большей или
∗ Работа выполнена при финансовой поддержке Министерства науки и высшего образования Российской Федерации в рамках государственного задания ¹ 0431-2022-0003.
меньшей степени подвержены первичному и/или вторичному засолению, а также другим эдафическим стрессам (4). Так, более 31 % почв в Астраханской области характеризуются высокой степенью засоления (концентрация ионов Na+ и SO 4 2 - достигает соответственно 7,1 и 12,5 мМ на 100 г почвы), а также около 20 % почв представлены солонцовыми комплексами (5). Кроме того, у томата насчитывается более 45 инфекционных болезней бактериальной, грибной, вирусной и вироидной этиологии, которые в различной степени распространены на территории Российской Федерации (6, 7). Как следствие, повышение устойчивости к абиотическим и биотическим стрессовым факторам — одно из приоритетных требований, предъявляемых к современным сортам и гибридам томата для реализации потенциальной урожайности. Поэтому необходимо постоянное расширение сортимента культуры с использованием как традиционных селекционных подходов, так и современных методов биотехнологии и генной инженерии (8-10).
Помимо большой практической значимости томат находит широкое применение в качестве модельного объекта в различных фундаментальных исследованиях, затрагивающих вопросы вегетативного развития и репродуктивной биологии (11, 12), изучения механизмов устойчивости растений к абиотическим и биотическим стрессам (9, 10, 13), ассоциативного симбиоза с микроорганизмами (14, 15), мейотической рекомбинации (16) и многие другие. Выбор такого модельного объекта обусловлено тем, что томат — относительно неприхотливое в выращивании самоопыляемое растение с коротким вегетационным периодом, высоким репродуктивным потенциалом, способное также к быстрому вегетативному размножению и отдаленной гибридизации с некоторыми видами рода Solanum (17). Культурный томат и родственные ему дикорастущие виды — диплоиды, кариотип которых представлен 24 хромосомами (2 n = 2½12 = 24). Кроме того, томат служит удобным модельным объектом благодаря большому числу морфологических признаков, четко идентифицируемых на разных этапах онтогенеза (18, 19), а также наличию подробных генетических и молекулярных карт (20) (в особенности после полного секвенирования генома томата в 2012 году) (21) и разработанных эффективных и воспроизводимых протоколов культивирования изолированных тканей in vitro (10, 22). Перечисленные факторы обеспечили широкое распространение трансгенных растений томата в качестве экспериментальной модели для изучения роли экспрессии гетерологичных генов различного происхождения в основополагающих биологических процессах.
Накопленные многочисленные данные свидетельствуют, что различные генно-инженерные стратегии позволяют значительно ускорить создание новых генотипов томата с признаками, которые трудно или невозможно достичь с помощью традиционной селекции. Так, например, повышения устойчивости растений томата к фитопатогенам возможно добиться посредством гибридизации томата с родственными дикорастущими видами, например S. pimpinellifolium L. и S. habrochaites S. Knapp & D.M. Spooner, с последующими многочисленными беккроссами (23, 24). Однако в таких селекционных программах могут участвовать только сравнительно близкородственные виды, способные к скрещиванию с культурным томатом; при этом процесс занимает до 15-20 лет (25). Растения S. lycopersicum L. с повышенной устойчивостью к грибным и бактериальным патогенам можно получать с применением различных генно-инженерных стратегий: экспрессией гетерологичных генов PR-белков (pathogenesis-related proteins — белки, синте- зируемые в растительной клетке при атаке патогена) и антимикробных пептидов растительного, животного и грибного происхождения; метаболической инженерией фитоалексинов и гормонов; использованием генов устойчивости (R-генов); ингибированием токсичных продуктов патогена; лигни-фикацией растительной клеточной стенки; активацией защитных реакций растения и др.) (9). При этом длительность селекционного процесса значительно сокращается.
Генетической трансформации может быть подвергнут как ядерный, так и пластидный геном. Последний вариант имеет ряд несомненных преимуществ, поскольку чужеродные гены, интегрированные в пластидный геном, характеризуются повышенной экспрессией за счет особенностей пластидной ДНК (26, 27) — большого числа копий пластомных генов на клетку (особенно в случае гомоплазмии) (26, 28), высокого уровня экспрессии трансгена вне зависимости от положения инсерции в пластидной ДНК из-за отсутствия компактной укладки хроматина (30); отсутствия эпигенетических эффектов и обусловленного ими сайленсинга трансгенов (29, 30). Все эти факторы позволяют довести выход продукта экспрессии транспластом-ных генов до 40 % от количества общего растворимого белка (ОРБ) (28, 29, 31). Таким образом, транспластомные растения служат перспективными биореакторами для производства гетерологичных белков медицинского и ветеринарного назначения (32-34). Также в пластидах успешно экспрессируются полицистронные кассеты, что упрощает ко-трансформацию одной мишени множеством разных генов (29). Несомненным экологическим преимуществом локализации трансгена в пластидах является наследование пла-стома по материнской линии, что приводит к отсутствию трансгена в пыльце и невозможности генетического загрязнения популяции (29, 33).
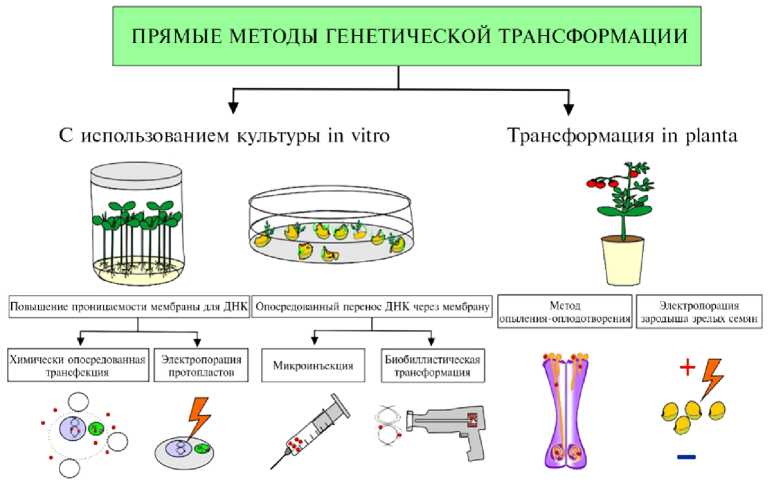
Классификация прямых методов генетической трансформации растений томата.
Существуют два принципиально отличающихся друг от друга подхода для введения чужеродной ДНК в геном томата (рис.). Первый (метод агробактериальной, или Agrobacterim-опосредованной, трансформации) базируется на естественном механизме заражения растений бактериальным патогеном рода Agrobacterim (A. tumefaciens или A. rhizogenes) и опосредованном им переносе чужеродной ДНК в растительный геном (35-37). Второй подход (см. рис.) основан на непосредственной доставке чужеродной ДНК в растительную клетку сквозь плазмалемму с помощью химических веществ (Ca2+, полиэтиленгликоль — ПЭГ) или физических воздействий (электрического импульса или повышенного давления) (так называемые прямые методы генетической трансформации томата). При этом трансгенные растения можно получать как классическим способом с использованием метода культуры изолированных органов и тканей in vitro, так и без него (трансформация in planta) (см. рис.). Вне зависимости от метода, применяемого для введения чужеродной ДНК в геном томата, процесс интеграции носит случайный характер (38).
Следует отметить, что каждый из прямых методов введения трансгена в геном томата имеет как преимущества, так и недостатки (39), однако все эти приемы применяются значительно реже, чем агробактериальная трансформация. Основные причины заключаются в их низкой эффективности (особенно в случае химически опосредованной трансфекции, электропорации протопластов и микроинъекции), необходимости наличия специализированного оборудования («генной пушки», микроманипулятора) и высококвалифицированного персонала, а также высокой копийности тандемно расположенных инсерций в растительной ДНК при биобаллистиче-ской трансформации, приводящей к низкой экспрессии или сайленсингу трансгена (39-41). Однако в последнее десятилетие в связи с развитием технологий сайт-специфичного редактирования генома растительных клеток (в том числе на модели томата) (42) с участием химерных белков и нуклеопротеидов, созданных на основе бактериальных или дрожжевых эндонуклеаз (43-46), прямые методы генетической трансформации находят все большее применение.
Кроме того, биобаллистическая, а также ПЭГ-опосредованная трансформация протопластов остаются неотъемлемым инструментарием при создании транспластомных растений томата (34, 47).
В настоящей обзорной статье мы систематизировали имеющиеся экспериментальные данные по генетической трансформации томата перечисленными выше прямыми методами, а также проанализировали различные факторы, определяющие эффективность процесса трансформации.
Химически опосредованная трансфекция. Трансформация клеток с помощью химических соединений, облегчающих перенос ДНК через мембрану, была впервые осуществлена в 1970-х годах. В частности, была показана принципиальная возможность введения меченой экзогенной ДНК в протопласты Ammi visnaga (L.) Lam. (48) и Nicotiana tabacum (L.) (49) с помощью различных химических соединений.
Кальций-фосфатный метод был впервые использован в 1973 году для генетической трансформации клеток человека чужеродной ДНК в работе F.L. Graham и A.J. Van der Eb (50). Суть метода заключается в следующем: к буферному раствору, содержащему ДНК, добавляют сначала раствор CaCl 2 , в результате чего отрицательно заряженные фосфатные группы молекул ДНК связываются с ионами Ca2+, а затем фосфатный буфер для получения осадка Ca 3 (PO 4 ) 2 . Полученный ДНК-содержащий раствор добавляют к суспензионной культуре изолированных протопластов, лишенных клеточной стенки, которая существенно затрудняет проникновение макромолекул внутрь клетки. В первой работе по трансформации протопластов томата описанным методом ее максимальная эффективность составила 2 %
(51). Трансгенная природа шести каллусных агрегатов была доказана, однако впоследствии получить из них полноценные регенерированные побеги не удалось. Авторами продемонстрировано, что выбор типа плазмиды для генетической трансформации оказывает определяющее влияние на эффективность получения трансгенных каллусных тканей.
Применение ПЭГ для переноса ДНК через плазмалемму впервые было апробировано для генетической трансформации протопластов двух видов табака (52). В состав буферного раствора для осаждения ДНК, помимо ПЭГ, входит также MgCl 2 . Впоследствии в результате ПЭГ-опосредо-ванной трансформации пластид томата плазмидой pSSH1 были получены транспластомные растения, устойчивые к селективному антибиотику спек-тиномицину. ЭТ, рассчитанная на основании рестрикционного картирования, составила 1,5½10 - 6 (53). S. Ray c соавт. (54) модифицировали метод химически опосредованной трансфекции пластидного генома томата за счет одновременного присутствия ПЭГ и CaCl 2 в буферном растворе для осаждения ДНК, а также добавления в раствор осмотика — маннитола. В результате с использованием плазмиды pCambia1302 в пластидную ДНК были интегрированы селективный и репортерный гены (соответственно nptII и gfp ). Авторы установили, что вследствие большей выживаемости протопластов ПЭГ-4000 предпочтительнее, чем ПЭГ-6000. Кроме того, важным критическим параметром для сохранения жизнеспособности протопластов оказалась длительность их инкубирования в буферном растворе после трансформации и до перенесения на питательную среду для индукции процессов морфогенеза. Оптимальное значение составило 24 ч, тогда как более продолжительное инкубирование негативно влияло жизнеспособность протопластов из-за токсического действия ПЭГ. Трансгенный статус полученных регенерантов был подтвержден полимеразной цепной реакцией (ПЦР), а также флуоресцентным анализом экспрессии репортерного гена; ЭТ авторы не определяли (54).
Микроинъекция. Метод был разработан для доставки макромолекул нуклеиновых кислот в клетки человека и животных (55, 56). При этом плазмалемма клетки механически прокалывается микроиглой — очень тонкой стеклянной пипеткой с наружным диаметром 1-2 мкм, внутри которой содержится растворенная ДНК. Трансформация выполняется специально обученным оператором под микроскопом, укомплектованным микроманипулятором.
Применение этого метода существенно осложняется наличием прочной клеточной стенки и крупной вакуоли у растительной клетки. Клеточная стенка затрудняет визуализацию ядра, а ее фрагменты засоряют микроиглу, поэтому метод микроинъекций разрабатывался для трансформации голых протопластов. Точность попадания микроиглы в определенные клеточные компартменты повышается за счет иммобилизации протопластов во время микроинъекции, например если клетки помещают на агаризован-ную среду (57) либо прикрепляют к предметному стеклу поли-L-лизином (58). H. Morikawa и Y. Yamada (59) разработали механический способ удержания протопластов с помощью дополнительных пипеток; ими также были применены флуоресцентные метки, обеспечивающие визуализацию введенной внутрь клетки ДНК (59). Точность микроманипуляций имеет критическое значение, поскольку при попадании микроиглы в вакуоль чужеродная ДНК разрушается гидролитическими ферментами; кроме того, нарушение целостности вакуоли может вызвать разрыв и гибель клетки из-за попадания в цитоплазму токсичных метаболитов (60).
В первых работах на модели протопластов, выделенных из гипокотилей Brassica napus L., была оценена выживаемость протопластов после введения в них чужеродной ДНК, а также их последующая способность к делению и формированию микрокаллуса с частотой соответственно до 70, 65 и 50 %. Авторами установлено, что на эффективность этих процессов влияют следующие факторы: генотип, возраст интактного экспланта, состав питательной среды для культивирования протопластов, а также рН буферного раствора (61). Однако трансгенные растения впоследствии были получены только для ограниченного числа культур, в том числе растений семейства Solanaceae —петунии (62), табака (63), а также для ячменя (64). Экспериментальная работа японских исследователей по генетической трансформации интактных каллусных клеток томата методом микроинъекции была опубликована одной из первых в 1988 году. Использовав плазмиду pE2KX, содержащую ген npt II , который обусловливает устойчивость к аминогли-козидным антибиотикам, авторы оптимизировали параметры, определяющие ЭТ, — размер микроиглы (применение микроиглы с наружным диаметром более 0,3 мкм приводит к необратимым повреждениям клеточных структур и гибели 90-95 % трансформированных клеток) и длительность процедуры микроинъекции (если она превышает 20 с, происходит необратимая структурно-функциональная дезорганизация ядра и других мембранных органелл). В результате селективного отбора выживших инъецированных клеток на питательной среде с добавлением канамицина частота формирования каллусных агрегатов спустя 1 мес культивирования составила 22 %, их трансгенная природа была подтверждена ПЦР-анализом по наличию гена npt II (65). Значения ЭТ, варьирующие в зависимости от изученных факторов в пределах 15-20 %, были сопоставимы с полученными на культуре протопластов люцерны (15-26 %) при введении различных плазмид (pTiC58, pMON8015, pMON120, pAL4404/pMON120) (66).
Электропорация. Суть метода заключается в том, что при воздействии электрического импульса в плазмалемме клетки образуются поры диаметром порядка 30 нм, существующие несколько минут, с последующим восстановлением нормального состояния мембраны. Кратковременное формирование пор достаточно для проникновения в цитоплазму клетки крупных водорастворимых макромолекул, в частности ДНК (67-70). Трансформация осуществляется с помощью прибора — электропоратора, который состоит из кюветы, пары электродов и генератора импульсов заданной формы (71). В кювету с буферным раствором добавляют суспензию трансформируемых клеток и векторные конструкции с целевыми генами, после чего сквозь раствор пропускают электрический импульс (обычно прямоугольный либо экспоненциальный) (72). Наибольшая ЭТ достигается при напряженности поля 1-20 кВ/см и длительности импульса 1-30 мс, однако для каждого типа клеток оптимальные значения подбираются эмпирически. Критические значения этих физических параметров для формирования пор и успешного транспорта ДНК сквозь плазмалемму клетки определяются ее собственным мембранным потенциалом, а также потенциалом, возникающим под действием внешнего электрического поля (73). При этом ЭТ в большинстве случаев имеет случайную составляющую, поскольку клетки в растворе находятся в неодинаковых условиях и, следовательно, приобретают различные потенциалы (74). ЭТ может быть увеличена за счет добавления в буферный раствор для электропорации различных компонентов, например ионов Ca2+ или Mg2+, ПЭГ для увеличения пермеабилизации мембран (75) или 4-(2-гидроксиэтил)-1-пиперазинэтансульфоновуой кислоты (HEPES) для поддержания оптимального значения рН (76).
Существенным ограничением широкого применения метода электропорации для растительных объектов является наличие клеточной стенки (77, 78). Применение культуры протопластов, лишенных клеточной стенки, значительно снижает ЭТ, поскольку последующее получение из них полноценных растений-регенерантов крайне затруднено для многих видов. Получение стабильных трансгенных линий ряда культур оказалось возможным в случае адаптации методики электропорации, разработанной для изолированных протопластов, для применения на интактных клетках. Такой подход показал свою эффективность при получении стабильно наследуемой инсер-ции трансгена в геноме кукурузы, однако для достижения положительного результата авторами продемонстрирована необходимость частичного нарушения клеточной стенки и увеличения пермеабилизации мембран у трансформируемых клеток (79).
О получении трансгенных растений томата методом электропорации впервые сообщили еще в 1989 году две независимые группы исследователей (80, 81). Так, C. Bellini с соавт. (80) вводили чужеродную ДНК в протопласты двух видов рода Solanum — S. lycopersicum L. и дикорастущего вида томата S. peruvianum L. ЭТ, рассчитанная как отношение числа устойчивых к селективному антибиотику трансформированных колоний к их общему числу, варьировала в пределах 0,3-2,5 %. Максимальная ЭТ достигалась при обработке протопластов трехкратным импульсом длительностью 100 мкс, создающим напряжение 250 В либо 300 В. Из трансформированных протопластов впоследствии были успешно получены растения-регенеранты (80). В работе M. Tsukada с соавт. (81) для генетической трансформации протопластов культурного томата сорта Petit Tomato использовали однократные импульсы экспоненциальной формы, возникающие в результате быстрого разряда предварительно заряженного конденсатора. Наилучшие результаты были достигнуты в случае напряженности электрического поля 1 кВ/см при разряде конденсатора 47 мкФ (81).
На модели протопластов дикого вида S. peruvianum L. была проведена оптимизация ряда физических и физиологических факторов, влияющих на процесс введения чужеродной ДНК, — состава буферного раствора для электропорации (Са2+- либо Mg2+-содержащий буферный раствор при наличии или отсутствии ПЭГ), концентрации клеток в суспензии протопластов, их предобработки (тепловой шок), а также параметров импульса (75). Максимальная эффективность процесса электропорации была достигнута при соблюдении следующих условий: применение Са2+-буферного раствора и теплового шока в течение 5 мин при температуре 45 ° С с последующим добавлением 8 % ПЭГ, а также прямоугольного импульса длительностью 30 мкс с напряженностью электрического поля 1,5 кВ/см.
Биобаллистическ ая трансформация. Этот прямой метод доставки макромолекул нуклеиновых кислот в растительные клетки был разработан Джоном Сэнфордом (John Sanford) в Корнельском университете (США) в 1984 году (82). Суть метода заключается в том, что молекулы ДНК наносят на поверхность носителей — золотых или вольфрамовых микрочастиц (83-95) (табл. 1) либо наночастиц (96), которые разгоняются до скорости 300-600 м/с за счет величины давления гелия. Ими осуществляют бомбардировку различных эксплантов, размещенных на мишени. Поскольку частицы, имеющие такую высокую скорость, успешно проникают сквозь клеточную стенку, биобаллистический метод пригоден для трансформации интактных клеток, что служит важным преимуществом, позволяя избежать этапов изоляции протопластов и последующего низкоэффективного морфогенеза (83).
1. Биобаллистический метод генетической трансформации томата и факторы, влияющие на его эффективность
|
Вид транс-формации1 и локализация транс-гена2 |
Эксплант, генотип |
Модель установки (производитель) |
Параметры биобаллистической трансформации |
Селективный и/или репор-терный ген4 |
ЭТ, % |
Примечание |
Ссылка |
||
|
расстояние между макроносителем частиц и стоп-экраном, см |
давление гелия, psi |
тип3 и размер микрочастиц, мкм |
|||||||
|
Т |
Суспензионная культура клеток томата сорта VFNT Cherry и Solanum pennellii Correll. |
PDS-1000/Heтм («Bio-Rad») |
8,5 |
нд |
W, 1,0 |
nptII , uidA ( gus ) |
1,2 (VFNT Cherry) и 45,0 |
ЭТ определяли как отношение числа устойчивых к канамицину каллусов к числу клеток, у которых наблюдалась транзиентная экспрессия репортер-ного гена после 48 ч обстрела |
(83) |
|
Т |
Листья, незрелые плоды диаметром 5-8 мм томата сорта VFNT Cherry LA 1221 |
PDS-1000/Heтм («Bio-Rad») |
8,5 |
1500, 1800 (оптимум — 1800) |
Au, 0,4-1,2 |
luc или uidA ( gus) |
нд |
Оптимизированы параметры биобал-листической трансформации для транзиентной экспрессии |
(89) |
|
СП |
Листья томата сорта IAC-Santa Clara |
PDS-1000/Heтм («DuPont») |
нд |
1100 |
Au, 0,6 |
aadA |
2,5* 1,15** |
* Отношение числа каллусов, устойчивых к спектиномицину, к общему числу трансформированных эксплантов ** Отношение числа регенерантов с подтвержденной посредством ПЦР инсерцией трансгена к исходному числу трансформированных эксплантов |
(28) |
|
СП |
Листья томата линии IPA-6 |
PDS-1000/Heтм (Bio-Rad) с Hepta Adaptor («Mologen») |
нд |
нд |
Au, 0,6 |
aadA |
нд |
Получение стабильно наследуемых транспластомных растений, экспрессирующих бактериальную ликопин-β-циклазу, с повышенной устойчивостью к гербицидам и 4-кратным увеличением содержания провитамина А в плодах |
(90) |
|
ся |
Фрагменты гипокотиля то- PDS-1000/Не™ 6,0 и 9,0 1350 мата сорта CastleRock («Bio-Rad») |
|
т |
Апикальная меристема, се- PDS-1000/Не™ 2,5-10,0 1100 мядоли и фрагменты гипо- («Bio-Rad») (оптимум — 7,5) котиля 5 томата линии IPA-3 |
|
ся |
Фрагменты гипокотиля то- PDS-1000/Не™ 6,0 и 9,0 1350 мата сорта CastleRock («Bio-Rad») |
|
ся |
Фрагменты гипокотиля с PDS-1000/Не™ 6,0 и 9,0 1350 семядолей (гиподольные («Bio-Rad») экспланты) томата сорта CastleRock |
|
т, ся |
Фрагменты листьев и пло- Gene Gun Не- нд 210 дов томата сорта Ferum Iios System («Bio-Rad») |
|
т |
Фрагменты различных ча- Scientz GJ- 1,0, 3,0 и 6,0 500, 650 и 1100 стей плода томата линии 1000 («Ningbo (оптимум — 6,0) (оптимум — 1100)5 F-144 Scientz Bio technology Со., Ltd») |
|
ся |
Каллусная ткань, получен- PDS-1000/Не™ 6,0, 9,0 и 12,0 1100 ная из листьев томата («Bio-Rad») (оптимум — 9,0) сорта Pusa Ruby |
Продолжение таблицы
|
Au, нд |
bar, uidA (gus) |
26,5 |
ЭТ определяли как отношение числа первичных трансформантов с подтвержденным посредством ПЦР статусом к общему числу регенерантов |
(91) |
|
W, 0,6 |
uidA (gus) |
от 22,69 |
ЭТ определяли как отношение числа |
(84) |
|
до 36,56 |
эксплантов с активностью GUS к общему числу трансформированных экплантов; максимальную ЭТ (36,56 %) отмечали при использовании фрагментов гипокотилей |
|||
|
Au, 1,0 |
hpt |
42,5 |
ЭТ определяли как отношение числа подтвержденных ПЦР-анализом трансгенных регенерантов к общему числу гигромицин-устойчивых регенерантов |
(92) |
|
Au, 1,0 |
hpt |
52,3 |
То же, что и (92) |
(93) |
|
W, 1,1 1ис или uidA (gus) |
нд Изучение активности плодоспеци- (85) фичного промотора гена SIPPC2, кодирующего фосфоенолпируваткар-боксилазу (ЕС 4.1.1.31) томата |
|
W, 1,0 uidA (gus) |
нд Наибольшее число окрашенных в ре- (86) зультате GUS-реакции синих точек составило 2456,91/см2 в районе места крепления плода томата к плодоножке |
|
нд nptll, uidA (gus) |
75,0 Максимальную ЭТ (75 %) отмечали в (102) случае, когда расстояние между макроносителем частиц и стоп-экраном составляло 9,0 см. |
Продолжение таблицы
Разработка прибора для обстрела микрочастицами (так называемой генной пушки) началась в 1984 году, и впоследствии идея была запатентована разработчиками (97). В дальнейшем первоначальная модель получила развитие в модификациях: He Biolistics Particle Delivery System (PDS-1000/Heтм; лицензирована фирмой «DuPont», США); некоммерческий вариант ускорителя Accel ™ Particle Gun (98); пушка для запуска частиц Particle Inflow Gun (99); микронацеливающее устройство, разработанное для трансформации апикальных меристем (100) и Helios Gene Gun («Bio-Rad», США). Последний из перечисленных вариантов не требует вакуумной камеры для удержания ткани-мишени и может быть использован как портативное устройство (101).
Биобаллистическая трансформация — наиболее широко применяемый прямой метод введения чужеродной ДНК в клетки томата, который используется как для изучения транзиентой экспрессии гетерологичных генов (83-87, 99), так и для получения трансгенных растений со стабильно наследуемой ядерной (85, 91-94, 102) или пластидной (28, 88, 90, 95) инсер-цией трансгена (см. табл. 1). Наиболее часто обстрел микрочастицами растительных тканей томата осуществляют с помощью установки PDS-1000/HeTM фирмы «DuPont» или «Bio-Rad» (28, 83, 84, 89-93, 94, 95) (см. табл. 1), значительно реже — серийными устройствами других производителей (86) либо устройствами, разработанными самостоятельно (103). Мишень с трансформируемыми изолированными клетками или эксплантами устанавливается в вакуумной камере с давлением порядка 0,1 атм. В момент сброса давления частицы выбрасываются из пушки в сторону мишени. Как правило, клетки, находящиеся в центре мишени, погибают из-за физического повреждения (104). Применяемая модель установки определяет следующую совокупность физических параметров биобаллистической трансформации, влияющих на эффективность процесса: концентрация ДНК, вид микрочастиц и их размер, варьирующий от 0,4 до 1,2 мкм, расстояние между макроносителем частиц и останавливающим экраном (от 6,0 до 9,0 мм), давление гелия (от 200 до 1800 psi), а также кратность выстрелов (один или несколько). Эти параметры оптимизируют с учетом возрастных и физиологических особенностей конкретных эксплантов, клетки которых подвергают обстрелу. Разработаны протоколы трансформации томата для получения стабильной и транзиентной экспрессии гетерологичных генов на модели суспензионных (83) и каллусных (87, 88, 102) культур клеток, апикальной меристемы (84), фрагментов гипокотиля (84, 91, 92, 94), семядолей (84), листьев (85, 89, 90, 95) и плодов различной степени зрелости (85, 86, 89). При этом ЭТ варьирует в широких пределах (от 1,2 до 80,0 %) в зависимости от вида трансформации (стабильная или транзиентная), особенностей генотипа, типа экспланта и многих других физиологических и физических факторов (см. табл. 1). Важно также отметить отсутствие универсальной методики определения ЭТ, в результате чего авторы предлагают кардинально разнящиеся способы расчета этого показателя (см. табл. 1, примечание), значения которого могут различаться даже на порядок.
В 1995 году стабильные трансгенные растения томата и его дикорастущего родственного вида S. pennellii Correll были впервые получены методом биобаллистической трансформации (83). Обстрелу подвергались клетки суспензионной культуры, которые впоследствии культивировали на питательной среде для индукции процессов морфогенеза с добавлением селективного агента — канамицина. В исследовании использовали вектор на основе дрожжевой хромосомы (YAC) и три типа плазмид, несущих гены uidA (gus) и nptII. Авторы продемонстрировали транзиентную экспрессию GUS во всех случаях, однако число отобранных канамицин-устойчивых каллусов у дикого вида было значительно больше, чем у культурного сорта при сопоставимом уровне транзиентной экспрессии. Отмеченный факт свидетельствует о том, что стабильная трансформация генотип-специфична, и причиной тому служит генетическая детерминированность различных генотипов и соматических тканей к морфогенезу in vitro (10, 22, 105, 106). В целом генотипы представителей рода Solanum можно расположить в следующем порядке по морфогенетическому потенциалу: близкородственные дикие виды томата S. pimpinellifolium L., S. peruvianum L. и S. glandulosum (L.) Mo-rong > S. lycopersicum L. (107); модельные генотипы, не имеющие практического значения > коммерчески значимые сорта и гибриды F1 (10); сорта S. lycopersicum L. > гибриды F1 S. lycopersicum L. (108).
В исследовании D. Ruma с соавт. (84) проведена оптимизация физических параметров биобаллистической трансформации при транзиентной экспрессии репортерного гена uidA ( gus ) в различных эксплантах томата линии IPA-3. Максимальная ЭТ, которая для апикальной меристемы, фрагментов гипокотилей и семядолей составила соответственно 34,12; 36,56 и 22,69 %, была достигнута при двукратном обстреле экспланта микрочастицами с расстояния 7,5 см и давлении гелия 1100 psi (84). Кроме того, авторы изучили биологические факторы, влияющие на частоту транзиентной экспрессии репортерного гена. На один выстрел оптимальное количество плаз-мидной ДНК составило 1,89 мкг из расчета на 1125 мкг микрочастиц. Использование ДНК в избыточных концентрациях снижает ЭТ из-за слипания микрочастиц (109). Прекультивирование эксплантов снижало ЭТ вследствие потери механической прочности клеточных стенок у компетентных клеток. Аналогичные результаты наблюдали в случае предобработки эксплантов томата 0,3 М раствором маннитола (84). В другой работе, напротив, предобработка листьев и плодов томата осмотиком (12 % маннитолом) перед обстрелом приводила к 30-кратному повышению уровня экспрессии гетерологичного гена люциферазы (89). В исследовании L. Sun с соавт. (86) при обстреле вольфрамовыми частицами плодов томата разной степени зрелости «генной пушкой» Scientz GJ-1000 («Scientz», КНР), в которой избыточное давление достигается не гелием, а азотом, максимальная ЭТ была достигнута при однократном обстреле эксплантов микрочастицами (концентрация ДНК — 0,83 мкг из расчета на один выстрел) с расстояния 6 см при давлении азота 1100 psi (86).
Чтобы с высокой частотой (26,5 %) получить трансгенные растения томата со стабильно наследуемым геном из эксплантов гипокотиля G.A. Abu-El-Heba с соавт. (91) предложили иные значения биобаллистической трансформации: концентрация ДНК — 1 мкг; двукратный обстрел микрочастицами золота при расстоянии между макроносителем и стоп-экраном 6 и 9 см соответственно для первого и второго выстрела; давление гелия — 1350 psi (91). Указанные значения успешно применены в ряде исследовательских работ для обстрела фрагментов гипокотилей, а также других типов растительной ткани томата (92, 93, 102) c учетом индивидуальных особенностей состава питательной среды для индукции морфогенеза.
Биобаллистическая трансформация — главный метод создания транс-пластомных растений томата, которые впервые были получены в начале ХХI века на основе коммерческого сорта IAC-Santa Clara (28). Особенность таких растений заключается в наследовании трансгена в поколениях по материнской линии. Отбор гомопластомных клеток и растений осуществляют на селективной питательной среде с добавлением спектиномицина в концентрациях 300-500 мг/л (28, 90, 95) при фотопериоде 16/8 ч (день/ночь) и пониженной освещенности (15 или 25 µE) (28, 95). Экспрессия целевого гена наблюдалась в разных типах пластид: хлоропластах листа, в хромопластах плодов и цветков (28).
Трансформация in planta . Трансформация in planta позволяет получать трансгенные растения, минуя длительный и трудоемкий этап культивирования изолированных органов и тканей in vitro. Главные преимущества трансформации растений in planta — относительная простота и быстрота, поскольку эти методы не подразумевают оснащения лаборатории специализированным биотехнологическим оборудованием вследствие отсутствия этапа культуры in vitro, тем самым исключая сомаклональную изменчивость (110, 111). Наилучшим материалом для трансформации in planta может считаться пыльца: ее можно получить в большом количестве, она содержит гаплоидные клетки и для трансформации можно применять практически любой из методов, апробированных ранее для трансформации протопластов и интактных клеток, — электропорацию, микроинъекцию, упаковку ДНК в липосомы, биобаллистический метод, а также Agrobacterium -опосредованную трансформацию (112).
При прорастании трансформированной пыльцы in planta экзогенная ДНК попадает в зародышевый мешок с последующим возможным формированием трансгенного диплоидного зародыша (113, 114). Этот прием, получивший название метода пыльцевых трубок, или pollen tube pathway, был разработан и впервые применен в 1974 году на растениях семейства Solanaceae — Petunia hybrida Vilm. (115, 116) и Nicotiana glauca Graham (115), а также на растениях ячменя (117). Впоследствии с помощью метода пыльцевых трубок были также получены трансгенные растения хлопчатника (118), ячменя (119), ржи (120) и других сельскохозяйственных культур, в том числе различных генотипов томата (121-126).
Первая работа, где была показана возможность генетической трансформации томата с использованием метода пыльцевых трубок, была опубликована в 1989 году (121). Продолжением этого исследования стали другие работы, в частности, были получены трансгенные гибриды S. lycopersicum L. сорта Факел и S. penelii Cor. (125). При этом генетическую трансформацию пыльцы и процесс опыления проводили одновременно. Свежевысушен-ную пыльцу помещали в питательную среду с добавлением 15 % сахарозы, 0,018 % борной кислоты и 0,04 % Ca(NO 3 ) 2 , к которой затем добавляли раствор плазмидной ДНК. Сразу после добавления ДНК проводили опыление трансформированной пыльцой предварительно кастрированных и изолированных цветков. ЭТ, рассчитанная как отношение числа канами-цин-устойчивых проростков, полученных из незрелых зародышей, к общему числу трансформированных зародышей, составила 2,2 %. Соотношение числа полученных из семян канамицин-устойчивых и канамицин-чувствительных проростков составило 3:1, что доказывает их трансгенный статус и свидетельствует об однолокусной модели наследования трансгена. Сравнительно низкая эффективность трансформации методом pollen tube pathway в значительной степени связана с нуклеазной активностью прорастающей пыльцы, в результате которой большая часть экзогенной ДНК деградирует (112).
Другая модификация метода пыльцевых трубок, основанная на более ранней работе Н.В. Турбина с соавт. (117), предполагает введение содержащего ДНК раствора в семязачаток уже оплодотворенного цветка. После опыления рыльце пестика удаляют и с помощью тонкой иглы вводят раствор, содержащий целевую последовательность ДНК (114). Этот метод также применялся для получения трансгенных растений томата (122-124). 530
Так, в работе R. Wang c соавт. (124) изучено влияние генотипа, концентрации плазмидной ДНК, а также компонентов буферного раствора для инъекции на ЭТ сортов томата Zhongshu 6, Liaoyuanduoli и Jinguan 9 при использовании векторной конструкции, содержащей селективный ген bar , обусловливающий устойчивость к фосфинотрицину, а также репортерный ген желтого флуоресцирующего белка ( yfp ). Авторами установлено отсутствие достоверных различий по ЭТ между изученными сортами. При этом наибольший выход трансгенных растений обеспечивало добавление в раствор для инъекции 600 нг/мкл плазмидной ДНК, а также 5 % сахарозы и 0,05 % сурфактанта Silvet-L-77 (124). Для описанной модификации метода pollen tube pathway критически значимы промежуток времени от опыления до трансформации и сохранность семязачатка при удалении пестика (123, 127). Установлено, что введение чужеродной ДНК следует осуществлять через 24 ч после опыления; уменьшение продолжительности этого периода снижает ЭТ (123). Что касается сохранности семязачатка, то применяются различные способы нарушения целостности пестика перед введением чужеродной ДНК, например полное удаление пестика перед инъекцией (123) либо удаление только его части (рыльца с частью столбика) (122). Вклад последнего фактора трудно оценить из-за низкой эффективности метода в целом. В зависимости от вышеперечисленных факторов ЭТ варьирует от 0,2 (124) до 1,4 % (128).
Разновидностью метода pollen tube pathway является агроинфильтрация чужеродной ДНК в различные генеративные органы растений (129, 130). Эффективность разных модификаций указанного метода достигает в среднем 3-4 %. Процедура активно используется для получения трансгенных растений арабидопсиса и других культур семейства Brassicaceae (131). Этот метод можно считать гибридным между pollen tube pathway и агробак-териальной трансформацией. На томате обсуждаемый метод был успешно применен для введения генов LFY и GUS (132).
Высокоэффективный протокол электропорации зрелых семян томата был предложен в работе Z. Hilioti с соавт. (133) для доставки систем геномного редактирования. Стерилизованные семена инкубировали в течение 12 ч в темноте при пониженной температуре (10 ° С) в растворе с добавлением 5 % сахарозы, 3 % H 3 BO 3 и 1,3 мM Ca(NO 3 ) 2 . Набухшие семена вакуумировали в буферном растворе (80 мM KCl, 5 мM CaCl 2 , 10 мM HEPES и 0,5 M маннитол) и выдерживали на льду в течение 1 ч. Электропорацию предобработанных семян проводили в присутствии буферного раствора с добавлением плазмидной ДНК (из расчета 50 мкг на 200 мкл буфера) в режиме трех импульсов длительностью по 4 мс каждый при напряженности поля 6,25 кВ/см. Авторами продемонстрировано, что 65 % растений томата, выращенных из трансфицированных семян, содержали различные мутации целевого гена, что свидетельствует об успешной трансформации зародышей используемым вектором, кодирующим нуклеазы ZFN, и их последующей экспрессии в электропорированных зародышах (133).
Использование прямых методов генетической трансформации томата для доставки систем геномного редактирования. За последние 5 лет отмечается бум исследовательских работ, посвященных направленному внесению изменений в геном томата с помощью систем геномного редактирования ZFNs (zinc-finger nucleases — нуклеазы «цинковых пальцев»), TALEN (transcription activator-like effector nucleases — эффекторные нуклеазы, подобные активаторам транскрипции) и CRISPR/Cas (clustered regularly interspaced short palindromic repeats — короткие палиндромные повторяющиеся элементы, расположенные группами и равномерно удаленные друг от друга) (43, 134). В зависимости от применяемой системы можно осуществлять различные варианты геномного редактирования — посредством нокаутных мутаций, возникающих в результате инсерций или делеций части нуклеотидов в участке редактирования за счет негомологичного объединения репарируемых концов или посредством гомологичной рекомбинации; нокин-редактирование гена (knock-in) за счет введения экзогенных олигонуклеотидов или более протяженных фрагментов ДНК после внесения разрывов и последующей гомологичной рекомбинации; внесение точечных однонуклеотидных замен за счет дезаминирования азотистых оснований (CBE — cytosine base editors, редактор оснований цитозина, ABE — adenine base editors, редактор оснований аденина). Механизмы действия систем геномного редактирования ZFNs, TALEN, CRISPR/Cas, CBE и ABE подробно рассмотрены в ряде обзорных статей (135-139).
На сегодняшний день различные методы редактирования генома томата широко применяются в исследованиях как фундаментального (140), так и ярко выраженного прикладного характера, например с целью повышения устойчивости растений к абиотическим (141, 142) и биотическим (143-146) стрессорам разной природы, повышения лежкости плодов и улучшения их качества (147-152), а также для ускорения процесса доместикации диких видов Solanum (153, 154). Доставка систем геномного редактирования осуществляется преимущественно посредством агробактериальной трансформации векторными конструкциями, экспрессирующими эти системы. Прямые методы введения чужеродной ДНК для получения растений томата с редактированным геномом также занимают значимое место в этом списке (табл. 2).
2. Прямые методы доставки систем геномного редактирования в растения томата
|
Система редактирования |
Способ доставки |
Генотип |
Ген |
Контролируемый признак |
Эффективность редактирования, % |
Ссылка |
|
TALEN |
ПЭГ - Ca2+-опо-средованная трансформация |
Micro-Tom ANT1 |
Ген, контролирующий биосинтез антоциана; признак фиолетовой окраски |
7,28 |
(155) |
|
|
CRISPR/Cas9 |
ПЭГ - Ca2+-опо-средованная трансформация |
Micro-Tom ANT1 |
Ген, контролирующий биосинтез антоциана; признак фиолетовой окраски |
2,75-8,8 |
(155) |
|
|
ПЭГ-опосредо-ванная трансформация |
Micro-Tom |
ССD7 ССD8 |
Гены, вовлеченные в биосинтез стриголактона |
30 90 |
(156) |
|
|
ПЭГ - Ca2+-опо-средованная трансформация |
Solanum per-uvianum L. |
SpRDR6 SpSGS3 SpPR-1 SpMlo1 SpProSys |
Гены, опосредующие устойчивость к фитопатогенам |
13,2 8,3 13,9 63,6 45,8 |
(157) |
|
|
ZFNs |
Электропорация зародышей зрелых семян |
Heinz 1706 |
LIL4 |
Транскрипционный фактор, контролирующий развитие семядольных и настоящих листьев, а также цветение и созревание плодов |
65 |
(133) |
|
Prime editing Примечан |
Биобаллистика Micro-Tom NanoLucM Обратная мутация, восста навливающая активность люциферазы NanoLuc ие. a — эффективность транзиентной экспрессии. |
0,26a |
(158) |
|||
Так, с помощью систем TALEN и CRISPR/Cas9 и ПЭГ-Ca2+-опо-средованной трансформации протопластов томата модельного сорта MicroTom была осуществлена точная вставка промотора CaMV35S между промо-торным и транскрибируемым участками гена ANT1, контролирующего биосинтез антоцианов. Обусловленная конститутивным промотором сверх- экспрессия гена ANT1 способствовала эктопическому накоплению пигмента в растительных тканях. В зависимости от типа генетической конструкции и системы геномного редактирования ЭТ варьировала от 2,75 до 8,8 %. Более двух третей инсерций трансгена оказались точными и стабильно наследовались в Т1 семенном поколении согласно менделевскому расщеплению (155).
ПЭГ-опосредованная доставка системы CRISPR/Cas9 в протопласты томатов позволила провести редактирование двух генов каротиноид-расщепляющих диоксигеназ ( ССD7 и ССD8 ), вовлеченных в биосинтез стриголактона. При этом применили мультиплексирование, то есть одновременное нацеливание нескольких гидовых РНК на оба гена. Для трансфекции протопластов авторы использовали многокомпонентный буферный раствор, содержащий 12,5 % ПЭГ-4000. В результате из 50 случайно отобранных каллусных агрегатов, образовавшихся в культуре протопластов, у одного (2 %) и пяти (10 %) выявили моноаллельные мутации (соответственно в генах CCD7 и CCD8 ), тогда как 13 (26 %) и 36 (72 %) каллусов содержали биаллельные мутации тех же генов (гомозиготное или гетерозиготное состояние) (156).
Y.C. Lin с соавт. (157) получили из культуры протопластов S. peruvi-anum L. независимые регенеранты с точечными мутациями, внесенными CRISPR/Cas9 в следующие гены, продукты экспрессии которых обусловливают устойчивость к фитопатогенам: SpRDR6 (РНК-зависимая РНК-полимераза 6), SpSGS3 (супрессор сайленсинга гена 3) (два ключевых гена РНК-сайленсинга, опосредующие защиту от вируса желтой курчавости листьев томата), SpPR-1 (pathogenesis-related protein-1), SpProSys (просистемин), а также SpMlo1 (один из представителей семейства генов локуса О, обусловливающий устойчивость к снежной плесени). В результате частота редактирования в зависимости от гена варьировала в пределах 8,3-63,6 % (157).
Использование зрелых зародышей семян томата сорта Heinz 1706 в качестве эксплантов для электропорации плазмидами с последовательностью ZFN позволило получить независимые линии с редактированным геном LIL4 , кодирующим транскрипционный фактор LEAFY COTYLEDON1-LIKE4, который контролирует развитие семядольных и настоящих листьев, а также цветение и плодоношение. Установлено, что 65 % растений томата, выращенных из трансфицированных семян, содержали различные мутации целевого гена (133).
Доставка систем геномного редактирования осуществляется также и с помощью биобаллистического метода. Примером служит редактирование генома томата сорта Micro-Tom посредством системы prime editing на основе нуклеазы Cas9, сшитой с обратной транскриптазой вируса лейкемии мышей (MuLV) и содержащей новый вариант гидовой РНК — prime editing guide RNA (pegRNA), которая не только направляет нуклеазу к нужному сайту ДНК, но и служит матрицей, кодирующей изменения (158). Эффективность редактирования генома с помощью этой технологии составила 0,26 % (показатель оценивали посредством восстановленной транзиентной экспрессии люциферазы через 7 сут после обстрела микрочастицами).
Итак, в настоящей обзорной статье рассмотрены различные прямые методы (химически опосредованная трансформация, микроинъекция, электропорация, биобаллистическая трансформация) введения чужеродной ДНК для получения трансгенных растений томата, а также факторы различной природы (физические, генетические и физиологические), влияющие на эффективность этого процесса. Первые три метода характеризуются низкой эффективностью, в связи с чем не нашли широкого применения.
Наиболее распространенным прямым способом генетической трансформации томата служит биобаллистический метод, с помощью которого с разной эффективностью получают растения со стабильно наследуемой инсер-цией трансгена как в ядерном, так и пластидном геноме. При этом следует отметить, что непосредственное сопоставление эффективности трансформации (ЭТ) не всегда возможно, поскольку разные авторы используют неодинаковые методики ее определения. Кроме того, в ряде исследовательских работ ЭТ не представлена вовсе, поскольку, по-видимому, полученные трансгенные растения томата были единичными или же задача определения ЭТ не ставилась. Следует отметить, что с широким развитием различных систем геномного редактирования прямые методы генетической трансформации томата используются все чаще, особенно при работе с культурой протопластов.
Список литературы Трансгенные растения томата (Solanum lycopersicum L.): прямые методы введения генов и факторы, влияющие на эффективность трансформации (обзор)
- FAO. FAOSTAT, 2021. Режим доступа: https://www.fa0.0rg/fa0stat/ru/#data/QCL. Без даты.
- Роньжина Е.С., Подлеснова В.С. Комплексная оценка качества томатов, выращиваемых в Калининградской области. Известия Горского государственного аграрного университета, 2021, 58-3: 30-35.
- Авдеев Ю.И. Технология томатов в открытом грунте Астраханской области. Картофель и овощи, 2014, 5: 7-9.
- Sorokin A., Bryzzhev A., Strokov A., Mirzabaev A., Johnson T., Kiselev S.V. The economics of land degradation in Russia. In: Economics of land degradation and improvement — a global assessment for sustainable development /E. Nkonya, A. Mirzabaev, J. von Braun (eds). Springer, Cham., 2016: 541-576 (doi: 10.1007/978-3-319-19168-3_18).
- Салина Ю.Б., Тютюма Н.В., Тютюма А.В. Засоление как критический фактор плодородия земель Астраханской области. Достижения науки и техники АПК, 2018, 32(12): 5-8 (doi: 10.24411/0235-2451-2018-11201).
- Лазарев А.М. Бактериальные болезни томата и меры борьбы с ними. СПб, 2015.
- Blancard D. Tomato diseases. Academic Press, San Diego, CA, USA, 2012.
- Кигашпаева О.П., Авдеев А.Ю. Брендовые астраханские сорта томата. Проблемы развития АПК региона, 2020, 2: 93-97 (doi: 10.15217/issn2079-0996.2020.2.93).
- Khaliluev M.R., Shpakovskii G.V. Genetic engineering strategies for enhancing tomato resistance to fungal and bacterial pathogens. Russian Journal of Plant Physiology, 2013, 60(6): 721-732 (doi: 10.1134/S1021443713050087).
- Gerszberg A., Hnatuszko-Konka K., Kowalczyk T., Kononowicz A.K. Tomato (Solanum lycoper-sicum L.) in the service of biotechnology. Plant Cell Tissue and Organ Culture, 2015, 120: 881902 (doi: 10.1007/s11240-014-0664-4).
- Anwar R., Fatima T., Mattoo A. Tomatoes: A model crop of Solanaceous plants. In: Oxford research encyclopedia of environmental science. Oxford University Press, Oxford, UK, 2019 (doi: 10.1093/acrefore/9780199389414.013.223).
- Chaban I., Khaliluev M., Baranova E., Kononenko N., Dolgov S., Smirnova E. Abnormal development of floral meristem triggers defective morphogenesis of generative system in transgenic tomatoes. Protoplasma, 2018, 255(6): 1597-1611 (doi: 10.1007/s00709-018-1252-y).
- Вершинина З.Р., Благова Д.К., Нигматуллина Л.Р. Лавина А.М., Баймиев А.Х., Чеме-рис А.В. Ассоциативный симбиоз трансгенных томатов с ризобиями повышает устойчивость растений к Fusarium oxysporum f. sp. lycopersici. Биотехнология, 2015, 31(3): 42-53.
- Arie T., Takahashi H., Kodama M., Teraoka T. Tomato as a model plant for plant-pathogen interactions. Plant Biotechnology, 2007, 24(1): 135-147 (doi: 10.5511/plantbiotechnology.24.135).
- Вершинина З.Р., Чубукова О.В., Масленникова Д.Р. Оценка глутатионового статуса в корнях трансгенных по генам psl и rapAl растений томата при действии Rhizobium legumi-nosarum. Физиология растений, 2021, 68(5): 524-532 (doi: 10.31857/S0015330321050225).
- Комахин Р.А., Комахина В.В., Милюкова Н.А., Голденкова-Павлова И.В., Фадина О.А., Жученко А.А. Трансгенные растения томата, экспрессирующие гены recA и NLS-recA-licBM3, как модель для изучения мейотической рекомбинации. Генетика, 2010, 46(12): 1635-1644.
- Rothan C., Diouf I., Causse M. Trait discovery and editing in tomato. The Plant Journal, 2019, 97(1): 73-90 (doi: 10.1111/tpj.14152).
- Salim M.M.R., Rashid M.H., Hossain M.M., Zakaria, M. Morphological characterization of tomato (Solanum lycopersicum L.) genotypes. Journal of the Saudi Society of Agricultural Sciences, 2020, 19: 233-240 (doi: 10.1016/j.jssas.2018.11.001).
- Renna M., D'Imperio M., Gonnella M., Durante M., Parente A., Mita G., Santamaria P., Serio F. Morphological and chemical profile of three tomato (Solanum lycopersicum L.) landraces of a semi-arid Mediterranean environment. Plants, 2019, 8(8): 273 (doi: 10.3390/plants8080273).
- Causse M., Giovannoni J., Bouzayen M., Zouine M. The Tomato Genome. Compendium of Plant Genomes. Springer, Berlin, Heidelberg, 2016 (doi: 10.1007/978-3-662-53389-5_3).
- The Tomato Genome Consortium. The tomato genome sequence provides insights into fleshy fruit evolution. Nature, 2012, 485: 635-641 (doi: 10.1038/nature11119).
- Bhatia P., Ashwath N., Senaratna T., Midmore D. Tissue culture studies of tomato (Lycoper-sicon esculentum). Plant Cell Tissue and Organ Culture, 2004, 78: 1-21 (doi: 10.1023/B:TICU.0000020430.08558.6e).
- Гавриш С.Ф. Опыт и перспективы селекции томата для защищенного грунта. Известия Тимирязевской сельскохозяйственной академии, 1992, 5: 147-161.
- Бухарова А.Р., Бухаров А.Ф. Отдаленная гибридизация овощных пасленовых культур. Мичуринск, 2008.
- Rommens C.M., Kishore G.M. Exploiting the full potential of disease-resistance genes for agricultural use. Current Opinion in Biotechnology, 2000, 11(2): 120-125 (doi: 10.1016/s0958-1669(00)00083-5).
- Bock R. Engineering plastid genomes: methods, tools, and applications in basic research and biotechnology. Annual Review of Plant Biology, 2015, 66: 211-241 (doi: 10.1146/annurev-arplant-050213-040212).
- Adem M., Beyene D., Feyissa T. Recent achievements obtained by chloroplast transformation. Plant Methods, 2017, 13(1): 30 (doi: 10.1186/s13007-017-0179-1).
- Ruf S., Hermann M., Berger I.J., Carrer H., Bock R. Stable genetic transformation of tomato plastids and expression of a foreign protein in fruit. Nature Biotechnology, 2001, 19(9): 870-875 (doi: 10.1038/nbt0901-870).
- Staub J.M., Maliga P. Long regions of homologous DNA are incorporated into the tobacco plastid genome by transformation. The Plant Cell, 1992, 4(1): 39-45 (doi: 10.1105/tpc.4.1.39).
- Kaplanoglu E., Kolotilin I., Menassa R., Donly C. Plastid transformation of Micro-Tom tomato with a hemipteran double-stranded RNA results in RNA interference in multiple insect species. International Journal of Molecular Sciences, 2022, 23(7): 3918 (doi: 10.3390/ijms23073918).
- Rogalski M., do Nascimento Vieira L., Fraga H.P., Guerra M.P. Plastid genomics in horticultural species: importance and applications for plant population genetics, evolution, and biotechnology. Frontiers in Plant Science, 2015, 6: 586 (doi: 10.3389/fpls.2015.00586).
- Saveleva N.V., Burlakovskiy M.S., Yemelyanov V.V., Lutova L.A. Transgenic plants as bioreactors to produce substances for medical and veterinary uses. Russian Journal of Genetics: Applied Research, 2016, 6(6): 712-724 (doi: 10.1134/S2079059716060071).
- Rozov S.M., Sidorchuk Y.V., Deineko E.V. Transplastomic plants: problems of production and their solution. Russian Journal of Plant Physiology, 2022, 69(2): 1-10 (doi: 10.1134/S1021443722020157).
- Zagorskaya A.A., Deineko E.V. Plant-expression systems: a new stage in production of bio-pharmaceutical preparations. Russian Journal of Plant Physiology, 2021, 68(1): 17-30 (doi: 10.1134/S1021443721010210).
- Gelvin S.B. Plant DNA repair and Agrobacterium T-DNA integration. International Journal of Molecular Sciences, 2021, 22(16): 8458 (doi: 10.3390/ijms22168458).
- Hwang H.H., Yu M., Lai E.M. Agrobacterium-mediated plant transformation: biology and applications. The Arabidopsis Book, 2017, 15: e0186 (doi: 10.1199/tab.0186).
- Anand A., Jones T.J. 2018 Advancing Agrobacterium-based crop transformation and genome modification technology for agricultural biotechnology. In: Agrobacterium Biology. Current Topics in Microbiology and Immunology. V. 418 /S.B. Gelvin (ed.). Springer, Cham., 2018: 489-507 (doi: 10.1007/82_2018_97).
- Chyi Y.S., Jorgensen R.A., Goldstein D., Tanksley S.D., Loaiza-Figueroa F. Locations and stability of Agrobacterium-mediated T-DNA insertions in the Lycopersicon genome. Molecular and General Genetics, 1986, 204(1): 64-69 (doi: 10.1007/BF00330188).
- Saifi S.K., Passricha N., Tuteja R., Kharb P., Tuteja N. In planta transformation: A smart way of crop improvement. In: Advancement in crop improvement techniques /N. Tuteja, R. Tuteja, N. Passricha (eds.). Woodhead Publishing, Sawston, Cambridge, 2020: 351-362 (doi: 10.1016/B978-0-12-818581-0.00021-8).
- Keshavareddy G., Kumar A.R.V., Ramu V.S. Methods of plant transformation-a review. International Journal of Current Microbiology and Applied Sciences, 2018, 7(7): 2656-2668 (doi: 10.20546/ijcmas.2018.707.312).
- Marenkova T.V., Loginova D.B., Deineko E.V. Mosaic patterns of transgene expression in plants. Russian Journal of Genetics, 2012, 48(3): 249-260 (doi: 10.1134/S1022795412030088).
- Xia X., Cheng X., Li R., Yao J., Li Z., Cheng Y. Advances in application of genome editing in tomato and recent development of genome editing technology. Theoretical and Applied Genetics, 2021, 134(9): 2727-2747 (doi: 10.1007/s00122-021-03874-3).
- Gaj T., Gersbach C.A., Barbas C.F. ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 2013, 31: 397-405 (doi: 10.1016/j.tibtech.2013.04.004).
- Khan Z., Khan S.H., Mubarik, Sadia B., Ahmad A. Use of TALEs and TALEN Technology for Genetic Improvement of Plants. Plant Molecular Biology Reporter, 2017, 35: 1-19 (doi: 10.1007/s11105-016-0997-8).
- Petolino J.F. Genome editing in plants via designed zinc finger nucleases. In Vitro Cellular & Developmental Biology - Plant, 2015, 51(1): 1-8 (doi: 10.1007/s11627-015-9663-3).
- Sardesai N., Subramanyam S. Agrobacterium: a genome-editing tool-delivery system. In: Agro-bacterium biology. Current Topics in microbiology and immunology. V. 418 /S.B. Gelvin (ed.). Springer, Cham., 2018: 463-488 (doi: 10.1007/82_2018_101).
- Ruf S., Bock R. Plastid Transformation in Tomato. In: Chloroplast biotechnology. Methods in molecular biology (methods and protocols). V. 1132 /P. Maliga (ed.). Humana Press, Totowa, NJ., 2014: 265-276 (doi: 10.1007/978-1-62703-995-6_16).
- Глеба Ю.Ю., Хасанов М.М., Слюсаренко А.Г., Бутенко Р.Г., Винецкий Ю.П. Проникновение 3Н-ДНК Bacillus subtilis в изолированные протопласты клеток табака Nicotiana taba-cum. Доклады АН СССР, 1974, 219(4-6): 1478-1481.
- Ohyama K., Gamborg O.L., Miller R.A. Uptake of exogenous DNA by plant protoplasts. Canadian Journal of Botany, 1972, 50(10): 2071-2080 (doi: 10.1139/b72-267).
- Graham F.L., Van der Eb A.J. A new technique for the assay of infectivity of human adenovirus 5 DNA. Virology, 1973, 52: 456-467 (doi: 10.1016/0042-6822(73)90341-3).
- Jongsma M., Koornneef M., Zabel P., Hille J. Tomato protoplast DNA transformation: physical linkage and recombination of exogenous DNA sequences. Plant Molecular Biology, 1987, 8(5): 383-394 (doi: 10.1007/BF00015816).
- Negrutm I., Shillito R., Potrykus I., Biasini G., Sala F. Hybrid genes m the analysis of transformation conditions. I. Setting up a simple method for direct gene transfer m plant protoplasts. Plant Molecular Biology, 1987, 8: 363-373 (doi: 10.1007/BF00015814).
- Nugent G.D., ten Have M., van der Gulik A., Dix P.J., Uijtewaal B.A., Mordhorst A.P. Plastid transformants of tomato selected using mutations affecting ribosome structure. Plant Cell Reports, 2005, 24: 341-349 (doi:10.1007/s00299-005-0930-3).
- Ray S., Lahiri S., Halder M., Mondal M., Choudhuri T.R., Kundu S. An efficient method of isolation and transformation of protoplasts from tomato leaf mesophyll tissue using the binary vector pCambia 1302. International Advanced Research Journal in Science, Engineering and Technology, 2015, 2: 146-150 (doi: 10.17148/IARJSET.2015.2631).
- Diacumakos E.G. Methods of micromanipulation of human somatic cells in culture. In: Methods in cell biology. V. 7 /D.M. Prescott (ed.). Academic Press, New York, 1974: 287-312 (doi: 10.1016/S0091-679X(08)61783-5).
- Capecchi M.R. High efficiency transformation by direct microinjection of DNA into cultured mammalian cells. Cell, 1980, 22(2): 479-488 (doi: 10.1016/0092-8674(80)90358-X).
- Griesbach R.J. Protoplast microinjection. Plant Molecular Biology Reporter, 1983, 1(4): 32-37 (doi: 10.1007/BF02712674).
- Steinbiss H.H., Stabel P. Protoplast derived tobacco cells can survive capillary microinjection of the fluorescent dye Lucifer Yellow. Protoplasma, 1983, 116(2): 223-227 (doi: 10.1007/BF01279844).
- Morikawa H., Yamada Y. Capillary microinjection into protoplasts and intranuclear localization of injected materials. Plant and Cell Physiology, 1985, 26(2): 229-236 (doi: 10.1093/oxfordjour-nals.pcp.a076901).
- Griesbach R.J., Sink K.C. Evacuolation of mesophyll protoplasts. Plant Science Letters, 1983, 30(3): 297-301 (doi: 10.1016/0304-4211(83)90168-2).
- Spangenberg G., Koop H.U., Lichter R., Schweiger H.G. Microculture of single protoplasts of Brassica napus. Physiologia Plantarum, 1986, 66(1): 1-8 (doi: 10.1111/j.1399-3054.1986.tb01223.x).
- Griesbach R.J. Chromosome-mediated transformation via microinjection. Plant Science, 1987, 50(1): 69-77 (doi: 10.1016/0168-9452(87)90032-X).
- Schnorf M., Neuhaus-Url G., Galli A., Iida S., Potrykus I., Neuhaus G. An improved approach for transformation of plant cells by microinjection: molecular and genetic analysis. Transgenic Research, 1991, 1(1): 23-30 (doi: 10.1007/BF02512993).
- Holm P.B., Olsen O., Schnorf M., Brinch-Pedersen H., Knudsen S. Transformation of barley by microinjection into isolated zygote protoplasts. Transgenic Research, 2000, 9(1): 21-32 (doi: 10.1023/A:1008974729597).
- Toyoda H., Matsuda Y., Utsumi R., Ouchi S. Intranuclear microinjection for transformation of tomato callus cells. Plant Cell Reports, 1988, 7(5): 293-296 (doi: 10.1007/BF00269921).
- Reich T.J., Iyer V.N., Miki B.L. Efficient transformation of alfalfa protoplasts by the intranuclear microinjection of Ti plasmids. Nature Biotechnology, 1986, 4(11): 1001-1004 (doi: 10.1038/nbt1186-1001).
- Okada K., Nagata T., Takebe I. Introduction of functional RNA into plant protoplasts by electro-poration. Plant and Cell Physiology, 1986, 27(4): 619-626 (doi: 10.1093/oxfordjournals.pcp.a077141).
- Chen C., Smye S.W., Robinson M.P., Evans J.A. Membrane electroporation theories: a review. Medical and Biological Engineering and Computing, 2006, 44(1-2): 5-14 (doi: 10.1007/s11517-005-0020-2).
- Kotnik T., Frey W., Sack M., Haberl Meglic S., Peterka M., Miklavcic D. Electroporation-based applications in biotechnology. Trends in Biotechnology, 2015, 33: 480-488 (doi: 10.1016/j.tibtech.2015.06.002).
- Kotnik T., Rems L., Tarek M., Miklav i D. Membrane electroporation and electropermeabili-zation: mechanisms and models. Annual Review of Biophysics, 2019, 48: 63-91 (doi: 10.1146/an-nurev-biophys-052118-115451).
- Rebersek M., Faurie C., Kanduser M., Corovic S., Teissié J., Rols M.P., Miklavcic D. Electro-porator with automatic change of electric fled direction improves gene electrotransfer in-vitro. BioMedical Engineering Online, 2007, 6(1): 1-11 (doi: 10.1186/1475-925X-6-25).
- Hanze J., Fischer L., Koenen M., Worgall S., Rascher W. Electroporation of nucleic acids into prokaryotic and eukaryotic cells by square wave pulses. Biotechnology Techniques, 1998, 12(2): 159-163 (doi: 10.1023/A:1008800903452).
- Kotnik T., Pucihar G., Miklavcic D. Induced transmembrane voltage and its correlation with electroporation-mediated molecular transport. Journal of Membrane Biology, 2010, 236: 3-13 (doi: 10.1007/s00232-010-9279-9) (doi: 10.1007/s00232-010-9279-9).
- Gurel F., Gozukirmizi N. Electroporation transformation of barley. In: Genetic transformation of plants. Molecular methods of plant analysis. V. 23 /J.F. Jackson, H.F. Linskens (eds.). Springer, Berlin, 2003: 69-89 (doi: 10.1007/978-3-662-07424-4_5).
- Nakata K., Tanaka H., Yano K., Takagi M. An effective transformation system for Lycopersicon peruvianum by electroporation. Japanese Journal of Breeding, 1992, 42(3): 487-495 (doi: 10.1270/jsbbs1951.42.487).
- Sherba J.J., Hogquist S., Lin H., Shan J.W., Shreiber D.I., Zahn J.D. The effects of electroporation buffer composition on cell viability and electro-transfection efficiency. Scientific Reports, 2020, 10(1): 3053 (doi: 10.1038/s41598-020-59790-x).
- Azencott H.R., Peter G.F., Prausnitz M.R. Influence of the cell wall on intracellular delivery to algal cells by electroporation and sonication. Ultrasound in Medicine & Biology, 2007, 33(11): 1805-1817 (doi: 10.1016/j.ultrasmedbio.2007.05.008).
- Ortiz-Matamoros M.F., Villanueva M.A., Islas-Flores T. Genetic transformation of cell-walled plant and algae cells: delivering DNA through the cell wall. Briefings in Functional (Genomics, 2018, 17(1): 26-33 (doi: 10.1093/bfgp/elx014).
- D'Halluin K., Bonne E., Bossut M., De Beuckeleer M., Leemans J. Transgenic maize plants by tissue electroporation. The Plant Cell, 1992, 4(12): 1495-1505 (doi: 10.1105/tpc.4.12.1495).
- Bellini C., Chupeau M.C., Guerche P., Vastra G., Chupeau Y. Transformation of Lycopersicon peruvianum and Lycopersicon esculentum mesophyll protoplasts by electroporation. Plant Science, 1989, 65(1): 63-75 (doi: 10.1016/0168-9452(89)90208-2).
- Tsukada M., Kusano T., Kitagawa Y. Introduction of foreign genes into tomato protoplasts by electroporation. Plant and Cell Physiology, 1989, 30(4): 599-603 (doi: 10.1093/oxfordjournals.pcp.a077781).
- Sanford J.C., Klein T.M., Wolf E.D. Delivery of substances into cells and tissues using a particle bombardment process. Particulate Science and Technology, 1987, 5: 27-37 (doi: 10.1080/02726358708904533).
- Van Eck J.M., Blowers A.D., Earle E.D. Stable transformation of tomato cell cultures after bombardment with plasmid and YAC DNA. Plant Cell Reports, 1995, 14(5): 299-304 (doi: 10.1007/BF00232032).
- Ruma D., Dhaliwal M.S., Kaur A., Gosal S.S. Transformation of tomato using biolistic gun for transient expression of the ß-glucuronidase gene. Indian Journal of Biotechnology, 2009, 8(4): 363369.
- Guillet C., Aboul-Soud M.A., Le Menn A., Viron N., Pribat A., Germain V., Just D., Baldet P., Rousselle P., Lemaire-Chamley M., Rothan C. Regulation of the fruit-specific PEP carboxylase SlPPC2 promoter at early stages of tomato fruit development. PLoS ONE, 2012, 7(5): e36795 (doi: 10.1371/journal.pone.0036795).
- Sun L., Liu S., Ren J., Cui M., Wang L., Leng P. Optimization of particle bombardment conditions by ß-glucuronidase (GUS) reporter system in tomato fruit. African Journal of Biotechnology, 2011, 10(4): 675-683 (doi: 10.5897/AJB10.578).
- Столбиков А.С., Саляев Р.К., Рекославская Н.И., Третьякова А.В. Генетическая трансформация каллусов томата биобаллистическим методом с использованием гена GUS, кодирующего синтез ß-глюкуронидазы. Известия Иркутского государственного университета. Серия: Биология. Экология, 2015, 13: 2-8.
- Столбиков А.С., Саляев Р.К., Рекославская Н.И. Пластидная трансформация каллусов растений томата биобаллистическим методом при разработке вакцин против онкогенной папилломы человека на основе растительной экспрессионной системы. Мат. Всероссийской научно-практической конференции с международным участием и школы молодых ученых «Механизмы устойчивости растений и микроорганизмов к неблагоприятным условиям среды». Иркутск, 2018: 1378-1380.
- Baum K., Grqning B., Meier I. Improved ballistic transient transformation conditions for tomato fruit allow identification of organ-specific contributions of I-box and G-box to the RBCS2 promoter activity. The Plant Journal, 1997, 12(2): 463-469 (doi: 10.1046/j.1365-313X.1997.12020463.x).
- Wurbs D., Ruf S., Bock R. Contained metabolic engineering in tomatoes by expression of carot-enoid biosynthesis genes from the plastid genome. The Plant Journal, 2007, 49(2): 276-288 (doi: 10.1111/j.1365-313X.2006.02960.x).
- Abu-El-Heba G.A., Hussein G.M., Abdalla N.A. A rapid and efficient tomato regeneration and transformation system. Landbauforschung Völkenrode, 2008, 58: 103-110.
- Abbas D.E., Abdallah N.A., Madkour M.M. Production of transgenic tomato plants with enhanced resistance against the fungal pathogen Fusarium oxysporum. Arabian Journal of Biotechnology, 2009, 12(1): 73-84.
- Abdallah N.A., Shah D., Abbas D., Madkour M. Stable integration and expression of a plant defensin in tomato confers resistance to fusarium wilt. GM Crops, 2010, 1(5): 344-350 (doi: 10.4161/gmcr.1.5.15091).
- Jiménez V.A.O., Varela G.B., Domínguez M.R., Sacudo R.B., Rojas R.T., Hernández M.E.T. Development of a regeneration and genetic transformation protocol for tomato (Solanum lycoper-sicum L.) cv. Rutgers. Agrociencia, 2019, 53(5): 725-740.
- Kaplanoglu E., Kolotilin I., Menassa R., Donly C. Transplastomic tomato plants expressing insect-specific double-stranded RNAs: a protocol based on biolistic transformation. In: RNAi strategies for pest management. Humana, NY, 2022: 235-252 (doi: 10.1007/978-1-0716-1633-8_18).
- Rajkumari N., Alex S., Soni K.B., Anith K.N., Viji M.M., Kiran A.G. Silver nanoparticles for biolistic transformation in Nicotiana tabacum L. 3 Biotech, 2021, 11(12): 497 (doi: 10.1007/s13205-021-03043-9).
- Sanford J.C., Wolf E.D. Apparatus for transporting substances into living cells and tissues. US Patent № 5 371 015, December 6 1994.
- McCabe D., Christou P. Direct DNA transfer using electric discharge particle acceleration (AC-CELL™ technology). Plant Cell, Tissue and Organ Culture, 1993, 33(3): 227-236 (doi: 10.1007/BF02319006).
- Finer J.J., Vain P., Jones M.W., McMullen M.D. Development of the particle inflow gun for DNA delivery to plant cells. Plant Cell Reports, 1992, 11(7): 323-328 (doi: 10.1007/BF00233358).
- Sautter C., Waldner H., Neuhaus-Url G., Galli A., Neuhaus G., Potrykus I. Micro-targeting: high efficiency gene transfer using a novel approach for the acceleration of micro-projectiles. Nature Biotechnology, 1991, 9(11): 1080-1085 (doi: 10.1038/nbt1991-1080).
- Finer J.J., Finer K.R., Ponappa T. Particle bombardment mediated transformation. In: Plant biotechnology. Current topics in microbiology and immunology. V. 240 /J. Hammond, P. McGarvey, V. Yusibov (eds.). Springer, Berlin, Heidelberg, 2000: 59-80 (doi: 10.1007/978-3-642-60234-4_3).
- Chaithra N., Gowda R.P.H., Guleria N. Transformation of tomato with Cry2ax1 by biolistic gun method for fruit borer resistance. International Journal of Agriculture, Environment and Biotechnology, 2015, 8(4): 795-803 (doi: 10.5958/2230-732X.2015.00088.1).
- Жимулев И.Ф. Общая и молекулярная генетика. Новосибирск, 2003.
- Russell J.A., Roy M.K., Sanford J.C. Major improvements in biolistic transformation of suspension-cultured tobacco cells. In Vitro Cellular & Developmental Biology - Plant, 1992, 28(2): 97105 (doi: 10.1007/BF02823026).
- Trujillo-Moya C., Gisbert C., Vilanova S., Nuez F. Localization of QTLs for in vitro plant regeneration in tomato. BMC Plant Biology, 2011, 11: 140 (doi: 10.1186/1471-2229-11-140).
- Pino L.E., Lombardi-Crestana S., Azevedo M.S., Scotton D.C., Borgo L., Quecini V., Figueira A., Peres L.E. The Rg1 allele as a valuable tool for genetic transformation of the tomato 'Micro-Tom' model system. Plant Methods, 2010, 6(1): 23 (doi: 10.1186/1746-4811-6-23).
- Lech M., Miczyñski K., Pindel A. Comparison of regeneration potentials in tissue cultures of primitive and cultivated tomato species (Lycopersicon sp.). Acta Societatis Botanicorum Poloniae, 1996, 65(1-2): 53-56 (doi: 10.5586/asbp.1996.009).
- Khaliluev M.R., Bogoutdinova L.R., Baranova G.B., Baranova E.N., Kharchenko P.N., Dol-gov S.V. Influence of genotype, explant type, and component of culture medium on in vitro callus induction and shoot organogenesis of tomato (Solanum lycopersicum L.). Biology Bulletin, 2014, 41(6): 512-521 (doi: 10.1134/S1062359014060041).
- Klein T.M., Gradziel M.E., Fromm M., Sanford J.C. Factors influencing gene delivery into Zea mays cells by high-velocity microprojectiles. Nature Biotechnology, 1988, 6: 559-563 (doi: 10.1038/nbt0588-559).
- Krishna H., Alizadeh M., Singh D., Singh U., Chauhan N., Eftekhari M., Sadh R.K. Somaclonal variations and their applications in horticultural crops improvement. 3 Biotech, 2016, 6(1): 54 (doi: 10.1007/s13205-016-0389-7).
- Ranghoo-Sanmukhiya V.M. Somaclonal variation and methods used for its detection. In: Propagation and genetic manipulation of plants /I. Siddique (ed.). Springer, Singapore, 2021: 59-68 (doi: 10.1007/978-981-15-7736-9_1).
- Чесноков Ю.В. Наследственные изменения, вызванные переносом экзогенной ДНК в высшие растения посредством прорастающей пыльцы. Докт. дис. Минск, 2000.
- Чесноков Ю.В. Перенос чужеродных генов в зародышевый мешок высших растении посредством прорастающей пыльцы. Биополимеры и клетка, 1992, 8(2): 53-58.
- Kaur R.P., Devi S. In planta transformation in plants: a review. Agricultural Reviews, 2019, 40(3): 159-174 (doi: 10.18805/ag.R-1597).
- Hess D., Lorz H., Weissert E.-W. Die Aufnahme bakterieller DNA in quellende und keimende Pollen von Petunia hybrid und Nicotiana glauca [Uptake of bacterial DNA into swelling and germinating pollen grains of Petunia hybrid and Nicotiana glauca], Zeitschrift fur Pflanzenphysiologie, 1974, 74(1): 52-63 (doi: 10.1016/S0044-328X(74)80204-7).
- Hess D., Gresshoff P.M., Fielits U., Gleiss D. Uptake of protein and bacteriophage into swelling and germinating pollen of Petunia hybrida. Zeitschrift fur Pflanzenphysiologie, 1975, 74(4): 371376 (doi: 10.1016/S0044-328X(75)80164-4).
- Турбин Н.В., Сойфер В.Н., Картель Н.А. и др. Генетические изменения признака waxy у ячменя под влиянием экзогенной ДНК дикого типа. Сельскохозяйственная биология, 1974, 9(6): 204-215.
- Zhou G.Y., Weng J., Zeng Y., Huang J., Qian S., Liu G. Introduction of exogenous DNA into cotton embryos. In: Methods in enzymology. V. 101. Academic Press, 1983: 433-481 (doi: 10.1016/0076-6879(83)01032-0).
- Картэль М.А., Забянькова K.I. Генетычныя змены i магчымы мехашзм ix узшкнення пад дзеяннем ДНК у раслш. Весщ нацыянальнай акадэмп навук БеларуЫ. Серыя бiялагiчных навук, 1984, 6: 42-46.
- De la Pena А., Lorz H., Schell J. Transgenic rye plants obtained by injecting DNA into young floral tillers. Nature, 1987, 325(6101): 274-276 (doi: 10.1038/325274a0).
- Чесноков Ю.В., Седов Г.И., Виконская Н.А. Простой метод генетической трансформации двудольных растений. Известия АН МССР, 1989, 6: 61-62.
- Chen D.P., Yu L.J. Establishment of tomato transformation technique via the pollen tube pathway method. Northern Horticulture, 2010, 14: 131-135.
- Xiaoxia J., Li W., Yu L. Transgenic technology of pollen-tube pathway in tomato (Solanum lycopersicum M.). Molecular Plant Breeding, 2013, 11(4): 605-610.
- Wang R., Li R., Xu T., Li T. Optimization of the pollen-tube pathway method of plant transformation using the Yellow Cameleon 3.6 calcium sensor in Solanum lycopersicum. Biologia, 2017, 72(10): 1147-1155 (doi: 10.1515/biolog-2017-0127).
- Чесноков Ю.В., Седов Г.И., Виконская Н.А. Эффекты действия экзогенной ДНК у томата. I. Генетическая трансформация. Генетика, 1995, 31(5): 648-691.
- Wang Y., Shen J. Probing into cytological embryology mechanism of pollen-tube pathway transgenic technology. Acta Botanica Boreali-Occidentalia Sinica, 2005, 26: 628-634.
- Luo Z.-X., Wu R. A simple method for the transformation of rice via the pollen-tube pathway. Plant Molecular Biology Report, 1988, 6: 165-174 (doi: 10.1007/BF02669590).
- Jian C., Li K., Ou W. Research progress in pollen-tube pathway method in transgenic plants. Chinese Journal of Tropical Crops, 2012, 33(5): 956-961.
- Bechtold N., Ellis J., Pelletier G. In planta Agrobacterium mediated gene transfer by infiltration of adult Arabidopsis thaliana plants. Comptes Rendus de L'Acadümie des Sciences Serie Ill-Sciences De La Vie-Life Sciences, 1993, 316: 1194-1199.
- Bent A.F., Clough S.J. Agrobacterium germ-line transformation: transformation of Arabidopsis without tissue culture. In: Plant molecular biology manual /S.B. Gelvin, R.A. Schilperoort (eds.). Springer, Dordrecht, 1998: 17-30 (doi: 10.1007/978-94-011-5242-6_2).
- Bent A. Arabidopsis thaliana floral dip transformation method. In: Agrobacterium protocols. Methods in molecular biology. V. 343 /K. Wang (ed.). Humana Press, 2006: 87-104 (doi: 10.1385/159745-130-4:87).
- Yasmeen A., Mirza B., Inayatullah S., Safdar N., Jamil M., Ali S., Choudhry M.F. In planta transformation of tomato. Plant Molecular Biology Reporter, 2009, 27: 20-28 (doi: 10.1007/s11105-008-0044-5).
- Hilioti Z., Ganopoulos I., Ajith, S., Bossis I., Tsaftaris A. A novel arrangement of zinc finger nuclease system for in vivo targeted genome engineering: the tomato LEC1-LIKE4 gene case. Plant Cell Reports, 2016, 35(11): 2241-2255 (doi: 10.1007/s00299-016-2031-x).
- Кузьмина Ю.В. Методы редактирования генома для увеличения лёжкости плодов томата. Биотехнология и селекция растений, 2020, 3(1): 31-39 (doi: 10.30901/2658-6266-2020-1-o6).
- Kuluev B.R., Gumerova G.R., Mikhaylova E.V., Gerashchenkov G.A., Rozhnova N.A., Vershinina Z.R., Khyazev A.V., Matniyazov R.T., Baymiev A.K., Chemeris A.V. Delivery of CRISPR/Cas components into higher plant cells for genome editing. Russian Journal of Plant Physiology, 2019, 66(5): 694-706 (doi: 10.1134/S102144371905011X).
- Nemudryi A.A., Valetdinova K.R., Medvedev S.P., Zakian S.M. TALEN and CRISPR/Cas genome editing systems: tools of discovery. Acta Naturae, 2014, 6(3): 19-40.
- Gerashchenkov G.A., Rozhnova N.A., Kuluev B.R., Kiryanova O.Yu., Gumerova G.R., Knyazev A.V., Vershinina Z.R., Mikhailova E.V., Chemeris D.A., Matniyazov R.T., Baimiev A.Kh., Gubaidullin I.M., Baimiev A.Kh., Chemeris A.V. Design of guide RNA for CRISPR/Cas plant genome editing. Molecular Biology, 2020, 54(1): 24-42 (doi: 10.1134/S0026893320010069).
- Malzahn A., Lowder L., Qi Y. Plant genome editing with TALEN and CRISPR. Cell & Bioscience, 2017, 7(1): 21 (doi: 10.1186/s13578-017-0148-4).
- Zhan X., Lu Y., Zhu J.K., Botella J.R. Genome editing for plant research and crop improvement. Journal of Integrative Plant Biology, 2021, 63(1): 3-33 (doi: 10.1111/jipb.13063).
- Feder A., Jensen S., Wang A.Q., Courtney L., Middleton L., Van Eck J., Liu Y.S., Giovan-noni J.J. Tomato fruit as a model for tissue-specific gene silencing in crop plants. Horticulture Research, 2020, 7: 142 (doi: 10.1038/s41438-020-00363-4).
- Liu L., Zhang J., Xu J., Li Y., Guo L., Wang Z., Zhang X., Zhao B., Guo Y.D., Zhang N. CRISPR/Cas9 targeted mutagenesis of SlLBD40, a lateral organ boundaries domain transcription factor, enhances drought tolerance in tomato. Plant Science, 2020, 301: 110683 (doi: 10.1016/j.plantsci.2020.110683).
- Danilo B., Perrot L., Mara K., Botton E., Nogue F., Mazier M. Efficient and transgene-free gene targeting using Agrobacterium-mediated delivery of the CRISPR/Cas9 system in tomato. Plant Cell Reports, 2019, 38(4): 459-462 (doi: 10.1007/s00299-019-02373-6).
- Nekrasov V., Wang C., Win J., Lanz C., Weigel D., Kamoun S. Rapid generation of a transgenefree powdery mildew resistant tomato by genome deletion. Scientific Reports, 2017, 7(1): 482 (doi: 10.1038/s41598-017-00578-x).
- Ortigosa A., Gimenez-Ibanez S., Leonhardt N., Solano R. Design of a bacterial speck resistant tomato by CRISPR/Cas9-mediated editing of SlJAZ2. Plant Biotechnology Journal, 2019, 17: 665673 (doi: 10.1111/pbi.13006).
- Martinez M.I.S., Bracuto V., Koseoglou E., Appiano M., Jacobsen E., Visser R.G., Wolters A.A., Bai Y. CRISPR/Cas9-targeted mutagenesis of the tomato susceptibility gene PMR4 for resistance against powdery mildew. BMC Plant Biology, 2020, 20(1): 284 (doi: 10.1186/s12870-020-02497-y).
- Yoon Y.J., Venkatesh J., Lee J.H., Kim J., Lee H.E., Kim D.S., Kang B.C. Genome editing of eIF4E1 in tomato confers resistance to pepper mottle virus. Frontiers in Plant Science, 2020, 11: 1098 (doi: 10.3389/fpls.2020.01098).
- Ito Y., Sekiyama Y., Nakayama H., Nishizawa-Yokoi A., Endo M., Shima Y., Nakamura N., Kotake-Nara E., Kawasaki S., Hirose S., Toki S. Allelic mutations in the Ripening-inhibitor locus generate extensive variation in tomato ripening. Plant Physiology, 2020, 183: 80-95 (doi: 10.1104/pp.20.00020).
- Li S., Xu H.J.L., Ju Z., Cao D.Y., Zhu H.L., Fu D.Q., Grierson D., Qin G.Z., Luo Y.B., Zhu B.Z. The RIN-MC fusion of MADS-box transcription factors has transcriptional activity and modulates expression of many ripening genes. Plant Physiology, 2018, 176: 891-909 (doi: 10.1104/pp.17.01449).
- Hunziker J., Nishida K., Kondo A., Kishimoto S., Ariizumi T., Ezura H. Multiple gene substitution by Target-AID base-editing technology in tomato. Scientific Reports, 2020, 10(1): 20471 (doi: 10.1038/s41598-020-77379-2).
- Nonaka S., Arai C., Takayama M., Matsukura C., Ezura H. Efficient increase of gamma-amino-butyric acid (GABA) content in tomato fruits by targeted mutagenesis. Scientific Reports, 2017, 7: 7057 (doi: 10.1038/s41598-017-06400-y).
- Deng L., Wang H., Sun C.L., Li Q., Jiang H.L., Du M.M., Li C.B., Li C.Y. Efficient generation of pink-fruited tomatoes using CRISPR/Cas9 system. Journal of Genetics and Genomics, 2018, 45: 51-54 (doi: 10.1016/j.jgg.2017.10.002).
- D'Ambrosio C., Stigliani A.L., Giorio G. CRISPR/Cas9 editing of carotenoid genes in tomato. Transgenic Research, 2018, 27(4): 367-378 (doi: 10.1007/s11248-018-0079-9).
- Zsögön A., Cermak T., Naves E.R., Notini M.M., Edel K.H., Weinl S., Freschi L., Voytas D.F., Kudla J., Peres L.E.P. De novo domestication of wild tomato using genome editing. Nature Biotechnology, 2018, 36(12): 1211-1216 (doi: 10.1038/nbt.4272).
- Li T., Yang X., Yu Y., Si X., Zhai X., Zhang H., Dong W., Gao C., Xu C. Domestication of wild tomato is accelerated by genome editing. Nature Biotechnology, 2018, 36: 1160-1163 (doi: 10.1038/nbt.4273).
- Cermak T., Baltes N.J., Cegan R., Zhang Y., Voytas D.F. High-frequency, precise modification of the tomato genome. Genome Biology, 2015, 16(1): 232 (doi: 10.1186/s13059-015-0796-9).
- Nicolia A., Andersson M., Hofvander P., Festa G., Cardi T. Tomato protoplasts as cell target for ribonucleoprotein (RNP)-mediated multiplexed genome editing. Plant Cell, Tissue and Organ Culture, 2021, 144(2): 463-467 (doi: 10.1007/s11240-020-01954-8).
- Lin Y.C. DNA-free CRISPR-Cas9 gene editing of wild tetraploid tomato Solanum peruvianum using protoplast regeneration. Plant Physiology, 2022, 188(4): 1917-1930 (doi: 10.1093/plphys/kiac022).
- Lu Y., Tian Y., Shen R., Yao Q., Zhong D., Zhang X., Zhu J. K. Precise genome modification in tomato using an improved prime editing system. Plant Biotechnology Journal, 2021, 19(3): 415417 (doi: 10.1111/pbi. 13497).


