Участие ретроэлементов в хромоанагенезе при развитии злокачественных новообразований
Автор: Мустафин Р.Н.
Журнал: Сибирский онкологический журнал @siboncoj
Рубрика: Обзоры
Статья в выпуске: 5 т.23, 2024 года.
Бесплатный доступ
цель исследования - определить роль ретроэлементов в механизмах хромоанагенеза при развитии злокачественных новообразований. материал и методы. Поиск соответствующих источников проводился в системах Scopus, Web of Science, PubMed, Elibrary с включением публикаций с февраля 2002 г. по декабрь 2023 г. Из 864 найденных научных статей об эпигенетических и молекулярно-генетических механизмах хромоанагенеза 64, содержащие информацию о вовлеченности в эти процессы ретроэлементов, были использованы для написания систематического обзора.
Ретроэлементы, канцерогенез, комплексные хромосомные перестройки, транспозиции, хромоанагенез, хромоплексия, хромотрипсис
Короткий адрес: https://sciup.org/140307913
IDR: 140307913 | УДК: 616-006.04:576.316 | DOI: 10.21294/1814-4861-2024-23-5-146-156
Текст обзорной статьи Участие ретроэлементов в хромоанагенезе при развитии злокачественных новообразований
Современные возможности секвенирования генома человека в злокачественных новообразованиях (ЗНО) позволили выявить наличие комплексных хромосомных перестроек, к которым относится хромотрипсис (ХТ) [1]. Термин был введен в 2011 г. P.J. Stephens et al. для характеристики комплексных хромосомных перестроек (КХП) при хроническом лимфолейкозе [2]. ХТ характеризуется развитием сложных структурных вариаций [3] с обширными изменениями в геноме и с различным количеством копий ДНК, которые ограничены одной или несколькими хромосомами. В процессе ХТ могут происходить фрагментация и последующая сборка одной хроматиды из аберрантной ядерной структуры (микроядра) [1]. В результате образуются новые мозаичные хромосомы, в которых отдельные фрагменты вновь соединены между собой в случайном порядке и ориентации. Наблюдаются также слияния теломер с последующим образованием хромосомного моста [3] при экзогенных и эндогенных повреждениях ДНК [4].
Для характеристики КХП был предложен термин «хромоанагенез», который включает в себя не только ХТ, но и хромоанасинтез (ХА) и хромоплексию (ХП) [5]. Механизмами образования КХП являются негомологичные соединения концов (НГСК), неаллельная гомологичная рекомбинация (НАГР), остановка вилки репликации, переключение матрицы (fork stalling and template switching – FoSTeS) и опосредованная микрогомологией, индуцированная разрывами репликация
(microhomology-mediated break-induced replication – MMBIR) [6]. Данные механизмы относятся к системам репарации ДНК, которые в норме необходимы для поддержания целостности и стабильности генома. Они направлены на защиту генома от различных повреждений, таких как несоответствие А-G или Т-С, образование пиримидиновых димеров, одноцепочечные или двуцепочечные разрывы ДНК [4]. Предполагается, что ХТ происходит из-за физической изоляции хромосом в микроядрах. Эти события ограничиваются ошибочно сегрегированной хромосомой в пределах одного клеточного деления [1]. Однако есть данные о том, что при заключении хромосомы в микроядро ХТ-подобные перестройки могут возникать за три клеточных цикла, так как микроядро является последствием ошибок сегрегации хромосом при митозе [7].
Сборка фрагментов ДНК при ХТ может сопровождаться удалением части из них, тогда как дупликация практически полностью отсутствует. Для ХТ характерны множественные разрывы хромосом. В результате чередуются участки генома, сохранившие и утратившие гетерозиготность вследствие перестроек, затрагивающих один гаплотип (одну из двух гомологичных хромосом). Характерны также случайный порядок и ориентация фрагментов ДНК в производных хромосомах и возможность восстановления производной хромосомы путем соединения точек разрыва [4]. При ХТ определяются незначительная микрогомология или ее отсутствие в местах соединения двуцепочечных разрывов ДНК [8]. Расположение двуцепочечных разрывов в высокоповторяющихся последовательностях обнаружено только в отдельных случаях конституционального ХТ [9, 10].
Частота встречаемости ХТ при разных типах ЗНО значительно отличается. Например, исследование рака толстой кишки с помощью полногеномного секвенирования и профилирования массива SNP показало наличие ХТ почти в каждом образце рака [11]. ХТ определяется более чем в 33 % случаев остеосарком и глиобластом [3]. Проведенное в 2020 г. полногеномное секвенирование 2 658 различных ЗНО человека показало наличие ХТ более чем в 50 % образцов опухолей. При этом было отмечено, что ХТ способствует амплификации онкогенов и инактивации генов репарации ДНК [12]. Помимо ХТ, ограниченного главным образом одной хромосомой, в ЗНО наблюдаются более широкомасштабные структурные перестройки генома с вовлечением большого количества хромосом, названные ХП [13]. Они характерны для 90 % образцов рака предстательной железы и представляют собой КХП между хромосомами и внутри них в виде замкнутых цепей. При этом происходят несколько последовательных КХП (от 3 до 40, с участием 6 и более хромосом) в виде множественных соединений разрывов, зависимых от последовательностей нуклеотидов, которые формируются скоординированно [14]. Нужно от- метить, что при ХП происходит активация онкогенов и инактивация генов супрессоров опухолей (ГСО) [13]. Однако, в отличие от ХТ (для которого характерно преимущественное поражение одной хромосомы, хотя могут происходить нарушения и в нескольких [15]), при ХП происходит одновременное нарушение регуляции многочисленных генов в нескольких хромосомах [16] (рис. 1).
Для ЗНО характерен также ХА – образование кластеров перестроек с изменением числа копий ДНК путем MMBIR во время репликации ДНК. После распада участков хромосом при ХА или их разрушения при ХТ происходит сшивание фрагментов хромосом в случайном порядке и ориентации. При ХП же сохраняется первоначальная ориентация участков хромосом, а новые сшивки характеризуются низкой микрогомологией или ее отсутствием. После ХА и ХП места соединений могут быть фланкированы областями с микрогомологией и вырезанием значительного участка в точке разрыва с инсерцией последовательностей, не соответствующих матричной ДНК. При ХТ этот ответ преимущественно обусловлен НГСК [17].
Отличием различных типов хромоанагенеза является также то, что точки разрывов при ХП группируются в активно транскрибируемой ДНК с высоким содержанием GC и связаны с областями генома, содержащими активно транскрибируемые
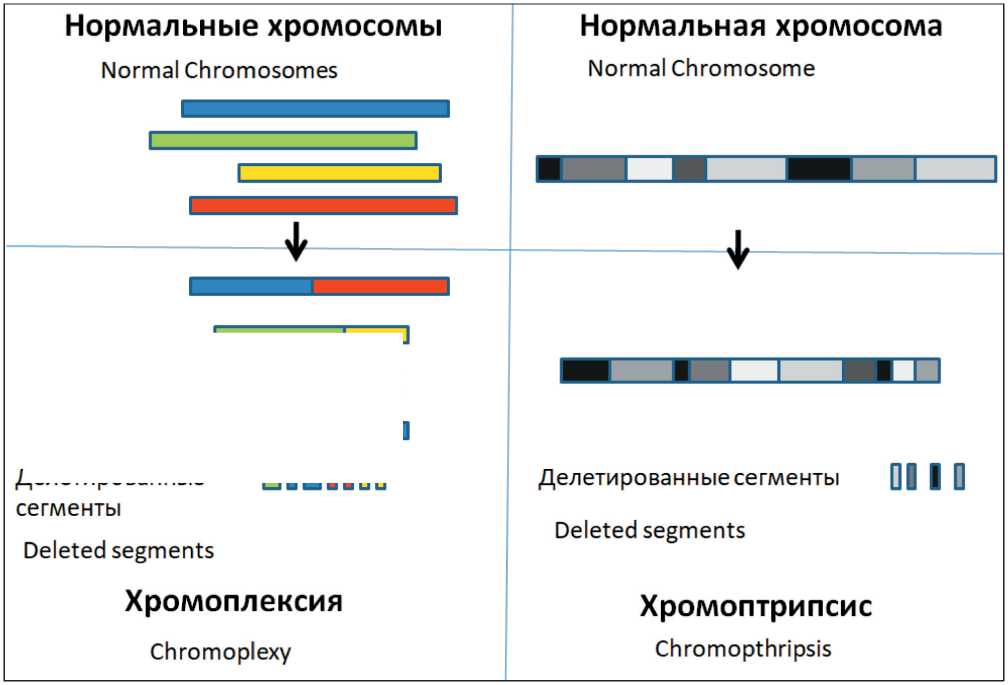
Рис. 1. Схема механизмов хромотрипсиса и хромоплексии. При хромоплексии происходят широкомасштабные структурные перестройки генома с вовлечением большого количества хромосом и сохранением первичной ориентации их участков. При хромотрипсисе сложные структурные вариации затрагивают одну хромосому с фрагментацией и последующей сборкой одной хроматиды из микроядра (аберрантной ядерной структуры). Примечание: рисунок выполнен автором
Fig. 1. Scheme of chromothripsis and chromoplexy mechanisms. Chromoplexy involves large-scale structural rearrangements of the genome involving a large number of chromosomes and preservation of the primary orientation of their loci. In chromothripsis, complex structural variations affect one chromosome with fragmentation and subsequent assembly of one chromatid from a micronucleus (aberrant nuclear structure). Note: created by the author
гены, и открытой конфигурацией хроматина [3]. ХП и ХТ характеризуются случайными деле-циями и слиянием сегментов хромосом с низким числом копий, опосредованных НГСК, но при ХТ происходят сотни разрывов в пределах одной или нескольких хромосом [15]. При ХП последовательные перестройки не кластеризованы и исчисляются десятками, с вовлечением нескольких хромосом. Кроме того, ХТ возникает как единичное клональное событие, т.е. комплексные перестройки возникают однократно в клетке, которая становится источником целого клона с измененными хромосомами при развитии ЗНО. При хромоплексии же КХП возникают более одного раза на разных стадиях развития опухоли в клетках, которые становятся источниками клонов клеток с измененными хромосомами (в некоторых из них может также возникать ХП, что приводит к образованию субклонов) [13]. В табл. 1 представлены отличительные особенности ХТ, ХП и ХА.
Помимо ЗНО, хромоанагенез, в особенности ХТ, может возникать в половых клетках, зиготах и в раннем эмбриогенезе, приводя к формированию врожденных пороков развития [11]. В клетках пациентов с наследственными болезнями, обусловленными нарушениями репарации ДНК (атаксия-телеангиэктазия, синдромы Неймеген и Блума), наблюдается хромосомная нестабильность в виде множества последовательных событий [4]. Несмотря на значительные изменения структуры хромосом при таких КХП, геном человека может проявлять толерантность к экстремальным хромосомным перестройкам без заметных фенотипических эффектов. Встречаются случаи, когда герминальный ХТ обнаруживали у людей без признаков патологии с развитием тяжелых врожденных пороков развития у их детей [18, 19]. Причинами такого феномена могут быть отсутствие очевидных фенотипических проявлений генов, содержащих CNV (copy number variation – вариа-
Таблица 1/ table 1
Дифференциальные характеристики различных типов хромоанагенеза differential characteristics of different types of chromoanagenesis
|
Название процесса/ Designation |
Характер изменений/ CHANGES nature |
Вовлеченные хромосомы/ Involved chromosomes |
Особенности процесса/ Features of the process |
Результат процесса/ Result |
|
S и 2 & 2 о X и |
Сложные структурные вариации, сотни разрывов/ Complex structural variations, hundreds of gaps |
Одна, реже несколько хромосом/ One, rarely several chromosomes |
Однократная фрагментация и последующая случайная сборка одной хроматиды из аберрантной ядерной структуры (микроядра)/ Single fragmentation and subsequent random assembly of one chromatid from an aberrant nuclear structure (micronucleus) |
Образование новой мозаичной хромосомы, хромосомного моста/ Formation of a new mosaic chromosome, a chromosomal bridge |
|
s га s ■ о X Z- |
Широкомасштабные структурные перестройки генома, десятки разрывов/ Largescale structural rearrangements of the genome, dozens of gaps |
Большое количество хромосом/ large number of chromosomes |
Несколько последовательных скоординированных перестроек хромосом с сохранением первичной ориентации участков хромосом/ Several successive coordinated rearrangements of chromosomes with preservation of the primary orientation of chromosome regions |
Нарушение регуляции многих генов в разных хромосомах/ Dysregulation of multiple genes on different chromosomes |
|
сл ^ сл 2 га О о |
Изменение числа копий ДНК путем MMBIR во время репликации ДНК/ Change in DNA copy number by MMBIR during DNA replication |
Несколько хромосом/ Several chromosomes |
Распад участков хромосом с их сшиванием в случайном порядке и ориентации, образование кластеров перестроек хромосом/ Disintegration of chromosome sections with their stitching together in random order and orientation, formation of chromosome rearrangement clusters |
Образование новых мозаичных хромосом/ Formation of new mosaic chromosomes |
|
ел га |
Структурные перестройки хромосом/ Structural rearrangements of chromosomes |
От одной до нескольких хромосом/ From one to tens of chromosomes |
Единовременные или многократные перестройки хромосом/ Single or multiple chromosome rearrangements |
Формирование новых мозаичных хромосом с нарушением регуляции генов/ Formation of new mosaic chromosomes with disruption of gene regulation |
Примечание: таблица составлена автором.
Note: created by the author.
ция числа копий), компенсация функций генов с помощью механизмов изменения транскрипции с использованием транскрипционных факторов или эпигенетических процессов (метилирование ДНК и модификации гистонов). Одним из возможных объяснений отсутствия клинических проявлений ХТ в описанных случаях является также компенсация структурно-функциональных изменений в геноме, обусловленная участием в данных перестройках транспозонов [19].
Роль ретроэлементов в развитии злокачественных опухолей
Транспозоны – это специфические участки ДНК, которые способны перемещаться в новые локусы генома. Они классифицируются на ретроэлементы (РЭ), транспозиция которых происходит с помощью промежуточной РНК и обратной транскриптазы, и ДНК-транспозоны, перемещающиеся путем «вырезания и вставки» или «катящегося кольца» с участием транспозазы. В геноме человека РЭ занимают более 35 % всех последовательностей ДНК: 8 % составляют содержащие LTR (long terminal repeats) РЭ и 27 % – не содержащие LTR (nonLTR-РЭ). Последние классифицируют на автономные LINE1 и неавтономные (SINE и SVA) ретроэлементы. LTR-РЭ включают в себя эндогенные ретровирусы HERV (human endogenous retroviruses) [20]. Компенсация структурно-функциональных изменений в геноме, обусловленная участием РЭ в хромосомных перестройках при ХТ, может быть связана с тем, что на протяжении эволюции РЭ играли ключевую роль в генной регуляции и преобразованиях геномов под действием стрессоров, что отражалось в адаптивных фенотипических изменениях организмов [20]. Поэтому индуцированные РЭ события могут быть компенсированы генными сетями, находящимися под регуляторным влиянием РЭ, что на уровне целого организма не вызывает существенных изменений. Более того, имеются примеры, когда преобразования геномов, вызванные активностью РЭ, используются для регуляции функции клеток в онтогенезе. Так, отдельные нейроны головного мозга здорового человека отличаются наличием множества вставок РЭ, которые влияют на особенности их функционирования. При этом ассоциированные с LINE1 соматические варианты нейронов обусловлены как MMBIR, так и инсерциями, т.е. LINE1-ассоциированные области перестройки генома являются горячими точками для соматического мозаицизма, которые влияют на 44–63 % нейронов ЦНС [21].
Подобно хромоанагенезу [12], характерной особенностью более половины различных ЗНО является активация РЭ, которые вызывают образование химерных онкогенов и инактивируют ГСО [22], что наблюдается также при ХТ [12] и ХП [13] и может быть обусловлено влиянием РЭ. Действительно, в среднем для различных типов
ЗНО определена практически идентичная частота встречаемости ХТ (50 %) [12] и вовлечения РЭ в онкоэкзаптацию (активацию РЭ, вызывающую образование химерных онкогенов и инактивацию ГСО) (49,7 %) [23]. При онкоэкзаптации мутации, обусловленные влиянием РЭ, становятся драйверными, приводя к образованию клона клеток для дальнейшего онкогенеза. Данное свойство можно использовать для планирования таргетной терапии ЗНО с использованием РЭ в качестве объектов воздействия. Это позволило бы предотвратить клональную эволюцию ЗНО вследствие приобретения ими адаптивных свойств при участии РЭ [22], что связано с ролью РЭ не только в патологических процессах, но также в адаптации к различным условиям внешней среды, в том числе и к стрессу [20]. Соответственно, РЭ могут стать основой для образования клонов опухолевых клеток с новыми свойствами, способствующими прогрессированию ЗНО. Необходимо отметить, что эволюционно молодые эндогенные ретровирусы HERV-K (HML-2) при их активации в ЗНО экспрессируются в онкогенные белки, такие как Np9, коактиватор β-катенина, ERK, Akt и Notch [24].
Образование химерных онкогенов в результате инсерции РЭ в области протоонкогенов обусловлено их использованием в качестве альтернативных промоторов без нарушения открытой рамки считывания новых последовательностей, полученных от РЭ [25–27], расположенных в интронах. В результате новые белковые продукты характеризуются инициирующим и усиливающим воздействием на канцерогенез [28–31]. Помимо этого, РЭ являются инструментами для возникновения ретрогенов с онкогенными свойствами, поскольку для данного процесса используются обратная транскриптаза и эндонуклеаза РЭ. Например, из гена NANOG в эволюции возник таким способом ретроген NANO-GP8, который в геноме человека стимулирует рост и размножение опухолевых клеток [32].
Примером химерных генов, сформированных в результате аномальной экспрессии из областей РЭ, инсертированных в интрон нормального протоонкогена, являются LTR2-FABP7 (из протоонкогена FABP7 ) [29], LTR-ALK (из гена ALK ) [31], LTR-ERBB4 [30], LINE1-MET . Анализ генома человека позволил обнаружить около 1 000 различных генов, содержащих в своем составе остатки последовательностей инсертированных в эволюции LINE, обладающих потенциалом образования альтернативных промоторов, с которых могут транскрибироваться аномальные химерные онкогены [28]. При метастазирующем колоректальном раке были выявлены такие свойства для генов MET, RAB3IP, CHRM3 [26]. При лимфоме Ходжкина сходные механизмы образования онкогенов описаны для инсерций эндогенных ретровирусов в область гена CSF1R [27] и IRF5 [25].
РЭ являются сенсорами экзогенных и эндогенных воздействий, в том числе гормональных изменений. Такая активация РЭ может объяснить механизмы прогрессирования рака простаты, характеризующегося слиянием гена эритробласт-ного трансформирующего онкогена ERG с геном изоформы 2 трансмембранной сериновой протеазы TMPRSS2. Слияние происходит вследствие разрыва двухцепочечной ДНК, индуцированного стимуляцией рецепторов андрогенов и генотоксическим стрессом [33]. Описанные изменения могут быть охарактеризованы как комплексные хромосомные перестройки, активное участие в которых принимают РЭ [34–40], НГСК [41, 42] и MMBIR [21, 36], которые являются основными механизмами всех форм хромоанагенеза [3, 4]. Необходимо остановиться на рассмотрении перечисленных данных более подробно.
Роль транспозоновв развитии хромоанагенеза
Доказано, что активированные non-LTR РЭ могут приводить к развитию конституционального ХТ. Потенциальный сайт расщепления эндонуклеазы LINE1 способствует двуцепочечным разрывам, вызывая в последующем репарацию посредством гомологичной рекомбинации внутри данных областей. При этом обнаруживаются инсерции 5’-усеченных SVA в одном из соединений точек разрыва в области, напоминающей консенсусный сайт расщепления LINE1 эндонуклеазы (5’-TTTT/ AA-3’). Была определена микрогомология 5’-конца вставки SVA с последовательностью в области точки разрыва дистального фрагмента [10]. Поскольку обратная транскрипция SVA при помощи продуктов экспрессии ORF2p LINE1-элементов может функционировать как «молекулярный пластырь» [43] двуцепочечных разрывов (соединять их с помощью своих транскриптов), а опосредованные LINE1 инсерции вызывают делеции протяженных участков генома, данные РЭ являются источниками ХТ [21].
Роль РЭ в формировании ХТ обусловлена генерированием двуцепочечных разрывов ДНК. При этом характерна ориентация фрагментов ДНК для воздействия на производную хромосомную структуру (инверсии, опосредованные рекомбинацией РЭ, происходят при ошибочном спаривании Alu), в противоположной ориентации с последующей рекомбинацией. Кроме того, РЭ функционируют в качестве «молекулярных пластырей», поскольку их РНК посредством микрогомологичных взаимодействий могут использоваться для соединения фрагментов ДНК. РЭ способны образовывать мостики дистальных фрагментов ДНК (РНК ретроэлемента с помощью микрогомологичных взаимодействий может служить средством связывания для дистальных последовательностей нуклеотидов ДНК) [44]. Необходимо отметить также, что активация
РЭ наблюдается в те же периоды онтогенеза (эмбриональное развитие) [45] и при патологических состояниях (ЗНО) [22, 23], что и ХТ. В большинстве образцов колоректального рака [11] обнаруживают ХТ, для которого ранним событием, ассоциированным с плохим прогнозом, является гипометилирование LINE1. При этом значительное снижение уровней метилирования LINE1 наблюдается при развитии опухолей в более молодом возрасте [46]. Проведенный в 2014 г. метаанализ показал характерное гипометилирование LINE1 и при других ЗНО человека [47].
При ХТ индуцированных LINE1-элементами двуцепочечных разрывов ДНК больше, чем ин-серций РЭ в области этих разрывов, что свидетельствует об их роли в развитии ХТ. Кроме того, активированные РЭ способствуют крупным хромосомным перестройкам, как это было показано для LINE1 и SVA, т.е. эндонуклеазная активность РЭ может служить инициатором хромосомных перестроек при ХТ. В постмитотических соматических клетках [43] (т. е. в высокодифференцированных, не способных к дальнейшему делению клетках, таких как нейроны головного мозга) LINE способствуют образованию CNV за счет НГСК [17]. При этом НГСК, опосредованное РЭ, играет роль не только в репарации патологических двунитевых разрывов ДНК при хромосомных транслокациях, но также в работе иммунной системы – в V(D)J рекомбинации при ранней дифференцировке лимфоцитов для формирования антител и Т-клеточных рецепторов [48].
Причинами КХП при хромоанагенезе являются активации РЭ, вызывающих транслокации и другие события геномной нестабильности, наблюдаемые при ЗНО. Об этом свидетельствует роль LINE в НГСК [41], характерном для ХТ [4]. Кроме того, при плоскоклеточном раке головы и шеи происходят КХП в хромосомах 12р, 8р, 3q, 14q, 6p, 4q, Xq, 8q, в точках разрыва которых располагаются главным образом LINE и SINE [39]. В клетках млекопитающих инвертированные Alu-повторы служат горячими точками КХП [40]. В геномах приматов за последние 40 млн лет происходили массивные экспансии Alu, приводящие к значительным КХП, в том числе с вовлечением генов онкосупрессоров, таких как BRCA1 [38]. Так, в тридцати локусах на 12 хромосомах человека выявлены комплексные LTR-РЭ, являющиеся источниками крупных геномных преобразований и сегментарных дупликаций [37]. РЭ могут служить потенциальными источниками для MMBIR, как это было показано в отношении Alu-Alu-опосредованных изменений в локусе 17p12 человека при болезни Шарко– Мари–Тута [36]. В развитии MMBIR описано также участие LINE1 [21].
В экспериментах на опухолевых клеточных культурах человека HeLa, линиях клеток кур с нокдауном по LINE1 и на рыбках данио было показано, что дефицит белков НГСК снижает частоту ретротранспозиций, так как НГСК участвует в механизмах перемещений LINE1 [41]. Данные особенности также указывают на вовлечение ретроэлементов в ХТ, для которого характерно соединение точек разрывов с незначительной микрогомологией или без нее [4]. В эксперименте при переносе человеческих LINE1 и интронов группы II в бактерии количество этих РЭ увеличивалось путем репарации ДНК в механизмах НГСК [42]. Кластеризованные CNV, обнаруживаемые с помощью анализа хромосомных микрочипов, часто регистрируются как герминальный ХТ. Это связано с тем, что большинство зарегистрированных CNV, опосредованных Alu, представлено простыми делециями и дупликациями, а опосредованные Alu-Alu перестройки обусловлены НАГР (характерного механизма ХТ). С помощью полногеномного секвенирования было показано, что данные перестройки могут быть опосредованы элементами Alu и LINE [4]. Действительно, Alu вносят значительный вклад в нестабильность генома, генерируя Alu-опосредованные
Таблица 2/table 2
Роль ретроэлементов в развитии злокачественных новообразований с участием механизмов хромоанагенеза
the role of retroelements in cancer development involving the mechanisms of chromoanagenesis
Механизм канцерогенеза/ Mechanism of carcinogenesis
Вовлеченность хромоанагенеза [43]/ Involvement of chromoanagenesis [43]
Роль ретроэлементов/ Role of retroelements
Двуцепочечные разрывы ДНК/ DNA double-strand breaks
Причина хромотрипсиса [43]/ Cause of chromothripsis [43]
Вызывают разрывы ДНК [43], которых больше, чем инсерций [47]/
Cause DNA [43] breaks, which are more numerous than insertions [47]
Инактивация генов супрессоров опухолей/ Inactivation of tumor suppressor genes
Геномные перестройки при хромотрип- Инсерция в области генов с образованием сисе [12] и хромоплексии [13]/ дефектного продукта гена [22, 23]/
Genomic rearrangements in chromothrip- Insertion in a gene region with defective gene sis [12] and chromoplexy [13] product formation [22, 23]
Активация онкогенов/ Activation of oncogenes
Геномные перестройки при хромотрип-сисе [12] и хромоплексии [13]/
Genomic rearrangements in chromothrip-sis [12] and chromoplexy [13]
Инсерция в протоонкоген с образованием химерного онкогена [22, 23, 25–31]/
Insertion into the proto-oncogene region with chimeric oncogene formation [22, 23, 25–31]
Экспрессия онкогенных белков из собственных генов ERV [24]/
Expression of oncogenic proteins from ERV self- genes [24]
Комплексные хромосомные перестройки/ Сomplex chromosomal rearrangements
Являются причинами хромотрипсиса, хромоплексии и хромоанасинтеза [1, 3, 8, 13, 14, 17]/
Are the causes of chromothripsis, chromoplexy and chromoanasynthesis [1, 3, 8, 13, 14, 17]
Драйверы перестроек вследствие наличия множества копий в геноме с комплементарными последовательностями [34–40, 43]/ Drivers of rearrangements due to the presence of multiple copies in the genome with complementary sequences [34–40, 43]
Неаллельная гомологичная рекомбинация/ Nonallelic homologous recombination
Негомологичные соединения концов/ Nonhomologous end joining
Причина хромотрипсиса [6]/ cause of chromothripsis [6]
LINE1, SVA [10] и Alu [49] являются источниками последовательностей для этого механизма/
LINE1, SVA [10] and Alu [49] are sequences source for this mechanism
Причина хромотрипсиса [4]/ Cause of chromothripsis [4]
Используются как «молекулярные пластыри» двуцепочечных разрывов ДНК [21, 41, 42]/ Used as “molecular band-aid” for double-stranded DNA breaks [21, 41, 42]
Репликация, опосредованная микрогомологией, индуцированная разрывами/ Microhomology-mediated break-induced replication
Причина хромоанасинтеза [17] LINE1 [21] и Alu [36] являются источниками и хромотрипсиса [6]/ последовательностей для данных механизмов/
Cause of chromoanasynthesis [17] and LINE1 [21] and Alu [36] are sources of sequenc- chromothripsis [6] es for these mechanisms
Кластеризированные CNV/Сlustered CNVs
Результат хромотрипсиса [4]/ Result of chromothripsis [4]
Образуются с помощью последовательностей LINE и Alu [4]/
Formed using LINE and Alu sequences [4]
Примечание: таблица составлена автором.
Note: created by the author.
CNV. Большинство из них представляет собой простые делеции и дупликации, происходящие за счет НАГР [49]. В основе формирования CNV могут лежать и другие механизмы образования хромосомных перестроек, такие как НГСК, НАГР, FoSTeS, MMBIR [6]. В таблице 2 представлены механизмы канцерогенеза с участием ХТ, ХП и ХА, в которых играют роль ретроэлементы.
Влияние хромоанагенеза и ретроэлементов на супрессоры опухолей
Активация РЭ при развитии ЗНО вызывает геномную нестабильность не только вследствие образования химерных онкогенов [22, 23, 25–31], экспрессии онкогенных белков [24], но также из-за инсерций в горячие точки мутагенеза ГСО [50–54], белковые продукты которых подавляют активность РЭ [55–58]. Кроме того, LINE1 контролирует транскрипцию гена WT1 [59], а герминальные мутации гена TP53 вызывают ХТ [4, 17] в развитии опухолей при наследственных опухолевых синдромах. В то же время РЭ инициируют ХТ и канцерогенез вследствие их влияния на ГСО, которые отличаются наличием горячих точек мутагенеза для РЭ [50–54]. Роль ХТ в инициировании образования опухолей при наследственных опухолевых синдромах можно объяснить активацией РЭ вследствие герминальной мутации одного аллеля онкосупрессорного гена, который в норме вызывает сайленсинг РЭ. Например, при синдроме Ли–Фраумени герминальная мутация в гене TP53 служит причиной ХТ [17]. Для гена TP53 доказана также роль в инактивации РЭ, что обусловлено ингибирующим воздействием его белкового продукта р53 в качестве транскрипционного фактора на сайты связывания РЭ [58]. Это свидетельствует о косвенной взаимосвязи ретроэлементов с ХТ, поскольку в случае синдрома Ли–Фраумени мутация в гене TP53 является первостепенным событием и может привести к активации РЭ. Регуляторная область 5’UTR элементов LINE1 взаимодействует с белковым продуктом гена RB1 , приводя к сайленсингу данного РЭ [57]. Белковые продукты ГСО BRCA1 [56] и ATM [55] подавляют экспрессию LINE1.
Можно предположить, что регуляторное влияние ГСО на ретроэлементы обусловлено их филогенетической взаимосвязью. Этим же объясняется и наличие горячих точек инсерционного мутагенеза в ГСО в связи с наличием комплементарных последовательностей. Например, в эволюции приматов за последние 40 млн лет произошло 8 крупных комплексных перестроек вокруг гена BRCA1 в локусе 17q21. Данные изменения были обусловлены массивными экспансиями Alu в геномах приматов [34]. Горячая точка мутагенеза для инсерций Alu выявлена в экзоне 5 ГСО PTEN при синдроме Коудена, в гене STK11 – при синдроме Пейтца– Йегерса [60]. Причиной 20–30 % случаев синдрома
Гиппеля–Линдау [53] и 10–20 % синдрома Линча [54] являются крупные делеции, обусловленные гомологичными рекомбинациями между Alu. При туберозном склерозе также описаны герминальные делеции онкосупрессорного гена TSC2 , связанные с гомологичной рекомбинацией, обусловленной Alu-элементами, расположенными в интронах гена [52]. Для семейной ретинобластомы описаны унаследованные мутации вследствие инсерцией LINE1 в интрон 14 ГСО RB1 . В результате был сформирован неканонический акцепторный сайт сплайсинга с образованием патологического мРНК продукта и дефектного белка [60]. Поскольку герминальные мутации в ГСО могут служить причиной ХТ [10], описанная выше взаимосвязь РЭ с протоонкогенами и ГСО свидетельствует о механизме взаимного потенцирования в канцерогенезе. Герминальная инактивация одного аллеля ГСО ведет к усиленной экспрессии РЭ [55–58], которые инсертируют в различные ГСО, содержащих горячие точки инсерционного мутагенеза [50–53]. Помимо устранения сайленсинга РЭ вследствие утраты контроля со стороны белков супрессоров опухолей, активация РЭ происходит под влиянием других факторов. К ним относится старение, ассоциированное с развитием ЗНО и характеризующееся прогрессирующим повышением экспрессии РЭ [61]. Также при различных ЗНО происходят глобальные эпигенетические изменения с гипометилированием, что также стимулирует РЭ [46, 47]. Кроме того, РЭ являются высокочувствительными структурами генома, активируемыми на стрессовые воздействия [20].
Заключение
ХТ, ХА и ХП являются одними из драйверных событий опухолевой эволюции. В отличие от традиционных взглядов на последовательное накопление мутаций, способствующих развитию ЗНО, крупномасштабные структурные изменения при хромоанагенезе способствуют прерывистой эволюции опухоли. Причиной данных явлений, согласно описанным в статье свидетельствам, служит активация РЭ. Действительно, РЭ обладают всеми свойствами, необходимыми для хромоанагенеза (рис. 2). Доказана роль активации ретроэлементов (под влиянием эпигенетических и средовых факторов, старения и инактивации ГСО) в качестве драйверов геномной нестабильности при опухолевом прогрессировании, способность РЭ вызывать крупные делеции ДНК и хромосомные перестройки, роль РЭ в развитии КХП, НГСК, MMBIR, которые являются ключевыми причинами хромоанагенеза. В связи с этим перспективным направлением в изучении механизмов ХТ, ХА и ХП может стать определение роли РЭ в данных процессах, поскольку возможно таргетное воздействие на активность транспозонов с помощью некодирующих РНК. Данное направление иссле-

АКТИВИРОВАННЫЕ РЕТРОЭЛЕМЕНТЫ/ACTIVATED RETROELEMENTS

мРНК mRNA
образование двуцепочечных разрывов ДНК formation of double-stranded DNA breaks
образование мостиков дистальных фрагментов ДНК formation of bridges of distal DNA fragments
Протоонкогены c ретроэлементами в качестве альтернативного промотора Proto-oncogenes with retroelements as an alternative promoter
усиленная экспрессия онкогена enhanced oncogene expression
Протоонкогены с ретроэлементами в области интрона Proto-oncogenes with retroelements in the intron region
экспрессия химерного онкогена chimeric oncogene expression
белки с онкогенными свойствами proteins with oncogenic properties
функционирование в качестве молекулярных «пластырей» functioning as molecular "band-aids"
инсерции в различные области генома insertions into various genome regions
КОМПЛЕКСНЫЕ ХРОМОСОМНЫЕ ПЕРЕСТРОЙКИ COMPLEX CHROMOSOMAL REARRANGEMENTS
АКТИВАЦИЯ ПРОЛИФЕРАЦИИ КЛЕТОК ACTIVATION OF CELL PROLIFERATION
КАНЦЕРОГЕНЕЗ CARCINOGENESIS
Рис. 2. Потенциальные механизмы участия ретроэлементов в комплексных хромосомных перестройках.
Примечание: рисунок выполнен автором
Fig. 2. Potential mechanisms of the influence of retroelements on complex chromosomal rearrangements. Note: created by the author дований злокачественных новообразований может стать основой для обнаружения новых методов эффективной противоопухолевой терапии, по- скольку позволит предотвратить прогрессирование развития ЗНО вследствие клональной эволюции, вызванной активацией РЭ.
Список литературы Участие ретроэлементов в хромоанагенезе при развитии злокачественных новообразований
- Zhang C.Z., Spector A., Cornils H., Francis J.M., Jackson E.K., Liu S., Meyerson M. Chromothripsis from DNA damage in micronuclei. Nature. 2015; 522(7555): 179-84. https://doi.org/10.1038/nature14493.
- Stephens P.J., Greenman C.D., Fu B., Yang F., Bignell G.R., Mudie L.J., Pleasance E.D., Lau K.W., Beare D., Stebbings L.A., McLaren S., Lin M.L., McBride D.J., Varela I., Nik-Zainal S., Leroy C., Jia M., Menzies A., Butler A.P., Teague J.W., Quail M.A., Burton J., Swerdlow H., Carter N.P., Morsberger L.A., Iacobuzio-Donahue C., Follows G.A., Green A.R., Flanagan A.M., Stratton M.R., Futreal P.A., Campbell P.J. Massive genomic rearrangement acquired in a single catastrophic event during cancer development. Cell. 2011; 144(1): 27-40. https://doi.org/10.1016/j.cell.2010.11.055.
- Marcozzi A., Pellestor F., Kloosterman W.P. The Genomic Characteristics and Origin of Chromothripsis. Methods Mol Biol. 2018; 1769: 3-19. https://doi.org/10.1007/978-1-4939-7780-2_1.
- Nazaryan-Petersen L., Bjerregaard V.A., Nielsen F.C., Tommerup N., Tümer Z. Chromothripsis and DNA Repair Disorders. J Clin Med. 2020; 9(3). https://doi.org/10.3390/jcm9030613.
- Holland A.J., Cleveland D.W. Chromanagenesis and cancer: mechanisms and consequences of localized, complex chromosomal rearrangements. Nat Med. 2012; 18(11): 1630-8. https://doi.org/10.1038/nm.2988.
- Hastings P.J., Ira G., Lupski J.R. A microhomology-mediated breakinduced replication model for the origin of human copy number variation. PLoS Genet. 2009; 5(1). https://doi.org/10.1371/journal.pgen.1000327.
- Ly P., Cleveland D.W. Rebuilding Chromosomes After Catastrophe: Emerging Mechanisms of Chromothripsis. Trends Cell Biol. 2017; 27(12): 917-30. https://doi.org/10.1016/j.tcb.2017.08.005.
- Slamova Z., Nazaryan-Petersen L., Mehrjouy M.M., Drabova J., Hancarova M., Marikova T., Novotna D., Vlckova M., Vlckova Z., Bak M., Zemanova Z., Tommerup N., Sedlacek Z. Very short DNA segment can be detected and handled by the repair machinery during germline chromothriptic chromosome reassembly. Hum Mutat. 2018; 39(5): 709-16. https://doi.org/10.1002/humu.23408.
- Nazaryan L., Stefanou E.G., Hansen C., Kosyakova N., Bak M., Sharkey F.H., Mantziou T., Papanastasiou A.D., Velissariou V., Liehr T., Syrrou M., Tommerup N. The strength of combined cytogenetic and mate-pair sequencing techniques illustrated by a germline chromothripsis rearrangement involving FOXP2. Eur J Hum Genet. 2014; 22(3): 338-43. https://doi.org/10.1038/ejhg.2013.147.
- Nazaryan-Petersen L., Bertelsen B., Bak M., Jønson L., Tommerup N., Hancks D.C., Tümer Z. Germline Chromothripsis Driven by L1-Mediated Retrotransposition and Alu/Alu Homologous Recombination. Hum Mutat. 2016; 37(4): 385-95. https://doi.org/10.1002/humu.22953.
- Kloosterman W.P., Hoogstraat M., Paling O., Tavakoli-Yaraki M., Renkens I., Vermaat J.S., van Roosmalen M.J., van Lieshout S., Nijman I.J., Roessingh W., van ‘t Slot R., van de Belt J., Guryev V., Koudijs M., Voest E., Cuppen E. Chromothripsis is a common mechanism driving genomic rearrangements in primary and metastatic colorectal cancer. Genome Biol. 2011; 12(10). https://doi.org/10.1186/gb-2011-12-10-r103.
- Cortés-Ciriano I., Lee J.J., Xi R., Jain D., Jung Y.L., Yang L., Gordenin D., Klimczak L.J., Zhang C.Z., Pellman D.S.; PCAWG Structural Variation Working Group; Park PJ; PCAWG Consortium. Comprehensive analysis of chromothripsis in 2,658 human cancers using whole-genome sequencing. Nat Genet. 2020; 52(3): 331-41. https://doi.org/10.1038/s41588-019-0576-7. Erratum in: Nat Genet. 2023; 55(5): 893. https://doi.org/10.1038/s41588- 020-0634-1. Erratum in: Nat Genet. 2023; 55(6): 1076. https://doi.org/10.1038/s41588-023-01315-z.
- Shen M.M. Chromoplexy: a new category of complex rearrangements in the cancer genome. Cancer Cell. 2013; 23(5): 567-9. https://doi.org/10.1016/j.ccr.2013.04.025.
- Baca S.C., Prandi D., Lawrence M.S., Mosquera J.M., Romanel A., Drier Y., Park K., Kitabayashi N., MacDonald T.Y., Ghandi M., van Allen E., Kryukov G.V., Sboner A., Theurillat J.P., Soong T.D., Nickerson E., Auclair D., Tewari A., Beltran H., Onofrio R.C., Boysen G., Guiducci C., Barbieri C.E., Cibulskis K., Sivachenko A., Carter S.L., Saksena G., Voet D., Ramos A.H., Winckler W., Cipicchio M., Ardlie K., Kantoff P.W., Ber ger M.F., Gabriel S.B., Golub T.R., Meyerson M., Lander E.S., Elemento O., Getz G., Demichelis F., Rubin M.A., Garraway L.A. Punctuated evolution of prostate cancer genomes. Cell. 2013; 153(3): 666-77. https://doi.org/10.1016/j. cell.2013.03.021.
- Rausch T., Jones D.T., Zapatka M., Stütz A.M., Zichner T., Weischenfeldt J., Jäger N., Remke M., Shih D., Northcott P.A., Pfaff E., Tica J., Wang Q., Massimi L., Witt H., Bender S., Pleier S., Cin H., Hawkins C., Beck C., von Deimling A., Hans V., Brors B., Eils R., Scheurlen W., Blake J., Benes V., Kulozik A.E., Witt O., Martin D., Zhang C., Porat R., Merino D.M., Wasserman J., Jabado N., Fontebasso A., Bullinger L., Rücker F.G., Döhner K., Döhner H., Koster J., Molenaar J.J., Versteeg R., Kool M., Tabori U., Malkin D., Korshunov A., Taylor M.D., Lichter P., Pfster S.M., Korbel J.O. Genome sequencing of pediatric medulloblastoma links catastrophic DNA rearrangements with TP53 mutations. Cell. 2012; 148(1-2): 59-71. https://doi.org/10.1016/j.cell.2011.12.013.
- Wang K., Wang Y., Collins C.C. Chromoplexy: a new paradigm in genome remodeling and evolution. Asian J Androl. 2013; 15(6): 711-2. https://doi.org/10.1038/aja.2013.109.
- Poot M. Genes, Proteins, and Biological Pathways Preventing Chromothripsis. Methods Mol Biol. 2018; 1769: 231-51. https://doi.org/10.1007/978-1-4939-7780-2_15.
- Pagter M.S., Roostmalen M.J., Baas A.F., Renkens I., Duran K.J., van Binsbergen E., Yaraki M.T., Hochstenbach R., Veken L.T., Cuppen E., Kloosterman W.P. Chromothripsis in healthy individuals afects multiple protein-coding genes and can result in severe congential abnormalities in ofspring. Am J Hum Genet. 2015; 96(4): 651-6. https://doi.org/10.1016/j.ajhg.2015.02.005.
- Bertelsen B., Nazaryan-Petersen L., Sun W., Mehrjouy M.M., Xie G., Chen W., Hjermind L.E., Taschner P.E.M., Tumer Z. A germline chromothripsis event stably segregating in 11 individuals through three generations. Genet Med. 2016; 18(5): 454-500. https://doi.org/10.1038/gim.2015.112.
- Mustafn R.N., Khusnutdionova E.K. The role of transposable elements in the ecological morphogenesis under the infuence of stress. Vavilov Journal of Genetics and Breeding. 2019; 23(4): 380-89. https://doi.org/10.18699/VJ19.506.
- Erwin J.A., Paquola A.C., Singer T., Gallina I., Novotny M., Quayle C., Bedrosian T.A., Alves F.I., Butcher C.R., Herdy J.R., Sarkar A., Lasken R.S., Muotri A.R., Gage F.H. L1-associated genomic regions are deleted in somatic cells of the healthy human brain. Nat Neurosci. 2016; 19(12): 1583-91. https://doi.org/10.1038/nn.4388. Erratum in: Nat Neurosci. 2017; 20(10): 1427. https://doi.org/10.1038/nn1017-1427a. Erratum in: Nat Neurosci. 2018; 21(7): 1016. https://doi.org/10.1038/s41593-018-0131-3.
- Rodriguez-Martin B., Alvarez E.G., Baez-Ortega A., Zamora J., Supek F., Demeulemeester J., Santamarina M., Ju Y.S., Temes J., GarciaSouto D., Detering H., Li Y., Rodriguez-Castro J., Dueso-Barroso A., Bruzos A.L., Dentro S.C., Blanco M.G., Contino G., Ardeljan D., Tojo M., Roberts N.D., Zumalave S., Edwards P.A., Weischenfeldt J., Puiggròs M., Chong Z., Chen K., Lee E.A., Wala J.A., Raine K.M., Butler A., Waszak S.M., Navarro F.C.P., Schumacher S.E., Monlong J., Maura F., Bolli N., Bourque G., Gerstein M., Park P.J., Wedge D.C., Beroukhim R., Torrents D., Korbel J.O., Martincorena I., Fitzgerald R.C., Van Loo P., Kazazian H.H., Burns K.H.; PCAWG Structural Variation Working Group; Campbell P.J., Tubio J.M.C.; PCAWG Consortium. Pan-cancer analysis of whole genomes identifes driver rearrangements promoted by LINE-1 retrotransposition. Nat Genet. 2020; 52(3): 306-19. https://doi.org/10.1038/s41588-019-0562-0. Erratum in: Nat Genet. 2023; 55(6): 1080. https://doi.org/10.1038/s41588-023-01319-9.
- Jang H.S., Shah N.M., Du A.Y., Dailey Z.Z., Pehrsson E.C., Godoy P.M., Zhang D., Li D., Xing X., Kim S., O’Donnell D., Gordon J.I., Wang T. Transposable elements drive widespread expression of oncogenes in human cancers. Nat Genet. 2019; 51(4): 611-7. https://doi.org/10.1038/s41588- 019-0373-3. Erratum in: Nat Genet. 2019; 51(5): 920. https://doi.org/10.1038/s41588-019-0416-9.
- Chen T., Meng Z., Gan Y., Wang X., Xu F., Gu Y., Xu X., Tang J., Zhou H., Zhang X., Gan X., Van Ness C., Xu G., Huang L., Zhang X., Fang Y., Wu J., Zheng S., Jin J., Huang W., Xu R. The viral oncogene Np9 acts as a critical molecular switch for co-activating β-catenin, ERK, Akt and Notch1 and promoting the growth of human leukemia stem/progenitor cells. Leukemia. 2013; 27(7): 1469-78. https://doi.org/10.1038/leu.2013.8.
- Babaian A., Romanish M.T., Gagnier L., Kuo L.Y., Karimi M.M., Steidl C., Mager D.L. Onco-exaptation of an endogenous retroviral LTR drives IRF5 expression in Hodgkin lymphoma. Oncogene. 2016; 35(19): 2542-46. https://doi.org/10.1038/onc.2015.308.
- Hur K., Cejas P., Feliu J., Moreno-Rubio J., Burgos E., Boland C.R., Goel A. Hypomethylation of long interspersed nuclear element-1 (LINE-1) leads to activation of proto-oncogenes in human colorectal cancer metastasis. Gut. 2014; 63(4): 635-46. https://doi.org/10.1136/gutjnl-2012-304219.
- Lamprecht B., Walter K., Kreher S., Kumar R., Hummel M., Lenze D., Köchert K., Bouhlel M.A., Richter J., Soler E., Stadhouders R., Jöhrens K., Wurster K.D., Callen D.F., Harte M.F., Giefng M., Barlow R., Stein H., Anagnostopoulos I., Janz M., Cockerill P.N., Siebert R., Dörken B., Bonifer C., Mathas S. Derepression of an endogenous long terminal repeat activates the CSF1R proto-oncogene in human lymphoma. Nat Med. 2010; 16(5): 571-9. https://doi.org/10.1038/nm.2129.
- Cervantes-Ayalc A., Esparza-Garrido R.R., Velazquez-Floes M.A. Long Interspersed Nuclear Elements 1 (LINE1): The chimeric transcript L1-MET and its involvement in cancer. Cancer Genet. 2020; 241: 1-11. https://doi.org/10.1016/j.cancergen.2019.11.004.
- Lock F.E., Rebollo R., Miceli-Royer K., Gagnier L., Kuah S., Babaian A., Sistiaga-Poveda M., Lai C.B., Nemirovsky O., Serrano I., Steidl C., Karimi M.M., Mager D.L. Distinct isoform of FABP7 revealed by screening for retroelement-activated genes in difuse large B-cell lymphoma. Proc Natl Acad Sci USA. 2014; 111(34): 3534-43. https://doi.org/10.1073/pnas.1405507111. Erratum in: Proc Natl Acad Sci USA. 2015; 112(33): 4630. https://doi.org/10.1073/pnas.1512789112.
- Scarfò I., Pellegrino E., Mereu E., Kwee I., Agnelli L., Bergaggio E., Garaffo G., Vitale N., Caputo M., Machiorlatti R., Circosta P., Abate F., Barreca A., Novero D., Mathew S., Rinaldi A., Tiacci E., Serra S., Deaglio S., Neri A., Falini B., Rabadan R., Bertoni F., Inghirami G., Piva R.; European T-Cell Lymphoma Study Group. Identifcation of a new subclass of ALK-negative ALCL expressing aberrant levels of ERBB4 transcripts. Blood. 2016; 127(2): 221-32. https://doi.org/10.1182/blood-2014-12-614503.
- Wiesner T., Lee W., Obenauf A.C., Ran L., Murali R., Zhang Q.F., Wong E.W., Hu W., Scott S.N., Shah R.H., Landa I., Button J., Lailler N., Sboner A., Gao D., Murphy D.A., Cao Z., Shukla S., Hollmann T.J., Wang L., Borsu L., Merghoub T., Schwartz G.K., Postow M.A., Ariyan C.E., Fagin J.A., Zheng D., Ladanyi M., Busam K.J., Berger M.F., Chen Y., Chi P. Alternative transcription initiation leads to expression of a novel ALK isoform in cancer. Nature. 2015; 526(7573): 453-7. https://doi.org/10.1038/nature15258.
- Fairbanks D.J., Fairbanks A.D., Ogden T.H., Parker G.J., Maughan P.J. NANOGP8: evolution of a human-specifc retro-oncogene. G3 (Bethesda). 2012; 2(11): 1447-57. https://doi.org/10.1534/g3.112.004366.
- Haffner M.C., Aryee M.J., Toubaji A., Esopi D.M., Albadine R., Gurel B., Isaacs W.B., Bova G.S., Liu W., Xu J., Meeker A.K., Netto G., De Marzo A.M., Nelson W.G., Yegnasubramanian S. Androgen-induced TOP2B-mediated double-strand breaks and prostate cancer gene rearrangements. Nat Genet. 2010; 42(8): 668-75. https://doi.org/10.1038/ng.613.
- Symer D.E., Connelly C., Szak S.T., Caputo E.M., Cost G.J., Parmigiani G., Boeke J.D. Human l1 retrotransposition is associated with genetic instability in vivo. Cell. 2002; 110(3): 327-38. https://doi.org/10.1016/s0092-8674(02)00839-5.
- Vogt J., Bengesser K., Claes K.B., Wimmer K., Mautner V.F., van Minkelen R., Legius E., Brems H., Upadhyaya M., Högel J., Lazaro C., Rosenbaum T., Bammert S., Messiaen L., Cooper D.N., Kehrer-Sawatzki H. SVA retrotransposon insertion-associated deletion represents a novel mutational mechanism underlying large genomic copy number changes with non-recurrent breakpoints. Genome Biol. 2014; 15(6). https://doi.org/10.1186/gb-2014-15-6-r80.
- Choi B.O., Kim N.K., Park S.W., Hyun Y.S., Jeon H.J., Hwang J.H., Chung K.W. Inheritance of Charcot-Marie-Tooth disease 1A with rare nonrecurrent genomic rearrangement. Neurogenetics. 2011; 12(1): 51-8. https://doi.org/10.1007/s10048-010-0272-3.
- Ji X., Zhao S. DA and Xiao-two giant and composite LTRretrotransposon-like elements identifed in the human genome. Genomics. 2008; 91(3): 249-58. https://doi.org/10.1016/j.ygeno.2007.10.014.
- Jin H., Selfe J., Whitehouse C., Morris J.R., Solomon E., Roberts R.G. Structural evolution of the BRCA1 genomic region in primates. Genomics. 2004; 84(6): 1071-82. https://doi.org/10.1016/j.ygeno.2004.08.019.
- Ribeiro I.P., Carreira I.M., Esteves L., Caramelo F., Liehr T., Melo J.B. Chromosomal breakpoints in a cohort of head and neck squamous cell carcinoma patients. Genomics. 2020; 112(1): 297-303. https://doi.org/10.1016/j.ygeno.2019.02.009.
- Wang H., Li Y., Truong L.N., Shi L.Z., Hwang P.Y., He J., Do J., Cho M.J., Li H., Negrete A., Shiloach J., Berns M.W., Shen B., Chen L., Wu X. CtIP maintains stability at common fragile sites and inverted repeats by end resection-independent endonuclease activity. Mol Cell. 2014; 54(6): 1012-21. https://doi.org/10.1016/j.molcel.2014.04.012.
- Suzuki J., Yamaguchi K., Kajikawa M., Ichiyanagi K., Adachi N., Koyama H., Takeda S., Okada N. Genetic evidence that the non-homologous end-joining repair pathway is involved in LINE retrotransposition. PLoS Genet. 2009; 5(4). https://doi.org/10.1371/journal.pgen.1000461.
- Lee G., Sherer N.A., Kim N.H., Rajic E., Kaur D., Urriola N., Martini K.M., Xue C., Goldenfeld N., Kuhlman T.E. Testing the retroelement invasion hypothesis for the emergence of the ancestral eukaryotic cell. Proc Natl Acad Sci U S A. 2018; 115(49): 12465-70. https://doi.org/10.1073/pnas.1807709115.
- Mustafin R.N., Khusnutdinova E.K. Rol' retroelementov v razvitii nasledstvennykh opukholevykh sindromov. Uspekhi molekulyarnoi onkologii. 2021; 8(4): 42-52. https://doi.org/10.17650/2313-805X-2021-8-4-42-52.
- Hancks D.C. A Role for Retrotransposons in Chromothripsis. Methods Mol Biol. 2018; 1769: 169-81. https://doi.org/10.1007/978-1-4939-7780-2_11.
- Mustafn R.N. The role of transposable elements in the diferentiation of stem cells. Mol. Genet. Microbiol. Virol. 2019; 34: 67-74. https://doi.org/10.17116/molgen20193702151.
- Baba Y., Huttenhower C., Nosho K., Tanaka N., Shima K., Hazra A., Schernhammer E.S., Hunter D.J., Giovannucci E.L., Fuchs C.S., Ogino S. Epigenomic diversity of colorectal cancer indicated by LINE-1 methylation in a database of 869 tumors. Mol Cancer. 2010; 9: 125. https://doi.org/10.1186/1476-4598-9-125.
- Barchitta M., Quattrocchi A., Maugeri A., Vinciguerra M., Agodi A. LINE-1 hypomethylation in blood and tissue samples as an epigenetic marker for cancer risk: a systematic review and meta-analysis. PLoS One. 2014; 9(10). https://doi.org/10.1371/journal.pone.0109478.
- Lieber M.R. The mechanism of double-strand DNA break repair by the nonhomologous DNA end-joining pathway. Annu Rev Biochem. 2010; 79: 181-211. https://doi.org/10.1146/annurev.biochem.052308.093131.
- Gu S., Yuan B., Campbell I.M., Beck C.R., Carvalho C.M., Nagamani S.C.S., Erez A., Patel A., Bacino C.A., Shaw C.A., Stankiewicz P., Cheung S.W., Bi W., Lupski J.R. Alu-mediated diverse and complex pathogenic copy-number variants within human chromosome 17 at p13.3. Hum Mol Genet. 2015; 24(14): 4061-77. https://doi.org/10.1093/hmg/ddv146.
- Borun P., de Rosa M., Nedoszytko B., Walkowiak J., Plawski A. Specifc Alu elements involved in a signifcant percentage of copy number variations of the STK11 gene in patients with Peutz-Jeghers syndrome. Fam Cancer. 2015; 14(3): 455-61. https://doi.org/10.1007/s10689-015-9800-5.
- Crivelli L., Bubien V., Jones N., Chiron J., Bonnet F., BaroukSimonet E., Couzigou P., Sevenet N., Caux F., Longy M. Insertion of Alu elements at a PTEN hotspot in Cowden syndrome. Eur J Hum Genet. 2017; 25(9): 1087-91. https://doi.org/10.1038/ejhg.2017.81.
- Dabora S.L., Nieto A.A., Franz D., Jozwiak S., Ouweland A.V.D., Kwiatkowski D.J. Characterisation of six large deletions in TSC2 identifed using long range PCR suggests diverse mechanisms including Alu mediated recombination. J Med Genet. 2000; 37(11): 877-83. https://doi.org/10.1136/jmg.37.11.877.
- Franke G., Bausch B., Hoffmann M.M., Cybulla M., Wilhelm C., Kohlhase J., Scherer G., Neumann H.P.H. Alu-Alu recombination underlies the vast majority of large VHL germline deletions: Molecular characterization and genotype-phenotype correlation in VHL patients. Hum Mutat. 2009; 30(5): 776-86. https://doi.org/10.1002/humu.20948.
- Hitchins M.P., Burn J. Alu in Lynch syndrome: a danger SINE. Cancer Prev Res (Phila). 2011; 4(10): 1527-30. https://doi.org/10.1158/1940-6207. CAPR-11-0417.
- Coufal N.G., Garcia-Perez J.L., Peng G.E., Marchetto M.C.N., Muotri A.R., Mu Y., Carson C.T., Macia A., Moran J.V., Gage F.H. Ataxia telangiectasia mutated (ATM) modulates long interspersed element-1 (L1) retrotransposition in human neural stem cells. Proc Natl Acad Sci USA. 2011; 108(51): 20382-87. https://doi.org/10.1073/pnas.1100273108.
- Mita P., Sun X., Fenyo D., Kahler D.J., Li D., Agmon N., Wudzinska A., Keegan S., Bader J.S., Yun C., Boeke J.D. BRCA1 and S phase DNA repair pathways restrict LINE-1 retrotransposition in human cells. Nat Struct Mol Biol. 2020; 27(2): 179-91. https://doi.org/10.1038/s41594-020-0374-z.
- Montoya-Durango D.E., Ramos K.A., Bojang P., Ruiz L., Ramos I.N., Ramos K.S. LINE-1 silencing by retinoblastoma proteins is efected through the nucleosomal and remodeling deacetylase multiprotein complex. BMC Cancer. 2016; 16: 38. https://doi.org/10.1186/s12885-016-2068-9.
- Tiwari B., Jones A.E., Caillet C.J., Das S., Royer S.K., Abrams J.M. P53 directly repress human LINE1 transposons. Genes Dev. 2020; 34(21-22): 1439-51. https://doi.org/10.1101/gad.343186.120.
- Ramos K.S., Montoya-Durango D.E., Teneng I., Nanez A., Stribinskis V. Epigenetic control of embryonic renal cell diferentiation by L1 retrotransposon. Birth Defects Res A Clin Mol Teratol. 2011; 91(8): 693-702. https://doi.org/10.1002/bdra.20786.
- Mustafin R.N. Vliyanie retroelementov na onkogeny i onkosupressory v kantserogeneze. Sovremennaya onkologiya. 2021; 23(4): 666-73. [Mustafn R.N. Infuence of retroelements on oncogenes and tumor suppressors in carcinogenesis: A review. Journal of Modern Oncology. 2021; 23(4): 666-73. (in Russian)]. https://doi.org/10.26442/18151434.2021.4.201199.
- Gorbunova V., Seluanov A., Mita P., McKerrow W., Fenyö D., Boeke J.D., Linker S.B., Gage F.H., Kreiling J.A., Petrashen A.P., Woodham T.A., Taylor J.R., Helfand S.L., Sedivy J.M. The role of retrotransposable elements in ageing and age-associated diseases. Nature. 2021; 596(7870): 43-53. https://doi.org/10.1038/s41586-021-03542-y.


