Учет взаимодействий между атомами Лиганда в задаче Докинга с помощью потенциала усредненных энергий
Автор: Лизунов А.Ю., Зайцева Н.И., Зосимов В.В.
Журнал: Труды Московского физико-технического института @trudy-mipt
Рубрика: Нанофизика и нанотехнологии
Статья в выпуске: 1 (21) т.6, 2014 года.
Бесплатный доступ
Предлагается универсальный алгоритм учета нековалентных взаимодействий между атомами лиганда при решении задачи докинга. Предложенный алгоритм может быть реализован с любой эмпирической функцией оценки межатомного взаимодействия. Алгоритм реализован в программе Алгокомб на основе функции оценки межатомного взаимодействия Тарасова-Мурышева. На примере стандартного теста Рогнана показано существенное улучшение качества решения задачи докинга для гибких лигандов при учете внутренних взаимодействий лиганда с помощью предложенного алгоритма.
Докинг, внутренние взаимодействия лиганда, оценочная функция, оценочная функция на основе усредненных энергий, нековалентные взаимодействия, гибкие лиганды
Короткий адрес: https://sciup.org/142185978
IDR: 142185978 | УДК: 577.32
Текст научной статьи Учет взаимодействий между атомами Лиганда в задаче Докинга с помощью потенциала усредненных энергий
В современном процессе разработки лекарств широко используется компьютерное моделирование (см., например, [3]). В частности, если известна, пространственная структура, белка, модуляция работы которого вызывает требуемый терапевтический эффект, широко применяется компьютерное моделирование взаимодействия белка-мишени и лиганда. — малой органической молекулы, потенциального лекарства. Моделирование позволяет проводить поиск лигандов, образующих наиболее прочный комплекс с заданным белком, то есть являющихся более эффективными лекарствами. Так, в задаче виртуального скрининга требуется на. основе расчетов отсортировать набор лигандов по прочности образования комплекса. с заданным белком. Для успешного решения задачи виртуального скрининга, необходимо эффективно решать задачу докинга — задачу поиска взаимного пространственного расположения лиганда, и белка, обеспечивающего наиболее прочную связь между ними. Большая часть современных программ при решении задачи докинга считает пространственную структуру белка жесткой, то есть считается, что атомы белка имеют фиксированные координаты, а. гибкость лиганда, достигается за. счет изменения торсионных углов в связях, допускающих вращение [4], [5]. Такой подход к докингу используется и в данной работе.
Физической величиной, точно выражающей «прочность» комплекса, белок-лиганд, является изменение свободной энергии системы белок-лиганд-растворитель при образовании комплекса, белка, с лигандом, однако с целью упрощения моделирования для оценки прочности комплекса, белок-лиганд часто используют оценку энергии образования комплекса: чем ниже энергия, тем прочнее комплекс. При этом энтропийная составляющая свободной энергии либо вообще не учитывается, либо учитывается с помощью грубых оценок, либо учитывается косвенно.
Точный расчет энергии образования комплекса, белок-лиганд должен включать в себя расчеты энергии взаимодействия лиганда, с белком, изменения внутренней энергии белка, изменения внутренней энергии лиганда, а. также изменения энергии взаимодействия с окружающим растворителем, причем все эти энергии должны вычисляться средствами квантовой механики. Проведение точного расчета, энергии комплекса, технически невозможно из-за. сложности системы. На практике программы докинга, вычисляют некоторую функцию, оценивающую взаимодействие лиганда, и белка, в заданной пространственной конфигурации, эту оценочную функцию мы будем сокращенно обозначать ОФ. Отметим, что для эффективного решения задач докинга и виртуального скрининга ОФ не обязательно должна совпадать с истинной энергией взаимодействия, достаточно того, чтобы ОФ и истинное значение энергии являлись согласованными по возрастанию функциями, то есть конфигурации белок-лиганд с большей истинной энергией взаимодействия должно сопоставляться большее значение ОФ. Именно этого свойства пытаются добиться от ОФ.
По способу вычисления оценочные функции принято делить на три класса: ОФ на основе силовых полей молекулярной механики, эмпирические ОФ и ОФ на основе усредненных энергий. Каждый из типов ОФ обладает своими достоинствами и недостатками, про первые два типа ОФ можно прочитать, например, в [3], [4], [5]. Разрабатываемая нами ОФ принадлежит к типу ОФ на основе усредненных энергий, на этом типе мы остановимся подробнее.
Оценочные функции на основе усредненных энергий (их также называют статистические ОФ или ОФ на основе знаний) конструируются на основе эмпирических усредненных потенциалов межатомного взаимодействия (сокращенно - УПМВ) между атомами различных типов, вычисляемых на основе частот встречаемости атомов на заданном расстоянии в экспериментальных данных. Впервые идея получения оценок энергии на основе частот встречаемости пар атомов на различных расстояниях появилась в работах [23], [24], [25] и использовалась для решения задачи сворачивания белка. В простейшем варианте УПМВ вычисляются исходя из статистики расстояний между атомами различных типов в экспериментальных данных (кристаллических структурах), предполагая, что плотность распределения атомов связана с энергией соотношением Максвелла-Больцмана, а энергия взаимодействия пары атомов заданных типов зависит только от расстояния между ними: U ij (т) = -кТ 1п(шу (г)/4тгг2), г де д у — типы атомов в лиганде и в белке, иду (т) — плотность распределения пар атомов белка и лиганда в зависимости от расстояния, U ij (т) — потенциал взаимодействия пары атомов в зависимости от расстояния. Итоговая ОФ в простейшем случае вычисляется как сумма УПМВ по всем парам атомов из лиганда и белка:
ОФ = £ ^ і (рМ і ) ( г рі ). (1)
l E lig,p E prot
Главным достоинством ОФ на основе усредненных энергий является неявный учет всех типов взаимодействий, влияющих на образование комплекса белок-лиганд, включая ван-дер-ваальсовы и гидрофобные взаимодействия, образование водородных связей и другие, не выделяемые явно, типы взаимодействий. Неявно учитывается взаимодействие с растворителем, а по утверждению некоторых авторов, ОФ на основе усредненных потенциалов неявным образом учитывает и энтропийную составляющую свободной энергии [16].
Недостатки ОФ на основе усредненных энергий можно разделить на два типа: принципиальные недостатки и недостатки потенциалов межатомного взаимодействия. Принципиальные недостатки - это в первую очередь заложенное в саму конструкцию УПМВ усреднение энергии по всем параметрам, определяющим энергию взаимодействия между атомами, за исключением расстояния. Например, пренебрежение пространственной ориентацией электронных орбиталей атомов приводит к пренебрежению направленностью водородных связей, поэтому пара атомов донора и акцептора водородной связи получит высокую оценку энергии взаимодействия, если находится на типичном расстоянии водородной связи, даже если углы между ковалентными связями этих атомов не позволяют образовать водородную связь при заданном взаимном расположении молекул. Также к принципиальным недостаткам простейших ОФ на основе усредненных энергий относится отсутствие учета внутренней энергии лиганда.
Недостатки УПМВ возникают из-за несовершенства используемого метода расчета УПМВ, например, из-за несовершенства типизации атомов или из-за того, что истинная зависимость плотности распределения пар атомов от энергии взаимодействия между ними в экспериментальных данных не точно соответствует распределению Максвелла. В качестве примера фактора, искажающего распределение Максвелла, можно привести наличие ковалентных связей между атомами нековалентно взаимодействующих молекул. Если пара атомов не участвует во взаимодействии, но эти атомы ковалентно связаны с участниками сильного нековалентного взаимодействия (например, водородной связи), то эти два атома могут пространственно сближаться независимо от энергии взаимодействия между ними, а за счет энергии взаимодействия их соседей. Таким образом, наличие ковалентных связей может приводить к искусственным максимумам в плотности распределения, не связанным с энергетической выгодностью расположения данных двух атомов.
Для исправления недостатков разрабатываются различные улучшенные ОФ на основе усредненных энергий, например, для улучшения УПМВ может использоваться дополнительная итеративная процедура оптимизации параметров на основе экспериментальных данных [2], [12].
-
2. Учет нековалентных взаимодействий между атомами лиганда и взаимодействия лиганда с растворителем в известных оценочных функциях на основе усредненных энергий
Расчет усредненного потенциала межатомных взаимодействий основан на усреднении энергии образования комплекса по всем параметрам, за исключением расстояний между атомами лиганда и белка. С одной стороны, благодаря этому ОФ на основе усредненных энергий неявно учитывает все взаимодействия, в том числе и взаимодействие системы белок-лиганд с растворителем и изменение внутренней энергии лиганда. При конструировании некоторых ОФ авторы ограничиваются этим неявным учетом взаимодействий лиганд-лиганд и лиганд-растворитель, так, ОФ РМҒ [16], TM-Score [2], PLASS [19] и М-Score [21] не содержат никаких дополнительных слагаемых, кроме суммы усредненных межатомных взаимодействий между атомами лиганда и белка, и вычисляются по простейшей формуле (1). С другой стороны, из-за усреднения энергии образования комплекса белок-лиганд по всем параметрам, кроме расстояний между атомами лиганда и белка, теряется информация о зависимости энергии комплекса от других параметров. Поэтому в некоторых ОФ на основе усредненных энергий для уточнения значения ОФ к выражению, вычисляемому по формуле (1), добавляются дополнительные слагаемые для более точного учета взаимодействия с растворителем и учета изменения внутренней энергии лиганда. Так, в ОФ PLP [13] и ASP [20] используют дополнительное слагаемое для учета торсионной энергии вращательных связей, а в ОФ DrugScore [17], Bleep [18] и ITScore [И] используют слагаемое для явного учета влияния растворителя.
В таблице 1 приведено краткое описание известных ОФ на основе усредненных энергий. Рассмотрим их с точки зрения явного учета внутренней энергии лиганда и взаимодействия лиганда с растворителем.
Gehlhaar и коллеги разработали модель межмолекулярного распознавания для докинга гибких лигандов в белковый сайт связывания — ОФ PLP [13]. Разработанная ОФ учитывает меж- и внутримолекулярные взаимодействия. Межмолекулярные взаимодействия оцениваются с помощью УПМВ, внутримолекулярные — с помощью слагаемых для оценки торсионной энергии для sp3-sp3 и sp2-sp3 связей и дополнительных штрафов в 10 000 единиц оценки за самопересечения лиганда (в случае, если расстояние между парой несвязанных атомов лиганда оказывается меньше 2.35 А).
В ОФ ASP [20] используются дополнительные слагаемые оценки внутренней энергии лиганда и штрафы за самопересечение лиганда, взятые из эмпирической ОФ ChemScore [22]. Дополнительные слагаемые, как и в ОФ PLP, учитывают только изменение торсионной энергии вращательных связей и самопересечения лиганда.
Таблица!
Известные ОФ на основе усредненных энергий
|
Название ОФ |
Формула для вычисления ОФ |
Краткое описание особенностей |
Авторы, год |
|
PLP |
Е ир1(гр1)+ /,р + Е А (1- rot - cos(ny - До)) + +Nda.s h * Penalty |
|
Gehlhaar, 1995 [13] |
|
SMoG1996 SMoG2001 |
Е U p t ([Г р 1 ]) + /,р +Nr ot 1 * F- rot 1 + +Nr ot 2 * F -ot 2 |
— нет полноценного учета зависимости УПМВ от расстояния, различается всего два состояния: расстояние менее 4.5 А и менее 3.5 А; — учет энтропийных потерь при замораживании вращательных связей лиганда, отдельно учитываются связи sp3-sp3 и sp2-sp3; |
DeWitte, 1996 [14] Ischenko, 2002 [15] |
|
РМҒ |
Е U p/ (Г р/ ) /,P |
— впервые вводится корректирующий множитель для объема сферического слоя, влияющий на оценку плотности атомов в зависимости от расстояния при вычислении УПМВ; |
Muegge, Martin, 1999 [16] |
|
DrugScore |
(1 — 7) Е U p/ (r p tH +7 Е ДW / (SAS, / SASo) + +7 Е ^W p (SAS, p SASo) |
— вводится дополнительное слагаемое, оценивающее энергию взаимодействия с растворителем. Для описания контакта с растворителем для каждого атома вычисляется параметр SAS (solvent-accessible surface). — явно указывается, что потенциал не учитывает внутренние взаимодействия лиганда; |
Gohlke, Hendlich, Klebe, 2000 [17] |
|
Bleep-1 Bleep-2 |
Е Up/ (T p t ) + /,p +д Е U /w (r /w )+ /,w +д Е U pw (г pw ) p,w |
— для учета взаимодействия с растворителем искусственно добавляются молекулы воды, как на стадии вычисления УПМВ, так и при вычислении ОФ; |
Mitchell, 1999 [18] |
|
TM-Score |
EUp/ (г р/ ) /,р |
— при вычислении УПМВ используется комбинированная процедура статистического анализа расстояний между атомами и эмпирического подбора коэффициентов; |
Muryshev, 2003 [2] |
|
PLASS |
Eupl (гр/) /,р |
— при вычислении УПМВ используется оригинальный способ корректировки объема, доступного для взаимодействия; |
Ozrin, 2004 [19] |
|
ASP |
£ Upl (r pl )+AE l t g + l,p +^E clash |
— к сумме УПМВ добавлены слагаемые для оценки изменения внутренней энергии лиганда и пересечений лиганда с белком. Дополнительные слагаемые взяты из эмпирической ОФ ChemScore [22], изменение внутренней энергии учитывает только энергию торсионных связей и самопересечения лиганда; |
Mooij, 2005 [20] |
|
M-score |
£ U pl (r pi ) i,p |
— при вычислении УПМВ атомы белка «размазываются» в окрестности их положения, известного из рентгеноструктурного анализа; |
Yang, 2006 [21] |
|
Algo_score |
£№ Wx l X £ U pl (r pl )] + p +S clash + S tars |
— вводится понятие обусловленности для атома лиганда в заданной точке пространства. Обусловленность выражает правдоподобие обнаружения атома в данном месте по отношению к белку. При подсчете ОФ взаимодействие каждого атома лиганда с атомами белка умножается на обусловленность. — используются эмпирические слагаемые для учета изменения торсионной энергии вращательных связей лиганда и штраф за самопересечение лиганда; |
Ramensky, 2007 [1] |
|
ITScore ITScore/SE |
£ U pl (r pl) + ++£ v^SAS ) + +aAS conf + 3 ^S aib |
|
Huang, 2006 [10] 2010 [11] |
При вычислении ОФ В1еер-2 [18] к рассматриваемой конформации комплекса белок-лиганд искусственно добавляются молекулы воды и при суммировании УПМВ учитываются взаимодействия лиганда и белка с молекулами водві. При вычислении УПМВ для ОФ Bleep использовались данные рентгеноструктурного анализа для большого разнообразия комплексов белок-лиганд с учетом гомологии [18], при этом к комплексам белок-лиганд также искусственно добавлялись молекулы воды. Разработаны две версии Bleep, в более ранней версии Bleep-1 молекулы воды явно не учитывались.
В ОФ DrugScore [17] к сумме попарных межатомных взаимодействий белка и лиганда добавляется слагаемое, учитывающее взаимодействие с растворителем. Для учета взаимодействия с растворителем вводится дополнительный одноатомный потенциал, зависящий от параметра SAS — solvent available surface - площади поверхности контакта атома с растворителем.
В улучшенной версии ITScore [10] — ITScore/SE [11], так же, как и в DrugScore, с помощью параметра SAS учитывается взаимодействия лиганда и белка с растворителем. Кроме того, добавляются два дополнительных слагаемых для учета энтропийного вклада в свободную энергию.
3. Новый метод оценки внутренних нековалентных взаимодействий между атомами лиганда
В данной работе предложен и реализован способ улучшения ОФ на основе усредненных энергий за счет учета внутренних взаимодействий лиганда и улучшенной оценки взаимодействия лиганда с растворителем. Основная идея нового метода состоит в использовании тех же УПМВ, с помощью которых оценивается энергия взаимодействия между атомами лиганда и белка, для оценки энергии нековалентных взаимодействий между атомами ли ганда и неявной оценки взаимодействия атомов лиганда с растворителем. Предложенный способ может быть применен с любой ОФ на основе усредненных энергий.
Новый метод был реализован на примере УПМВ Тарасова-Мурышева [2] и ОФ Algo_score, используемой в программе Алгокомб [1]. Исходно Algo_score вычислялся по формуле
So id = ^СЕ (І г ,р з ) + Sclash + S tars i
г , 3
здесь Е(Іг,р з ) — УПМВ Тарасова-Мурышева для пары атомов из лиганда и из белка, С г — коэффициент обусловленности для данного положения атома лиганда или 1, если Е (І г ,Р з ) > 0, то есть если атому лиганда энергетически невыгодно находиться в данном 3
месте. Коэффициент обусловленности принимает значения от 0 до 1 и показывает, встречается ли атом с тем же типом, что Іг, в похожем белковом окружении среди атомов лигандов из белок-лигандных комплексов базы данных PDB [6], более подробно об ОФ Algo_score и о коэффициенте обусловленности смотрите в [1]. Sc i ash ~ штраф за пересечение молекул, возникающий, если в рассматриваемой позиции лиганда пара атомов оказывается на расстоянии, меньшем суммы их ван-дер-ваальсовых радиусов; Stars - оценка торсионных напряжений в лиганде, вычисляемая в соответствии с потенциалом Amber [7].
Из формулы (2) видно, что внутримолекулярные взаимодействия лиганда в ОФ Algo_score оцениваются только как сумма штрафов за самопересечения лиганда и торсионной энергии вращательных связей. Во многих случаях такой оценки изменения внутренней энергии лиганда оказывается достаточно, например, если лиганд имеет мало вращательных связей или если структура сайта белка-мишени не позволяет атомам лиганда сблизиться и вступить в нековалентное взаимодействие. Однако для более гибких лигандов нековалентные взаимодействия между атомами лиганда, такие как водородные связи и гидрофобные взаимодействия, могут существенно влиять на свободную энергию комплекса. В качестве примера можно привести олигопептиды, активность которых бывает связана со «склонностью» олигопептида образовывать структуру альфа-спирали (см., например, [8]), что в свою очередь обусловлено образованием внутримолекулярных водородных связей.
Измененное значение ОФ Algo_score отличается от исходного добавлением суммы оценок попарных взаимодействий между атомами лиганда и вычисляется по формуле
S new = S old + а^Е (І г ,І3 ) = Vcf. (І г ,р3 )+ S clash + S tars + а^ Е (І г ,І3 ), (3)
г,з г,з г,з здесь Said и Snew — оценки энергии взаимодействия лиганда и белка в заданной пространственной конфигурации, вычисленные с помощью исходной и модифицированной версии ОФ Algo_score соответственно; Е(Іг,Із) — УПМВ Тарасова-Мурышева для пары атомов лиганда Іг, І3. При вычислении последнего слагаемого суммирование ведется по всем атомам лиганда, расстояние между которыми по графу молекулы не менее 5 ковалентных связей, причем хотя бы одна из этих связей является вращательной. Коэффициент учета внутренних взаимодействий альфа вводится для того, чтобы компенсировать умножение того же самого УПМВ Тарасова-Мурышева на коэффициент обусловленности при подсчете взаимодействий между атомами лиганда и белка (2). Коэффициент обусловленности принимает значения от 0 до 1, поэтому в отсутствие коэффициента альфа возникала бы переоценка внутренних взаимодействий лиганда по отношению к взаимодействиям лиганда с белком.
В программе Алгокомб также предусмотрена возможность вычислять другую ОФ (directed mixed score [1]), учитывающую не только положения атомов лиганда, но и ориентацию его ковалентных связей, мы будем называть эту ОФ Algoscoredirected. Вычисление ОФ Algo score directed отличается от вычисления Algo_score (2) дополнительной переоценкой позиции лиганда в зависимости от того, встречаются ли фрагменты рассматриваемого лиганда в заданной пространственной ориентации в похожем белковом окружении среди лигандов из базы данных PDB. Вычисление ОФ Algo score directed требует гораздо большей вычислительной работы, поэтому обычно она используется на заключительной стадии, чтобы пересчитать ОФ лучших позиций, найденных на основе базовой ОФ (2). Использование процедуры пересчета ОФ с учетом ориентации заметно улучшает результаты докинга [1].
4. Влияние учета нековалентных взаимодействий между атомами лиганда на качество докинга. Тест Рогнана
Одним из критериев оценки качества моделирования взаимодействия лиганда с белком является способность решать задачу докинга, то есть способность находить правильное положение гибкого лиганда в активном сайте белка-мишени. В качестве правильного положения, как правило, берется положение лиганда, известное из рентгеноструктурного анализа данного комплекса белок-лиганд. Для выбора оптимальной позиции программа докинга ищет среди всех положений лиганда в сайте белка позицию, на которой достигается минимум функции, оценивающей энергию системы белок-лиганд. Лиганд рассматривается как набор атомов, соединенных ковалентными связями, гибкость лиганда достигается за счет вращения связей с низким барьером торсионной энергии, такие связи мы будем называть вращательными.
Чтобы исследовать, как влияет учет нековалентных взаимодействий между атомами лиганда на решение задачи докинга, проводился докинговый тест Рогнана [9]. Тест Рогнана является одним из общепринятых тестов эффективности программ докинга, в этом тесте рассматриваются 100 комплексов белок-лиганд, пространственная структура которых с высокой точностью расшифрована методом рентгеноструктурного анализа. С помощью тестируемой программы выполняется докинг лигандов в соответствующие им белки, и найденное положение лиганда сравнивается с положением, известным из рентгеноструктурного анализа, — нативным положением. Считается, что для данного комплекса докинг прошел успешно, если среднеквадратичное отклонение положений атомов лиганда в найденной позиции от нативных положений не превосходит 2 А. Итоговая эффективность программы докинга оценивается как доля комплексов, для которых докинг прошел успешно.
Среди 100 комплексов теста Рогнана представлены разнообразные по гибкости лиганды, так как эффект от учета внутренних взаимодействий лиганда ожидался более существенным на гибких лигандах, отдельно рассматривалась группа из 32 гибких лигандов, имеющих 5 или более вращательных связей.
Чтобы оценить влияние изменений, внесенных в оценочную функцию, на решение задачи докинга, был проведен ряд запусков теста Рогнана с различными параметрами. Были проведены две серии запусков теста Рогнана - без пересчета ОФ с учетом ориентации лиганда и с пересчетом. В каждой серии запусков коэффициент учета внутренних взаимодействий лиганда альфа из формулы (3) принимал значения 0, 0.5, 0.75, 1. Параметр альфа задает вес внутримолекулярных взаимодействий лиганда по отношению к взаимодействиям между лигандом и белком, расчет со значением коэффициента альфа, равным нулю, совпадает с расчетом по старой оценочной функции (2), в которой не учитываются взаимодействия между атомами лиганда. Сводные результаты запусков теста Рогнана приведены в таблицах 2 и 3. В таблицах приведено количество комплексов, для которых найденное с помощью программы докинга положение лиганда оказалось неправильным, то есть среднеквадратичное отклонение координат атомов лиганда в найденном положении от о координат атомов лиганда в нативном положении превосходило 2 А.
Т а б л и ц а 2
Результаты теста Рогнана в зависимости от коэффициента учета внутренних взаимодействий лиганда. Процедура пересчета ОФ с учетом ориентации не использовалась
|
Значение коэффициента учета внутренних взаимодействий альфа |
Неправильно найденных позиций (из 100) |
Неправильно найденных позиций среди гибких лигандов (из 32) |
|
0 |
22 |
9 |
|
0.5 |
17 |
2 |
|
0.75 |
15 |
2 |
|
1 |
19 |
4 |
Т а б л и ц а 3
Результаты теста Рогнана в зависимости от коэффициента учета внутренних взаимодействий лиганда. Использовалась процедура пересчета ОФ с учетом ориентации
|
Значение коэффициента учета внутренних взаимодействий альфа |
Неправильно найденных позиций (из 100) |
Неправильно найденных позиций среди гибких лигандов (из 32) |
|
0 |
19 |
8 |
|
0.5 |
14 |
3 |
|
0.75 |
13 |
2 |
|
1 |
16 |
3 |
4.1. Значимость внутренних взаимодействий при докинге гибких лигандов
Для лигандов с малым числом вращательных связей учёт внутренних взаимодействий не должен сильно влиять на результат докинга, так как атомам лиганда сложно сблизиться на расстояние, необходимое для взаимодействия. Если у лиганда нет вращательных связей, то учет внутренних взаимодействий вообще не должен влиять на результат докинга. Приведенная на рис. 1 диаграмма зависимости доли неудачных расчетов докинга от количества вращательных связей в лиганде подтверждает это предположение.
Для гибких лигандов, напротив, учет внутренних взаимодействий лиганда может оказать существенное влияние на результат докинга, что также подтверждается диаграммой на рис. 1 и данными в последних столбцах таблиц 2 и 3. Отметим, что реализованный алгоритм учета внутренних взаимодействий позволил для гибких лигандов (с числом вращательных связей более 4) снизить число ошибок при докинге в 4 раза с 8 до 2 (см. табл. 3).
Из таблиц 2 и 3 также можно сделать вывод, что процедура пересчета ОФ с учетом ориентации, реализованная в исходной версии программы Алгокомб, улучшает результат докинга жестких молекул, но фактически не улучшает результат докинга для гибких молекул. Количество неверно найденных позиций среди гибких лигандов при использовании процедуры пересчета ОФ с учетом ориентации (но без учета внутренних взаимодействий лиганда) снизилось всего на одну с 9 до 8. В результате докинг с учетом внутренних взаимодействий лиганда, но без пересчета ОФ с учетом ориентации, дает в тесте Рогнана лучший результат (85% успеха), чем докинг без учета внутренних взаимодействий, но с пересчетом ОФ с учетом ориентации (81% успеха).
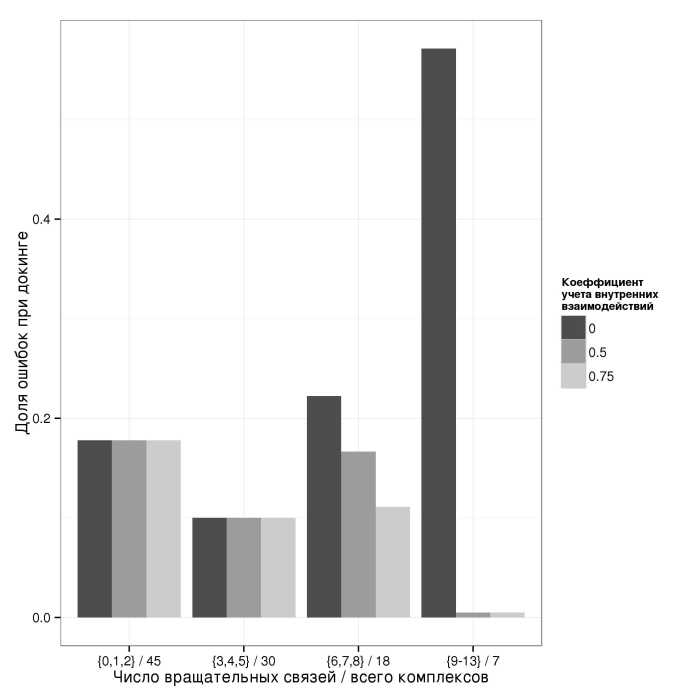
Рис. 1. Зависимость доли неправильно найденных с помощью процедуры докинга позиций от числа вращательных связей в лиганде на примере комплексов из теста Рогнана
4.2. Оптимальное значение коэффициента учета внутренних взаимодействий лиганда
Согласно реализованной модели взаимодействия лиганда с белком в итоговую ОФ вносят вклад как взаимодействия между атомами лиганда и белка, так и взаимодействия атомов лиганда между собой. Энергия межатомного взаимодействия и в том, и в другом случаях оценивается с помощью УПМВ Тарасова-Мурышева [2] (ТМ-потенциала), но при оценке взаимодействия атома лиганда с белком энергетический ТМ-потенциал умножается на коэффициент обусловленности C t (см. формулу (2)), показывающий, встречается ли атом лиганда соответствующего типа в похожем белковом окружении среди известных структур белок-лигандных комплексов из PDB [6]. Коэффициент обусловленности может принимать значение от 0 до 1. Таким образом, из формулы (2) видно, что энергетический ТМ-потенциал взаимодействия атома лиганда с белком умножается на некоторый коэффициент, меньший 1. Если бы мы учитывали внутренние нековалентные взаимодействия между атомами лиганда с помощью того же самого энергетического ТМ-потенциала без понижающего коэффициента, вклад внутренних взаимодействий между атомами лиганда был бы завышен по отношению к вкладу взаимодействий между атомами лиганда и белка, поэтому в формулу (3) был введен коэффициент альфа учета внутренних взаимодействий лиганда.
Расчеты показали, что наилучший результат в тесте Рогнана достигается при значении коэффициента учета внутренних взаимодействий лиганда около 0.75 (табл. 2 и 3). Этот результат согласуется с предположением, что наилучшие результаты докинга должны достигаться при значении коэффициента альфа, близком к усредненному значению коэффициента обусловленности на атомах лиганда в лучшей позиции, так как в лучшей позиции лиганда коэффициенты обусловленности должны быть близки к 1, но все же меньше 1.
4.3. Сравнение положений лиганда, найденных при докинге с учетом и без учета внутренних взаимодействий лиганда
Сравнение результатов докинга гибких лигандов с учетом и без учета взаимодействий между атомами лиганда показало, что улучшение качества докинга при учете внутренних взаимодействий лиганда было связано в основном с правильным учетом гидрофобных контактов, образующихся между разными частями лиганда.
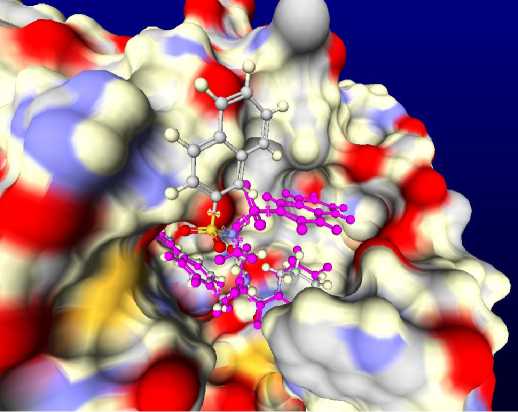
Рис. 2. Расположение лиганда, в активном сайте комплекса, ldwd. Фиолетовым цветом выделено положение лиганда, найденное при учете внутренних взаимодействий между атомами лиганда, и не сильно отличающееся от нативного положения, известного из рентгеноструктурного анализа.
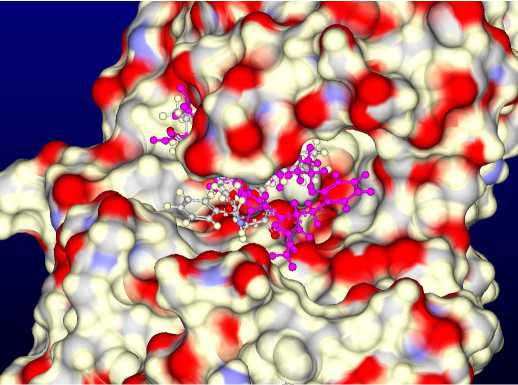
Рис. 3. Расположение лиганда, в активном сайте комплекса, leed. Фиолетовым цветом выделено положение лиганда, найденное при учете внутренних взаимодействий между атомами лиганда, и не сильно отличающееся от нативного положения, известного из реитгеиоструктуриого анализа.
На. рис. 2 и рис. 3 приведены положения лигандов в сайтах связывания комплексов с PDB-идентификаторами ldwd и leed, найденные с учетом и без учета, внутренних взаимодействий лиганда. Фиолетовым цветом выделено положение, найденное с учетом внутренних взаимодействий лиганда, и близкое к нативному положению, известному из рентгеноструктурного анализа. В обоих случаях нахождение правильного положения лиганда, связанно с учетом внутренних гидрофобных взаимодействий между атомами лиганда: в комплексе ldwd между нафтильной и циклогексильной группами, а. в комплексе leed между бензольным кольцом и циклогексильной группой.
5. Выводы
Предложенный алгоритм учета внутренних нековалентных взаимодействий между атомами лиганда существенно улучшил качество решения задачи докинга. В тесте Рогнана при учете внутренних взаимодействий число неправильно позиционированных лигандов уменьшилось на 32 процента (с 19 до 13). Особенно заметно улучшение позиционирования на гибких лигандах: для лигандов, имеющих 5 или более вращательных связей (всего 32 комплекса), число ошибок уменьшилось на 75 процентов (с 8 до 2).
Анализ положений лигандов показал, что улучшение результатов докинга было достигнуто в основном для лигандов, образующих в нативной конформации внутренние гидрофобные контакты. Внутренних водородных связей между атомами лиганда среди лигандов с улучшившимся результатом докинга нет, но этот факт объясняется спецификой выбора комплексов в тестовом наборе Рогнана. Для подтверждения значимости учета внутренних водородных связей между атомами лиганда для решения задачи докинга планируется сделать отдельную подборку тестовых комплексов.
Предложенный алгоритм учета изменения внутренней энергии лиганда и косвенного учета энергии взаимодействия лиганда с растворителем с помощью добавления суммы взаимодействий между атомами лиганда, оцененных с помощью УПМВ, является универсальным и может быть легко применен с любой другой ОФ на основе усредненных энергий.
Список литературы Учет взаимодействий между атомами Лиганда в задаче Докинга с помощью потенциала усредненных энергий
- Ramensky V., Sobol A., Zaitseva N., Rubinov A., Zosimov V. A novel approach to local similarity of protein binding sites substantially improves computational drug design results//Proteins. -2007. -V. 69(2). -P. 349-357
- Muryshev A.E., Tarasov D.N., Butygin A.V., Butygina O.Y., Aleksandrov A.B. & Nikitin S.M. A novel scoring function for molecular docking//J. of Comp.-Aided Mol. Design. -2003. -V. 17. -P. 597-605
- Хёльтье Х.Д. [и др.]. Молекулярное моделирование: теория и практика. -М.: БИНОМ. Лаборатория знаний, 2009
- Cheng T., Li X., Li Y., Liu Z., Wang R. Comparative assessment of scoring functions on a diverse test set//J. Chem. Inf. Model. -2009. Apr. -V. 49(4). -P. 1079-1093
- Plewczynski D., La𝑧´niewski M., Augustyniak R., Ginalski K. Can we trust docking results? Evaluation of seven commonly used programs on PDBbind database//J. Comput. Chem. -2011. -V. 32(4). -P. 742-55
- Электронная база данных трехмерных структур белковых комплексов Protein Data Bank (PDB). URL: http://www.rcsb.org/pdb/
- Scott J. Weiner, Peter A. Kollman, Paul Weiner, et al. A new force field for molecular mechanical simulation of nucleic acids and proteins//J. Am. Chem. Soc. -1984. -V. 106(3). -P. 765-784
- Andrew M. Petros, David G. Nettesheim, Stephen W. Fesik, et al. Rationale for Bcl-xL/Bad peptide complex formation from structure, mutagenesis, and biophysical studies//Protein Science. -2000. -V. 9. -P. 2528-2534
- Kellenberger E., Rodrigo J., Muller P. and Rognan D. Comparative evaluation of eight docking tools for docking and virtual screening accuracy//Proteins: Structure, Function, and Bioinformatics. -2004. -V. 57. -P. 225-242
- Huang S.Y., Zou X.Q. An Iterative Knowledge-Based Scoring Function to Predict Protein-Ligand Interactions: I. Derivation of Interaction Potentials//J. Comput. Chem. -2006. -V. 27. -P. 1866-1875
- Huang S.Y., Zou X.Q. Inclusion of solvation and entropy in the knowledge-based scoring function for protein-ligand interactions//J. Chem. Inf. Model. -2010. -V. 50. -P. 262-273
- Qiancheng Shen, Hualiang Jiang, et al. Knowledge-Based Scoring Functions in Drug Design: 2. Can the Knowledge Base Be Enriched?//J. Chem. Inf. Model. -2011. -V. 51. -P. 386-397
- Gehlhaar D.K., Verkhivker G.M., Rejto P.A., Sherman C.J., Fogel D.B., Fogel L.J., Freer S.T. Molecular recognition of the inhibitor AG-1343 by HIV-1 protease: conformationally flexible docking by evolutionary programming//Chem. Biol. -1995. -V. 2. -P. 317-324
- DeWitte R.S., Shakhnovich E.I. SMoG: de novo design method based on simple, fast and accurate free energy estimates. 1. Methodology and supporting evidence//J. Am. Chem. Soc. -1996. -V. 118. -P. 11733-11744
- Ischenko A.V., Shakhnovich E.I. Small Molecule Growth 2001 (SMoG2001): An improved knowledgebased scoring function for protein-ligand interaction//J. Med. Chem. -2002. -V. 45. -P. 2770-2780
- Muegge I., Martin Y.C. A general and fast scoring function for protein-ligand interactions: a simplified potential approach//J. Med. Chem. -1999. -V. 42(5). -P. 791-804
- Gohlke H., Hendlich M., Klebe G. Knowledge-based scoring function to predict protein-ligand interactions//J. Mol. Biol. -2000. -V. 295(2). -P. 337-356
- Mitchell J.B.O., Laskowski R.A., Alex A.,Thornton J.M. BLEEP-potential of mean force describing protein-ligand interactions: I. Generating potential//J. Comp.Chem. -1999. -V. 20(2). -P. 1165-1176
- Ozrin V.D., Subbotin M.V., Nikitin S.M. PLASS: protein-ligand affinity statistical score-a knowledge-based force-field model of interaction derived from the PDB//J. Comput. Aided Mol. Des. -2004. -V. 18(4). -P. 261-270
- Mooij W.T., Verdonk M.L. General and targeted statistical potentials for protein-ligand interactions//Proteins. -2005. -V. 61(2). -P. 272-287
- Yang C.Y., Wang R., Wang S. M-score: a knowledge-based potential scoring function accounting for protein atom mobility//J. Med. Chem. -2006. -V. 49(20). -P. 5903-5911
- Eldridge M.D., Mee R.P. et al. Empirical scoring functions: I. The development of a fast empirical scoring function to estimate the binding affinity of ligands in receptor complexes//J. of Comp.-Aided Mol. Design. -1997. -V. 11. -P. 425-445
- Seiji Tanaka and Harold A. Scheraga. Medium-and Long-Range Interaction Parameters between Amino Acids for Predicting Three-Dimensional Structures of Proteins//Macromolecules. -1976. -V. 9(6). -P. 945-950
- Manfred J. Sippl. Calculation of conformational ensembles from potentials of mena force: An approach to the knowledge-based prediction of local structures in globular proteins//J. Mol. Biol. -1990. -V. 213(4). -P. 859-883
- Manfred J. Sippl. Boltzmann’s principle, knowledge-based mean fields and protein folding. An approach to the computational determination of protein structures//J. Comput. Aided Mol. Des. -1993. -V. 7. -P. 473-501


