Валидация геномного прогноза племенной ценности быков-производителей по признакам молочной продуктивности дочерей на примере популяции черно-пестрого и голштинского скота
Автор: Сермягин А.А., Белоус А.А., Контэ А.Ф., Филипченко А.А., Ермилов А.Н., Янчуков И.Н., Племяшов К.В., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетическая структура популяций
Статья в выпуске: 6 т.52, 2017 года.
Бесплатный доступ
Быстрое развитие молекулярно-генетических методов в селекции животных за последние десять лет привело к повышению интенсивности отбора в популяциях. Расширение перечня учитываемых хозяйственно полезных признаков молочного скота позволило увеличить возможности селекции по улучшению здоровья коров и изучению природы метаболического синтеза компонентов молока. Целью настоящей работы была валидация оценок геномной племенной ценности по признакам молочной продуктивности с использованием ядра популяции скота черно-пестрой и голштинской пород на основе полногеномных данных. Было проведено моделирование отбора в 124 стадах из Московской и Ленинградской областей на основании результатов геномной оценки племенной ценности в сравнении с оценкой по предкам и качеству потомства. Установлено, что отбор производителей на основе геномной информации корректирует оценку по родословной и уточняет прогноз племенной ценности животного, полученный по качеству потомства. Значения повторяемости геномных оценок племенной ценности были получены посредством валидации информации по родословной и геному для 100 быков с наличием данных как минимум по 300 дочерям. Выборка составила ядро создаваемой референтной региональной группы молочного скота. Для расчета аддитивной матрицы родства использовали 1050 предков. После контроля качества генотипирования в анализ было взято 39818 SNP. Процедура оценки животных основывалась на методах REML (restricted maximum likelihood estimation), BLUP SM (BLUP Sire Model), GBLUP (genomic BLUP). Среднегодовой генетический тренд по признакам молочной продуктивности в исследуемых популяциях составил за период с 1987 по 2006 год +60 кг молока по удою, +2,5 кг по количеству молочного жира, +1,5 кг по содержанию молочного белка. Повторяемость оценок, полученных по геному, ранжировалась от 0,371 до 0,606 для признаков молочной продуктивности, что в среднем превышало прогноз племенной ценности предков на 0,147. Точность оценки, полученной по качеству потомства, варьировала от 0,879 до 0,900 или была выше прогноза по геному на 0,405 ед. Изучены подходы по принципу формирования референтной популяции на основе анализа многомерного шкалирования и генетических дистанций, которые составили между региональными популяциями Fst = 0,0025. Показано разложение неравновесия по сцеплению между маркерами на дистанциях до 1000 т.п.н. Установлено, что на расстояниях от 5 до 70 т.п.н. степень сцепления достигала максимальных величин от 0,20 до 0,54. В рамках изучения метаболических путей синтеза компонентов молока получены селекционно-генетические параметры и MLS-оценки по расширенному составу молока: лактозе (h2 = 0,18), сухому веществу (h2 = 0,10), сухому обезжиренному молочному остатку (h2 = 0,19), точке замерзания молока (h2 = 0,06), оценке числа соматических клеток (h2 = 0,10) и концентрации мочевины (h2 = 0,04). По ряду признаков установлены значения аддитивной генетической изменчивости, указывающие на объективные возможности их использования в селекции молочного скота. Для получения достоверных полногеномных ассоциаций будет проводиться дальнейшее накопление базы компонентного состава молока коров по дополнительным спектрам. В процессе комплексных исследований обоснованы подходы по использованию геномных оценок, расширению референтной популяции и списка признаков количественного и качественного состава молока.
Геномная оценка племенной ценности, молочная продуктивность, референтная популяция, неравновесие по сцеплению, наследуемость, компонентный состав молока
Короткий адрес: https://sciup.org/142214093
IDR: 142214093 | УДК: 636.2:636.082:575.113.1 | DOI: 10.15389/agrobiology.2017.6.1148rus
Текст научной статьи Валидация геномного прогноза племенной ценности быков-производителей по признакам молочной продуктивности дочерей на примере популяции черно-пестрого и голштинского скота
Развитие современных методов и подходов требует переосмысления существующей практики племенного дела в стадах крупного рогатого скота. Система селекции животных в рамках программ разведения пород становится определяющей, поскольку во многом зависит от давления отбора
* В работе использовано оборудование ЦКП «Биоресурсы и биоинженерия сельскохозяйственных животных» ФГБНУ ФНЦ ВИЖ им. Л.К. Эрнста. Исследования выполнены в рамках проекта ¹ 17-44-500324, поддержанного РФФИ и Правительством Московской области. Использованы полногеномные профили быков, полученные при выполнении работ по проекту РНФ ¹ 14-36-00039.
и инструментов оценки племенной ценности по таким категориям, как отцы и матери быков-производителей, а также отцы и матери высокопродуктивных коров. Неотъемлемым звеном в работе с отечественным скотом должна стать комплексная проверка животных по собственной продуктивности, родословной, суммарному влиянию генетических маркеров (оценка по геному), качеству потомства.
Проект по внедрению геномной оценки в практику молочного скотоводства, стартовавший в начале XXI века, открыл геномную эру в селекции (1, 2). Получены первые результаты геномной оценки быков голштинской и джерсейской пород (3, 4). В настоящее время по крайней мере 32 страны принимают участие в организации международной системы геномной оценки (multiple across country evaluation, GMACE). Однако только в 11 из них прогноз геномной племенной ценности по молочной продуктивности имеет официальный статус для использования в селекционных программах с молочным скотом (5-7).
Практическая эффективность подходов геномной селекции зависит от множества факторов, главные из которых — объем референтной популяции, вклад информации по каждому генотипированному животному и его потомкам по ряду поколений, природа генетической изменчивости изучаемых признаков (наследуемости), а также плотность геномного сканирования, распределение локусов количественных признаков (QTL) и степень неравновесного сцепления (LD) маркеров с ними (8). Однако при достижении этих параметров, в частности по числу животных в референтной популяции (например, свыше 1 млн гол. в США), возникают совершенно иные задачи по разработке алгоритмов для расчета геномных оценок. Так, алгоритм APY используется для формирования ядра популяции из проверенных по качеству потомства и молодых (ремонтных) генотипиро-ванных животных и предусматривает инвертирование геномной матрицы меньшего размера. Для голштинской породы оптимальное число составляет не менее 12 тыс. гол. в зависимости от эффективного размера популяции (9). Вопрос оперативного доступа к информации и времени, которое компьютер затрачивает на процедуру, также оказывается определяющим: от 2 ч при использовании APY-алгоритма до 1 мес при стандартном подходе для получения геномной матрицы родства (GRM) вида G-1 (10).
Кроме того, ведутся исследования по идентификации отдельных каузальных нуклеотидов (точечных мутаций) для количественных признаков (QTN), которые наравне со множеством известных некодирующих полиморфных замен (SNP) могут повысить точность геномного прогноза. Поиски направлены на оптимизацию числа SNP с высокой долей изменчивости каузальных вариантов для построения GRM при учете информации по большому числу генотипов и приближения точности оценок к 100 % (11). Так, причинные SNP, достоверно ассоциированные с хозяйственно полезными признаками молочного скота, были детектированы на хромосомах 5-й, 6-й, 9-й, 14-й, 15-й и 20-й. Наибольшую генетическую дисперсию по показателям молочной продуктивности имели полиморфизмы, расположенные близко либо внутри генов DGAT1 (BTA14), GHR (BTA20), ABCG2 (BTA6) (12-16). В то же время для низконаследуемых признаков (возраст 1-го отела и продолжительность сервис-периода) также были обнаружены регионы на 21-й хромосоме (47-59 млн.п.н.) с генами FAM181A, SLC24A4 и NKX21 (17). Следует отметить, что по признакам состава молока и мяса (протеиновая фракция, жирные кислоты) получены результаты, указывающие на тесную связь с некоторыми QTL-регионами в геноме (18, 19). Это направление исследований весьма актуально для по- нимания процессов синтеза первичных структурных элементов и поиска метаболических путей регуляции количественных признаков у животных.
При селекционном контроле состава молока коров наибольший интерес представляют экономические показатели доли жира и белка, количества соматических клеток. В последнее время значимое место отводится индикаторам здоровья животных в стадах, которые можно опосредованно определить по составу исходного продукта (20). Введение дополнительных параметров оценки количественного и качественного состава молока коров (следы ацетона и β -гидроксимасляной кислоты, содержание мочевины) должно положительно отразиться на реализации генетической ценности животного за счет нормализации обмена веществ (ацидоз, кетоз), улучшения фертильности, удлинения срока продуктивного использования и качества получаемой продукции (21).
Анализ генетических параметров содержания лактозы, сухого вещества (СВ) и сухого обезжиренного молочного остатка (СОМО) в молоке коров показал перспективность использования этих признаков в селекции: для лактозы — h2 = 0,281-0,340, СВ — h2 = 0,298, СОМО — h2 = 0,20-0,30 (22, 23). Для концентрации мочевины в молоке, характеризующей баланс между энергией рациона и потреблением протеина животным, также показана детерминированность генотипом (h2 от 0,072 до 0,130-0,220) (24). Другие индивидуальные параметры, такие как точка замерзания (ТЗ), позволяют оценить регламент процесса доения на предмет избыточного содержания воды в молоке. В связи со значительным влиянием на ТЗ паратипических факторов (возраст животного, месяц года, стадия лактации, размер стада, система доения) показатель наследуемости не превышал h2 = 0,120 (25, 26; A. Costa с соавт., 2017, персональное сообщение). Контроль компонентного состава молока коров по большему числу показателей позволит глубже понять процессы обмена веществ в организме с целью поиска генетических механизмов регуляции метаболизма.
Создание системы геномной оценки — один из приоритетов интеграции российского племенного животноводства в мировую структуру с целью повышения конкурентоспособности отечественного племенного материала. Эта задача не решается простым воспроизведением зарубежных моделей из-за существенных различий в факторах среды (кормление, содержание, технологии, климатические условия), генетической структуре популяций (генетическое разнообразие, LD), методах и способах измерения хозяйственно полезных признаков. В настоящее время происходит накопление информации о племенном учете, расширение российской референтной популяции скота черно-пестрой и голштинской пород, а также апробирование современных методик оценки генотипа животных. Лучшие мировые практики в этой области могут быть эффективно инкорпорированы при достижении сопоставимого количества и объема информации.
Методологию геномного прогноза отрабатывали в Московской и Ленинградской областях — регионах с высокими результатами по продуктивности и племенной работе в молочном скотоводстве. В статье представлены результаты по моделированию отбора быков-производителей посредством валидации геномного прогноза племенной ценности, впервые выполненной на российской популяции голштинизированного черно-пестрого и голштинского скота. Показано, что в рамках региональной компоненты значения генетических оценок уточняются показателями предков, которые, в свою очередь, корректируются по большому числу SNP-маркеров. Описана аддитивная изменчивость по дополнительным показателям состава молока коров, отражающим качественные и количественные параметры продукции.
Цель работы — валидация геномной племенной ценности по признакам молочной продуктивности с использованием ядра популяции черно-пестрого и голштинского скота на основе полногеномных данных.
Методика. Исследования проводили на популяциях голштинизиро-ванного черно-пестрого и голштинского скота в Московской (МО) и Ленинградской (ЛО) областях. Из каждого региона отобрали по 50 быков-производителей (годы рождения 1987-2006), генотипированных с помощью биочипа Illumina Bovine SNP50K v2 BeadChip («Illumina», США) с плотностью 54609 SNP. В выборку вошли быки, имеющие не менее 300 дочерей-первотелок с известными показателями по удою за 305 сут лактации, массовой доле жира (МДЖ) и белка (МДБ), количеству молочного жира (МЖ) и белка (МБ). Выборка стала основой ядра референтной популяции, на которой моделировали процедуры оценки быков по предкам (родословной), качеству потомства и полногеномным данным.
Для расчета прогноза племенной ценности (EBV) использовали BLUP Sire Model (BLUP SM) по ранее апробированной модели смешанного типа (15). Общая база данных включала информацию по 77375 коровам-первотелкам из 124 племенных стад (35190 гол. из 77 стад МО, 42185 гол. из 47 стад ЛО). Посредством обнуления EBV для случайно отобранных 10 быков-производителей из каждой региональной популяции (всего 20 гол.), были пересчитаны оценки по средней племенной ценности предков (PA) и получен геномный прогноз (DGV, непосредственная геномная племенная ценность) согласно методу GBLUP (genomic BLUP) (27). Валидируемая выборка состояла из 68175 гол. (31485 гол. из МО, 36690 гол. из ЛО). Вариационноковариационные компоненты оценивали методом ограниченного максимального правдоподобия (restricted maximum likelihood estimation, REML) с учетом популяционных констант наследуемости: по удою — 0,180, МДЖ — 0,221, МЖ — 0,177, МДБ — 0,173, МБ — 0,142. EBV быков-производителей получали с помощью программы семейства BLUPF90, включая REMLF90 (28).
Общее число предков в родословной быков-производителей составляло 1050 гол. Расчет генетических дистанций между популяциями (Fst) и многомерное шкалирование (MDS) c оценкой неравновесия по сцеплению между маркерами осуществляли с использованием пакетов Plink 1.07 и Plink 1.9 (29). После контроля качества генотипирования в анализ взяли 39818 SNP. Для получения значений LD вычисления проводили по каждой хромосоме с последующим усреднением в скользящем окне размером 1000 т.п.н. (--chr … --r2 --ld-window 1000 --ld-window-kb 1000 --ld-window-r2 0) (30).
Использовав базу данных о расширенном составе молока (7784 коровы из 7 племенных стад, Московская обл., 2016 год), созданную по результатам инфраспектрометрического анализа (прибор CombiFoss F+, «FOSS», Дания), оценили имеющиеся показатели с целью изучения метаболических путей и механизмов регуляции синтеза основных компонентов молока. Учитывали селекционно-генетические параметры количественных (содержания лактозы, %; сухое вещество, %; СОМО, %) и качественных признаков (точка замерзания молока, ° C; число соматических клеток, SCS; количества мочевины, мг/100 мл). Оценку средних по показателям проводили методом наименьших квадратов (MLS) в программе Statistica 7.0, учитывая взаимодействия стадо—месяц контрольного события, возраст в лактациях и влияние отца-быка.
Результаты. Для сопоставления оценок племенной ценности быков по регионам и выделения генетических групп животных, в том числе с целью прогнозирования эффекта отбора и результативности подбора производителей разных генераций, провели сравнительный анализ по годам рождения.
Установлен поступательный рост генетического потенциала групп быков
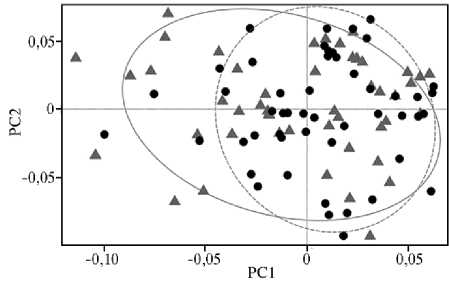
Рис. 1. Пространственное расположение популяций гол-штинизированных черно-пестрых и голштинских быков-производителей из хозяйств Московской ( ▲ ) и Ленинградской ( • ) областей по главным компонентам PC1 и PC2.
регионах. При детальном анализе методом
вне зависимости от региона происхождения и числа оцененных потомков. По Московской области среднегодовой генетический тренд составил +37 кг молока, +1,7 и +0,8 кг продукции молочного жира и белка, в Ленинградской области он был выше более чем в 2 раза — соответственно +84, +3,3 и +2,3 кг. Мы полагаем, что рост в значительной степени был связан с объемами и интенсивностью использования племенных ресурсов голштинской породы в многомерного шкалирования было показано, что объединенные региональные популяции скота на при- мере быков-производителей значительно не отличались друг от друга гене- тически (рис. 1). Индекс фиксации (Fst) при этом составил 0,0025, что полностью подтверждает идентичность животных по происхождению от скота голштинского черно-пестрого корня.
Дистанция, т.п.н.
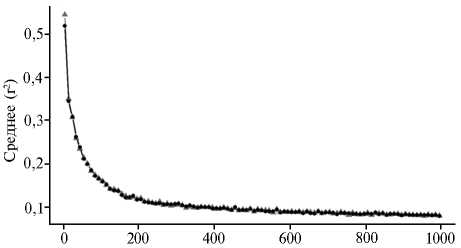
Рис. 2 Разложение неравновесия по сцеплению между маркерами на дистанции 1000 т.п.н. для популяций голштинизированных черно-пестрых и голштинских быков-производителей из хозяйств Московской ( ▲ ) и Ленинградской областей ( • ).
Разложение неравновесия по сцеплению между маркерами (SNP) показало тесный характер наследования полиморфизмов на расстоянии от 0-5 до 60-70 т.п.н. с r2 = 0,201-0,544 для МО и r2 = 0,199-0,518 для ЛО. На дистанциях 110-120 т.п.н. наблюдалась умеренная взаимосвязь по сцеплению от 0,151 (МО) до 0,153 (ЛО), что потенциально указывает на возможность одинаково эффективно картировать QTL в популяциях молочного скота разного происхождения (рис. 2). Минимальные LD отмечали на расстоянии между маркерами 1000 т.п.н. (r2 = 0,080).
1. Валидация оценок племенной ценности голштинизированных черно-пестрых и голштинских быков-производителей (Московская и Ленинградская обл.)
|
Признак |
PA |
DGV |
EBV |
|
Удой за 305 сут первой лактации |
0,495 |
0,606 |
0,900 |
|
Массовая доля жира |
0,272 |
0,371 |
0,897 |
|
Количество молочного жира |
0,208 |
0,468 |
0,899 |
|
Массовая доля белка |
0,403 |
0,468 |
0,896 |
|
Количество молочного белка |
0,338 |
0,535 |
0,879 |
Прим еч ани е. PA — племенная ценность по предкам, DGV — непосредственная геномная племенная ценность, EBV — племенная ценность по потомству. Для PA и DGV расчет повторяемости произведен на основе рангового коэффициента корреляции, для EBV расчет точности произведен на основе вариационных компонентов по методу REML.
Мы валидировали прогноз геномной племенной ценности в сравнении с другими оценками (табл. 1): по родословной повторяемость в среднем 1152
составляла 0,343, по геному — 0,490, по потомству — 0,894. Это обеспечило превышение значений непосредственной геномной племенной ценности (DGV) по сравнению с оценкой по предкам на +0,147 ед., или на 14,7 %. Сравнение для PA и DGV проводили с оценкой по EBV согласно ранговому коэффициенту корреляции. Точность EBV рассчитывали на основе вариационных компонентов генетической изменчивости методом REML. Максимальные значения точности геномных оценок получили для удоя (0,606), МБ (0,535), МДБ (0,468), МЖ (0,468). По жирномолочности показатель прогнозной оценки был минимальным. Мы связываем это с разным направлением и интенсивностью селекции голштинизированного черно-пестрого (прежде всего голштинского) скота в МО (удой, процент жира и белка) и ЛО (удой, сумма молочной продукции). Можно говорить об эффективности прогнозных оценок по геному для уточнения племенной ценности по предкам и корректировки показателей оценки по качеству потомства при недостаточном его числе. Полученные прогнозные значения позволили кратко обосновать закономерности наследования хозяйственно полезных признаков в популяции голштинизированного черно-пестрого и голштинского скота центральных регионов России. Важный элемент внедрения результатов геномной оценки в селекционный процесс — разработка критериев отбора животных в референтную популяцию, объединение родственных групп скота в единую информационную систему, оценка эффективности привлеченной информации по комплексу признаков, а также валидация оценок и отбор молодых быков с геномным прогнозом для заказных спариваний.
Чтобы найти дополнительные параметры для количественной и качественной оценки состава молока коров, мы определили генетические взаимосвязи и изменчивость по ряду показателей (табл. 2). Некоторые из них обусловлены паратипическими факторами, однако изучение генетической составляющей может помочь выявить комплексные механизмы регуляции проявления таких признаков как на популяционном, так и на геномном уровне.
2. Селекционно-генетические параметры и MLS-оценка состава молока от голштинизированных черно-пестрых и голштинских коров по дополнительным признакам (Московская и Ленинградская области)
|
Показатель |
MLS |
Показатель |
|||||
|
лактоза |
СВ |
СОМО |
ТЗ 1 |
SCS 1 |
мочевина |
||
|
Лактоза |
4,65±0,01 |
0,18н |
- 0,07 |
0,24 |
0,09 |
- 0,43 |
- 0,07 |
|
СВ |
12,31±0,06 |
- 0,08 |
0,10н |
0,24 |
- 0,13 |
0,08 |
- 0,19 |
|
СОМО |
8,99±0,02 |
- 0,03 |
0,48 |
0,19н |
0,17 |
0,04 |
- 0,23 |
|
ТЗ |
- 0,586±0,003 |
- 0,36 |
0,19 |
0,41 |
0,06н |
- 0,03 |
- 0,34 |
|
SCS |
3,9±0,1 |
- 0,73 |
0,07 |
- 0,01 |
0,25 |
0,10н |
0,03 |
|
Мочевина |
27,9±0,4 |
- 0,16 |
- 0,15 |
0,19 |
- 0,53 |
- 0,06 |
0,04н |
П р и м е ч а н и е. СВ — сухое вещество, СОМО — сухой обезжиренный молочный остаток, ТЗ — точка замерзания молока, SCS — оценка числа соматических клеток; по диагонали — наследуемость (н), ниже диагонали — генетические корреляции, выше — фенотипические корреляции.
Наследуемость показателей расширенного состава молока коров значительно варьировала. В близких к стандартным величинам пределах находились процент лактозы (0,18), СОМО (0,19) и SCS (0,10). Для СВ и ТЗ наблюдалась пониженная аддитивная изменчивость (соответственно 0,10 и 0,06), а для мочевины были получены низкие значения, что, по нашему мнению, связано с отсутствием баланса рациона по энергии (недостаток) и протеину (избыток) и небольшим числом наблюдений. Значительное влияние паратипических факторов не позволило установить достоверные различия по межгрупповой изменчивости для дочерей быков-производителей. Обнаружилась тесная генетическая взаимосвязь между содержанием лактозы в молоке и числом соматических клеток (rg = -0,73), что может быть обусловлено использованием молочного сахара бактерия- ми. Умеренные корреляции установлены между содержанием сухого вещества в молоке и СОМО (rg = 0,48), а также между точкой замерзания и СОМО (rg = 0,41), концентрацией мочевины (rg = -0,53) и лактозы (rg = -0,36). С увеличением числа соматических клеток в молоке его точка замерзания снижается (rg = 0,25), что может свидетельствовать об отрицательном влиянии патогенной микрофлоры на физические свойства молока. Эти исследования будут продолжены при расширении базы данных по контрольным суточным показателям за 2013-2017 годы.
Таким образом, прогнозирование племенной ценности (EBV) быков-производителей по однонуклеотидным полиморфизмам эффективнее оценки по предкам. Популяционно-генетический анализ подтвердил обоснованность использования объединенной референтной выборки для определения EBV вследствие идентичности происхождения животных, входящих в ядро популяции. Наибольшее сцепление между маркерами наблюдалось на расстояниях от 5 до 120 т.п.н. при минимальном r2 = 0,15, что обеспечивает устойчивую передачу наследственной информации по ряду локусов количественных признаков на уровне породы вне зависимости от популяционной принадлежности. Дополнительные показатели состава молока могут стать индикаторами контроля за состоянием здоровья животных, качеством продукции, расширят возможности селекции. Анализ полногеномных ассоциаций поможет создать карту метаболических путей синтеза молока у коров отечественной популяции голштинизированного черно-пестрого и голштинского скота.
Список литературы Валидация геномного прогноза племенной ценности быков-производителей по признакам молочной продуктивности дочерей на примере популяции черно-пестрого и голштинского скота
- Schaeffer L.R. Strategy for applying genome-wide selection in dairy cattle. J. Anim. Breed. Genet., 2006, 123: 218-223.
- Смарагдов М.Г. Тотальная геномная селекция с помощью SNP как возможный ускоритель традиционной селекции. Генетика, 2009, 45(1): 725-728.
- García-Ruiz A., Cole J.B., VanRaden P.M., Wiggans G.R., Ruiz-Lópeza F.J., Van Tassell C.P. Changes in genetic selection differentials and generation intervals in US Holstein dairy cattle as a result of genomic selection. PNAS, 2016, 113(28): E3995-E4004 ( ) DOI: 10.1073/pnas.1519061113
- Taylor J.F., Taylor K.H., Deckera J.E. Holsteins are the genomic selection poster cows. PNAS, 2016, 113(28): 7690-7692 ( ) DOI: 10.1073/pnas.1608144113
- Interbull Centre. Режим доступа: http://www.interbull.org/static/web/g_proddoc1708r.pdf. Дата обращения: 01.11.2017.
- Vandenplas J., Gengler N. Strategies for comparing and combining different genetic and genomic evaluations: A review. Livest. Sci., 2015: 121-130 ( ) DOI: 10.1016/j.livsci.2015.09.012
- Sullivan P.M. Defining a parameter space for GMACE. Interbull Bulletin, 2016, 50: 85-93.
- Hayes B.J., Bowman P.J., Chamberlain A.J., Goddard M.E. Invited review: Genomic selection in dairy cattle: Progress and challenges. J. Dairy Sci., 2009, 92: 433-443 ( ) DOI: 10.3168/jds.2008-1646
- Misztal I., Legarra A. Invited review: efficient computation strategies in genomic selection. Animal, 2017, 11(5): 731-736 ( ) DOI: 10.1017/S1751731116002366
- Masuda Y., Misztal I., Tsuruta S., Legarra A., Aguilar I., Lourenco D.A.L., Fragomeni B., Lawlor T.L. Implementation of genomic recursions in single-step genomic BLUP for US Holsteins with a large number of genotyped animals. J. Dairy Sci., 2016, 99: 1968-1974 ( ) DOI: 10.3168/jds.2015-10540
- Fragomeni B.O., Lourenco D.A.L., Masuda Y., Legarra A., Misztal I. Incorporation of causative quantitative trait nucleotides in single-step GBLUP. Genet. Sel. Evol., 2017, 49: 59 ( ) DOI: 10.1186/s12711-017-0335-0
- Смарагдов М.Г. Анализ расположения локусов, влияющих на показатели молока, в хромосомах крупного рогатого скота. Генетика, 2008, 44(6): 829-834.
- Смарагдов М.Г. Связь полиморфизма гена DGAT1 у быков-производителей с молочной продуктивностью коров. Генетика, 2011, 47(1): 126-132.
- Смарагдов М.Г. Исследование связи аллелей генов рецептора гормона роста и рецептора пролактина у быков-производителей с молочной продуктивностью их дочерей. Генетика, 2012, 48(9): 1085-1090.
- Sermyagin A.A., Gladyr’ E.A., Kharitonov S.N., Ermilov A.N., Strekozov N.I., Brem G., Zinovieva N.A. Genome-wide association study for milk production and reproduction traits in Russian Holstein cattle population. Agricultural Biology, 2016, 51(2): 182-193 ( ) DOI: 10.15389/agrobiology.2016.2.182eng
- Weller J.I., Ezra E., Ron M. Invited review: A perspective on the future of genomic selection in dairy cattle. J. Dairy Sci., 2017, 100: 8633-8644 ( ) DOI: 10.3168/jds.2017-12879
- Nayeri S., Sargolzaei M., Abo-Ismail M.K., May N., Miller S.P., Schenkel F., Moore S.S., Stothard P. Genome-wide association for milk production and female fertility traits in Canadian dairy Holstein cattle. BMC Genet., 2016, 17: 75 ( ) DOI: 10.1186/s12863-016-0386-1
- Huang W., Peñagaricano F., Ahmad K.R., Lucey J.A., Weigel K.A., Khatib H. Association between milk protein gene variants and protein composition traits in dairy cattle. J. Dairy Sci., 2012, 95: 440-449 ( ) DOI: 10.3168/jds.2011-4757
- Saatchi M., Garrick D.J., Tait Jr. R.G., Mayes M.S., Drewnoski M., Schoon-maker J., Diaz C., Beitz D.C., Reecy J.M. Genome-wide association and prediction of direct genomic breeding values for composition of fatty acids in Angus beef cattle. BMC Genomics, 2013, 14: 730 ( ) DOI: 10.1186/1471-2164-14-730
- Filipejová T., Kováčik J., Kirchnerová K., Foltýs V. Changes in milk composition as a result of metabolic disorders of dairy cows. Potravinárstvo, 2011, 5(1): 10-16 (doi: 10.5219/113).
- Hanuš O., Genčurová V., Zhang Y., Hering P., Kopecký J., Jedelská R., Dolínková A., Motyčka Z. Milk acetone determination by the photometrical method after microdiffusion and via FT infra-red spectroscopy. J. Agrobiol., 2011, 28(1): 33-48 ( ) DOI: 10.2478/v10146-011-0004-9
- Jonkus D., Paura L. Estimation of genetic parameters for milk urea and milk production traits of Latvian Brown cows. Agriculturae Conspectus Scientificus, 2011, 76(3): 227-230.
- Czajkowska A., Sitkowska B., Piwczynski D., Wójcik P., Mroczkowski S. Genetic and environmental determinants of the urea level in cow’s milk. Arch. Anim. Breed., 2015, 58: 65-72 ( ) DOI: 10.5194/aab-58-65-2015
- Rzewuska K., Strabel T. The genetic relationship between reproduction traits and milk urea concentration. Anim. Sci. Pap. Rep., 2015, 33(3): 243-256.
- Henno M., Ots M., Joudu I., Kaart T., Kart O. Factors affecting the freezing point stability of milk from individual cows. Int. Dairy J., 2008, 18: 210-215 ( ) DOI: 10.1016/j.idairyj.2007.08.006
- Otwinowska-Mindur A., Ptak E., Grzesiak A. Factors affecting the freezing point of milk from Polish Holstein-Friesian cows. Ann. Anim. Sci., 2017, 17(3): 873-885 ( ) DOI: 10.1515/aoas-2016-0088
- VanRaden P.M. Efficient methods to compute genomic predictions. J. Dairy Sci., 2008, 91: 4414-4423 ( ) DOI: 10.3168/jds.2007-0980
- Misztal I., Tsuruta S., Strabel T., Auvray B., Druet T., Lee D.H. BLUPF90 and related programs (BGF90). Proc. 7th World Congress on genetics applied to livestock production. Montpellier, 2002, 28(28-27): 21-22.
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M., Bender D., Maller J., Sklar P., de Bakker P., Daly M.J., Sham P.C. PLINK: a toolset for whole-genome association and population-based linkage analysis. Am. J. Hum. Genet., 2007, 81(3): 559-575 ( ) DOI: 10.1086/519795
- Pérez O’Brien A.M., Utsunomiya Y.T., Mészáros G., Bickhart D.M., Liu G.E., Van Tassell C.P., Sonstegard T.S., da Silva M.V.B., Garcia J.F., Sölkner J. Assessing signatures of selection through variation in linkage disequilibrium between taurine and indicine cattle. Genet. Sel. Evol., 2014, 46: 19 ( ) DOI: 10.1186/1297-9686-46-19


