Валидация панели SNP-маркеров для контроля происхождения локальных российских пород овец
Автор: Денискова Т.Е., Доцев А.В., Гладырь Е.А., Сермягин А.А., Багиров В.А., Хомподоева У.В., Ильин А.Н., Брем Г., Зиновьева Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Геномное сканирование, контроль происхождения
Статья в выпуске: 6 т.50, 2015 года.
Бесплатный доступ
Создание панелей для контроля происхождения овец на основе наиболее высокоинформативных SNP (single nucleotide polymorphism) с частотой минорных аллелей MAF ≥ 0,3 - актуальная проблема овцеводства. Международным обществом генетики животных (International Society for Animal Genetics, ISAG) предлагается использование панели происхождения из 88 аутосомных SNP, разработанной Международным консорциумом по геномике овец (International Sheep Genomics Consortium, ISGС). Однако эта панель базируется на результатах оценки ДНК-профилей только североамериканских, новозеландских и австралийских пород овец. Российские породы не были включены в исследование, и вопрос о возможности применения панели ISAG для подтверждения достоверности их происхождения остается открытым. Мы провели полногеномное исследование SNP у четырех аборигенных российских пород овец - романовской (ROM, n= 22), забайкальской тонкорунной (ZBL, n = 7), полугрубошерстной бурятской овцы Буубей (BUB, n= 15) и тувинской короткожирнохвостой (TUV, n = 16) с использованием Ovine SNP50k BeadChip. Обработку полученных данных проводили для общего числа маркеров (54241 SNP) и для 88 аутосомных SNP, рекомендованных ISAG. Для оценки информативности панели в контроле происхождения для каждой из пород и для всей выборки в целом определяли долю MAF≥ 0,3, среднюю частоту MAF, вероятность совпадения генотипов (PI) и вероятности исключения в качестве родителей (P1, P2, P3). Универсальность панели оценивали посредством сравнения степени генетической дифференциации пород на основании всего спектра SNP и панели ISAG, используя в качестве критериев значения индекса F st (AMOVA) при парном сравнении и результаты анализа главных компонент (PCA плот). Для расчетов использовали программное обеспечение Plink 1.09 и GenAlEx 6.5. По результатам контроля качества всей выборки было отобрано 47385 SNP со средним значением MAF = 0,292±0,131. Значение MAF для 88 SNP происхождения составило 0,380±0,091. Большая часть SNP (81,8 %) оказались информативными (MAF ≥ 0,3). Доля информативных SNP различалась между породами и составляла 56,8 % у ROM, 63,4 % - у ZBL, 71,6 % - у BUB и 72,7 % - y TUV. При этом 21 SNP (23,9 %) были высокоинформативны во всех четырех породах; 37 SNP (42,0 %), 17 SNP (19,3 %) и 10 SNP (11,4 %) оказались информативными соответственно в трех, двух или только в одной породе. Маркер DU196132_525.1 был мономорфен у овец TUV (MAF = 0). Три SNP с MAF st при использовании 88 SNP по сравнению с полногеномными SNP-профилями при сохранении характера выявленных связей подтвердили высокую универсальность панели ISAG. Низкая породная зависимость SNP панели подтверждалась формированием на PCA плоте слабо консолидированных перекрывающихся массивов, соответствующих отдельным породам. Вероятность совпадения генотипов по 88 SNP составила от 4,32×10 -33 у TUV до 7,48×10 -33 у BUB. Вероятность исключения в качестве родителя составила P1 ≥ 99,99 % для всех четырех пород. Значение P2 оказалось максимальным для TUV (P2 ≥ 99,99 %), для трех остальных пород - P2 ≥ 99,98 %, значение P3 для всех пород было A 99,99 %. Полученные нами данные, хотя и обнаруживают некоторую породную зависимость панели ISAG, в целом показывают ее пригодность для контроля достоверности происхождения четырех аборигенных российских пород овец.
Генотипирование snp, локальные породы овец, панель isag, контроль происхождения
Короткий адрес: https://sciup.org/142133638
IDR: 142133638 | УДК: 636.32/.38:575.113:577.2.08:51-76 | DOI: 10.15389/agrobiology.2015.6.746rus
Текст научной статьи Валидация панели SNP-маркеров для контроля происхождения локальных российских пород овец
Генетическое совершенствование пород сельскохозяйственных животных основано на оценке и отборе индивидуумов, обладающих наибольшей племенной ценностью (estimated breeding value, EBV) по селекционно значимым признакам. Достоверность EBV непосредственно связана с точностью записей родословных, поэтому ошибки в идентификации родителей приводят к снижению генетического прогресса и, как следствие, к экономическим потерям (1). Информация о родословной необходима для контроля степени инбридинга — одного из важных элементов системы управления генетическими ресурсами животных (2).
Микросателлиты были открыты в 1984 году и известны также под названием коротких тандемных повторов (STR, short tandem repeats), или коротких повторов последовательностей (SSR, short sequence repeats) (3). Благодаря высокой степени полиморфизма, менделевскому типу наследования и равномерному распределению по всему геному они более 25 лет оставались наиболее широко используемым типом ДНК-маркеров для контроля происхождения сельскохозяйственных животных разных видов (4-8).
Развитие высокопроизводительных методов анализа генома привело к созданию и использованию панелей на основе полиморфизмов единичных нуклеотидов (single nucleotide polymorphism, SNP) при идентификации происхождения животных (9-13). Хотя SNP-маркеры менее полиморфны по сравнению с STR (большинство SNP биаллельны), этот недостаток полностью нивелируется благодаря возможности проводить одновременный анализ нескольких десятков и даже сотен SNP с относительно небольшими затратами. Преимущества анализа SNP состоят в отсутствии особых требований к качеству ДНК (анализ SNP проводится, как правило, посредством получения коротких ампликонов длиной менее 100 п.н.), автоматизации процесса генотипирования, легкости в интерпретации результатов, возможности прямого сопоставления данных между лабораториями.
Обеспечение высокой достоверности контроля происхождения животных на основе SNP требует использования большего числа локусов по сравнению с STR. В исследованиях, проведенных на человеке (14, 15), свиньях (16), мясном и молочном крупном рогатом скоте (17, 18), было показано, что для достижения сходной информативности на 1 STR требуется 3-6 SNP. При этом авторы указывают, что повышение информативности SNP панелей может достигаться за счет увеличения числа исследуемых полиморфизмов и приближения средней частоты минорных аллелей (minor allele frequency, MAF) к 0,5. По данным K.G. Dodds с соавт. (19), для достижения информативности, аналогичной таковой у широко используемых панелей на основе STR, необходимо в 3-4 раза больше SNP, в то время как в случае панелей на основе доминантных маркеров (например, ISSR и AFLP) последних должно быть в 17 раз больше.
Среди подходов, используемых для определения происхождения индивидуумов на основе ДНК (простое исключение, распределение по категориям и фракциям, реконструкция генотипа) (20, 21), наиболее распространен метод исключения родителей. Он основан на принципе, согласно которому у потомков в каждом локусе должны быть только аллели, имеющиеся у родителей, и вероятность исключения (probability of exclusion, PE) — это вероятность того, что заявленный индивид исключается в качестве родителя (22). Этот метод требует высокой точности генотипирования (> 99 %) и значений MAF ≥ 0,3. Ключевым фактором, определяющим эффективность систем контроля происхождения, служит выбор SNP.
Разработка панелей SNP маркеров для анализа происхождения овец была начата после создания Международным консорциумом по геномике овец (International Sheep Genomics Consortium, ISGС) чипа средней плотности Ovine SNP50K BeadChip, включающего 54241 SNP (23). В настоящее время для овец создано шесть панелей, различающихся набором и числом SNP. Консорциумом ISG предложена панель, включающая 88 SNP (24), содружеством CSIRO (Australia’s Commonwealth Scientific and Industrial Research Organization, Австралия) и Центром SheepCRC (Sheep Cooperative Research Centre, Австралия) — 382 SNP (25), научно-исследовательским институтом AgResearch (Новая Зеландия) — 84 и 300 SNP (12). M.P. Heaton с соавт. (13) разработали панель SNP для оценки глобального разнообразия пород, включающую 163 SNP, и выделили среди них набор из 109 SNP для использования на североамериканских породах овец.
В качестве официальной панели для тестирования происхождения домашних овец Международным обществом генетики животных (ISAG, International Society for Animal Genetics) рекомендованы 88 аутосомных маркеров, предложенные Консорциумом ISG и дополненные одним Y-хромосомным SNP (26). Выбор SNP для включения в панель осуществлялся по результатам тестирования 22 пород овец из Африки, Азии и Европы с использованием технологии Golden Gate (10). Отобранные SNP были оценены на расширенной выборке из 74 пород с использованием технологии Infinium (23). В последующем была показана возможность анализа этой панели с помощью технологий Fluidigm и Sequenom (27).
Следует отметить, что вышеназванные SNP панели контроля происхождения не были протестированы на российских породах овец. Среди 74 пород, используемых для оценки информативности официальной панели ISAG, присутствовали овцы североамериканской популяции российской аборигенной романовской породы. Однако ограниченность выборки и отсутствие ясности в ее происхождении не позволяют говорить о представительности породы в целом.
Целью нашей работы стала оценка информативности панели ISAG из 88 аутосомных маркеров, принятой в качестве официальной панели для тестирования происхождения домашних овец, при контроле происхождения российских локальных пород.
Методика. Исследуемая выборка была представлена четырьмя локальными российскими породами овец — романовской (ROM, n = 22), забайкальской тонкорунной (ZBL, n = 7), аборигенной полугрубошерст-ной бурятской овцой Буубей (BUB, n = 15) и тувинской короткожирнохвостой (TUV, n = 16).
Геномную ДНК выделяли с помощью колонок Nexttec («Nexttec™ Biotechnologie GmbH», Германия) в соответствии с рекомендациями производителя. Скрининг SNP выполняли с использованием чипа Ovine SNP50K BeadChip («Illumina Inc.», CША). Обработку полученных данных проводили как для общего числа прогенотипированных маркеров (54241 SNP), так и для 88 аутосомных SNP, рекомендованных ISAG для подтверждения происхождения домашних овец (далее панель ISAG, или панель происхождения). Пороговым критерием для определения аллеля в качестве информативного было выбрано значение MAF ≥ 0,3.
Контроль качества SNP и статистический анализ полученных данных, в том числе определение MAF, расчет индекса фиксации Fst при парном сравнении по B.S. Weir с соавт. (28) и анализ главных компонент (principal component analysis, PCA) осуществляли в программе Plink 1.07 (29) . Для анализа отбирали SNP, локализованные на аутосомных хромосомах и прошедшие контроль качества по следующим параметрам: по показателю GenCall, указывающему достоверность результатов генотипирования (GC > 0,5), частоте минорных аллелей (MAF > 0,01), тесту на равновесие Харди-Вайнберга (HWE > 1e-6) и уровню генотипирования (GENO > 0,01).
Визуализацию данных, в том числе построение PCA плотов и графиков распределения SNP-маркеров по группам в зависимости от MAF, осуществляли с помощью языка программирования R (30).
Вероятность совпадения генотипов (probability of identity, PI) для каждого локуса рассчитывали, используя формулу (31):
PI = 2 ½ ( ∑ p i 2)2 - ∑ p i 4, где p i — частота i - го аллеля в локусе.
Значения PI для числа несцепленных маркеров k определяли как произведение индивидуальных PI каждого маркера.
Вероятность исключения (probability of exclusion, PE) для каждого локуса определяли для трех частных случаев.
-
1. Исключение одного родителя, если известны генотипы обоих родителей и потомка (P1) (32):
-
2. Исключение родителя, если известны генотипы одного родителя и потомка (P2) (33):
-
3. Исключение обоих родителей, если известны генотипы родителей и потомка (P3) (33):
P1 = 1 - 2 ½ ∑ p i 2+ ∑ p i 3+ 2 ½ ∑ p i 4 - 3 ½ ∑ p i 5 - 2 ½ ( ∑ p i 2)2+ 3 ½ ∑ p i 2½ ∑ p i 3.
P2 = 1 - 4 ½ ∑ p i 2+ 2 ½ ( ∑ p i 2)2+ 4 ½ ∑ p i 3 - 3 ½ ∑ p i 4.
P3 = 1 + 4 ½ ∑ p i 4 - 4 ½ ∑ p i 5 - 3 ½ ∑ p i 6 - 8 ½ ( ∑ p i 2)2+ 8 ½ ( ∑ p i 2) ½ ( ∑ p i 3) + 2 ½ ( ∑ p i 3)2.
Значения P1, P2, P3 (P) для k -го числа несцепленных маркеров определяли по формуле:
P = 1 - (1 - P 1 ) ½ (1 - P 2 ) ½ (1 - P 3 )…½ (1 - P k ).
Для расчетов PI, P1, P2 и P3 использовали программное обеспечение GenAlEx 6.5 (34).
Результаты. Результативность полногеномного SNP генотипирования (call rate) варьировала между породами от 91,5 % (BUB и TUV) до 91,8 % (ROM) и 91,9 % (ZBL) и для всей выборки овец составила 91,7 %. Все исследованные животные прошли контроль на соответствие критерию animal call rate ≥ 90 %. Поскольку исследования предусматривали анализ только аутосомных маркеров, из него были исключены 1828 SNP, локализованных на половых хромосомах. Не прошли контроль по критериям качества чтения (GC Score ≥ 0,5) и степени кластеризации (GC Score ≥ 0,3) 1371, 1469, 1665 и 1636 SNP соответственно у ROM, ZBL, BUB и TUV. Затем у исследованных пород были исключены соответственно 3479, 3663, 3743 и 3786 полиморфизмов как не соответствующие критерию SNP call rate > 90 %. Все оставшиеся маркеры удовлетворяли критерию х 2 на равновесие в популяции по Харди-Вайнбергу, и для дальнейшего анализа были отобраны 47563 (87,7 %) SNP у ROM, 47281 (87,2 %) у ZBL, 47005 (86,7 %) у BUB и 46991 (86,6 %) у TUV. Для анализа всей выборки отобрали 47385 полиморфизмов (87,4 % от числа исследованных).
При оценке результатов генотипирования SNP, включенных в панель ISAG, контроль качества (SNP call rate = 99,2 %) прошли 87 из 88 локусов, за исключением DU426312_454.1, который был успешно ге-нотипирован только у 55 % животных. Однако для того, чтобы иметь данные, сопоставимые с полученными в других исследованиях, этот SNP включили в дальнейший анализ.
В исследованной выборке овец большая часть маркеров происхождения (81,8 % SNP) оказались информативными (MAF ≥ 0,3). Так, 51,1 и 30,7 % SNP встречались в диапазоне частот соответственно от 0,4 до 0,5 и от 0,3 до 0,4. Низкоинформативными были 1,1 % SNP, встречающихся с частотой от 0 до 0,1 %. Распределение всех исследованных SNP по груп- пам в зависимости от MAF было более равномерным: для MAF > 0,0-< 0,1; ≥ 0,1-< 0,2; ≥ 0,2-< 0,3; ≥ 0,3-< 0,4 и ≥ 0,4-≤ 0,5 доли SNP составили соответственно 9,2; 17,8; 21,2; 24,6 и 26,2 %.
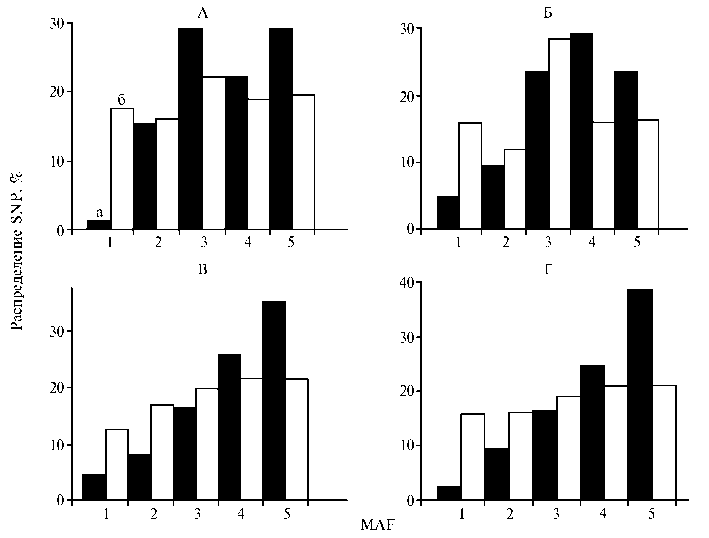
Рис. 1. Распределение исследованных SNP (single nucleotide polymorphism) у овец пород романовская (А) , забайкальская тонкорунная (Б) , Буубей (В) и тувинская короткожирнохвостая (Г) в зависимости от средней частоты минорных аллелей (minor allele frequency, MAF): 1 — > 0,0-< 0,1, 2 — ≥ 0,1-< 0,2, 3 — ≥ 0,2-< 0,3, 4 — ≥ 0,3-< 0,4, 5 — ≥ 0,4- ≤ 0,5; а — 88 SNP, включенных в панель ISAG (International Sheep Genomics Consortium), б — 47385 SNP, отобранных по результатам контроля качества для пород.
Доля информативных SNP различалась между породами и составляла 56,8 % у ROM, 63,4 % — у ZBL, 71,6 % — у BUB и 72,7 % — y TUV (рис. 1). Полиморфизмы с MAF < 0,1 встречались во всех исследованных породах, причем наибольший процент малоинформативных маркеров отмечался у ZBL и BUB (по 4,5 %), а наименьший — у ROM (1,1 %). Этот факт, возможно, объясняется тем, что среди животных, участвовавших в тестировании панели происхождения (13, 26), были 10 овец романовской породы. Однако эти овцы были представлены только североамериканской популяцией, которая не может характеризовать породу в целом. У овец TUV 2,3 % маркеров имели MAF < 0,1, при этом маркер DU196132_525.1 был мономорфным.
Средние значения MAF составили для ROM — 0,332±0,110, для ZBL — 0,335±0,118, для BUB — 0,347±0,121 и для TUV — 0,360±0,109, в целом по выборке — 0,380±0,091. При использовании полногеномной панели средние значения MAF для ROM, ZBL, BUB и TUV равнялись соответственно 0,261±0,146; 0,272±0,146; 0,269±0,143 и 0,272±0,142, для всей выборки — 0,292±0,131.
Из 88 маркеров происхождения для 21 SNP (23,9 %) отмечали высокую информативность во всех четырех породах (MAF ≥ 0,3); 37 (42,0 %), 17 (19,3 %) и 10 SNP (11,4 %) оказались информативными соответственно в трех, двух и только в одной породе. Три SNP были неинформативны для всех четырех пород (табл. 1).
1. SNP (single nucleotide polymorphism) происхождения и среднеарифметические значения MAF (minor allele frequency) для четырех локальных российских пород
|
SNP |
Хромосома |
MAF |
|||||
|
ROM |
ZBL |
BUB |
TUV |
все породы* |
|||
|
SNP-маркеры для четырех |
локальных пород |
о в е ц (MAF < 0,3) |
|||||
|
DU232924 |
365.1 |
1-я |
0,068 |
0,071 |
0,067 |
0,156 |
0,092 |
|
DU501115 |
497.1 |
2-я |
0,182 |
0,143 |
0,200 |
0,094 |
0,158 |
|
DU372582 |
268.1 |
9-я |
0,159 |
0,071 |
0,067 |
0,188 |
0,133 |
|
SNP-маркеры |
для трех локальных |
п о р од ов ец (MAF < 0,3) |
|||||
|
DU305004 |
417.1 |
4-я |
0,250 |
0,125 |
0,393i |
0,156 |
0,242 |
|
DU453259 |
440.1 |
5-я |
0,114 |
0,125 |
0,231 |
0,344i |
0,203 |
|
DU530067 |
219.1 |
7-я |
0,114 |
0,375i |
0,250 |
0,125 |
0,183 |
|
DU213735 |
493.1 |
8-я |
0,318i |
0,250 |
0,286 |
0,250 |
0,417 |
|
DU417675 |
79.1 |
11-я |
0,386i |
0,250 |
0,179 |
0,219 |
0,358 |
|
DU275428 |
276.1 |
13-я |
0,273 |
0,438i |
0,286 |
0,250 |
0,292 |
|
DU223894 |
556.1 |
14-я |
0,364i |
0,250 |
0,286 |
0,281 |
0,408 |
|
DU264531 |
279.1 |
19-я |
0,273 |
0,438i |
0,286 |
0,281 |
0,317 |
|
DU258053 |
237.1 |
19-я |
0,295 |
0,563i |
0,250 |
0,219 |
0,300 |
|
DU380983 |
440.1 |
21-я |
0,250 |
0,188 |
0,036 |
0,406i |
0,233 |
П р и м еч а ни е. ROM — романовская порода, ZBL — забайкальская тонкорунная порода, BUB — Буу-бей, TUV — тувинская короткожирнохвостая порода. Индексом «i» обозначены информативные SNP, ос- тальные SNP— неинформативные.
* Для MAF представлено не среднеарифметическое значение, а величина, рассчитанная при объединении всех четырех пород в один пул.
Сравнительный анализ PCA плотов, построенных по результатам исследований 47385 SNP и 88 SNP (рис. 2), показал довольно четкую дифференциацию пород при применении всего спектра маркеров, в то время как при использовании панели происхождения формировались мало консолидированные частично перекрывающиеся массивы. Анализ значений Fst при парном сравнении как меры генетических различий (табл. 2) ожидаемо подтвердил снижение степени дифференциации при использовании панели происхождения по сравнению с полногеномными SNP-профилями при сохранении характера выявленных генетических связей между породами (r2 = 0,95). Следовательно, полученные данные хоть и подтверждают сохранение породной зависимости SNP панели ISAG, в целом указывают на ее относительно высокую универсальность.
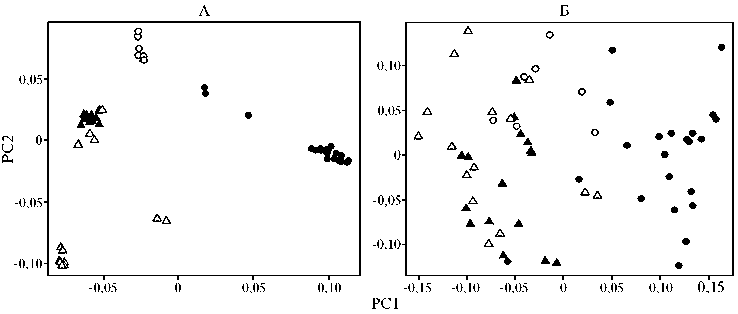
Рис. 2. Анализ главных компонент, выполненный на основе SNP (single nucleotide polymorphism) у овец пород романовская ( • ) , забайкальская тонкорунная ( о ) , Буубей (А) и тувинская короткожирнохвостая ( А) : А — PCA плот по 47385 детектируемым OvineSNP50K BeadChip маркерам, Б — PCA (principal component analysis) плот по 88 маркерам, включенным в панель ISAG (International Sheep Genomics Consortium).
Расчет вероятности совпадения генотипов (PI) по 88 SNP показал высокую информативность панели: для ROM, ZBL, BUB и TUV значения PI составили соответственно 5,86½10-33, 5,24½10-33, 7,48½10-33 и 4,32½10-33.
Вероятность исключения в качестве родителя P1 оказалась ≥ 99,99 % для всех четырех пород. Значение P2 было максимальным для TUV (P2 ≥ 99,99 %), для трех остальных пород — P2 ≥ 99,98 %. Значение P3 для всех пород было ≥ 99,99 %.
2. Индекс фиксации Fst при парном сравнении для SNP панели происхождения и полногеномных SNP-профилей у сравниваемых пород
|
Порода |
1 ROM 1 |
ZBL 1 |
BUB 1 |
TUV |
|
ROM |
0,0544 |
0,0578 |
0,0493 |
|
|
ZBL |
0,0797 |
0,0437 |
0,0418 |
|
|
BUB |
0,0834 |
0,0537 |
0,0261 |
|
|
TUV |
0,0820 |
0,0542 |
0,0318 |
П р и м еч а ни е. Над диагональю представлены значения, рассчитанные на основании данных анализа 88 SNP (single nucleotide polymorphism) маркеров, включенных в панель ISAG (International Sheep Genomics Consortium); под диагональю — значения, рассчитанные по 47385 SNP маркерам, отобранным по результатам контроля качества для пород.
Создание панелей для контроля происхождения овец на основании наиболее высокоинформативных SNP — актуальная проблема овцеводства. Так, M.P. Heaton с соавт. (13) с этой целью использовали 2915 овец 74 породных групп, предоставленных консорциумом ISG, выполнили анализ 47693 аутосомных SNP с помощью множественных критериев и выбрали 163 SNP с желательными свойствами для тестирования происхождения. В среднем каждый из отобранных SNP оказался высокоинформативным (MAF ≥ 0,3) в 48±5 породных группах. Для создания панели контроля происхождения североамериканских пород овец из 163 SNP были выбраны 109. Результативность сканирования и точность для этих 109 SNP составила более 99 %.
Число требующихся SNP зависит от MAF и используемой маркерной панели. Посредством математического моделирования E. Baruch и J.I. Weller (35) установили, что для достижения вероятности исключения, равной 99 %, нужно от 15 до 54 SNP. Число необходимых SNP возрастает при ограничении количества информации (если известен генотип только одного вероятного родителя) и значении MAF < 0,1. При тестировании на российских породах международной панели происхождения, в состав которой входят 88 аутосомных маркеров, доля высокоинформативных SNP в среднем по всей выборке составила 82 %. Необходимо отметить, что такое значение складывается не только из отдельных SNP, высокоинформативных во всех породах, но и за счет маркеров, информативных в двух или трех породах, на что указывают различия для MAF ≥ 0,3 между породами — от 50,0 до 61,4 %.
Согласно J.W. Kijas с соавт. (27), отбор SNP для создания панели происхождения должен быть проведен со смещением в сторону высоких значений MAF (0,3-0,5) с целью ее унификации, то есть обеспечения возможности использовать панель на широком спектре пород. Наши исследования в целом подтвердили это правило и для российских локальных пород: 56,8; 63,4; 71,6 и 72,7 % животных пород ROM, ZBL, BUB и TUV характеризовались значениями MAF, большими или равными пороговому значению 0,3.
Одним из критериев информативности панели служит определение числа SNP, требующихся для получения высокой вероятности исключения родителей. Нами показано, что при наличии данных о генотипах обоих родителей минимальное число маркеров, необходимое для их исключения в качестве родителей с вероятностью более 99,99 % (критерий P3), для каждой из исследованных пород составляло 27. Для достижения аналогичной вероятности исключения одного из родителей при наличии информации об обоих (P1), требовалось не менее 43 SNP. Если генотип одного из родителей был неизвестен (Р2), минимальное необходимое число маркеров возрастало до 66.
Таким образом, основываясь на множестве критериев, в том числе на вероятности совпадения генотипов и вероятности исключения в качестве родителя (родителей), можно сделать вывод, что международная панель ISAG (International Sheep Genomics Consortium), включающая 88 аутосомных маркеров, может быть использована для контроля достоверности происхождения четырех российских локальных пород (романовской, забайкальской тонкорунной, Буубей и тувинской короткожирнохвостой). Для создания наиболее информативной панели с целью оценки происхождения всего спектра пород, разводимых в России, и их роли в обеспечении глобального биоразнообразия домашних овец в дальнейшем следует ориентироваться на увеличение числа исследованных пород, а также на анализ других SNP панелей, предложенных научным сообществом.
Л И Т Е Р А Т У Р А
Sel’skokhozyaistvennaya biologiya [ Agricultural Biology ], 2015, V. 50, ¹ 6, pp. 746-755
VALIDATION OF THE SNP PANEL FOR PARENTAGE ASSIGNMENT IN LOCAL RUSSIAN SHEEP BREEDS
Acknowledgements:
The equipment of Bioresources and Bioengineering Center of L.K. Ernst All-Russian Research Institute of Animal Husbandry was used.
Supported by the Russian Science Foundation, project ¹ 14-36-00039
Список литературы Валидация панели SNP-маркеров для контроля происхождения локальных российских пород овец
- Dodds K.G., Tate M.L., Sise J.A. Genetic evaluation using parentage information from genetic markers. J. Anim. Sci., 2005, 83: 2271-2279.
- Зиновьева Н.А., Харзинова В.Р., Сизарева Е.И., Гладырь Е.А., Костюнина О.В., Луговой С.И., Тапиха В.А., Гамко Л.Н., Овсеенко Е.В., Шавырина К.М., Эрнст Л.К. Оценка вклада различных популяций в генетическое разнообразие свиней корня крупной белой породы. Сельскохозяйственная биология, 2012, 6: 35-42 ( , 10.15389/agrobiology.2012.6.35eng) DOI: 10.15389/agrobiology.2012.6.35rus
- Tautz D., Renz M. Simple sequences are ubiquitous repetitive components of eukaryotic genomes. Nucl. Acids Res., 1984, 12: 4127-4138 ( ) DOI: 10.1093/nar/12.10.4127
- Dakin E.E., Avise J.C. Microsatellite null alleles in parentage analysis. Heredity, 2004, 93: 504-509 ( ) DOI: 10.1038/sj.hdy.6800545
- Зиновьева Н.А., Харзинова В.Р., Логвинова Т.И., Гладырь Е.А., Сизарева Е.И., Чинаров Ю.И. Микросателлитные профили как критерии определения чистопородности и оценки степени гетерогенности подборов родительских пар в свиноводстве. Сельскохозяйственная биология, 2011, 6: 47-53.
- Новгородова И.П., Волкова В.В., Гладырь Е.А., Селионова М.И., Растоваров Е.И., Фисинин В.И., Зиновьева Н.А. Изучение информативности микросателлитов кур G. gallus для характеристики аллелофонда индеек M. gallopavo. Достижения науки и техники АПК, 2011, 10: 66-67.
- Киселева Т.Ю., Подоба Б.Е., Заблудовский Е.Е., Терлецкий В.П., Воробьев Н.И., Кантанен Ю. Анализ 30 микросателлиных маркеров у шести локальных популяций крупного рогатого скота. Сельскохозяйственная биология, 2010, 6: 20-25.
- Хлесткина Е.К. Молекулярные маркеры в генетических исследованиях и селекции. Вавиловский журнал генетики и селекции, 2013, 17(4/2): 1044-1054.
- Werner F.A., Durstewitz G., Habermann F.A., Thaller G., Kramer W., Kollers S., Buitkamp J., Georges M., Brem G., Mosner J., Fries R. Detection and characterization of SNPs useful for identity control and parentage testing in major European dairy breeds. Anim. Genet., 2004, 35: 44-49 ( ) DOI: 10.1046/j.1365-2052.2003.01071.x
- Kijas J.W., Townley D., Dalrymple B.P., Heaton M.P., Maddox J.F., McGrath A., Wilson P., Ingersoll R.G., McCulloch R., McWilliam S., Tang D., McEwan J., Cockett N., Oddy V.H., Nicholas F.W., Raadsma H. A genome wide survey of SNP variation reveals the genetic structure of sheep breeds. PLoS ONE, 2009, 4: e4668 ( ) DOI: 10.1371/journal.pone.0004668
- Hayes B.J. Technical note: Efficient parentage assignment and pedigree reconstruction with dense single nucleotide polymorphism data. J. Dairy Sci., 2011, 94: 2114-2117 ( ) DOI: 10.3168/jds.2010-3896
- Clarke S.M., Henry H.M., Dodds K.G., Jowett T.W.D., Manley T.R., Anderson R.M., McEwan J.C. A high throughput single nucleotide polymorphism multiplex assay for parentage assignment in New Zealand sheep. PLoS ONE, 2014, 9(4): e93392 ( ) DOI: 10.1371/journal.pone.0093392
- Heaton M.P., Leymaster K.A., Kalbfleisch T.S., Kijas J.W., Clarke S.M., McEwan J., Maddox J.F., Basnayake V., Petrik D.T., Simpson B., Smith T.P.L., Chitko-McKown C.G., the International Sheep Genomics Consortium. SNPs for parentage testing and traceability in globally diverse breeds of sheep. PLoS ONE, 2014, 9(4): e94851 ( ) DOI: 10.1371/journal.pone.0094851
- Gill P. An assessment of the utility of single nucleotide polymorphisms (SNPs) for forensic purposes. Int. J. Legal Med., 2001, 114: 204-210 ( ) DOI: 10.1007/s004149900117
- Li L., Li C.-T., Li R.-Y., Liu Y., Lin Y. SNP genotyping by multiplex amplification and microarrays assay for forensic application. Forensic Sci. Int., 2006, 162: 74-79 ( ) DOI: 10.1016/j.forsciint.2006.06.010
- Rohrer G.A., Freking B.A., Nonneman D. Single nucleotide polymorphisms for pig identification and parentage exclusion. Anim. Genet., 2007, 38(3): 253-258 ( ) DOI: 10.1111/j.1365-2052.2007.01593.x
- Heaton M.P., Harhay G.P., Bennett G.L., Stone R.T., Grosse W.M., Casas E., Keele J.W., Smith T.P.L., Chitko-McKown C.G., Laegreid W.W. Selection and use of SNP markers for animal identification and paternity analysis in U.S. beef cattle. Mammalian Genome, 2002, 13: 272-281 ( ) DOI: 10.1007/s00335-001-2146-3
- Fisher P.J., Malthus B., Walker M.C., Corbett G., Spelman R.J. The number of single nucleotide polymorphisms and on-farm data required for whole-herd parentage testing in dairy cattle herds. J. Dairy Sci., 2009, 92: 369-374 ( ) DOI: 10.3168/jds.2008-1086
- Dodds K.G. The number of markers required for parentage assignment. Proc. Association for the Advancement of Animal Breeding and Genetic. Melbourne, Victoria, Australia, 2003, 15: 39-42.
- Jones A.G., Small C.M., Paczolt K.A., Ratterman N.L. A practical guide to methods of parentage analysis. Molecular Ecology Resources, 2010, 10: 6-30 ( ) DOI: 10.1111/j.1755-0998.2009.02778.x
- Jones A.G., Ardren W.R. Methods of parentage analysis in natural populations. Mol. Ecol., 2003, 12: 2511-2523 ( ) DOI: 10.1046/j.1365-294x.2003.01928.x
- Chakraborty R., Stivers D.N. Paternity exclusion by DNA markers: effects of paternal mutations. J. Forensic Sci., 1996, 41: 671-677 ( ) DOI: 10.1520/JFS13976J
- Kijas J.W., Lenstra J.A., Hayes B., Boitard S., Porto Neto L.R., San Cristobal M., Servin B., McCulloch R., Whan V., Gietzen K., Paiva S., Barendse W., Ciani E., Raadsma H., McEwan J., Dalrymple B. Genome wide analysis of the world’s sheep breeds reveals high levels of historic mixture and strong recent selection. PLoS ONE, 2012, 10: e1001258 ( ) DOI: 10.1371/journal.pone.0094851
- Consortium ISG (2013) ISGC SNP Loci for Parentage Assignment (http://www.sheephap-map.org/news/parentage_page.php, http://sheephapmap.org/news/89_CorePanel.pdf).
- Bell A., Henshall J., Gill S., Gore K., Kijas J. Success rates of commercial SNP based parentage assignment in sheep. Proc. 20th Biennial Conference of the Association for the Advancement of Animal Breeding and Genetics 2013/N.L. Villalobos (ed.). Napier, New Zealand, 2013: 278-282.
- ISAG (2012) (http://www.isag.us/Docs/AppGenSheepGoat2012.pdf).
- Kijas J.W., McEwan J., Clarke S., Henry H., Maddox J., McCulloch R., Driver F., Ilic K., Heaton M. Development of a SNP Panel for Parentage Assignment in Sheep. International Sheep Genomics Consortium, 2012 (http://sheep-hapmap.org/news/PAG_2012_ParentagePoster.pdf).
- Weir B.S., Cockerham C.C. Estimating F-statistics for the analysis of population structure. Evolution, 1984, 38: 1358-1370.
- Purcell S., Neale B., Todd-Brown K., Thomas L., Ferreira M.A.R., Bender D., Maller J., Sklar P., de Bakker P.I.W., Daly M.J., Sham P.C. PLINK: a tool set for whole-genome association and population-based linkage analyses. Am. J. Hum. Genet., 2007, 81: 559-575 ( ) DOI: 10.1086/519795
- R Development Core Team. R: A language and environment for statistical computing. 2009. R Foundation for statistical computing. Vienna, Austria, 2005 (ISBN 3-900051-07-0) (http://www.Rproject.org).
- Waits L.P., Luikart G., Taberlet P. Estimating the probability of identity among genotypes in natural populations: cautions and guidelines. Mol. Ecol., 2001, 10: 249-256 ( ) DOI: 10.1046/j.1365-294X.2001.01185.x
- Jamieson A. The effectiveness of using co-dominant polymorphic allelic series for (1) checking pedigrees and (2) distinguishing full-sib pair members (in Festschrift in Honour of Dr Clyde J. Stormont). Anim. Genet., 1994, 25(Suppl. 1): 37-44.
- Jamieson A., Taylor S.C.S. Comparisons of three probability formulae for parentage exclusion. Anim. Genet., 1997, 28: 397-400 ( ) DOI: 10.1111/j.1365-2052.1997.00186.x
- Peakall R., Smouse P.E. GenAlEx 6.5: genetic analysis in Excel. Population genetic software for teaching and research -an update. Bioinformatics, 2012, 28: 2537-2539 ( ) DOI: 10.1093/bioinformatics/bts460
- Baruch E., Weller J.I. Estimation of the number of SNP genetic markers required for parentage verification. Anim. Genet., 2008, 39: 474-479 ( ) DOI: 10.1111/j.1365-2052.2008.01754.x


