Вариабельность фрагмента гена pho1a у дикорастущих клубнеобразующих и неклубнеобразующих видов (Solanum subgenus Potatoe) и сортов картофеля S. tuberosum L
Автор: Шмелькова Е.О., Слугина М.А., Мелешин А.А., Кочиева Е.З.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетические основы селекции
Статья в выпуске: 5 т.53, 2018 года.
Бесплатный доступ
В клубнях картофеля крахмал - основной метаболит, поэтому структурно-функцио-нальный анализ генов его биосинтеза и распада и оценка их вариабельности имеет как фундаментальное, так и прикладное значение. Конечное содержание крахмала в гетеротрофных органах растений (плодах, семенах, клубнях) зависит не только от скорости биосинтеза составляющих крахмала - амилозы и амилопектина, но и от работы катаболических ферментов. Ферменты биосинтеза крахмала достаточно хорошо описаны, в то время как о механизмах разрушения крахмала мало что известно. На сегодняшний день накапливается все больше данных о важной роли крахмалфосфорилаз в этом процессе. Фосфорилазы крахмала широко распространены среди представителей растительного мира. Структура и полиморфизм гена крахмалфосфорилазы слабо изучены у двудольных растений. В клубнях картофеля разрушение крахмала осуществляется за счет L-формы крахмалфосфорилазы, которую кодирует ген Pho1a ( STP23 ). В настоящей работе у растений картофеля впервые проведен анализ участка гена Pho1a (экзоны II-IV), кодирующего регуляторную часть функционального гликозилтрансферазного домена, в состав которого входят глюкозо-6-фосфат-связывающий сайт, сайт связывания с пиродоксальфосфатом и активный сайт связывания с глюкозой...
Крахмалфосфорилаза, нуклеотидная и аминокислотная вариабельность, дикорастущие виды, сорта и линии картофеля, подрод potatoe
Короткий адрес: https://sciup.org/142216597
IDR: 142216597 | УДК: 633.49:631.522/.524:577.21 | DOI: 10.15389/agrobiology.2018.5.907rus
Текст научной статьи Вариабельность фрагмента гена pho1a у дикорастущих клубнеобразующих и неклубнеобразующих видов (Solanum subgenus Potatoe) и сортов картофеля S. tuberosum L
Основным резервным источником углерода и энергии в клетках растений можно считать крахмал. Конечное содержание крахмала в гетеротрофных органах (плодах, семенах, и клубнях) зависит не только от скорости биосинтеза составляющих крахмала — амилозы и амилопектина, но и от работы катаболических ферментов. Ферменты биосинтеза крахма-
Работа выполнена в рамках гранта РФФИ ¹ 17-29-08017 и КПНИ «Развитие селекции и семеноводства картофеля».
ла достаточно хорошо описаны, в то время как разрушение крахмала — процесс мало изученный (1). Подробнее всего катаболизм описан в листьях, есть данные для эндоспермов злаковых, меньше всего сведений получено для клубней картофеля (2).
В целом ферменты, разрушающие крахмал, можно подразделить на две категории: гидролитические (амилазы EC 3.2.1.1, EC 3.2.1.2, 4-α-глю-канотрансфераза EC 2.4.1.25, мальтаза EC 3.2.1.20, изоамилазы EC 3.2.1.68) и фосфоролитические (α-гликанфосфорилаза EC 2.4.1.1, мальтозофосфо-рилаза EC 2.4.1.8, α-глюкан-Н2О-дикиназа EC 2.7.9.4, фосфоглюкан-Н2О-дикиназа EC 2.7.9.5) (3-5). Их сравнительная активность может изменяться в зависимости от стадии развития растения или условий среды. Какая из групп ферментов имеет большее значение, спорный вопрос. Предполагается, что процесс инициируется за счет фосфорилирования гликанов, что делает поверхность крахмального зерна более гидрофильной и доступной для гидролитических ферментов, дополнительно создавая селективные белок-углеводные и белок-белковые взаимодействия (5-8).
Среди фосфоролитических ферментов наибольшего внимания заслуживают крахмалфосфорилазы — растительные аналоги α-гликанфос-форилаз (EC 2.4.1.1) (9). Основополагающая роль крахмалфосфорилазы заключается в катализе распада крахмала за счет замещения углерода на фосфор в гликозидной связи с образованием глюкозо-1-монофосфата (10, 11). Однако в случае недостатка фосфата крахмалфосфорилазы могут осуществлять обратную реакцию синтеза крахмала (12).
Крахмалфосфорилазы широко распространены среди представителей растительного мира (13). Наличие множества изоформ — характерная черта крахмалфосфорилаз, которые различаются кинетическими свойствами и локализацией в клетке (13). Большинство высших растений с известными геномами и транскриптомами имеют два типа крахмалфосфорилаз — пластидную (Pho1/L-form/L-SP) и цитозольную (Pho2/H-form/r H-SP) (14). Пластидный белок размером около 105 кДа имеет низкую аффинность к разветвленным гликанам, в то время как цитозольная форма размером около 90 кДа характеризуется высокой аффинностью к линейным и разветвленным гликанам и даже к гетерогликанам (15-17). Впервые различные формы растительных крахмалфосфорилаз были обнаружены в клубнях картофеля ( Solanum tuberosum L.) и семенах гороха ( Pisum sativum L.) (18, 19). Структура и полиморфизм генов крахмалфосфорилаз достаточно широко изучены у представителей класса Однодольные: ячменя ( Hordeum vulgare L.) (20), риса ( Oryza sp.) (21), кукурузы ( Zea mays L.) (6, 22, 23). При этом аналогичных данных для двудольных крайне мало: известны полноразмерная последовательность гена батата (24) и кДНК четырех сортов картофеля (25).
В клубнях картофеля крахмал — основной метаболит, поэтому структурно-функциональный анализ генов крахмалфосфорилаз и оценка их вариабельности имеет не только фундаментальное, но и прикладное значение, поскольку сможет пролить свет на функции этих белков, а также использоваться в селекции новых сортов с повышенным содержанием крахмала, устойчивых к холодовому осахариванию.
В клубнях картофеля разрушение крахмала осуществляется за счет L-формы крахмалфосфорилазы, которая кодируется геном Pho1a ( STP23 ). Полноразмерная последовательность этого гена у картофеля на сегодняшний день недоступна. В GenBank NCBI размещена лишь соответствующая мРНК S. tuberosum (NM 001288286.1), однако известно, что ген находится на 3-й хромосоме (26), имеет длину 16.4 т.п.н. и состоит из 15 экзонов и
14 интронов (27).
В настоящей работе впервые проанализирован аллельный полиморфизм участка гена Pho1a со II по IV экзон на широкой выборке образцов, включающих как дикорастущие виды картофеля, так и сорта и линии S. tuberosum. Это позволило выявить 11 новых аллельных вариантов, которые могут быть использованы в дальнейшем в селекционных программах.
Целью представленного исследования стала оценка вариабельности гена крахмалфосфорилазы Pho1a в области II-IV экзонов у клубнеоразую-щих и неклубнеобразующих видов картофеля ( Solanum , подрод Potatoe ), а также у отечественных и зарубежных сортов и линий культивируемого вида S. tuberosum.
Методика . Для изучения аллельного полиморфизма отобрали 96 образцов картофеля, включая 15 дикорастущих видов подсекций Potatoe и Estolonifera , один из которых — неклубнеобразующий ( S. etuberosum ), а также 67 сортов и 14 линий культивируемого картофеля S. tuberosum. Для дополнительной оценки полиморфизма были взяты последовательности экзонов риса Oryza sativa (AK063766.1), кукурузы Zea mays (NM 001309854.1), ячменя Hordeum vulgare (JQ277327.1) и батата Ipomoea batatas (L.) Lam. (M64362.1), доступные в базе данных GenBank NCBI. Семена дикорастущих видов получали из коллекций Всероссийского института генетических ресурсов растений им. Н.И. Вавилова (г. Санкт-Петербург, Россия) и CGN (Centre for Genetic Resources, Вагенинген, Нидерланды). Сорта и линии предоставлены Всероссийским НИИ картофельного хозяйства им. А.Г. Лорха (Московская обл., Россия).
Ядерную ДНК выделяли из молодых листьев калий-ацетатным методом с дополнительной очисткой смесью фенола с хлороформом (28). Для амплификации изучаемого фрагмента была разработана праймерная комбинация Pho2F (5´-CTGAACATGAAGCAAGCGTA-3´) — Pho4R (5´-GGCTATGGACTTAGGTACA-3´). Реакционная смесь для проведения ПЦР содержала 1½ буфер (50 мM Tris-HCl, pH 8,0, 50 мM КCl, 1,5 М MgCl2, 20 мМ dNTPs), по 10 мкМ соответствующего праймера, 0,25 ед. Taq ДНК-полимеразы («Диалат Лтд», Россия) и 100 нг геномной ДНК картофеля. Температурно-временной профиль ПЦР был следующим: первый цикл — 5 мин при 95 °С; 30 с при 94 °С, 40 с при 55 °С, 1 мин при 72 °С (35 циклов); финальная элонгация — 1 мин при 72 °С. ПЦР проводили с использованием реактивов фирмы «Диалат Лтд» (Россия) в амплификаторе BioRad С1000 («Bio-Rad Laboratories, Inc.», США).
Продукты ПЦР визуализировали при помощи электрофореза в 1 % агарозном геле LE 2 Аgarose («Хеликон», Россия) в 1½ ТВЕ-буфере, окрашенном бромистым этидием, и документировали в системе BioDocII («Biometra GmbH», Германия). Продукты амплификации секвенировали с использованием тех же праймеров в ЦКП «Биоинженерия» на платформе Applied Biosystems 3730 DNA Analyzer («Applied Biosystems», США).
Выравнивание и анализ полиморфизма нуклеотидных и аминокислотных последовательностей осуществляли в программе MEGA 7.0 (29). В качестве референсной использовали последовательность мРНК S. tuberosum из базы данных GenBank NCBI (NM 001288286.1). Функциональный эффект аминокислотных замен рассчитывали в программе Provean .
Результаты. Для анализа полиморфизма гена Pho1a был выбран фрагмент со II по IV экзон, кодирующий N-концевую регуляторную часть функционального гликозилтрансферазного домена (ID 10136827), в состав которого входят глюкозо-6-фосфат-связывающий сайт, сайт связывания с пиродоксальфосфатом и активный сайт связывания с глюкозой (20). Подбор сайтов отжига праймеров основывался на возможности амплификации Pho1a у филогенетически отдаленных видов, а также на способности дискриминировать целевую изоформу гена от паралогов Pho1b и Pho2. Фрагмент гена был амплифицирован и секвенирован с помощью разработанной праймерной пары Pho2F-Pho4R.
У всех сортов и селекционных линий S. tuberosum последовательности имели одинаковую протяженность — 670 п.н. Длина полученных последовательностей Pho1a у видов составляла от 666 п.н. ( S. vernei , S. lig-nicaule ) до 672 п.н. ( S. pinnatisectum ). Длины последовательностей варьировали за счет наличия инсерций и делеций, локализованных исключительно в интронах.
Всего в составе исследуемых последовательностей у 96 образцов картофеля выявили 59 точечных нуклеотидных замен (SNPs), из которых 58 SNPs обнаружили в последовательностях у дикорастущих видов. Общая степень полиморфизма для всей выборки составила 8,75 %, а для набора видов клубнеобразующего и неклубнеобразующего картофеля — 8,6 %. У анализируемых сортов культивируемого картофеля S. tuberosum было найдено только два полиморфных сайта (0,29 %), у селекционных линий замены полностью отсутствовали (табл. 1). Подавляющее большинство замен оказались локализованы в интронных последовательностях. Вариабельность исследуемых экзонных последовательностей составила 5,95 % за счет наличия 15 SNPs, 14 из которых были найдены у видов и только одна замена — у сортов S. tuberosum.
1. Число и доля вариабельных сайтов в составе исследуемых последовательностей Pho1a (II-IV экзоны) у дикорастущих видов, а также сортов и линий Solanum tuberosum
|
Анализируемая последовательность |
Дикорастущие виды и сорт Приор |
Cорта |
Селекционные линии |
Все образцы |
|
Полноразмерный ген |
58 (8,60 %) |
2 (0,29 %) |
0 |
59 (8,75 %) |
|
кДНК |
14 (5,55 %) |
1 (0,39 %) |
0 |
15 (5,95 %) |
|
Белок |
3 (3,57%) |
1 (1,19 %) |
0 |
3 (3,57 %) |
Основываясь на наличии вышеназванных замен в экзонных последовательностях, можно выделить 11 аллельных вариантов (табл. 2, представлена online на сайте журнала . Девять из них характерны для дикорастущих видов, причем семь уникальны для исследуемой выборки. Аллельный вариант Pho1a_A1 был характерен только для неклубнеобразующего картофеля S. etuberosum, однако, несмотря на его эволюционную отдаленность от клубнеобразующих видов (30), различия касались только одной SNP. Также не было выявлено множественных SNPs, отличающих представителей видов надсерии Rotata от эволюционно более древних видов надсерии Stellata (см. табл. 2 на сайте журнала . Более того, у некоторых видов Stellata (S. poly-adenium, S. chacoense, S. lignicaule) обнаружили тот же аллельный вариант Pho1a_A2, что и у большинства анализируемых сортов S. tuberosum.
У культивируемого картофеля S. tuberosum анализируемые последовательности были представлены двумя аллельными вариантами. Аллельный вариант Pho1a_A10 обнаружили у девяти сортов российской и зарубежной селекции (Bintje, Red Scarlett, Ушконыр, Карасайский, Аврора, Алладин/Aladin, Чернский, Пламя, Удача). Сорта отличались от диких видов наличием нуклеотидной замены G150A (индекс соответствует номеру в последовательности кДНК).
Все 14 селекционных линий картофеля были мономорфными по фрагменту Pho1a и имели одну общую с большинством сортов аллель Pho1a_A2, которую также обнаружили у ряда дикорастущих видов (см. табл. 2). Одинаковый аллельный вариант Pho1a_A2 у дикорастущих видов и большинства сортов можно, скорее всего, объяснить использованием образцов дикорастущих видов в скрещиваниях. Так, сорта Чародей, Холмогорский, Сударыня, Очарование, Сиреневый туман — гибриды, полученные с участием S. demissum, S. vernei, S. stoloniferum.
Попытка связать присутствие определенного аллельного варианта с качественными характеристиками клубней (содержание крахмала, холодостойкость, холодовое осахаривание) не увенчалось успехом.
Провести сравнительный анализ полиморфизма этого участка гена крахмалфосфорилазы у картофеля и других растений достаточно тяжело, так как последовательность Pho1a изучена у небольшого числа видов однодольных (6, 20-23), а для двудольных такие данные крайне ограничены (24). Ранее был изучен полиморфизм исследованного участка у дикорастущих и культивируемых видов томата ( Solanum , секция Lycopersicon ). Всего в составе таких последовательностей у 13 образцов 11 видов томата детектировано 24 SNPs, из них 7 в последовательностях экзонов, 17 — в последовательностях интронов, общий полиморфизм составил 3,6 % (М.А. Слугина, неопубликованные данные). Интересно, что в последовательностях интронов у трех видов дикорастущего картофеля ( S. limbaniense , S. bulbocastanum , S. pinnatisectum ) найдены инделы, общие со всеми видами томата, что еще раз указывает на филогенетическое родство томата и картофеля.
Сравнение фрагмента Pho1a (II-IV экзоны) у изучаемых образцов картофеля с последовательностями у других видов, взятыми из базы данных NCBI, показало достаточно консервативную структуру этого участка гена у анализируемых однодольных и двудольных видов растений. При этом гомология первичных экзонных последовательностей была достаточно высокой, а интронных — очень низкой. На исследуемом участке локализация интронов совпадала, но их длины различались. Проведенный анализ позволил выявить 87 SNPs (33, 98 %) (рис., А, размещен на сайте журнала . Наибольшая гомология (89,45 %) наблюдалась у картофеля с бататом. Самыми отдаленными оказались последовательности фрагмента Pho1a картофеля и кукурузы (23,05 %).
А
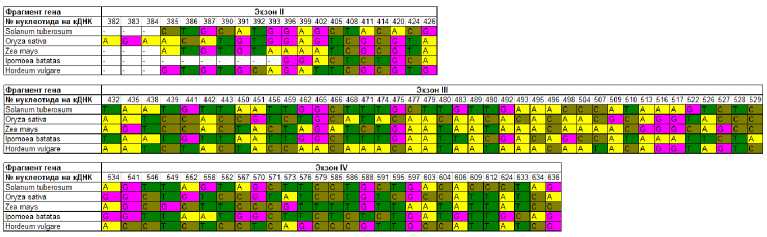
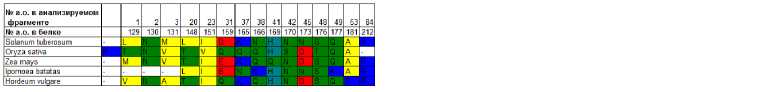
SNPs в экзонных последовательностях фрагмента гена Pho1a (II-IV экзоны) у видов Solanum tuberosum L. (NM 001288286.1), Oryza sativa L. (AK063766.1), Zea mays L. (NM 001309854.1), Ipomoea batatas (L.) Lam. (M64362.1), Hordeum vulgare L. (JQ277327.1) (А) и замещения аминокислот в анализируемом участке белка (Б) .
Белок Pho1 имеет размер 966 а.о. и состоит из четырех участков: N-концевого транзитного пептида, который осуществляет доставку белка в пластиды, регуляторного домена, центральной инсерции L78 (отличает эту изоформу от Pho2) и каталитического домена. Вместе регуляторный домен L78 и каталитический домен формируют функциональный гликозилтрансферазный домен, функция которого заключается в переносе фрагментов сахара с активированной молекулы донора на молекулу акцептора с последующим образованием гликозидной связи (20, 25). При выравнивании последовательностей известных крахмалфосфорилаз одно- и двудольных растений показано, что регуляторный и каталитический домены наиболее консервативны (20).
Кодирующие последовательности исследованного фрагмента (экзон II—экзон IV) гена крахмалфосфорилазы Pho1a картофеля были транслированы, что позволило оценить их аминокислотную вариабельность. Последовательность, доступная для анализа, составила 84 а.о. (позиции со 129-й до 212-й а.о.), входила в состав регуляторного домена и включала глю-козо-6-фосфат- и пиродоксальфосфат-связывающие сайты, а также активный сайт связывания с глюкозой (20).
Только четыре из 15 экзонных SNPs в анализируемом фрагменте приводили к заменам аминокислотных остатков. При этом A/T31 и G/T33 входили в состав одного кодона и приводили к замещению по одной и той же аминокислотной позиции: в положении 139 метионин был замещен на изолейцин (M139I) или на лейцин (M139L). Замещение M139I было характерно для группы сортов российской и зарубежной селекции. Среди них Ушконыр, Карасайский, Аврора, Bintje, Чернский, Red Scarlett, Aladin, Пламя, Удача. В остальных исследованных сортах и селекционных линиях аминокислотный полиморфизм не обнаружили. Интересно, что для клонированных и секвенированных ранее полноразмерных кДНК сортов картофеля Diana, Theresa, Saturna и Satina аминокислотный полиморфизм на участке со II по IV экзон не выявлен (25). То есть наличие вариантов последовательности крахмалфосфорилазы показано на более широкой выборке сортов и линий S. tuberosum , представленной в настоящей статье.
Значительную вариабельность фрагмента Pho1a выявили у дикорастущих видов. Было детектировано замещение M139L, характерное для дикорастущего клубнеобразующего картофеля S. circaefolium. Уникальную для выборки аминокислотную замену T157S обнаружили у образца S. ver-nei. Однако, согласно расчетам в программе Provean (31), обе замены носят нейтральный характер и не приводят к изменению заряда белка. Замена R212S у неклубнеобразующего картофеля S. etuberosum , в отличие от вышеописанных, является радикальной, что может привести к изменению структуры белка Pho1a, а значит, повлиять на его функциональность.
Выравнивание полученных аминокислотных последовательностей с транслированными последовательностями (II-IV экзоны) у риса, кукурузы, ячменя и батата выявило 14 аминокислотных замен, среди которых три были радикальными (см. рис., Б, .
В целом крайне низкая аминокислотная вариабельность анализируемого регуляторного домена Pho1a у дикорастущих видов и сортов картофеля согласуется с данными, полученными для крахмалфосфорилаз других видов растений (20), что свидетельствует о важной роли этого домена для связывания с субстратами и кофакторами и тем самым объясняет сохранение его консервативности в ходе эволюции. Выявленные в регуляторном домене замещения аминокислот потенциально могут влиять на активность фермента. В дальнейшем необходимо продолжить поиск ассоциаций детек-912
тированных замен с содержанием крахмала в клубнях, учитывая перспективность применения таких ассоциаций в селекционных программах.
Таким образом, мы провели анализ участка гена Pho1a со II по IV экзон у 15 дикорастущих видов, 67 образцов культивируемого картофеля и 14 селекционных линий и описали его нуклеотидный и аминокислотный полиморфизм. Несмотря на консервативность исследуемых экзонов, было описано 59 нуклеотидных замен на указанном фрагменте, из которых 15 локализовались в экзонах, а три приводили к аминокислотному замещению. Уникальная замена SNP636, приводящая к замещению аргинина на серин R/S, обнаруженная у неклубнеобразующего картофеля S. etuberosum является радикальной и потенциально может вызывать изменение конформации белка. Было детектировано 11 аллельных вариантов, и дальнейший поиск их ассоциированных с содержанием крахмала в клубнях картофеля может найти применение в селекционных программах. Влияние выявленных аминокислотных замен в функционально-значимом районе и их потенциального влияния на активность фермента крахмалфосфори-лазы требует дополнительного изучения.
Список литературы Вариабельность фрагмента гена pho1a у дикорастущих клубнеобразующих и неклубнеобразующих видов (Solanum subgenus Potatoe) и сортов картофеля S. tuberosum L
- Smith A., Zeeman S., Smith S. Starch degradation. Annu. Rev. Plant Biol., 2005, 56: 73-98 (doi: 10.1146/annurev.arplant.56.032604.144257).
- Zeeman S., Kossmann J., Smith A. Starch: its metabolism, evolution, and biotechnological modification in plants. Annu. Rev. Plant Biol., 2010, 61: 209-234 ( ) DOI: 10.1146/annurev-arplant-042809-112301
- Kossmann J., Lloyd J. Understanding and influencing starch biochemistry. Crit. Rev. Plant Sci., 2000, 19: 171-226 ( ) DOI: 10.1146/annurev-arplant-042809-112301
- Blennow A., Nielsen T., Baunsgaard L., Mikkelsen R., Engelsen S. Starch phosphorylation: a new front line in starch research. Trends Plant Sci., 2002, 7(10): 445-450 ( ) DOI: 10.1016/S1360-1385(02)02332-4
- Fettke J., Eckermann N., Kötting O., Ritte G., Steup M. Novel starch-related enzymes and carbohydrates. Cell. Mol. Biol., 2007, 52: OL883-OL904.
- Yu Y., Mu H., Wasserman B., Carman G. Identification of the maize amyloplast stromal. 112-kD protein as a plastidic starch phosphorylase. Plant Physiol., 2001, 125: 351-359 ( ) DOI: 10.1104/pp.125.1.351
- Hejazi M., Fettke J., Haebel S., Edner C., Paris O., Frohberg C., Steup M., Ritte G. Glucan, water dikinase phosphorylates crystalline maltodextrins and thereby initiates solubilization. Plant J., 2008, 55(2): 323-333 ( ) DOI: 10.1111/j.1365-313X.2008.03513.x
- Orzechowski S. Starch metabolism in leaves. Acta Biochim. Pol., 2008, 55: 435-445.
- Breton C., Šnajdrová L., Jeanneau C., Koča J., Imberty A. Structures and mechanisms of glycosyltransferases. Glycobiology, 2006, 16(2): 29R-37R ( ) DOI: 10.1093/glycob/cwj016
- Schneider E., Becker J., Volkmann D. Biochemical properties of potato phosphorylase change with its intracellular localization as revealed by immunological methods. Planta, 1981, 151(2): 124-134 ( ) DOI: 10.1007/BF00387813
- Kamrani M., Baghban Kohnehrouz B., Gholizadeh A. Effect of RNAi-mediated gene silencing of starch phosphorylase L and starch-associated R1 on cold-induced sweetening in potato. Journal of Horticultural Science and Biotechnology, 2016, 91(6): 625-633 ( ) DOI: 10.1080/14620316.2016.1208544
- Cuesta-Seijo J.A., Ruzanski C., Krucewicz K., Meier S., Hägglund P., Svensson B., Palcic M.M. Functional and structural characterization of plastidic starch phosphorylase during barley endosperm development. PLoS ONE, 2017, 12(4): e0175488 ( ) DOI: 10.1371/journal.pone.0175488
- Nighojkar S., Kumar A. Starch phosphorylase: biochemical, molecular and biotechnological aspects. Genet. Eng. Biotechnol., 1997, 17: 189-202.
- Nakano K., Fukui T. The complete amino acid sequence of potato alpha-glucan phosphorylase. J. Biol. Chem., 1986, 261: 8230-8236.
- Fettke J., Eckermann N., Poeste S., Pauly M., Steup M. The glucan substrate of the cytosolic (Pho2) phosphorylase isozyme from Pisum sativum L.: identification, linkage and subcellular localization. Plant J., 2004, 39(6): 933-946 ( ) DOI: 10.1111/j.1365-313X.2004.02181.x
- Fettke J., Eckermann N., Tiessen A., Geigenberger P., Steup M. Identification, subcellular localization and biochemical characterization of water-soluble heteroglycans (SHG) in leaves of Arabidopsis thaliana L.: distinct SHG reside in the cytosol and in the apoplast. Plant J., 2005, 43(4): 568-586 ( ) DOI: 10.1111/j.1365-313X.2005.02475.x
- Fettke J., Poeste S., Eckermann N., Tiessen A., Pauly M., Geigenberger P., Steup M. Analysis of cytosolic heteroglycans from leaves of transgenic potato (Solanum tuberosum L.) plants that under-or overexpress the Pho 2 phosphorylase isozyme. Plant Cell Physiol., 2005, 46(12): 1987-2004 ( ) DOI: 10.1093/pcp/pci214
- Hanes C. The breakdown and synthesis of starch by an enzyme system from pea seeds. Proc. Roy Soc. London Ser. B, 1940, 128: 421-450.
- Hanes C. The reversible formation of starch from glucose-1-phosphate catalysed by potato phosphorylase. Proc. Roy Soc. London Ser. B, 1940, 129: 174-208.
- Ma J., Jiang Q.T., Zhang X.W., Lan X.J., Pu Z.E., Wei Y.M., Liu C., Lu Z.-X. Structure and expression of barley starch phosphorylase genes. Planta, 2013, 238(6): 1081-1093 ( ) DOI: 10.1007/s00425-013-1953-6
- Satoh H., Shibahara K., Tokunaga T., Nishi A., Tasaki M., Hwang S.-K., Okita T., Kaneko N., Fujita N., Yoshida M., Hosaka Y., Sato A., Utsumi Y., Ohdan T., Nakamura Y. Mutation of the plastidial a-glucan phosphorylase gene in rice affects the synthesis and structure of starch in the endosperm. Plant Cell, 2008, 20(7): 1833-1849 ( ) DOI: 10.1105/tpc.107.054007
- Mu H.H., Yu Y., Wasserman B.P., Carman G.M. Purification and characterization of the maize amyloplast stromal 112-kDa starch phosphorylase. Arch. Biochem. Biophys., 2001, 388(1): 155-164 ( ) DOI: 10.1006/abbi.2000.2267
- Subasinghe R.M. Role and regulation of starch phosphorylase and starch synthase IV in starch biosynthesis in maize endosperm amyloplasts. PhD Thesis. The University of Guelph, Canada, 2013.
- Lin Y.C., Chang S.C., Juang R.H. Plastidial a-glucan phosphorylase 1 complexes with disproportionating enzyme 1 in Ipomoea batatas storage roots for elevating malto-oligosaccharide metabolism. PLoS ONE, 2017, 12(5): e0177115 ( ) DOI: 10.1371/journal.pone.0177115
- Nader Nieto C. Structural and functional characterization of natural alleles of potato (Solanum tuberosum L.) starch phosphorylases associated with tuber quality traits. University of Cologne, Cologne, 2011.
- Chen X., Salamini F., Gebhardt C. A potato molecular-function map for carbohydrate metabolism and transport. Theor. Appl. Genet., 2001, 102(2-3): 284-295 ( ) DOI: 10.1007/s001220051645
- Schreiber L., Nader-Nieto A.C., Schönhals E.M., Walkemeier B., Gebhardt C. SNPs in genes functional in starch-sugar interconversion associate with natural variation of tuber starch and sugar content of potato (Solanum tuberosum L.). G3: Genes, Genomes, Genetics, 2014, 4(10): 1797-1811 ( ) DOI: 10.1534/g3.114.012377
- Edwards K., Johnstone C., Thompson C. A simple and rapid method for the preparation of plant genomic DNA for PCR analysis. Nucleic Acids Res., 1991, 19(6): 1349 ( ) DOI: 10.1093/nar/19.6.1349
- Kumar S., Stecher G., Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets. Mol. Biol. Evol., 2016, 33(7): 1870-1874 ( ) DOI: 10.1093/molbev/msw054
- Hawkes J.G. The potato: evolution, biodiversity and genetic resources. Belhaven Press, Oxford, 1990 ( ) DOI: 10.1086/417081
- Choi Y., Sims GE., Murphy S., Miller JR., Chan AP. Predicting the functional effect of amino acid substitutions and indels. PLoS ONE, 2012, 7(10): e46688 ( ) DOI: 10.1371/journal.pone.0046688


