Влияние конформации спирали H38 на стабильность предреакционного состояния пептидилтрансферазного центра рибосомы: молекулярно-динамическое исследование
Автор: Макарова Т.М.
Журнал: Вестник Южно-Уральского государственного университета. Серия: Химия @vestnik-susu-chemistry
Рубрика: Физическая химия
Статья в выпуске: 2 т.16, 2024 года.
Бесплатный доступ
Молекулярно-динамическое моделирование комплекса 70S рибосомы E. coli в каноническом А/А, Р/Р-состоянии, содержащее стоп-пептид ErmBL, позволило наблюдать формирование и дестабилизацию предреакционного состояния пептидилтрансферазного центра (ПТЦ). При отсутствии антибиотиков в рибосоме, субстраты пептидилтрансферазной реакции (ПТР), а именно аминогруппа Lys-aa-тРНК и сложный эфир Asp-Р-тРНК, оказываются стабильно сближены на протяжении 200 нс траектории так, что атомы азота и углерода, между которыми предполагается образование новой пептидной связи, удерживаются на расстоянии не более 4,5 Å. В присутствии же эритромицина в рибосомном туннеле это расстояние за то же время моделирования может увеличиваться до 6 и более Å, при этом искажается сайт связывания ССА-конца А-тРНК. Важную роль в позиционировании А-тРНК играет спираль Н38 23S рРНК: паттерны ее связывания с А-тРНК и соседней спиралью Н84, участвующей в позиционировании уже Р-тРНК, заметно отличаются. В траектории, где субстраты ПТР наиболее сближены, спирали Н38 и Н84 сцеплены стэкинг-взаимодействием между остатками U890 и G2308, а остаток А896 образует стэкинг-контакт с G19 А-тРНК. Присутствие эритромицина в рибосомном туннеле разрушает стэкинг-контакт между Н38 и Н84, а положение А-тРНК изменяется настолько, что она взаимодействует с остатком А896 Н38 другим основанием, С56. Такое искажение связывания А-тРНК приводит к разрушению предреакционной структуры ПТЦ, что объясняет действие антибиотика. Важно понимать, что положение Н38 стоит дополнительно оптимизировать для получения предреакционных структур рибосом из-за недостатка структурных данных относительно этой спирали.
Рибосома, эритромицин, пептидилтрансферазный центр, молекулярная динамика
Короткий адрес: https://sciup.org/147244657
IDR: 147244657 | УДК: 577.217.347, | DOI: 10.14529/chem240217
Текст научной статьи Влияние конформации спирали H38 на стабильность предреакционного состояния пептидилтрансферазного центра рибосомы: молекулярно-динамическое исследование
Одним из средств, используемых бактериями для защиты рибосомы от антибиотиков, является адаптивное метилирование остатков рибосомных РНК, участвующих в связывании антибиотиков и реализации их воздействия на рибосому. Примером может служить метилтрансфераза Erm [1], чей биосинтез регулируется лидерным пептидом ErmBL. Этот фермент вводит две метильные группы на экзоциклическую аминогруппу основания A2058 23S рибосомной РНК, что подавляет связывание макролидов в рибосомном туннеле. Матричная РНК, кодирующая стоп-пептид ErmBL и метилтрансферазу Erm, образует две шпильки: первая включает конец кода ErmBL, вторая – скрывает открытую рамку считывания Erm. Синтез лидерного пептида ErmCL приводит к расплетанию первой шпильки и перестройке вторичной структуры матричной РНК: вторая шпилька также расплетается, а её 5’-концевой участок формирует новую шпильку с 3’-концевым участком первой. При этом освобождается открытая рамка считывания Erm и становится возможен его синтез. В отсутствие антибиотика синтез ErmBL быстро завершается, так что открытая рамка считывания Erm скоро скрывается обратно. В присутствии антибиотика синтез ErmBL останавливается, так что открытая рамка считывания Erm надолго становится доступна другим рибосомам, которые начинают вырабатывать метилтрансферазу, обеспечивающую устойчивость рибосом к антибиотикам.
В присутствии эритромицина биосинтез лидерного пептида ErmBL производит аминокислотную последовательность M1LVFQMRNVDK11, где последний остаток Lys11 так и остается связанным с аминоацил-тРНК, так как именно на нем не происходит реакции транспептидации. Критически важными для останова пептидилтрансферазной реакции (ПТР) в пептиде являются остатки Arg7 и Val9-Lys11, то есть находящиеся в самой верхней части рибосомного туннеля. При этом стэкинг-контакт между Phe4 и основанием U2609 не важен для останова, но важен контакт Arg7-U2586 [2].
Известна структура комплекса рибосомы E. coli , связавшей эритромицин и пептид ErmBL, полученная методом криоэлектронной микроскопии (PDB ID 5JTE), и даже есть работы с расчетом молекулярной динамики, основывающиеся на этой структуре [3], хотя и использующие довольно простую модель воды SPC/E и несколько устаревшее ныне силовое поле parm99. Но до сих пор никто не пытался анализировать удаленные изменения от воздействия связавшихся в рибосомном туннеле антибиотиков и растущих пептидов. Более того, в упомянутой работе эффект антибиотика был сведен к ограничению подвижности пептида, смещению А76 пептидил-тРНК на 1–2 Å и невозможности для остатков U2506 и U2585 занять активированное положение, тогда как сам комплекс тРНК с рибосомой не анализировался. Мы же полагаем, что изменения в пеп-тидилтрансферазном центре (ПТЦ) и в структуре связывания аминоацил-тРНК могут быть более существенными и включать дестабилизацию последней, поскольку из тех же экспериментальных данных известно, что пептид ErmBL стабилизирует предаккомадационное состояние аминоацил-тРНК, в котором ССА-конец не вошел в контакт с А-сайтом [2]. Цель данного исследования состояла в том, чтобы найти аллостерические изменения в рибосоме от воздействия эритромицина с ErmBL в туннеле и построить структурные модели с «выключенным» состоянием ПТЦ, включающие модель удаленных участков, что могло бы приблизить нас к поиску аллостерических эффекторов на ПТЦ.
В этом исследовании мы смоделировали несколько траекторий комплекса рибосомы с Lys-A-тРНК и ErmBL-P-тРНК в присутствии и отстутствие эритромицина. Мы смоделировали по 3 траектории длиной 200 нс для каждого из видов систем с тем, чтобы проанализировать первичные изменения структуры при запуске МД. И этот анализ дал интересные результаты.
Экспериментальная часть
Моделируемая система. Исходная система была построена на основе структуры рибосомы E. coli , несущей эритромицин А и лидерный пептид ErmBL (PDB ID: 5JTE), в которую были добавлены модифицированные гетероциклические основания в соответствии с банком данных [4]. Подготовка системы состояла из процедур, описанных ниже:
-
• Установка недостающих белка L1 и нуклеотидов 884–893 23S рРНК из другой структуры рибосомы E. coli (PDB ID: 4V7D [5]); рибосомных белков L10 и L31 из структуры рибосомы E. coli (PDB ID: 5AFI [6]); 1–10 остатков рибосомного белка bL27 из структуры T. thermophilus (PDB id: 1VY4 [7] (с мутагенезом G6A и L7G по нумерации E. coli ); остатки 16–17 Lys-тРНКLys в A-сайте из структуры Lys-тРНК и некоторых недостающих атомов в белковых остатках путем выравнивания полных аминокислотных структур.
-
• Оптимизация структуры вновь введенных фрагментов методом наискорейшего спуска в вакууме при фиксации координат атомов остальной части рибосомы.
-
• Добавление ионов калия K+ с улучшенными параметрами для воды TIP4P EW [8] вблизи отрицательно заряженных групп [9] для нейтрализации заряда системы и дальнейшая оптимизация ионов K+ в вакууме при фиксации рибосомы и ее лигандов.
-
• Установка рибосомы в ромбическую ячейку размером 27 × 28 × 25 нм, заполненную молекулами воды TIP4PEW, обеспечивающими слой чистого растворителя толщиной не менее 2 нм с каждой стороны, и добавление ионов K+, Mg2+ и Cl- в количестве, соответствующем концентрации KCl в 100 мМ и MgCl 2 в 6 мМ (без учета противоионов K+ и Mg2+ из исходной структуры).
-
• Оптимизация структуры растворителя и ионов с последующим расчётом молекулярной динамики протяжённостью 5 нс, в ходе которой на все атомы рибосомы, кроме атомов водорода, были наложены позиционные ограничения с константой жесткости 1000 кДж/моль∙нм.
-
• Оптимизация системы короткой молекулярной динамикой с позиционными ограничениями для Cα-атомов для аминокислотных остатков или атомов фосфора рибонуклеотидных остатков вплоть до стабилизации RMSD (обычно 5–10 нс).
Условия моделирования
Молекулярно-динамическое моделирование выполнялось посредством пакета GROMACS 5.1.4 [10, 11] и силового поля AMBER-ff14SB [12]. Пространственные структуры и молекулярные электростатические потенциалы неканонических остатков и эритромицина были получены с помощью квантово-химических расчетов методом Хартри – Фока с базисным набором 6-31G*. Точечные заряды были рассчитаны с использованием модели RESP [13]. Моделирование проводилось при T = 310 K, поддерживаемой термостатом масштабирования скоростей с дополнительным стохастическим членом [14] и периодом релаксации 0,1 пс, и изотропным постоянным давлением, контролируемым баростатом Берендсена [15] с периодом релаксации 5 пс. По всем трём измерениям действовали периодические граничные условия. Радиус обрезания для ван-дер-ваальсовых и кулоновских взаимодействий составлял 1 нм. Электростатические взаимодействия обрабатывались с помощью сети частиц Эвальда [16] с шагом сетки 0,125 нм и интерполяцией четвертого порядка. Шаг интегрирования во всех расчетах составлял 2 фс. Алгоритм LINCS [17] применялся для ограничения длины ковалентных связей атомов водорода. Остальные условия МД-моделирования были идентичны описанным в работе [20].
Методы анализа траекторий. Встречаемость водородных связей и стэкинг-взаимодействий оценивали на конечных участках траекторий с установившейся величиной RMSD в соответствии с определенными геометрическими критериями. Для водородных связей это:
-
• расстояние между донором и акцептором водорода не более 3,5 Å,
-
• угол акцептор – донор – водород не более 30°.
Что касается стэкинг-взаимодействия, то среднее арифметическое координат трёх определенных атомов в ароматической плоскости (C2, C4 и C6 для пиримидиновых оснований и N1, N3 и C8 для пуриновых, аналогично для ароматических аминокислот) принималось за центр азотистого основания, а плоскость, содержащая эти три точки, считалась «плоскостью основания», поэтому требования для распознавания стэкинг-взаимодействия в определенном кадре траектории были следующими:
-
• расстояние между центрами не более 5,5 Å,
-
• угол между плоскостями не более 30°,
-
• угол между плоскостью первого основания и отрезком между центрами более 45°; в противном случае копланарные основания, противостоящие друг другу как пара оснований, могут быть ошибочно определены как стэкинг.
Средние встречаемости взаимодействий и стандартные отклонения рассчитывали по трём соответствующим значениям для траекторий систем, содержащих эритромицин. Консервативность остатков РНК оценивали по базе данных SILVA [1].
Обсуждение результатов
Анализ молекулярно-динамических траекторий показал значительную подвижность пептида в туннеле и вариабельность его контактов даже в присутствии антибиотика. Особенно ярко эта вариабельность проявляется для первых 6 остатков аминокислот, для которых не обнаруживалось почти никаких воспроизводящихся связей в присутствии ли эритромицина или в его отсутствии. Так, в большинстве траекторий разрушается стэкинг-взаимодействие Phe4 ErmBL с U2609 23S рРНК, что согласуется с биохимическими данными о том, что мутации по этим остаткам не влияют на эффективность останова трансляции. Остаток Arg7 заякоривается своей боковой группой за фосфаты остатков 23S рРНК в верхней части туннеля, в первую очередь это фосфатные группы U2441, C2063, A2439. Также он образует водородные связи с одним из экзоцик-лических кислородов остатка U2586 23S рРНК, которые есть лишь в одной из трех траекторий без эритромицина. Взаимодействие этих остатков важно для останова ПТР. Остаток Asn7 своей остовной аминогруппой образует водородные связи с А2062 (в присутствии эритромицина – чаще). Боковая группа не образует стабильных и воспроизводящихся связей, что коррелирует с данными о том, что этот остаток можно заменить на аланин без снижения эффекта останова ПТР. Амидная группа Val9 формирует остовную связь с остовным кислородом Arg7 только в присутствии эритромицина. Вероятно, эта связь вносит свой вклад в стабилизацию конформации верхней части пептида.
Сам эритромицин в ходе моделирования траектории молекулярной динамики под действием пептида несколько смещается, образуя контакты с G2502 и С2610 преимущественно кладинозой. При этом сохраняется довольно слабая водородная связь между экзоциклической аминогруппой А2058 и дезозамином вследствие его небольшого смещения под действием пептида относительно положения в структуре. Основные взаимодействия пептида и эритромицина в туннеле представлены в табл. 1. Но главный эффект от связывания пептида в туннеле проявлялся на удаленных от него функциональных сайтах.
В отсутствие эритромицина стабилизация структуры ПТЦ и тРНК в рибосоме происходила достаточно быстро, в течение первых 40–60 нс, хотя пептид был более подвижен, иногда занимая то пространство, где ранее находился эритромицин, что можно отследить по графикам RMSD как целой рибосомы, так и области туннеля и связывания тРНК. В этих траекториях атомы потенциальной новой пептидной связи, а именно азот аминогруппы лизина на Lys-А-тРНК и карбоксильный атом углерода аспарагиновой кислоты на ErmBL-тРНК, именуемые в дальнейшем атакующим и атакуемым атомами соответственно, оставались сближенными на 3–4,5 Å (рис. 1, А). При этом первую траекторию можно считать «лучшей» по этому показателю: в ней означенное расстояние в среднем составляло 3,4 ± 0,2 Å. Аминогруппа при этом находилась примерно перпендикулярно плоскости сложноэфирной группы, что удобно для нуклеофильной атаки (рис. 2, А). Это положение обеспечивалось связями Р- и А-тРНК в районе ПТЦ, в том числе образованием стопки последовательных стэкинг-контактов ССА-конца А-тРНК с А-спиралью (Н92): основаниями U2555, C2556, G2557 и далее.
Таблица 1
Вcтречаемость нековалентных взаимодействий пептида ErmBL и эритромицина с рибосомой в различных состояниях
|
Донор |
Акцептор |
no Ery |
Ery I |
Ery Ia |
Ery Ib |
Ery III |
|
Водородные связи |
||||||
|
A2058/N6–H |
Ery/DES/O 2’ |
0 ± 0 |
7 |
18 |
15 |
25 |
|
A2059/N6–H |
Ery/DES/O 2’ |
0 ± 0 |
0 |
2 |
14 |
0 |
|
Ery/CLD/O 4 –H |
U2506/O4 |
0 ± 0 |
16 |
74 |
92 |
0 |
|
A2062/N6–H |
ErmBL/Phe4/O |
25 ± 43 |
0 |
38 |
0 |
94 |
|
A2062/O 2′ –H |
ErmBL/Asn8/O |
23 ± 40 |
21 |
79 |
1 |
25 |
|
A77/O 2′ –H |
ErmBL/Asp10/Oγ |
7 ± 13 |
25 |
1 |
19 |
0 |
|
A77/O 2′ –H |
ErmBL/Asp10/Oγ′ |
24 ± 42 |
50 |
2 |
16 |
86 |
|
ErmBL/Arg7/N δ –H |
C2442/O phosph1 |
0 ± 0 |
0 |
7 |
31 |
0 |
|
ErmBL/Arg7/N ω –H |
A2062/O 3′ |
13 ± 22 |
26 |
59 |
0 |
0 |
|
ErmBL/Arg7/N ω –H |
A2062/O 4′ |
0 ± 0 |
15 |
1 |
13 |
0 |
|
ErmBL/Arg7/N ω –H |
A2439/O phosph2 |
37 ± 34 |
0 |
0 |
0 |
39 |
|
ErmBL/Arg7/N ω –H |
C2063/O phosph1 |
44 ± 42 |
0 |
8 |
86 |
74 |
|
ErmBL/Arg7/N ω –H |
C2442/O phosph2 |
0 ± 0 |
11 |
21 |
68 |
0 |
|
ErmBL/Arg7/N ω′ –H |
A2439/O phosph2 |
34 ± 56 |
0 |
0 |
0 |
28 |
|
ErmBL/Arg7/N ω′ –H |
U2441/O phosph2 |
83 ± 17 |
64 |
99 |
95 |
97 |
|
ErmBL/Arg7/N ω′ –H |
U2586/O 2′ |
2 ± 3 |
14 |
0 |
0 |
0 |
|
ErmBL/Arg7/N–H |
U2586/O2 |
28 ± 49 |
33 |
0 |
0 |
69 |
|
ErmBL/Arg7/N–H |
U2586/O4 |
0 ± 0 |
0 |
31 |
86 |
0 |
|
ErmBL/Asn8/N–H |
A2062/N3 |
13 ± 18 |
89 |
20 |
64 |
0 |
|
ErmBL/Asn8/N–H |
A2062/O2′ |
20 ± 35 |
2 |
21 |
26 |
58 |
|
ErmBL/Lys11/N–H |
ErmBL/Asp10/Oγ |
20 ± 35 |
24 |
60 |
0 |
30 |
|
ErmBL/Lys11/N–H |
ErmBL/Asp10/Oγ′ |
6 ± 10 |
62 |
10 |
0 |
9 |
|
ErmBL/Lys11/N £ -H |
C2452/N3 |
16 ± 27 |
1 |
29 |
1 |
4 |
|
ErmBL/Val9/N–H |
ErmBL/Arg7/O |
0 ± 0 |
27 |
4 |
47 |
0 |
|
G2061/N2–H |
ErmBL/Asp10/Oγ |
31 ± 8 |
57 |
0 |
0 |
5 |
|
G2061/N2–H |
ErmBL/Asp10/Oγ′ |
36 ± 28 |
13 |
58 |
9 |
0 |
|
U1781/N3–H |
ErmBL/Leu2/O |
0 ± 0 |
51 |
0 |
0 |
0 |
|
U2585/N3–H |
ErmBL/Lys11/O |
42 ± 41 |
0 |
0 |
0 |
0 |
|
U2586/N3–H |
ErmBL/Gln5/O |
0 ± 0 |
88 |
80 |
98 |
0 |
|
A77/O 2′ –H |
ErmBL/Asp10/O |
0 ± 0 |
0 |
0 |
46 |
0 |
Окончание табл. 1
|
Донор |
Акцептор |
no Ery |
Ery I |
Ery Ia |
Ery Ib |
Ery III |
|
ErmBL/Lys11/N–H |
C2507/O phosph1 |
0 ± 0 |
0 |
3 |
82 |
0 |
|
A2058/N6–H |
Ery/DES/O 2’ |
0 ± 0 |
7 |
18 |
15 |
25 |
|
A2059/N6–H |
Ery/DES/O 2’ |
0 ± 0 |
0 |
2 |
14 |
0 |
|
Ery/CLD/O 4 –H |
U2506/O4 |
0 ± 0 |
16 |
74 |
92 |
0 |
|
G2505/N1–H |
Ery/CLD/O 3 |
0 ± 0 |
0 |
0 |
0 |
84 |
|
L22/Lys90/N £ -H |
Ery/LCE/O 12 |
0 ± 0 |
0 |
0 |
0 |
86 |
|
A2062/N6–H |
ErmBL/Phe4/O |
25 ± 43 |
0 |
38 |
0 |
94 |
|
A2062/O 2′ –H |
ErmBL/Asn8/O |
23 ± 40 |
21 |
79 |
1 |
25 |
Примечание. No Ery – статистика по трем стабильным участкам траекторий без эритромицина (среднее ± стандартное отклонение), Ery I – состояние первой траектории с эритромицином после 80 нс, Ery IIa – состояние второй траектории с эритромицином между 40 нс и 140 нс, Ery IIb – состояние второй траектории с эритромицином после 160 нс, Ery III – состояние третьей траектории с эритромицином после 220 нс.
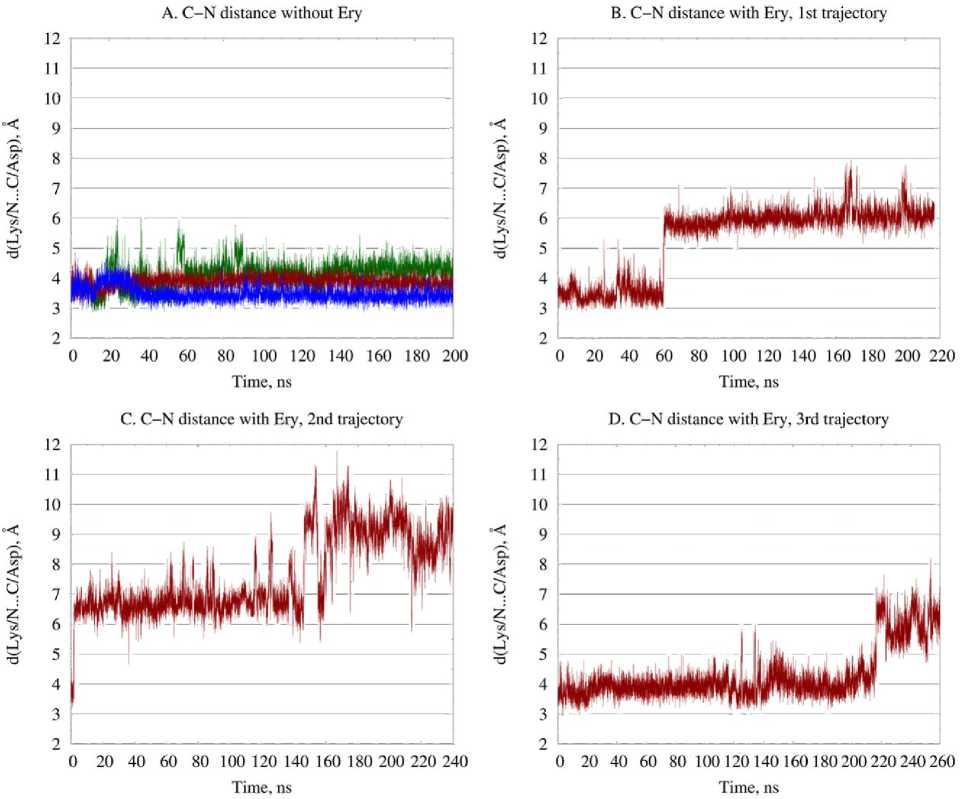
Рис. 1. Графики расстояний между атомами потенциальной пептидной связи между Asp10 и Lys11 в стоп-пептиде ErmBL, образование которой не происходит при наличии эритромицина в туннеле: A. Расстояние С–N для траекторий без эритромицина. Синим цветом показана первая, багровым – вторая, зеленым – третья траектории. B. Расстояние С–N в первой траектории с эритромицином. С. Расстояние С–N во второй траектории с эритромицином.
D. Расстояние С–N в третьей траектории с эритромицином
Встречаемость взаимодействий в А-спирали представлена в табл. 2.
Таблица 2
Вcтречаемость нековалентных взаимодействий в рибосомном туннеле и ближайшей окрестности в различных траекториях
|
Донор |
Акцептор |
no Ery |
Ery I |
Ery Ia |
Ery Ib |
Ery III |
|
Водородные связи |
||||||
|
C2611/N4–H |
G2505/O6 |
0 ± 0 |
54 |
12 |
44 |
0 |
|
A2450/N6–H |
C2063/N3 |
3 ± 3 |
93 |
43 |
24 |
16 |
|
G2446/N2–H |
C2065/O2 |
8 ± 8 |
36 |
30 |
81 |
92 |
|
G830/N1–H |
m2G2445/O 2′ |
41 ± 12 |
2 |
0 |
0 |
3 |
|
L4/Gly64/N–H |
A2060/O phosph 1 |
0 ± 0 |
81 |
71 |
75 |
64 |
|
L4/Lys63/N £ -H |
G2061/O phosph 1 |
0 ± 0 |
46 |
36 |
50 |
0 |
|
L4/Lys63/N £ -H |
G2061/O phosph 2 |
0 ± 0 |
31 |
29 |
44 |
38 |
|
L4/Thr65/N–H |
A2060/O phosph 1 |
2 ± 2 |
65 |
44 |
35 |
26 |
|
Стэкинг-взаимодействия |
||||||
|
A–tRNA/C74 |
U2555 |
98 ± 1 |
8 |
85 |
47 |
98 |
|
U1944 |
U1955 |
70 ± 14 |
0 |
86 |
16 |
0 |
|
G2557 |
C2556 |
92 ± 1 |
94 |
94 |
96 |
0 |
|
C2559 |
A2560 |
54 ± 7 |
0 |
10 |
2 |
0 |
|
A2614 |
A2577 |
37 ± 4 |
12 |
0 |
0 |
0 |
|
A945 |
A2448 |
43 ± 13 |
0 |
0 |
0 |
0 |
|
C2063 |
G2061 |
26 ± 20 |
100 |
96 |
90 |
100 |
Примечание. No Ery – статистика по трем стабильным участкам траекторий без эритромицина (среднее ± стандартное отклонение), Ery I – состояние первой траектории с эритромицином после 80 нс, Ery IIa – состояние второй траектории с эритромицином между 40 нс и 140 нс, Ery IIb – состояние второй траектории с эритромицином после 160 нс, Ery III – состояние третьей траектории с эритромицином после 220 нс.
В случае же присутствия эритромицина в туннеле конформационные перестройки, происходившие в нем, приводили к серьезному разобщению атакующей и атакуемой группы. В разных траекториях это происходило за различное, но обозримое время моделирования: в первой траектории это происходило на 60–70 нс, во второй траектории – в первых же наносекундах, в третьей – после 220 нс моделирования, что можно отследить по графикам расстояний между атакующим и атакуемым атомам (см. рис. 1B–D). Во второй траектории разобщение атакующего атома азота и атакуемого атома углерода достигало еще больших величин после 140 нс.
Назовем участок резкого изменения расстояний между атакующим и атакуемым атомами в ПТЦ «переходом», которых наблюдалось всего четыре. Назовем эти переходы и состояния, формирующиеся после них, как I, IIa, IIb и III согласно номерам траекторий, где во второй наблюдалось два перехода. Каждый такой переход сопровождался существенным искажением связывания А–тРНК, в первую очередь, c так называемой А-спиралью (Н92). При каждом переходе так или иначе нарушались взаимодействия тРНК с этими спиралями. Так, в переходах I и IIa происходил полный выход ССА-конца тРНК из стэкинг-взаимодействия с А-спиралью, в результате чего последняя замещала CCА-конец тРНК основаниями G2553 и U2554, образуя стэкинг– взаимодействия уже с ними (рис. 2В). В переходах IIb и III стэкинг ССА-конца тРНК с А-спиралью хотя и сохранялся, но структура комплекса серьезно искажалась из-за разрыва стэкинг-контакта оснований C2556 и G2557, то есть надлома самой А-спирали чуть выше взаимодействия с ССА-концом тРНК (рис. 2D). Во всех этих случаях это приводило к разобщению субстратов ПТР, исключающему образование новой пептидной связи даже спонтанно.
При этом основной «ловушкой» для атакующей аминогруппы лизина в присутствии эритромицина являлась боковая карбоксильная группа Asp10, с которой атакующая аминогруппа образовывала водородную связь, чем стабилизировалось «арестованное» состояние ПТЦ. Впрочем, во второй траектории в состоянии IIb формировалась водородная связь уже между боковыми группами Lys11 и Asp10, что еще больше расталкивало реакционные центры, полностью отворачивая атакующую аминогруппу от ПТЦ. Таким образом, сами субстраты являются «проблемными» для ПТР, поскольку при малейшем нарушении структуры связывания склонны фиксировать себя в нереакционноспособном положении, что и объясняет специфичность останова трансляции на этих остатках. Хотя этот фактор является лишь вспомогательным, так как при замене Asp10 на некоторые другие остатки без карбоксильной группы останов также происходил, хотя и в меньшей степени [21]. Так что ключевую роль в останове играют именно искажения структуры реакционного комплекса в ПТЦ.
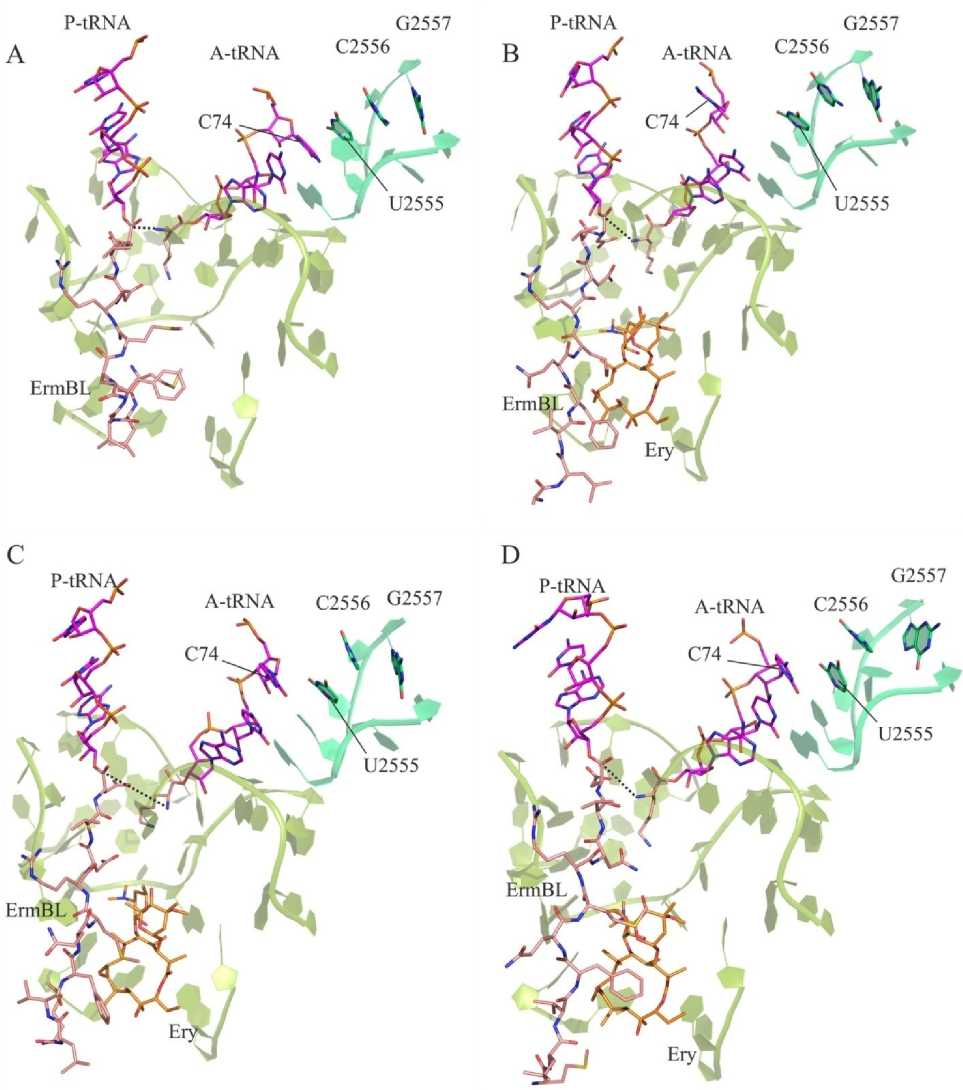
Рис. 2. Структура комплекса рибосомы с Lys-А-тРНК и ErmBL-Р-тРНК в отсутствие и присутствии эритромицина в туннеле. Малиновым цветом показаны ССА-концы тРНК, розовым – пептид ErmBL, ярко-салатовым выделена т. н. А-спираль (Н92), с вершиной которой ССА-конец А-тРНК образует стопку согласно структурным данным.
Черным пунктиром соединены атомы потенциальной новой пептидной связи. A. Центроид основного кластера окружения пептида ErmBL в объединенных траекториях без эритромицина. B. Центроид основного кластера окружения пептида ErmBL в первой траектории с эритромицином. С. Центроид основного кластера окружения пептида ErmBL во второй траектории с эритромицином. D. Центроид основного кластера окружения пептида ErmBL в третьей траектории с эритромицином
В частности, эритромицин «давит» на стопку A2058||A2059||m2A2503||G2061||C2063 и фиксирует ее в положении максимального взаимодействия с Н74 непосредственно рядом с ПТЦ. Это воздействие заметно смещает саму спираль Н74.
Но это не единственное различие в структуре связывания А-тРНК с рибосомой. При всех переходах изменялись взаимодействия локтя А-тРНК с A896 Н38 23S рРНК, именуемой также A-site finger: если в отсутствие эритромицина остаток А896 образовывал стэкинг-контакт преимущественно с G19 А-тРНК (рис. 3A) на самой вершине локтя, то при наличии эритромицина в системе контакт смещался к его уотсон-криковскому партнеру С56 А-тРНК, что означает заметное вращение самой А-тРНК (рис. 3B).
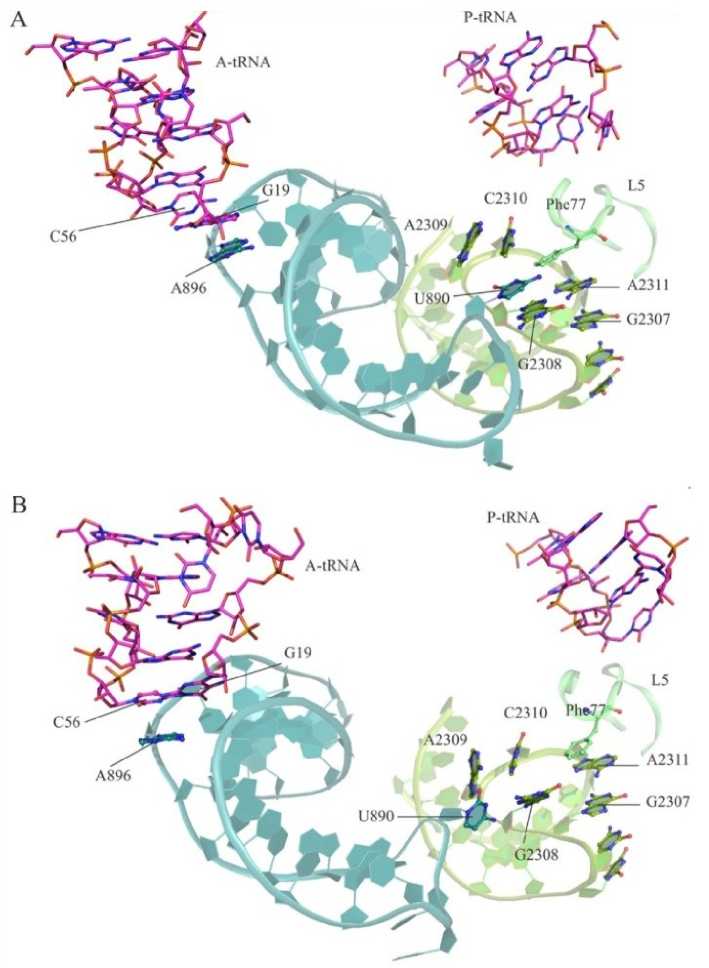
Рис. 3. Структура контакта между вершиной H38 (ASF) и Н84 в траекториях, содержащих ErmBL.
A. Центроид основного кластера окружения контакта Н38/Н84 в первой (лучшей по расстоянию С–N) траектории без эритромицина. B. Центроид основного кластера окружения контакта Н38/Н84 в объединенных траекториях с эритромицином
Изменение взаимодействий локтя А-тРНК с вершиной Н38 (или ASF, A-site finger) происходит на фоне изменений структуры этой вершины, ее как внутренних, так и внешних взаимодействий прежде всего со спиралью Н84. Эти две спирали образуют любопытный контакт, который функционально важен: если вершина Н38 связывает локоть А-тРНК, то Н84 через тонкую прослойку фрагмента белка L5 контактирует с локтем Р-тРНК. При локте Р-тРНК вершина Н84 формирует стопку оснований, куда встраивается Phe77 белка L5: A2309||C2310||Phe77||A2311|| G2307||C2306||U2305, при этом основание G2308 может выпетливаться и контактировать с вы-петливающимся из ASF U890. В исходной структуре и в некоторых участках траектории с эритромицином (например, в состоянии I, IIa) эти выпетливающие остатки сближались так, что могли образовать водородную связь (см. рис. 3В). В других структурах с эритромицином этот контакт и вовсе полностью расходился. В структуре же без эритромицина, которую можно считать «лучшей», с наиболее хорошо сближенными субстратами ПТР, эти выпетливающиеся остатки образовывали прочное стэкинг-взаимодействие (см. рис. 3А). Но в целом, спирали были более сближены в траекториях без эритромицина, чем с ним. Более того, в присутствии эритромицина так или иначе нарушалась стопка вершины Н84 при локте Р-тРНК (см. рис. 3В).
Таким образом, можно заключить, что присутствие эритромицина в туннеле приводит к нарушению контактов вершин Н38 и Н84 и, как следствие, нарушению взаимного позиционирования А- и Р-тРНК. Что касается аллостерического взаимодействия ПТЦ и ASF, то оно возможно, так как большой рычаг спирали Н38 основанием уходит в область, близкую к ПТЦ со стороны спирали Н74, которая смещается под воздействием эритромицина.
Также стоит упомянуть, что структура вершины Н38 плохо различима в структурах рибосомы, полученных методом рентгеноструктурного анализа, и крайне разупорядочена в структурах, полученных методом криоэлектронной микроскопии, и эта ситуация не меняется даже при большом разрешении метода [18]. Причем известно, что функциональное значение этой спирали заключается не только в поддержании связывания А-тРНК: анализ данных мутагенеза и химического зондирования дает основания полагать, что эта спираль участвует в передаче аллостерического сигнала между различными функциональными центрами, включая ПТЦ и центр связывания элонгационных факторов [19]. Нечеткость структурных данных говорит о том, что у структуры Н38 и ее контактов с Н84 есть не одно конформационное состояние, которое зависит от состояния рибосомы в целом. В большинстве структур эти спирали не взаимодействуют, но при запуске молекулярной динамики контакты между ними образуются даже там, где их не было: в частности, при получении структуры А/А, Р/Р-состояния рибосомы из предаккомадационного состояния (PDB ID: 5AFI) контакты между U890 H38 и G2308, G2309 H84 формировались по мере постадийной оптимизации структуры со вписанной в нее А-тРНК, несмотря на то, что U890 в исходной структуре находилась во внутриспиральных стэкинг-взаимодействиях.
Очевидно, что данное взаимодействие между H38 и H84 имеет особое влияние на взаимное расположение тРНК, в том числе в ПТЦ. Изменение этих взаимодействий меняет даже основание А-тРНК, с которым взаимодействует ASF. С другой стороны, структурные данные по контактам этих спиралей вызывают вопросы. Следовательно, для получения более четких картин комплексов рибосомы с различным состоянием ПТЦ (как активным, так и «выключенным») понадобится уточнение возможных структур этого участка 23S рРНК.
Заключение
Смоделирован комплекс рибосомы в каноническом А/А, Р/Р состоянии, содержащий стоп-пептид ErmBL как в присутствии антибиотика эритромицина, так и без него. Связывание эритромицина в рибосомном туннеле провоцирует значительное расхождение субстратов пептидил-трансферазной реакции, и таким образом, ингибирует ее. Разупорядочивание субстратов ПТР друг относительно друга связано с изменением паттерна связывания А-тРНК рибосомой, что проявляется как в А-сайте недалеко от ПТЦ, так и в области локтя А-тРНК, образующего стэ-кинг-контакт со спиралью Н38 23S рРНК. Вершина этой спирали не разрешена в исходной структуре, как и во многих других структурах рибосомы, однако ее конформация и контакты как с А-тРНК, так и со спиралью Н84 имеют значимое влияние на расположение А-тРНК в целом, и, как следствие, стабилизацию или разрушение предреакционного состояния пептидилтрансфераз-ного центра. Это необходимо иметь в виду при моделировании структур рибосом, где важно понять состояние ПТЦ, для чего необходимо производить дополнительную оптимизацию спирали Н38 перед запуском основного расчета молекулярной динамики.
Список литературы Влияние конформации спирали H38 на стабильность предреакционного состояния пептидилтрансферазного центра рибосомы: молекулярно-динамическое исследование
- Hahn J., Grandi G., Gryczan T.J. et al. // Molecular and General Genetics MGG. 1982. V. 186, No. 2. P. 204. DOI: 10.1007/bf00331851.
- Arenz S., Ramu H., Gupta P. et al. // Nature Communications. 2014. V. 5, No. 1. DOI: 10.1038/ncomms4501.
- Arenz S., Bock L.V., Graf M. et al. // Nature Communications. 2016. V. 7, No. 1. DOI: 10.1038/ncomms12026.
- Cannone J.J., Subramanian S., Schnare M.N. et al. // BMC Bioinformatics. 2002. V. 3, No. 1. P. 1-31. DOI: 10.1186/1471-2105-3-2.
- Brilot A.F., Korostelev A.A., Ermolenko D.N. et al. // Proc. Natl. Acad. Sci. U.S.A. 2013. V. 110, No. 52. P. 20994. DOI: 10.1073/pnas.1311423110.
- Fischer N., Neumann P., Konevega A.L. et al. // Nature. 2015. V. 520, P. 567. DOI: 10.1038/nature14275.
- Polikanov Y., Steitz T., Innis C. // Nature Struct. Mol. Biol. 2014. V. 21, P. 787. DOI: 10.1038/nsmb.2871.
- Joung I.S., Cheatham T.E. // J. Phys. Chem. B. 2008. V. 112, No. 30. P. 9020. DOI: 10.1021/jp8001614.
- Athavale S., Petrov A., Hsiao C. et al. // Plos One. 2012. V. 7, P. 1. DOI: 10.1371/journal.pone.0038024.
- van der Spoel D., Lindahl, E., Hess B. et al. // J. Comput. Chem.. 2005. V. 26, P. 1701. DOI: 10.1002/jcc.20291.
- van der Spoel D., Lindahl E., Hess B. et al. // J. Chem. Theory Comput. 2008. V. 4, P. 435. DOI: 10.1021/ct700301q.
- Maier J.A., Martinez C., Kasavajhala K. et al. // Journal of Chemical Theory and Computation. 2015. V. 11, No. 8. P. 3696. DOI: 10.1021/acs.jctc.5b00255.
- Bayly C.I., Cieplak P., Cornell W. et al. // J. Phys. Chem.. 1993. V. 97, No. 40. P. 10269. DOI: 10.1021/j100142a004.
- Bussi G., Donadio D., Parrinello M. // J. Chem. Phys. 2007. V. 126, P. 014107. DOI: 10.1063/1.2408420.
- Berendsen H., Postma J., van Gunsteren W. et al. // J. Chem. Phys. 1984. V. 81, P. 3684. DOI: 10.1063/1.448118.
- Darden T., York D., Pedersen L. // J. Chem. Phys. 1993. V. 98, P. 10089. DOI: 10.1063/1.464397.
- Hess B., Bekker H., Berendsen H.J.C. et al. // J. Comput. Chem. 1997. V. 18, No. 12. P. 1463. DOI: 10.1002/(SICI)1096-987X(199709)18:12<1463::AID-JCC4-3.0.CO;2-H
- Watson Z.L., Ward F.R., Meheust R. et al. // eLife. 2020. V. 9. DOI: 10.7554/elife.60482.
- Sergiev P.V., Kiparisov S.V., Burakovsky D.E. et al. // Journal of Molecular Biology. 2005. V. 353, No. 1. P. 116. DOI: 10.1016/j.jmb.2005.08.006.
- Shishkina A.V., Makarova T.M., Tereshchenkov A.G., Makarov G.I., Korshunova G.A., Bogdanov A.A. // Biochemistry (Moscow). 2015. V. 80, No. 11. P. 1500–1507.
- Wang S., Jiang K., Du X., Lu Y., Liao L., He Z., He W. // Frontiers in Microbiology. 2021. Vol. 12. 690744. doi: 10.3389/fmicb.2021.690744.


