Выделение и характеристика штамма - деструктора дибутилфталата Rhodococcus sp. 5a-k4
Автор: Пьянкова А.А., Краева А.В., Нечаева Ю.И., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 3, 2024 года.
Бесплатный доступ
Изучена способность бактериального штамма 5A-K4, выделенного из ризосферы растений бескильницы расставленной (Puccinellia distans (Jacq.) Parl.), произрастающих на территории промышленных разработок Верхнекамского месторождения солей (Пермский край), к росту на дибутилфталате (ДБФ) в качестве единственного источника углерода и энергии. Штамм 5A-K4 идентифицирован как представитель рода Rhodococcus. Анализ гена 16 S рРНК исследуемого штамма показал наибольший уровень сходства (99.86%) с гомологичным геном R. erythropolis NBRC 15567T. Штамм эффективно рос на ДБФ в качестве субстрата в присутствии до 30 г/л NaCl в среде культивирования. Показано, что Rhodococcus sp. 5A-K4 способен к росту на высоких концентрациях ДБФ (до 12 г/л). В геноме штамма выявлены гены dpeH и mpeH, продукты которых участвуют на начальных этапах разложения ДБФ. Нуклеотидные последовательности генов dpeH и mpeH гомологичны последовательностям генов а/p гидролаз штаммов - представителей классов Actinomycetes и Bacilli. На основании полученных данных предложен путь разложения ДБФ штаммом 5A-K4. Таким образом, ризосферный штамм-деструктор Rhodococcus sp. 5A-K4 может быть использован в качестве бактериального агента при разработке методов фиторемедиации почв, загрязненных фталатами.
Бескильница расставленная, ризосфера, дибутилфталат, деструкция, rhodococcus
Короткий адрес: https://sciup.org/147246121
IDR: 147246121 | УДК: 579.26: | DOI: 10.17072/1994-9952-2024-3-309-317
Текст научной статьи Выделение и характеристика штамма - деструктора дибутилфталата Rhodococcus sp. 5a-k4
Эфиры фталевой кислоты (ЭФК) – класс токсичных антропогенных соединений, которые преимущественно использовались в качестве добавок или пластификаторов для увеличения гибкости, прозрачности и долговечности пластиковых материалов [Lu М. et al., 2020]. Дибутилфталат (ДБФ) является одним из наиболее широко используемых ЭФК, применяется при производстве инсектицидов, упаковочных материалов, косметики, покрытий, одежды и изоляторов для электрооборудования [He, Niu, Lu, 2014]. ЭФК обладают потенциальной канцерогенностью, тератогенностью и мутагенностью [Gardner et al., 2016], в связи с чем, внесены Агентством по охране окружающей среды США, Европейским Союзом и Национальным центром экологического мониторинга Китая в список приоритетных загрязнителей [Lu М. et al., 2020].
В настоящее время ЭФК обнаружены во всем мире в различных средах, таких как вода, воздух, ил, почва и донные отложения [Gao, Wen, 2016]. Значительные концентрации фталатов выявлены в районах работы предприятий горнодобывающей промышленности в составе глинисто-солевых шламов, избыточных рассолов и отходов калийного производства, вследствие применения реагентов, продуктами трансформации которых являются фталаты [Бачурин, Одинцова, 2006].
Одним из основных путей деградации ЭФК в окружающей среде считается разложение с помощью микроорганизмов. Биодеградация ЭФК является эффективным, недорогим, безопасным и экологически чистым методом [Gao, Wen, 2016; Lu М. et al., 2020]. Среди штаммов, способных осуществлять деструкцию ДБФ, описаны бактерии рода Rhodococcus [Aleshchenkova et al., 1996; Li, Gu, 2005; Li et al., 2006; Chao, Cheng, 2007; Lu Y. et al., 2009; Jin et al., 2010; He, Niu, Lu, 2014; Yang et al., 2018; Wang et al., 2022]. Путь деградации эфиров фталевой кислоты у большинства аэробных бактерий, в том числе у представителей рода Rhodococcus , аналогичен и включает гидролиз диэфиров фталата до моноэфиров и последующую их деструкцию до орто -фталевой кислоты и бутанола [Chao et al., 2006; Wang et al., 2022]. Дальнейший путь разложения осуществляется через орто -фталевую кислоту, которая метаболизируется через образование протокатеховой кислоты и, далее – до основных продуктов жизнедеятельности микробной клетки [Vamsee-Krishna, Phale, 2008; Gao, Wen, 2016]. Однако, несмотря на растущее количество сообщений о штаммах, разлагающих ЭФК, генах и ферментах, связанных с деградацией, механизм гидролиза ЭФК требует дальнейшего изучения. Исследование механизмов деградации ЭФК, а также поиск эффективных штаммов-деструкторов будет способствовать разработке новых подходов к восстановлению окружающей среды.
Цель работы – характеристика штамма-деструктора дибутилфталата, выделенного из ризосферы растений бескильницы расставленной, произрастающих в районе промышленных разработок Верхнекамского месторождения солей (Пермский край).
Материалы и методы
Объекты исследования. В работе использован штамм 5A-K4, выделенный из ризосферы растений бескильницы расставленной (Puccinellia distans (Jacq.) Parl.), произрастающих на территории Верхнекамского промышленного региона (Пермский край). Образцы растений были отобраны на расстоянии 8 м от солеотвала (г. Соликамск, Пермский край, Россия), координаты участка отбора проб: N59.633686°, E56.760206°. Почва исследуемого участка супесчаная подзолистая, отличается повышенным содержанием хлоридов, сульфатов, бикарбонатов. Содержание ионов Na+, К+, Ca2+ составляло 17, 26, 88 мг/кг, соот- ветственно. В исследуемой почве выявлено наличие дибутилфталата/диоктилфталата в концентрации 1.0/1.4 мг/кг [Nazarov et al., 2024].
Среды и условия культивирования . Для культивирования бактерий использовали минеральную среду Раймонда ( МСР ) [Raymond, 1961]. В качестве субстратов использовали дибутилфталат (ДБФ), диметилфталат, диэтилфталат, орто- фталевую, протокатеховую, бензойную кислоты, нафталин, бифенил и бутанол в концентрации 1.0 г/л, а также салициловую и гентизиновую кислоты в концентрации 0.5 г/л. Для приготовления богатой среды Раймонда ( БСР ) в МСР добавляли 5 г/л триптона («VWR Life Science Amresco», США) и 2.5 г/л дрожжевого экстракта («Biospringer», Франция) в качестве ростовых субстратов. В работе использовали среды без добавления соли, а также среды с концентрацией NaCl до 70 г/л. Для приготовления плотных сред вносили агар («Helicon», Россия) до конечной концентрации 15 г/л.
Накопительное культивирование и выделение бактерий . 5 г образца ризосферной почвы ресус-пендировали в 10 мл МСР, затем 100 мкл полученной суспензии помещали в 15 мл МСР (30 г/л NaCl) с ДБФ в качестве единственного источника углерода и энергии (1 г/л) в колбу объемом 50 мл. Культивирование проводили на термостатируемом шейкере Environmental Shaker Incubator ES-20/60 («BioSan», Латвия) при 100 об./мин. в течение 3 месяцев, после чего осуществляли высев суспензии на агаризован-ную БСР с содержанием 30 г/л NaCl и инкубировали при 28°С до появления колоний.
Описание морфологии колоний бактерий проводили на седьмой день культивирования на агаризо-ванной БСР (30 г/л NaCl).
Выделение ДНК из чистой культуры бактерий проводили общепринятым методом [Ausbel et al., 1995].
Амплификацию гена 16S рРНК проводили с универсальными бактериальными праймерами 27F и 1492R [Lane, 1991] на амплификаторе C1000 TouchTM Thermal Cycler («Bio-Rad Laboratories», США).
Исследование генов деструкции ДБФ осуществляли путем амплификации фрагментов генов dpeH и mpeH , кодирующих дибутил- и монобутилэстеразу, соответственно, с последующим секвенированием и анализом полученных нуклеотидных последовательностей. Амплификацию генов dpeH и mpeH осуществляли с использовалнием праймеров: dpeH-F 5’-TAAGAAGGAGATATACA TATGCAGATCGTACTGGTGCACG-3’, dpeH-R 5’-GTGGTGGTGGTGGTGCTCGAGGGCAATGACGG GCGTCGAG-3’ и mpeH-F 5’-TAAGAAGGAGATATACATATGGATTCGATTTCACGCCC-3’, mpeH-R 5’-GTGGTGGTGGTGGTGCTCGAGGAGCGCCGTGATCCACCG-3’, соответственно [Lu et al., 2020].
Для детекции ПЦР-продуктов использовали горизонтальный электрофорез в 1%-ном агарозном геле с использованием 1× буфера ТВЕ (Трис – 10.8 г/л, борная кислота – 5.5 г/л, 0.5 М ЭДТА – 4 мл, вода дистиллированная – 79.7 мл/л) при комнатной температуре, напряжении 5–15 В/см в течение 20–40 мин. Агарозные гели окрашивали раствором бромистого этидия (0.5 мкг/мл) в течение 15 мин. и фотографировали в УФ-свете с помощью системы гельдокументирования BioDocAnalyze («Bio-Rad Laboratories», США). Для определения размеров полученных фрагментов использовали маркер длин ДНК 100+ bp DNA Ladder («Евроген», Россия).
Секвенирование и анализ генов. Определение нуклеотидных последовательностей генов 16S рРНК, dpeH и mpeH проводили с применением набора реактивов «GenSeq-100» («Синтол», Россия) на автоматическом секвенаторе Нанофор 05 («Синтол», Россия) согласно рекомендациям производителя. Анализ полученных последовательностей осуществляли с использованием программ Sequence Scanner v. 2.0, MEGA v. 7.0 . Поиск гомологичных последовательностей осуществляли по международным базам данных EzBioCloud и GenBank (http:/. Для построения филогенетических деревьев использовали метод «neighbor-joining» программы MEGA v. 7.0. Эволюционные расстояния рассчитывали с использованием метода «p-distance». Статистическую достоверность ветвления («bootstrap»-анализ) оценивали на основе 1 000 альтернативных деревьев (приведены значения выше 50%).
Рост бактерий на ДБФ оценивали при культивировании в жидкой МСР. Субстрат вносили до конечной концентрации 1.0, 3.0, 5.0, 7.0, 9.0, 10.0, 11.0, 12.0 г/л. В качестве инокулята добавляли культуру (ОП 600 =1.0), выращенную в жидкой МСР с ДБФ в концентрации 1.0 г/л. Культивирование осуществляли в колбах объемом 250 мл (объем среды – 100 мл) на термостатируемом шейкере Environmental Shaker Incubator ES-20/60 («BioSan», Латвия) при температуре 28°С и скорости вращения 140 об/мин. Оптическую плотность ( ОП ) культуральной жидкости определяли на спектрофотометре
UV-Visible BioSpec-mini («Shimadzu», Япония) при длине волны 600 нм в кювете с длиной оптического пути 1 см. Удельную скорость роста ( μ , ч-1) рассчитывали по стандартной формуле
μ = (lnB2 – lnB1)/(t2 – t1), где В1 и В2 – оптические плотности культуры в моменты времени t1 и t2, соответственно [Нетрусов, 2005].
Статистическая обработка результатов . Все эксперименты были выполнены в трехкратной повторности. Для обработки полученных данных использовали программу Microsoft Excel 2007.
Результаты и их обсуждение
Методом накопительного культивирования на МСР с 30 г/л NaCl и ДБФ в качестве субстрата из ризосферы растений бескильницы расставленной, отобранных с территории солеразработок (г. Соликамск, Пермский край), был изолирован бактериальный штамм 5A-K4.
При культивировании на богатой среде (БСР) штамм имел бежевые округлые непрозрачные колонии диаметром 2–3 мм с ровным краем, гладкой блестящей поверхностью, выпуклым профилем, мелкозернистой структурой и мягкой консистенцией. На основе анализа фрагмента гена 16S рРНК (735 п.н.) штамм 5A-K4 имел наибольшее сходство, на уровне 99.86%, со штаммом Rhodococcus erythropolis NBRC 15567T.
При культивировании на БСР при различных концентрациях NaCl в среде, показано, что штамм растет как на среде без добавления соли, так и при содержании NaCl до 50 г/л.
Поскольку штамм был выделен из накопительной культуры с ДБФ в качестве субстрата, была проверена его способность к росту на фталатах и на возможных продуктах их разложения ( орто -фталевой, протокатеховой кислотах и бутаноле). Установлено, что штамм способен к эффективному росту на среде с дибутилфталатом, диметилфталатом, диэтилфталатом, бутанолом и протокатеховой кислотой, но не растет на орто -фталевой кислоте. Также штамм может использовать в качестве единственного источника углерода бензойую, салициловую и гентизиновую кислоты.
Исследованы ростовые характеристики штамма при культивировании на ДБФ в качестве субстрата без соли, а также при различных концентрациях NaCl в среде (табл. 1). Наиболее высокие показатели максимальной ОП и удельной скорости штамм имел при росте на среде без соли. При культивировании в среде с добавлением 30 г/л NaCl штамм также демонстрировал эффективный рост (табл. 1). При добавлении в среду 40 г/л NaCl рост штамма на среде с ДБФ не наблюдался.
Таблица 1
Параметры роста штамма 5A-K4 в МСР с ДБФ (1.0 г/л) при разных концентрациях NaCl [Growth parameters of the strain 5A-K4 in MMR and DBP (1.0 g/L) at different concentrations of NaCl]
|
Концентрация NaCl, г/л |
Удельная скорость роста, ч-1 |
Максимальное значение ОП 600 |
|
без NaCl |
0.038±0.002 |
1.41 |
|
30 |
0.021±0.001 |
0.81 |
Так как штамм 5A-K4 демонстрировал эффективный рост на ДБФ в качестве субстрата (табл. 1), были исследованы ростовые характеристики штамма при культивировании с высокими концентрациями ДБФ в среде МСР без добавления соли. Показано, что штамм способен к росту при содержании ДБФ до 12 г/л (табл. 2). При увеличении концентрации ДБФ в среде культивирования лаг-фаза изменялась незначительно. Максимальное значение ОП зафиксировано при содержании 3 г ДБФ/л, а максимальное значение удельной скорости роста – при 1 г ДБФ/л (табл. 2).
Таблица 2
Параметры роста штамма 5A-K4 в МСР при разных концентрациях ДБФ [Growth parameters of the strain 5A-K4 in MMR at different concentrations of DBP]
|
Концентрация ДБФ, г/л |
Удельная скорость роста, ч-1 |
Максимальное значение ОП 600 |
|
1 |
0.084±0.004 |
1.13 |
|
3 |
0.060±0.003 |
1.89 |
|
5 |
0.047±0.002 |
1.76 |
|
7 |
0.042±0.003 |
1.57 |
|
9 |
0.028±0.002 |
1.38 |
|
10 |
0.016±0.002 |
1.15 |
|
11 |
0.010±0.001 |
1.02 |
|
12 |
0.007±0.001 |
0.72 |
Проведена амплификация генов dpeH и mpeH , продукты которых участвуют в двух последовательных стадиях гидролиза ДБФ [Lu et al., 2020]. С использованием праймеров, описанных [Lu M. et al., 2020], у исследуемого штамма 5A-K4 выявлено наличие обоих генов (рис. 1).
Проведены секвенирование и анализ амплифицированных генов dpeH и mpeH. Сравнение гена dpeH штамма 5A-K4 с гомологичными последовательностями из базы данных GenBank показало, что данная последовательность имеет наибольше сходство с последовательностями α/β гидролаз бактерий – представителей классов Actinomycetes и Bacilli. Сходство на уровне 99.85% сравниваемая последовательность имеет с геном α/β гидролазы штамма Rhodococcus pyridinivorans 5Ap, который был изолирован из ливийской почвы, загрязненной нефтью [Чернявская и др., 2016]. На филогенетическом дереве транслированных аминокислотных последовательностей (ТАП) данный штамм формирует отдельный кластер с исследуемым штаммом Rhodococcus sp. 5A-K4 (рис. 2). Также ген dpeH штамма 5A-K4 имеет сходство (99.54%) с гомологичными генами Bacillus subtilis DK01 и штаммами семейства Micrococcaceae (уровень сходства 99.39%) – Glutamicibacter sp. ZJUTW, Microbacterium paraoxydans 70447 и Microbacterium schleiferi A32-1 (рис. 2). Стоит отметить, что штамм Glutamicibacter sp. ZJUTW, выделенный из активного ила (Китай), использовался в составе бактериального консорциума для одновременного и эффективного разложения шести эфиров фталевой кислоты и демонстрировал наибольшую активность при деградации короткоцепочечных эфиров [Liu et al., 2023].
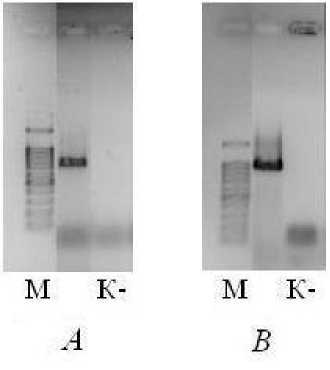
Рис. 1. Электрофореграмма результатов амплификации генов mpeH ( A ) и dpeH ( B ) штамма 5A-K4.
M – маркер 100+ bp DNA Ladder («Евроген», Россия), K- – отрицательный контроль
[Electropherogram of the mpeH ( A ) and dpeH ( B ) amplification results of the strain 5A-K4.
M – 100+ bp DNA Ladder marker (“Evrogen”, Russia), K- – negative control]
力 іЕ^ е Н ЛЕ2 ^ tf^ ^дгг^ sp. 5A4
加 I 山口 г идр 口 паз а еЛ 口曲酬比弊 用旧"由"也"期 в 口出 54 p (Q 口鹏汇 11516)
in L сф гидроп-ща Е&а 就 ін jzj-Mij DEE 1 (WSFLM643)
I 山卩 г ндриЕ.аза J 俄 Wrm5nЕ 財 Hum 戸占匕口: pti2Hls 7044*7 (QXE31721) 石 cdfl г itjijio Л-Щ.1 J й7сга& □ ctsnuyn 15aЛе 淞 г" A32-1 (QPED4173j
—сф гидр 口 паза GJumm2Msi w*r 5P. £ JUTWi : QEF09054 : i
__________I-------- 山卩 гидр 口 паза. 戍向□必比比也 sp. LW-X Y12 (С?х'Ш5б2ёГ ; і
SI------------------------------------------DpeH Micro bactenuj-гі 5P. PAE-1 (AZFty727)
Рис. 2. Положение dpeH -гена штамма Rhodococcus sp. 5A-K4 на филогенетическом дереве, построенном на основании сравнительного анализа ТАП генов α/β гидролаз.
Масштаб соответствует 5 аминокислотным заменам на каждые 1000 аминокислот. В скобках указаны номера в базе данных GenBank
[Positions of the gene dpeH of the strain Rhodococcus sp. 5A-K4 in the phylogenetic tree based on comparison of the amino acid sequences translated from α/β hydrolase genes.
The scale bar corresponds to 5 amino acid substitutions per 1000 amino acids. The GenBank accession numbers are given in parentheses]
Еще один кластер на филогенетическом дереве формируют ТАП α/β гидролаз штаммов Rhodococ-cus sp. LW-XY12 и Microbacterium sp. PAE-1. Уровень сходства нуклеотидной последовательности гена dpeH штамма 5A-K4 и последовательностей штаммов, входящих в этот кластер, составлял 98.47–98.93%. Штамм Rhodococcus sp. LW-XY12, изолированный из активного ила (Китай), способен к эффективному разложению ЭФК [Song et al., 2022]. Штамм Microbacterium sp. PAE-1, выделенный из сточных вод заво- да по производству пластмасс, способен к разложению ДБФ до орто-фталевой кислоты через монобутилфталат посредством двух последовательных стадий гидролиза с участием продуктов генов dpeH и mpeH [Lu M. et al., 2020]. Анализ полученной последовательности гена mpeH штамма 5A-K4 показал, что данная последовательность имеет наибольшее сходство с генами mpeH тех же штаммов (представителей классов Actinomycetes и Bacilli), с которыми близка и последовательность гена dpeH (рис. 2, 3). Кроме штаммов, с которыми выявлено сходство по гену dpeH, нуклеотидная последовательность гена mpeH штамма Rhodococcus sp. 5A-K4 имеет высокий процент сходства (98.97%) и с нуклеотидной последовательностью α/β гидролазы штамма Rhodococcus ruber YC-YT1, способного к эффективному разложению эфиров фталевой кислоты [Yang et al., 2018].
α/β гидролаза Rhodococcus sp. LW-XY12 (QXU56362)
α/β гидролаза Microbacterium schleiferi A32-1 (QPE04172)
-
54 MpeH Microbacterium sp. PAE-1 (AZP89726)
0.002
78 I 一 I α/β гидролаза Rhodococcus ruber YC-YT1 (AXY49591)
69 α/β гидролаза Microbacterium paraoxydans 70447 (QXE31720) α/β гидролаза Glutamicibacter sp. ZJUTW (QEP09053)
α/β гидролаза Bacillus subtilis DK01 (WGP04643)
I MpeH Rhodococcus sp. 5A-4
95 α/β гидролаза Rhodococcus pyridinivorans 5Ap (QOW01517)
Рис. 3. Положение mpeH -гена штамма Rhodococcus sp. 5A-K4 на филогенетическом дереве, построенном на основании сравнительного анализа ТАП генов α/β гидролаз.
Масштаб соответствует 2 аминокислотным заменам на каждые 1000 аминокислот. В скобках указаны номера в базе данных GenBank
[Positions of the gene mpeH of the strain Rhodococcus sp. 5A-K4 in the phylogenetic tree based on comparison of the amino acid sequences translated from α/β hydrolase genes.
The scale bar corresponds to 2 amino acid substitutions per 1000 amino acids. The GenBank accession numbers are given in parentheses]
Таким образом, у исследуемого штамма Rhodococcus sp . 5A-K4 показано наличие генов dpeH и mpeH , продукты которых участвуют в двух последовательных стадиях гидролиза ДБФ через монобутилфталат до орто- фталевой кислоты (рис. 4).
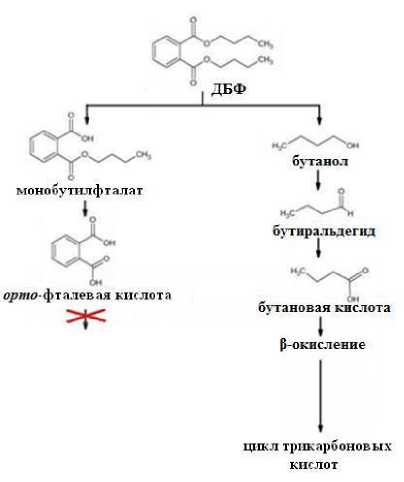
Рис. 4. Схема вероятного пути разложения ДБФ
[A scheme of probable degradation pathway of DBP]
Установлено, что штамм 5A-K4 не способен к росту на орто-фталевой кислоте – основном промежуточном метаболите разложения ДБФ у бактерий [Gao, Wen, 2016]. Было зафиксировано накопление орто-фталевой кислоты в культуральной жидкости при росте исследуемого штамма в МСР с ДБФ в качестве субстрата (данные не представлены). Поэтому можно предположить, что деструкция ДБФ у Rhodococcus sp. 5A-K4 идет по пути, представленному на рис. 4, через промежуточный продукт метаболизма ДБФ – бутанол [Mahajan et al., 2019]. Это предположение также подтверждается способностью штамма Rhodococcus sp. 5A-K4 к эффективному росту на бутаноле в качестве субстрата.
Заключение
В результате проведенных исследований из ризосферы растений бескильницы расставленной ( Puccinellia distans (Jacq.) Parl.), произрастающих на загрязненных/засоленных почвах района разработок Верхнекамского месторождения солей (Пермский край), изолирован штамм-деструктор дибутилфталата Rhodococcus sp. 5A-K4. Штамм способен к эффективному росту на среде с дибутилфталатом, а также диметил- и диэтилфталатом, бутанолом, протокатеховой, бензойной, салициловой и гентизиновой кислотами в качестве субстрата. В геноме штамма выявлены гены dpeH и mpeH , продукты которых участвуют в гидролизе ДБФ. Нуклеотидные последовательности генов dpeH и mpeH сходны с последовательностями генов α/β гидролаз бактерий – представителей классов Actinomycetes и Bacilli . Интересен факт, что штамм не растет на орто -фталевой кислоте, являющейся ключевым метаболитом при деструкции ДБФ у большинства изученных бактерий-деструкторов ЭФК. На основании полученных данных предложен путь разложения ДБФ штаммом 5A-K4 с использованием бутанола в качестве ключевого метаболита. Rhodococcus sp. 5A-K4 может использовать в качестве единственного источника углерода и энергии ДБФ при содержании 30 г/л NaCl в среде, а также способен к эффективному росту при концентрации ДБФ до 12 г/л. Таким образом, ризосферный деструктор ДБФ Rhodococcus sp. 5A-K4 представляет интерес для дальнейших исследований и возможности использования его в качестве бактериального агента при разработке методов фиторемедиации почв, загрязненных фталатами.
Список литературы Выделение и характеристика штамма - деструктора дибутилфталата Rhodococcus sp. 5a-k4
- Бачурин Б.А., Одинцова Т.А. Стойкие органические загрязнители в отходах горного производства // Современные экологические проблемы Севера. Апатиты: Изд-во Кольского НЦ РАН, 2006. Ч. 2. С. 7-9.
- Нетрусов А.И. Практикум по микробиологии. М.: Академия, 2005. 608 с.
- Чернявская М.И. и др. Первичный анализ генома бактерий-деструкторов нефти Rhodococcus pyridinivorans 5Ap // Труды БГУ. 2016. Т. 11, ч. 1. С. 219-223.
- Aleshchenkova Z.M. et al. The degradation of plasticizers by Rhodococcus erythropolis 40F // Mikrobiol. Z. 1996. Vol. 58(4). P. 34-38.
- Ausbel F.M. Short Protocols in Molecular Biology. 3rd ed. N.Y.: John Wiley & Sons, 1995. 450 p.
- Chao W.L., Cheng C.-Y. Effect of introduced phthalate-degrading bacteria on the diversity of indigenous bacterial communities during di-(2-ethylhexyl) phthalate (DEHP) degradation in a soil microcosm // Chemo-sphere. 2007. Vol. 67(3). P. 482-488.
- Chao W.L. et al. Degradation of di-butyl-phthalate by soil bacteria // Chemosphere. 2006. Vol. 63(8). P. 1377-1383.
- Gao D.-W., Wen Z.-D. Phthalate esters in the environment: a critical review of their occurrence, biodegradation, and removal during wastewater treatment processes // Science of the Total Environment. 2016. Vol. 541. P. 986-1001.
- Gardner S.T. et al. Assessing differences in toxicity and teratogenicity of three phthalates, diethyl phthalate, di-n-propyl phthalate, and di-n-butyl phthalate, using Xenopus laevis embryos // Journal of Toxicology and Environmental Health. 2016. Vol. 79(2). P. 71-82.
- He Z., Niu C., Lu Z. Individual or synchronous biodegradation of di-n-butyl phthalate and phenol by Rhodococcus ruber strain DP-2 // Journal of Hazardous Materials. 2014. Vol. 273. P. 104-109.
- Jin D.-C. et al. Biodegradation of di-n-butyl phthalate by Rhodococcus sp. JDC-11 and molecular detection of 3,4-phthalate dioxygenase gene // Journal of Microbiology and Biotechnology. 2010. Vol. 20(10) P. 1440-1445.
- Lane D.J. 16S/23S rRNA sequencing // Nucleic acid techniques in bacterial systematics. 1991. P. 115175.
- Li J. et al. Bioremediation of environmental endocrine disruptor di-n-butyl phthalate ester by Rhodococcus ruber // Chemosphere. 2006. Vol. 65(9). P. 1627-1633
- Li K., Gu J. Biodegradation of di-n-butyl phthalate by mangrove microorganism Rhodococcus ruber 1K // Ying Yong Sheng Tai Xue Bao. 2005. Vol. 16(8). P. 1566-1574.
- Liu T. et al. Synthetic bacterial consortia enhanced the degradation of mixed priority phthalate ester pollutants // Environmental Research. 2023. Vol. 235. P. 116-121.
- Lu M. et al. Degradation of dibutyl phthalate (DBP) by a bacterial consortium and characterization of two novel esterases capable of hydrolyzing PAEs sequentially // Ecotoxicology and Environmental Safety. 2020. Vol. 195. P. 1-9.
- Lu Y. et al. Biodegradation of dimethyl phthalate, diethyl phthalate and di-n-butyl phthalate by Rhodococcus sp. L4 isolated from activated sludge // Journal of Hazardous Materials. 2009. Vol. 168(2-3). P. 938-943.
- Mahajan R. et al. Biodegradation of di-n-butyl phthalate by psychrotolerant Sphingobium yanoikuyae strain P4 and protein structural analysis of carboxylesterase involved in the pathway // International Journal of Biological Macromolecules. 2019. Vol. 122. P. 806-816.
- Nazarov A.V. et al. Soil bacterial communities in the affected zone of salt dump (Solikamsk, Perm krai) // Eurasian Soil Science, 2024, Vol. 57(8). P. 1353-1361.
- Raymond R.L. Microbial oxidation of n-paraffinic hydrocarbons // Developments in Industrial Microbiology. 1961. Vol. 2(1). P. 23-32.
- Song X. et al. Biodegradation of phthalate acid esters by a versatile PAE-degrading strain Rhodococcus sp. LW-XY12 and associated genomic analysis // International Biodeterioration and Biodegradation. 2022. Vol. 170. P. 1-12.
- Vamsee-Krishna C., Phale P.S. Bacterial degradation of phthalate isomers and their esters // Indian Journal of Microbiology. 2008. Vol. 48. P. 19-34.
- Wang L. et al. Analysis of the performance of the efficient di-(2-ethylhexyl) phthalate-degrading bacterium Rhodococcus pyridinovorans DNHP-S2 and associated catabolic pathways // Chemosphere. 2022. Vol. 306. P. 135-142.
- Yang T. et al. Biodegradation of di-(2-ethylhexyl) phthalate by Rhodococcus ruber YC-YT1 in contaminated water and soil // International Journal of Environmental Research and Public Health. 2018. Vol. 15(5). P. 1-20.


