Выявление анцестральных характеристик генома у Rhizobium leguminosarum bv. Trifolii
Автор: Аксенова Т.С., Чирак Е.Р., Онищук О.П., Курчак О.Н., Афонин А.М., Пинаев А.Г., Андронов Е.Е., Проворов Н.А.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Эволюция симбиоза
Статья в выпуске: 3 т.55, 2020 года.
Бесплатный доступ
Клубеньковые бактерии вида Rhizobium leguminosarum - симбиотические азотфиксаторы, разделяющиеся на два биотипа: viciae и trifolii (D.C. Jordan с соавт., 1984). За функцию симбиотической азотофиксации отвечают симбиотические гены, эволюция которых зависит от растений-хозяев (J.P.W. Young с соавт., 1989). Недавно было показано, что по типу организации симбиотических регионов геномов ризобии, выделенные из вавиловии красивой Vavilovia formosa (Stev.) Fed . , возможно, близки к протосимбионту трибы FabeaeR. leguminosarum bv. viceae (E.R. Chirak с соавт., 2019; A.K. Kimeklis с соавт., 2019). Однако в эволюции R. leguminosarum имела место еще одна более ранняя дивергенция между биотипами viceae и trifolii, отправной точкой которой был протосимбионт всего вида R. leguminosarum , существовавший до его разделения на биовары. В своей работе мы представляем результаты секвенирования группы геномов R. leguminosarum bv. trifolii и сопоставления структуры их симбиотических регионов с соответствующими участками геномов R. leguminosarum bv. viciae , относящихся к анцестральным и «продвинутым» типам. Для этого мы сравнили полные геномы штаммов R. leguminosarum bv. trifolii (методика секвенирования Oxford Nanopore) и референсных штаммов R. leguminosarum bv. viceae , относящихся к анцестральным и «продвинутым» типам. Было показано, что в геномах штаммов симбионтов клевера находятся четыре из пяти анцестральных признаков: увеличенный размер межгенных областей в симбиотическом регионе, наличие в nod -опероне гена nodX , отсутствие гена nodT в sym -регионе и только одна копия оперона fixNOPQ , располагающаяся на плазмиде pSym. Исходя из полученных результатов, мы предполагаем, что протосимбионт R. leguminosarum мог быть также близок к ризобиям клевера, как и к ризобиям вавиловии.
Эволюция симбиоза, симбиотические гены, протосимбионт, полногеномное секвенирование
Короткий адрес: https://sciup.org/142226311
IDR: 142226311 | УДК: 631.461.52:575.22:575.85 | DOI: 10.15389/agrobiology.2020.3.489rus
Текст научной статьи Выявление анцестральных характеристик генома у Rhizobium leguminosarum bv. Trifolii
Клубеньковые бактерии вида Rhizobium leguminosarum — наиболее широко распространенные в умеренных широтах симбиотические азот-фиксаторы, которые разделяются на два контрастно различающихся по хозяйской специфичности биотипа: bv. viciae (симбионты вики, гороха, чины, чечевицы и вавиловии) и bv. trifolii (симбионты клевера) (1). За функцию симбиотической азотофиксации отвечают симбиотические гены ( sym -гены), эволюция которых во многом определяется растениями-хозяевами (2).
У ризобий выделяют три основные группы sym -генов: nod (синтез липохитоолигосахаридных сигнальных Nod-факторов, индуцирующих развитие клубеньков) (3-5), nif (синтез нитрогеназы) (5) и fix (поставка энергии для нитрогеназы и регуляции nif -генов) (5, 6). Эволюция симбиотического комплекса сначала происходила с помощью сборки групп генов, которые кодируют различные сигнальные и метаболические свойства, обеспечивающие функционирование симбиоза, у первичных ризобий, происходящих из несимбиотических диазотрофов, с последующим переносом собранных симбиотических конструкций в так называемые вторичные (производные) виды (7). Первичные ризобии — родственники современного рода Bradyrhizobium были близки к свободноживущему фототрофу Rhodopseudomonas и приобрели способность к фиксации азота посредством рекрутирования некоторых генов, контролирующих фотосинтез, в систему fix (8). Эта реорганизация привела к появлению фотосинтетически активных Bradyrhizobium spp., нодулирующих стебли тропических бобовых без
∗ Исследование выполнено при финансовой поддержке РФФИ в рамках научного проекта ¹ 18-34-00839 (сбор клубеньков и выделение штаммов) и РНФ ¹ 19-16-00081 (секвенирование геномов и биоинфор-матический анализ данных).
использования nod -генов. Возможность синтезировать Nod-факторы, за которые отвечают nod -гены, вероятно, впервые приобрели бактерии рода Bradyrhizobium , у которых фототрофность была замещена способностью использовать продукты фотосинтеза растений. У этих гетеротрофных ри-зобий обычно сохраняется экспрессия nif -генов ex planta, но они не способны к диазотрофному росту из-за низкой активности нитрогеназы (9). Наиболее изученные вторичные ризобии представлены родами Rhziobium , Sinorhizobium , Mezorhizobium и Neorhizobium . Эти бактерии лишены систем фотосинтеза и не могут экспрессировать гены нитрогеназы ex planta, их появление было результатом горизонтального переноса sym -генов из первичных ризобий в различные почвенные гетеротрофные бактерии (10).
Организация sym -генов подробно изучена у вида Rhizobium legumi-nosarum bv. viceae , штаммы которого существенно варьируют по специфичности, проявляемой в отношении разных форм растений-хозяев. Недавно было показано, что по типу организации симбиотических регионов ( sym -регионы) геномов ризобии R. leguminosarum bv. viceae можно разделить на две группы (10). Первая группа, выделенная из вавиловии красивой Vavilovia formosa (Stev.) Fed . (растения, предположительно близкого к последнему общему предку всей трибы Fabeae ) (11) и обладающая комплексом анцестральных черт генома, характеризуется расширенным sym -регионом, иногда разделенным между двумя симбиотическими плазмидами (pSym), наличием генов nodX и fixW в плазмидных sym -оперонах, отсутствием хромосомных копий fixNOPQ и расположением nodT вне оперонов nod -генов. Во второй, производной (или эволюционно «продвинутой») группе sym -регион более компактен, есть хромосомные копии fixNOPQ , nodT интегрирован в nod- оперон между nodN и nodO , а гены nodX и fixW утрачиваются. Переход из анцестральной формы в «продвинутую» связан с общей (структурной и функциональной) компактизацией генома, увеличением интенсивности азотофиксации и сужением хозяйской специфичности. Таким образом, показано, что ризобии, выделенные из вавиловии красивой, близки к протосимбионту трибы Fabeae R. leguminosarum bv. viceae . Однако в эволюции ризобий этой группы имела место еще одна более ранняя дивергенция между R. leguminosarum bv. viceae и R. leguminosarum bv. trifolii, отправной точкой которой был протосимбионт всего вида R. le-guminosarum , существовавший до его разделения с образованием биоваров viceae и trifolii . И хотя на сегодняшний день ничего не известно ни о растении-хозяине этого протосимбионта, ни тем более об организации его генома, очевидно, что для понимания эволюционных процессов конструирования этого протосимбионта очень полезным станет сопоставление симбиотических оперонов анцестральных вариантов R. leguminosarum bv. viceae и соответствующих участков в геномах R. leguminosarum bv. trifolii .
В представляемой работе мы приводим первые результаты секвенирования группы геномов Rhizobium leguminosarum bv. trifolii (Rlt) и сопоставления структуры их симбиотических регионов с соответствующими участками геномов R. leguminosarum bv. viciae (Rlv), относящихся к анце-стральному и продвинутому типам.
Целью исследования был поиск анцестральных симбиотических признаков в геноме Rhizobium leguminosarum bv. trifolii.
Методика. Клубеньковые бактерии выделяли из 50 образцов ризосферной почвы у трех видов клевера (Trifolium pratense L., T. repens L., T. hybridum L.), собранных в пгт. Вырица (Ленинградская обл., Гатчинский р-н). Для сбора образцов выбирали участки компактного произраста- ния цветущих растений (не дальше 0,2-0,3 м друг от друга), расстояния между участками отбора составляли не менее 5 м.
Из каждого образца готовили почвенные суспензии, которыми инокулировали стерильные проростки клевера двух видов — лугового ( T. pratense ) и ползучего ( T. repens ).
Растения выращивали в условиях гнотобиотического микровегета-ционного опыта на безазотной среде Красильникова-Кореняко. С каждого зеленого растения отбирали по одному клубеньку, который стерилизовали в 96 % спирте и дважды промывали стерильной водой. Клубеньки разрушали стеклянной палочкой в эппендорфе; 0,1 мл водной суспензии высевали на агаризованную среду 79 (12). На 3-и сут роста отдельные колонии пересевали в пробирки с бобовым агаром для хранения (12). Было получено 37 изолятов ризобий клевера, из которых отобрали пять и выращивали их в 5 мл жидкой среды 79 в течение 1 сут при температуре 28 ° С. Культуры использовали для выделения геномной ДНК по стандартной методике (13).
Для полногеномного секвенирования подготовку библиотек проводили по рекомендованному производителем протоколу 1D native barcoding genomic DNA с использованием наборов EXP-NBD104, EXP-NBD114 и SQK-LSK109 («Oxford Nanopore», Великобритания). Библиотеки секвенировали на приборе MinION («Oxford Nanopore», Великобритания) в соответствии с инструкцией производителя на ячейке R9.4. Бей-сколлинг raw-файлов fast5, полученных в результате секвенирования, проводили в программе Albacore v. 2.3.1 . Для демультиплексирования ридов использовали приложение Deepbiner v. 0.2.0 (14), для очистки — программу Porechop v. 0.2.3 . Сборку ридов осуществляли в Flye v. 2.6 . Полученные сборки корректировали в пакете Racon v. 1.3.2 , используя параметры -m 8 -x -6 -g -8 -w 500, а также в программе Medaka v. 0.10.0 . Аннотирование геномов проводили в программе Prokka (15). Геномы были депонированы в GenBank в биопроекте PRJNA611463 .
Извлечение последовательностей, конкатенацию и другие манипуляции с геномами в процессе информатической обработки проводили в пакете CLC Genomics Workbench v. 7.5.1 (). Последовательности выравнивали, используя программу Molecular Evolutionary Genetics Analysis (MEGA) X (18). Построение филогенетических деревьев методом максимального правдоподобия с bootstrap (1000 повторов) проводили в программе PhyML v. 3.3 (19). Выбор модели распределения определялся программой автоматически методом наименьшего BIC (Bayesian information criterion) (20). Полученные дендрограммы визуализировались в online приложении iTOL (21).
Результаты . Анализ геномов выполнили у пяти местных изолятов (3B, 9B, 22B, 23B и 31B) и пяти штаммов R. leguminosarum (табл. 1).
-
1. Штаммы Rhizobium leguminosarum , использованные в работе
Штамм Регион Растение-хозяин Номер в базе GenBank Ссылка
Rh izo b iu m legum in o saru m bv. trifo l i i (Rlt)
3B пгт. Вырица, Ленинградская обл., Россия Trifolium repens L. PRJNA611463 Эта работа
9B пгт. Вырица, Ленинградская обл., Россия Trifolium pratense L. PRJNA611463 Эта работа
Продолжение таблицы 1
|
22B |
пгт. Вырица, Ленинградская обл., Россия |
Trifolium pratense L. |
PRJNA611463 |
Эта работа |
|
23B |
пгт. Вырица, Ленинградская обл., Россия |
Trifolium pratense L. |
PRJNA611463 |
Эта работа |
|
31B |
пгт. Вырица, Ленинградская обл., Россия |
Trifolium pratense L. |
PRJNA611463 |
Эта работа |
|
WSM1689 Греция |
Trifolium uniflorum L. |
CP007045-CP007050 |
(17) |
|
|
Vaf10 |
Rh izo b iu m l egum in o saru m bv. Северная Осетия, Россия Vavilovia formosa (Stev.) Fed. |
v ic ia e (Rlv) CP016286-CP016293 |
(10) |
|
|
Vaf108 |
Дагестан, Россия |
Vavilovia formosa (Stev.) Fed. |
CP018228-CP018236 |
(10) |
|
TOM |
Турция |
Pisum sativum L. |
AQUC01000001-AQUC01000006 |
(16) |
|
248 |
Англия |
Vicia faba L. |
ARRT01000001-ARRT01000007 |
(16) |
Мы проанализировали геномы у пяти полученных изолятов Rlt (3B, 9B, 22B, 23B, 31B) и сравнили их с геномами штаммов Rlv (см. табл. 1). Для сравнения были выбраны штаммы Vaf108 и Vaf10 — симбионты вави-ловии красивой ( Vavilovia formosa ) (22), которая, вероятно, является ближайшим живым родственником общего предка трибы Fabeae (23), TOM — симбионт афганских культиваров гороха ( Pisum sativum L.) (24), 248 — симбионт Vicia faba L. и WSM1689 — симбионт T. uniflorum (см. табл. 1). Так как дивергенция биоваров R. leguminosarum определяется симбиотическими генами, в то время как хромосомный фон у этих биоваров общий (25), мы сосредоточили внимание на симбиотических регионах геномов.
Состав sym -оперонов. При сравнении sym -регионов полученных геномных последовательностей у изолятов Rlt 3B, 9B, 22B, 23B и 31B с sym -регионами штаммов Rlt WSM1689 и Rlv Vaf10, Vaf108, TOM и 248 были найдены различия в составе и расположении sym -генов между Rlt и Rlv (рис. 1). У штаммов 3B, 9B, 22B и 23B sym -гены организованы в sym -опероны, расположенные на pSym. Несколько отличается штамм 31B, у которого оперон fixNOQPGHIS расположен на хромосоме (рис. 2) и содержит два дополнительных гена ( fixLK ), не выявленных в других штаммах (см. рис. 2).
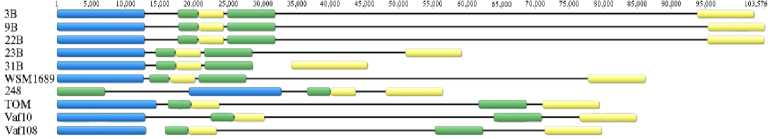
Рис. 1. Различия в структурной организации sym -оперонов в геномах изолятов Rhizobium legu-minosarum . Штаммы 3B, 9B, 22B, 23B, 31B и WSM1689 — R. leguminosarum bv. trifolii (Rlt), Vaf10, Vaf108, TOM и 248 — R. leguminosarum bv. viciae (Rlv). Синим цветом обозначены nod -опероны, зеленым — nif и желтым — fix . Шкала сверху — длина sym -регионов, п.н.
|
VaflO —(E--1 |
1---ВИН |
завоз |
НИВОШ |
— |
|||||||||||
|
1 |
|||||||||||||||
|
VaflOS —Ш----1 том —CD---1 |
ЗиВав |
— |
Виеоишйв |
)— |
|||||||||||
|
1—ЕИНхЕНЖВ—1 |
--Е@ |
0ОВИПВ |
|||||||||||||
|
ноикшты |
ШЕУРйТнш |
||||||||||||||
|
0,0 В I3 BUD |
|||||||||||||||
|
WSM16R9 — |
1------SB)- |
BGBH |
_ |
||||||||||||
|
3B —CD--1 |
'-----ЗИП 1-----ВВП |
11 |
|||||||||||||
|
---M61SJH |
|||||||||||||||
|
^"^^ ^^^T ^" —^ |
i---ВЁяШШЯ-- |
| |^j| |
ВЙГЭЙШПЯ |
-- |
|||||||||||
|
23B —B— - |
1--тар| |
в tuns' |
— |
||||||||||||
|
—JJ— - |
1-----ВВП |
□ВВП |
— |
]--ILlKlNlOtiLPJGlHHW- |
|||||||||||
Рис. 2. Схематическая структура sym -оперонов у изолятов Rhizobium leguminosarum . Штаммы 3B, 9B, 22B, 23B, 31B и WSM1689 — R. leguminosarum bv. trifolii (Rlt), Vaf10, Vaf108, TOM и 248 — R. leguminosarum bv. viciae (Rlv). Синим цветом обозначены nod -опероны, зеленым — nif и желтым — fix . Звездочкой (*) отмечен ген, расположенный в хромосоме.
На хромосомах штаммов Rlt 3B, 9B, 22B и 31B также найден ген nodT , но нет гена fixW . У штамма 23B ген nodT не выявлен, но из всех изученных штаммов Rlt только у него есть ген fixW , расположенный на отдельной плазмиде. В nod -оперонах всех Rlt штаммов обнаружен ген nodX , но отсутствует ген nodO .
Распределение sym -регионов в геноме. У штаммов Rlt структура sym -регионов оказалась разнообразной (см. рис. 1). У 31B в отличие от остальных рассматриваемых штаммов оперон fixNOQPGHIS расположен на хромосоме. У штаммов 3B, 9B и 22B сильно увеличено расстояние между оперонами nifHDKE и fixNOQPGHIS , а у штамма 23B оно
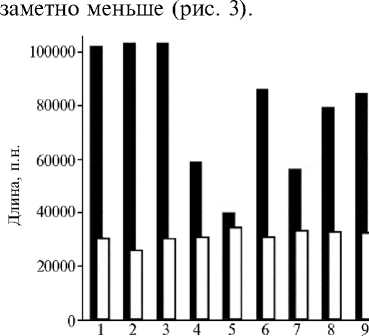
Рис. 3. Соотношение размеров sym -регионов (черные столбики) и sym -генов (белые столбики) в геномах изолятов Rhizobium leguminosarum bv. trifolii (Rlt) (1 — 3B, 2 — 9B, 3 — 22B, 4 — 23B, 5 — 31B, 6 — WSM1689) и R. leguminosarum bv. viciae (Rlv) (7 — 248, 8 — TOM, 9 — Vaf10, 10 — Vaf108).
Кроме того, организация sym -региона у штамма 23B наиболее компактная и сравнима по размерам с таковой у штамма 10 248 (симбионт V. faba) . Размеры sym -региона у штаммов 23B и 31B соответствуют характерным для эволюционно продвинутой группы, для которой типично компактное расположение sym -генов, тогда как штаммы 3B, 9B и 22B, у которых sym -регион расширен, можно отнести к предковой эволюционной группе .
Филогенетический анализ sym -генов. У штаммов Rlt и Rlv, помимо выявленных структурных особенностей симбиотического региона, мы проанализировали нуклеотидный полиморфизм трех групп генов — fix , nif и nod . Филогении соответствующих конкатенатов представлены на рисунке 4. Обособление симбионтов клевера в относительно компактном кластере показано для двух групп генов ( nif и nod ), в то время как штаммы Rlv образуют компактный кластер только по генам fix . Заслуживает внимание и тот факт, что в филогении по генам nif и nod продвинутые симбионты Rlv (248 и TOM) оказываются с достоверной статистической поддержкой в одном кластере с ризобиями клевера, в то время как в филогении по fix генам два штамма Rlt (WSM1689 и 31B) попадают в относительно компактный кластер с группой Rlv, включающей как продвинутые, так и анцестральные штаммы Rlv.
Биовары R. leguminosarum представлены симбионтами двух сильно различающихся триб бобовых. Разделение R. leguminosarum на биовары viciae и trifolii имеет давнюю эволюционную историю, и штаммы этих биоваров не образуют клубеньков при перекрестной инокуляции бобовых из триб Trifolieae и Fabeae. В биоваре viciae выделяются симбионты вави-ловии красивой, обладающие рядом анцестральных признаков, и предполагается, что они наиболее близки к протосимбионту трибы Fabeae — общему предку биовара R. leguminosarum bv. viceae (10). В представленном исследовании при сопоставлении геномов ризобий вавиловии, секвенирование которых было выполнено ранее, и геномов ризобий клевера, изученных в этой работе, нами получены данные, имеющие отношение к общему для всего вида R. leguminosarum протосимбионту, который существовал на эволюционно более раннем этапе — до разделения на биовары viceae и trifolii.
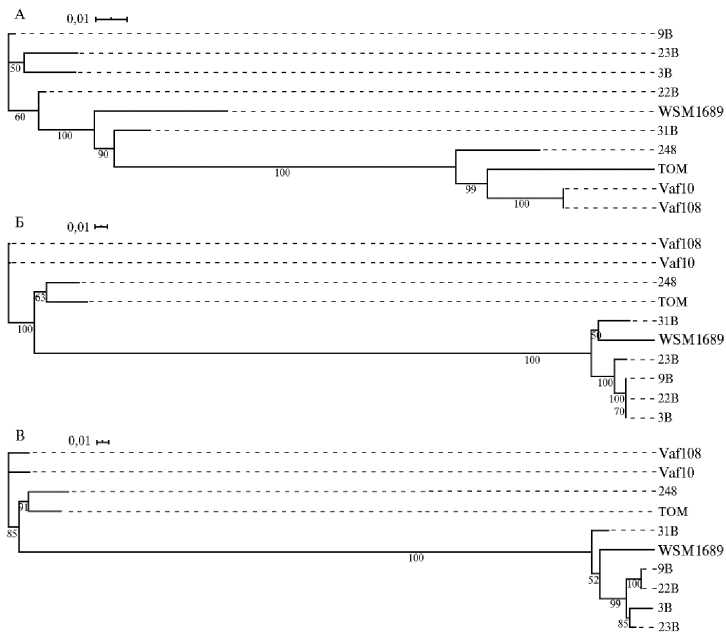
Рис. 4. Дендрограмма, построенная на основании конкатенированных последовательностей генов fixABCGHINOPQ (А) , nifABDEHKN (Б) и nodABCDEFIJLMN (В) из геномов Rhizobium leguminosarum . Штаммы 3B, 9B, 22B, 23B, 31B и WSM1689 — R. leguminosarum bv. trifolii (Rlt), Vaf10, Vaf108, TOM и 248 — R. leguminosarum bv. viciae (Rlv).
Один из анцестральных признаков — существенно больший размер межгенных областей в симбиотическом регионе из-за первичной «грубой» сборки на ранних этапах эволюции. Позднее происходит компактизация этих регионов (10). Важный результат выполненных нами исследований заключается в выявлении у изученных штаммов Rlt 3B, 9B и 22B sym -региона, размер которого намного больше, чем у симбионтов вавиловии (см. рис. 1, табл. 2).
Для всех штаммов Rlt характерно наличие в nod -опероне гена nodX , что у штаммов Rlv также служит признаком, маркирующим анцестральные генотипы (25). Значимость гена nodX для симбиоза с клевером не изучена, а его утрата у продвинутых штаммов Rlv связана с сужением хозяйской специфичности и повышением активности азотофиксации (26).
Еще один анцестральный признак — отсутствие гена nodT, кодирующего efflux-систему, которая обеспечивает эффективное высвобождение nod-фактора из клетки ризобий (27). Как было показано у штаммов Rlv, вероятный эволюционный сценарий связан с рекрутированием этого гена из хромосомы в симбиотический кластер посредством дупликации, неофункционализации и переноса. У штаммов Rlt ген nodT присутствует в одной копии только на хромосоме (исключение — изолят 31B, у которого nodT вообще не выявлен). Таким образом, по этому признаку геномы ри- зобий клевера демонстрируют соответствие еще более ранним этапам эволюции симбиотического кластера.
У штаммов Rlv к анцестральным признакам, связанным с функциональной избыточностью анцестральных генотипов, относится также наличие гена fixW . У изученных симбионтов клевера ген fixW найден только у штамма 31B, но не в fix -опероне на pSym, как у Rlv, а в отдельном несимбиотическом контиге. Функция fixW , как предполагалось ранее, может быть связана с глубокой дифференциацией бактероидов, характерной для ризобий. Проявление fixW и его значение для симбиоза подробно не изучены, но, скорее всего, fixW не влияет на хозяйскую специфичность (28).
Наконец, штаммы Rlt 3B, 9B, 22B и 23B, как и штаммы Rlv Vaf10 и Vaf108, обладают только одной копией генов fixNOPQ на pSym (в хромосоме этот оперон отсутствует) в отличие от остальных представителей Rlv, у которых в геноме две копии fixNOPQ — на pSym и в хромосоме. В случае штамма 31В выявлена одна копия fixNOPQ , но в хромосоме. Гены fixNOQP и их гомологи у грамотрицательных не фиксирующих азот бактерий кодируют высокоаффинную терминальную цитохромоксидазу типа cbb3, которая обеспечивает дыхание в микроаэрофильных условиях (29), например дыхание в клубеньках в условиях симбиоза. Скорее всего, дупликация кластера fixNOQP и его перенос в хромосому происходит на поздних стадиях эволюции R. leguminosarum (30). Предполагаемые эволюционные предшественники R. leguminosarum , имевшие близких общих предков с родом Agrobacterium (31), не имеют fix -генов или их гомологов, что указывает на то, что хромосомные копии fix -генов представляют собой поздние приобретения в эволюционной истории генома R. leguminosarum (10).
Выявленные нами различия в топологии филогений конкатенатов fixABCGHINOPQ , nifABDEHKN и nodABCDEFIJLMN свидетельствуют о независимой эволюции групп генов, контролирующих различные функции. Полученные данные позволяют распространить предположения, высказанные ранее о независимой эволюции этих групп генов внутри биовара viceae (10), на весть вид R. leguminosarum .
2. Анцестральные признаки генома у биотипов Rhizobium leguminosarum , найденные в совокупности (по результатам полногеномного секвенирования 10 штаммов)
|
Биотип Rhizobium |
Расширенный sym -регион |
В nod -опероне нет гена nodT |
Наличие nodX |
Наличие fixW |
Нет хромосомной копии fixNOPQ |
|
R. leguminosarum bv. trifolii |
+ |
+ |
+ |
+ |
|
|
R. leguminosarum bv. viciae |
+ |
+ |
+ |
+ |
+ |
П р и м е ч а н и е. «+» или «–» — соответственно наличие или отсутствие признака. Изученные штаммы: 3B, 9B, 22B, 23B, 31B и WSM1689 — R. leguminosarum bv. trifolii (Rlt), Vaf10, Vaf108, TOM и 248 — R. legumi-nosarum bv. viciae (Rlv).
Итак, анализ результатов полногеномного секвенирования показал, что из рассматриваемых штаммов Rhizobium leguminosarum bv. trifolii как минимум три (3B, 9B и 22B) обладают большой частью анцестраль-ных признаков (расширенный sym-регион, отсутствие хромосомной копии fixNOPQ и гена nodT в nod-опероне, наличие nodX), выявленных у ри-зобий вавиловии красивой Vavilovia formosa (Stev.) Fed. Однако эволюционная интерпретация полученных данных осложняется тем, что механизмы эволюции ризобий R. leguminosarum bv. viciae и R. leguminosarum bv. trifolii, несомненно, определяются не только особенностями растений-хозяев и их филогенеза, но и историей продвижения этих растений в раз- личные эколого-географические зоны. Не исключено, что признаки, идентифицированные в одной группе ризобий как анцестральные, в другой группе могут иметь иной эволюционный смысл. Несмотря на эти затруднения один из интересных результатов выполненного исследования, по нашему мнению, состоит в предположении, что ризобии клевера вместе с ризобиями вавиловии могут быть близки к протосимбионту R. legumi-nosarum. Такое допущение мы рассматриваем как одну из рабочих гипотез для дальнейших исследований.
Список литературы Выявление анцестральных характеристик генома у Rhizobium leguminosarum bv. Trifolii
- Jordan D.C. Family III. Rhizobiaceae. In: Bergey's manual of systematic bacteriology /N.R. Krieg, J.G. Holt (eds.). Williams and Wilkins, Baltimore, 1984: 234-242.
- Young J.P.W., Johnston A.W.B. The evolution of specificity in the legume-rhizobium symbiosis. Trends in Ecology & Evolution, 1989, 4(11): 341-349 ( DOI: 10.1016/0169-5347(89)90089-X)
- Göttfert M. Regulation and function of rhizobial nodulation genes. FEMS Microbiology Reviews, 1993, 10(1-2): 39-63 ( ). DOI: 10.1111/j.1574-6968.1993.tb05863.x
- Debellé F., Moulin L., Mangin B., Dénarié J., Boivin C. Nod genes and Nod signals and the evolution of the Rhizobium legume symbiosis. Acta Biochimica Polonica, 2001, 48(2): 359-365. ( ). DOI: 10.18388/abp.2001_3921
- Shamseldin A. The role of different genes involved in symbiotic nitrogen fixation. Global Journal of Biotechnology & Biochemistry, 2013, 8(4): 84-94 ( ). DOI: 10.5829/idosi.gjbb.2013.8.4.82103
- Fischer H.M. Genetic regulation of nitrogen fixation in rhizobia. Microbiological Reviews, 1994, 58(3): 352-386.
- Provorov N.A., Andronov E.E. Evolution of root nodule bacteria: reconstruction of the speciation processes resulting from genomic rearrangements in a symbiotic system. Microbiology, 2016, 85: 131-139 ( ).
- DOI: 10.1134/S0026261716020156
- Rey F.E., Harwood C.S. FixK, a global regulator of microaerobic growth, controls photosynthesis in Rhodopseudomonas palustris. Molecular Microbiology, 2010, 75(4): 1007-1020 ( ).
- DOI: 10.1111/j.1365-2958.2009.07037.x
- Wongdee J., Boonkerd N., Teaumroong N., Tittabutr P., Giraud E. Regulation of nitrogen fixation in Bradyrhizobium sp. strain DOA9 involves two distinct NifA regulatory proteins that are functionally redundant during symbiosis but not during free-living growth. Frontiers in Microbiology, 2018, 9: 1644 ( ).
- DOI: 10.3389/fmicb.2018.01644
- Chirak E.R., Kimeklis A.K., Karasev E.S., Kopat V.V., Safronova V.I., Belimov A.A., Aksenova T.S., Kabilov M.R., Provorov N.A., Andronov E.E. Search for ancestral features in genomes of Rhizobium leguminosarum bv. Viciae strains isolated from the relict legume Vavilovia formosa. Genes, 2019, 10(12): 990 ( ).
- DOI: 10.3390/genes10120990
- Mikič A., Smýkal P., Kenicer G., Vishnyakova M., Sarukhanyan N., Akopian J., Vanyan A., Gabrielyan I., Smýkalová I., Sherbakova E., Zorić L., Atlagić J., Zeremski-Škorić T., Ćupina B., Krstić Ð., Jajić I., Antanasović S., Ðorđević V., Mihailović V., Ivanov A., Ochatt S., Ambrose M. The bicentenary of the research on ‘beautiful' vavilovia (Vavilovia formosa), a legume crop wild relative with taxonomic and agronomic potential. Bot. J. Linn. Soc., 2013, 172(4): 524-531 ( ).
- DOI: 10.1111/boj.12060
- Биологическое разнообразие клубеньковых бактерий в экосистемах и агроценозах. Теоретические основы и методы (монография) /Под ред. М.Л. Румянцевой, Б.В. Симарова. СПб, 2011.
- Somasegaran P., Hoben H.J. Isolating and purifying genomic DNA of Rhizobia using a large-scale method. In: Handbook for Rhizobia: methods in legume-rhizobium technology. Springer, New York, 1994: 279-283 ( ).
- DOI: 10.1007/978-1-4613-8375-8_31
- Wick R.R., Judd L.M., Holt K.E. Deepbinner: Demultiplexing barcoded Oxford Nanopore reads with deep convolutional neural networks. PLoS Computational Biology, 2018, 14(11): e1006583 ( )
- DOI: 10.1371/journal.pcbi.1006583
- Seemann T. Prokka: rapid prokaryotic genome annotation. Bioinformatics, 2014, 30(14): 2068-2069 ( ).
- DOI: 10.1093/bioinformatics/btu153
- Reeve W., Ardley J., Tian R., Eshragi L., Yoon J.W., Ngamwisetkun P., Seshadri R., Ivanova N.N., Kyrpides N.C. A genomic encyclopedia of the root nodule bacteria: assessing genetic diversity through a systematic biogeographic survey. Standards in Genomic Sciences, 2015, 10: 14 ( ).
- DOI: 10.1186/1944-3277-10-14
- Terpolilli J., Rui T., Yates R., Howieson J., Poole P., Munk C., Tapia R., Han C., Markowitz V., Tatiparthi R., Mavrommatis K., Ivanova N., Pati A., Goodwin L., Woyke T., Kyrpides N., Reeve W. Genome sequence of Rhizobium leguminosarum bv. trifolii strain WSM1689, the microsymbiont of the one flowered clover Trifolium uniflorum. Standards in Genomic Sciences, 2013, 9: 527-539 ( ).
- DOI: 10.4056/sigs.4988693
- Kumar S., Stecher G., Li M., Knyaz C., Tamura K. MEGA X: molecular evolutionary genetics analysis across computing platforms. Molecular Biology and Evolution, 2018, 35(6): 1547-1549 ( ).
- DOI: 10.1093/molbev/msy096
- Guindon S., Dufayard J.F., Lefort V., Anisimova M., Hordijk W., Gascuel O. New algorithms and methods to estimate maximum-likelihood phylogenies: assessing the performance of PhyML 3.0. Systematic Biology, 2010, 59(3): 307-321 ( ).
- DOI: 10.1093/sysbio/syq010
- Schwarz G. Estimating the dimension of a model. Annals of Statistics, 1978, 6(2): 461-464 ( ).
- DOI: 10.1214/aos/1176344136
- Letunic I., Bork P. Interactive tree of life (iTOL) v3: an online tool for the display and annotation of phylogenetic and other trees. Nucleic Acids Research, 2016, 44(W1): W242-W245 ( ).
- DOI: 10.1093/nar/gkw290
- Safronova V.I., Kimeklis A.K., Chizhevskaya E.P., Belimov A.A., Andronov E.E., Pinaev A.G., Pukhaev A.R., Popov K.P., Tikhonovich I.A. Genetic diversity of rhizobia isolated from nodules of the relic species Vavilovia formosa (Stev.). Fed. Antonie van Leeuwenhoek, 2014, 105: 389-399 ( ).
- DOI: 10.1007/s10482-013-0089-9
- Макашева Р.Х., Дрозд А.М., Адамова О.П., Голубев А.А. Многолетний горох. Сборник трудов по прикладной ботанике, генетике и селекции, 1973, 51(1): 44.
- Ma S.W., Iyer V.N. New field isolates of Rhizobium leguminosarum biovar Viciae that nodulate the primitive pea cultivar Afghanistan in addition to modern cultivars. Applied and Environmental Microbiology, 1990, 56(7): 2206-2212.
- Kimeklis A.K., Chirak E.R., Kuznetsova I.G., Sazanova A.L., Safronova V.I., Belimov A.A., Onishchuk O.P., Kurchak O.N., Aksenova Т.S., Pinaev A.G., Andronov E.E., Provorov N.A. Rhizobia isolated from the relict legume Vavilovia Formosa represent a genetically specific group within Rhizobium leguminosarum biovar viciae. Genes, 2019, 10(12): 991 ( ).
- DOI: 10.3390/genes10120991
- Provorov N., Tikhonovich I. Genetic resources for improving nitrogen fixation in legume-rhizobia symbiosis. Genetic Resources and Crop Evolution, 2003, 50: 89-99 (doi: 10.1023/A:1022957429160).
- Alvarez-Ortega C., Olivares J., Martínez J.L. RND multidrug efflux pumps: What are they good for? Frontiers in Microbiology, 2013, 4: 7 ( ).
- DOI: 10.3389/fmicb.2013.00007
- Hontelez J.G., Lankhorst R.K., Katinakis P., van den Bos R.C., van Kammen A. Characterization and nucleotide sequence of a novel gene fixW upstream of the fixABC operon in Rhizobium leguminosarum. Mol. Gen. Genet., 1989,218: 536-544 ( ).
- DOI: 10.1007/BF00332421
- Talbi C., Sanchez C., Hidalgo-Garcia A., González E.M., Arrese-Igor C., Girard L., Bedmar E.J., Delgado M.J. Enhanced expression of Rhizobium etli cbb3 oxidase improves drought tolerance of common bean symbiotic nitrogen fixation. Journal of Experimental Botany, 2012, 63(14): 5035-5043 ( ).
- DOI: 10.1093/jxb/ers101
- Kopat V.V., Chirak E.R., Kimeklis A.K., Safronova V.I., Belimov A.A., Kabilov M.R., Andronov E.E., Provorov N.A. Evolution of fixNOQP genes encoding cytochrome oxidase with high affinity to oxygen in Rhizobia and related bacteria. Russ. J. Genet., 2017, 53: 766-774 ( ).
- DOI: 10.1134/S1022795417070067
- Berrada H., Fikri-Benbrahim K. Taxonomy of the rhizobia: current perspectives. British Microbiology Research Journal, 2014, 4(6): 616-639 ( ).
- DOI: 10.9734/BMRJ/2014/5635


