Взаимодействие лидерного пептида CMLAL с рибосомой E. coli, связавшей хлорамфеникол в неканоническом сайте
Автор: Макаров Г.И.
Журнал: Вестник Южно-Уральского государственного университета. Серия: Химия @vestnik-susu-chemistry
Рубрика: Физическая химия
Статья в выпуске: 2 т.16, 2024 года.
Бесплатный доступ
Методами докинга, взвешенной и равновесной молекулярной динамики была получена структура комплекса 70S A/A,P/P-рибосомы E. coli , связавшей антибиотик хлорамфеникол в неканоническом сайте недалеко от пептидилтрансферазного центра в присутствие известного стоп-пептида к нему - ClmAL. В ней хлорамфеникол удерживается в рибосоме за счет вклинивания своего нитрофенильного остатка в полость между остатками Ψ2504 и U2506 23S рРНК и образования гидрофобных контактов с ними, а также водородных связей с остатками G2505 и G2061 23S рРНК. Лидерный пептид ClmAL при этом образует множество стабильных водородных связей с остатками G2061, m2A2503, U2609 и C2610 23S рРНК в рибосомном туннеле. Молекулярно-динамическое моделирование этого тройного комплекса показало, что механизм действия антибиотика заключается в индуцировании расхождения субстратов пептидилтрансферазной реакции друг относительно друга на расстояние, исключающее реакцию транспептидации. Это расхождение субстратов пептидилтрансферазной реакции стабилизировалось взаимодействиями между аминогруппой боковой цепи остатка лизина в А-сайте с одной стороны и основанием C2063 и хлорамфениколом - с другой. При этом α-аминогруппа остатка лизина образует водородную связь с карбонильной группой остатка Ala-7 CmlAL. Это, совместно со специфическими взаимодействиями остатков пептида CmlAL с остатками рРНК рибосомного туннеля, объясняет останов трансляции в присутствие хлорамфеникола именно на данной последовательности и, как следствие, присутствие данной последовательности в генах, кодирующих белок-транспортер CmlA, отвечающий за резистентность рибосом к данному антибиотику.
Рибосома, хлорамфеникол, cmlal, стоп-пептид, молекулярная динамика
Короткий адрес: https://sciup.org/147244658
IDR: 147244658 | УДК: 577.217.347, | DOI: 10.14529/chem240219
Текст научной статьи Взаимодействие лидерного пептида CMLAL с рибосомой E. coli, связавшей хлорамфеникол в неканоническом сайте
Один из способов обеспечения устойчивости к хлорамфениколу, используемый бактериальными клетками, заключается в применении белка-транспортёра CmlA, который откачивает хлорамфеникол из клетки [1–3]. Как и в случае других систем устойчивости к антибиотикам, CmlA вырабатывается клеткой не постоянно, но только в присутствии хлорамфеникола. Трансляция CmlA регулируется лидерным пептидом CmlAL, подобно тому, как лидерные пептиды семейства Erm регулируют синтез соответствующей метилтрансферазы [4]. В мРНК старт-кодон лидерного пептида CmlAL доступен рибосоме, а последовательность Шайн – Дальгарно и старт-кодон регулируемого белка CmlA скрыт вторичной структурой, содержащей три шпильки (рис. 1). В отсутствие хлорамфеникола синтез лидерного пептида CmlAL начинается и проходит до конца, при этом вторичная структура мРНК не нарушается, так что синтез CmlA не идёт. В присутствии же хлорамфеникола CmlAL останавливает свой биосинтез, что вызывает конформационные изменения в мРНК, приводящие к открытию старт-кодона CmlA и его синтезу [5, 6].
Биохимические эксперименты, использующие метод обратной транскрипции на мРНК (toeprinting), установили, что в присутствии хлорамфеникола синтез CmlAL останавливается на последовательности M 1 STSKNAD 8 так, что следующий остаток лизина остаётся в A-сайте рибосомы [7]. Более того, эти эксперименты показали, что в присутствии обоих тРНК и пептида в рибосомном туннеле хлорамфеникол также присутствует в пептидилтрансферазном центре или верхней части рибосомного туннеля, однако каноническое связывание хлорамфеникола, установленное методами рентгеноструктурного анализа [8, 9], при этом невозможно. Однако хлорамфеникол может связываться в неканоническом сайте связывания, будучи при этом способен взаимодействовать с растущим пептидом [10]. Теперь мы предприняли попытку исследовать структуру возникающего при этом тройного комплекса пептид – рибосома – хлорамфеникол методами молекулярно-динамического моделирования.
Экспериментальная часть
Моделируемая система. Мы использовали структуру A/A,P/P-состояния рибосомы E. coli , смоделированную в работе [11]. Из этой структуры были выделены все остатки, у которых хотя бы один атом попадал в тетрагональную область 9 × 9 × 10 нм, включающую в себя весь пепти-дилтрансферазный центр и рибосомный туннель таким образом, что центр этой области находился в тоннеле, а туннель пролегал вдоль мысленной оси аппликат. При этом в ней также был установлен в неканоническом сайте связывания хлорамфеникол, так же, как было найдено в [10]:
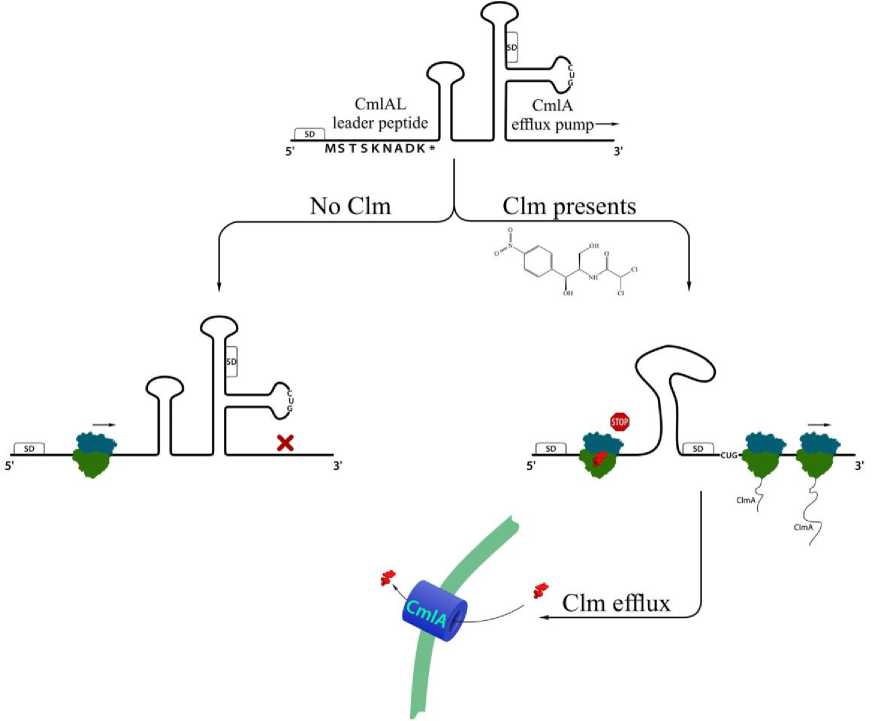
Рис. 1. Механизм регуляции трансляции белка-транспортёра CmlA лидерным пептидом CmlA между основаниями Ψ2504 и U2506. В этой системе устанавливали стоп-пептид CmlAL (M1STSKNAD8) в конформациях, предсказанных докингом в программе rDock [12]. В процессе докинга C-конец пептида был фиксирован, всего было выполнено 20000 запусков оптимизационного процесса – 20 попыток по 1000 запусков каждая, причём при каждой попытке генератор случайных чисел инициализировался случайным числом.
Моделируемая система помещалась в тетрагональную ячейку размерами 13,2 × 13,2 × 12,2 нм, заполненную молекулами воды TIP4PEW [13] таким образом, что грани были покрыты слоем воды около 0,9 нм. Остаточный отрицательный заряд был скомпенсирован ионами калия [14], причем располагали их вблизи отрицательно заряженных групп [15] (скрипт был любезно предоставлен А. Залевским). Молекулы воды были частично заменены ионами калия, магния и хлорид-анионами для достижения концентрации MgCl 2 в 7 мМ и KCl в 100 мМ, что препятствовало вымыванию координированных РНК ионов магния и калия из рибосомы в водную фазу. Суммарно итоговая система содержала 153 иона магния, 922 ионов калия, 127 хлорид-анионов. В ходе молекулярно-динамических симуляций все остатки, чьи атомы находились не далее 0,1 нм от грани моделируемого фрагмента рибосомы, были позиционно ограничены. Этот подход сохраняет локальную подвижность остатков рРНК стенок рибосомного тоннеля, необходимую для подстройки к связыванию лиганда.
При моделировании комплекса пептида SecM с полноразмерной A/A,P/P-рибосомой E. coli мы устанавливали пептид в смоделированной в конформации в указанную выше структуру рибосомы из [11] и хлорамфеникол по образцу структуры из [10]. При этом мы пользовались полученной в этой работе полной симуляционной ячейкой размером 23,7 × 26,7 × 24,7 нм, содержащей не только A/A,P/P-рибосому с координированными ионами, но и уравновешенную молекулярно–динамическими симуляциями воду и растворённые ионы в указанных выше концентрациях. При этом удалялись молекулы воды, перекрывающиеся с устанавливаемым пептидом.
Для оценки a priori взвешивающего потенциала для пептида CmlAL мы выполнили расчёт уравновешенной метадинамики с обменом потенциалами для системы, состоящей из позиционно ограниченного CCA-конца тРНК, ацилированного CmlAL, в солевом растворе с указанными выше концентрациями ионов. Размер симуляционной ячейки при этом составлял 2 × 4,5 × 2 нм.
Расчеты проводились при помощи пакета GROMACS [16, 17] версии 5.1.4, анализ полученных траекторий проводился при помощи версии 2019.4 этого же пакета. Молекулярно- механические модели канонических и неканонических нуклеотидных и аминокислотных остатков строились с использованием силового поля AMBER–ff14SB [18]. Пространственные структуры и молекулярные электростатические потенциалы для неканонических остатков биополимеров, подлежащих включению в силовое поле, получались при помощи квантово-химических расчетов методом HF/6–31G*. Точечные заряды были получены при помощи метода RESP [19].
Моделирование производилось при температуре 310 К, поддерживаемой термостатом масштабирования скоростей с добавочным стохастическим членом [20] с периодом привязки 0,1 пс и периодическими граничными условиями с изотропным постоянным давлением, полученным при помощи баростата Берендсена [21] с периодом привязки равным 5 пс. Электростатические взаимодействия обрабатывались с помощью сети частиц Эвальда [22] с шагом сетки 0,125 нм и четвертым порядком интерполяции. Координаты записывались в файл траектории каждые 25 пс, а шаг интегрирования составлял 2 фс. Состав и протяжённость полученных траекторий показан в табл. 1. Длины связей атомов водорода ограничивались при помощи алгоритма LINCS [23].
Методы анализа траекторий. Анализ траекторий, получаемых при молекулярнодинамическом моделировании, включал визуальный анализ посредством программы PyMOL, поиск и оценку частот встречаемости водородных связей и стэкинг-взаимодействий согласно геометрическим критериям, описанным в работе [24], кластеризацию полученных конформаций моделируемой системы методом GROMOS [25] и расчет энергий нековалентных взаимодействий между группами атомов, понимаемых как суммы энергий ван-дер-ваальсовых и кулоновских взаимодействий между ними:
E noncov = E VdW + E Coulomb .
Анализ полученных траекторий также включал сравнение с траекторией для рибосомы E. coli , пребывающей в каноническом A/A,P/P-состоянии, полученной ранее в работе [11]. Далее она будет обозначаться как «AP E. Coli» .
Таблица 1
Количество и протяжённость траекторий, полученных при моделировании взаимодействий лидерного пептида CmlAL с рибосомой E. coli , пребывающей в каноническом A,A/P,P-состоянии
|
Операция |
Количество траекторий |
Протяжённость, нс |
|
Равновесная молекулярная динамика конформаций CmlAL, найденных докингом |
6 |
100 |
|
Уравновешенная метадинамика с обменом потенциалами CmlAL в растворе |
8 |
400 |
|
Уравновешенная метадинамика с обменом потенциалами с a priori взвешивающим потенциалом |
1 |
200 |
|
Уравновешенная метадинамика с обменом потенциалами с a posteriori взвешивающим потенциалом |
1 |
400 |
|
Моделирование основных кластеров из уравновешенной метадинамики |
6 |
200 |
|
Моделирование комплекса CmlAL с 70S рибосомой в A,A/P,P-состоянии |
2 |
400 |
Обсуждение результатов
Мы выполнили докинг лидерного пептида CmlAL (M1STSKNAD8) в рибосомный туннель фрагмента рибосомы, описанного выше, зафиксировав C-конец пептида на 3’-конце P/P–тРНК. При этом хлорамфеникол находился в неканоническом сайте связывания. Всего было выполнено 20000 запусков оптимизационного процесса – 20 попыток по 1000 запусков каждая, причём при каждой попытке генератор случайных чисел инициализировался случайным числом, для того чтобы достичь вырождения выборки конформаций. Из них мы выбрали 6 конформаций, характеризующихся наивысшим сродством к рибосомному туннелю.
Для выбранных конформаций лидерного пептида CmlAL была рассчитана равновесная молекулярная динамика протяжённостью 100 нс, предназначенная для того, чтобы оценить стабильность предсказанной докингом конформации. При этом на сахарофосфатный остов остатков РНК были наложены позиционные ограничения; эта мера ограничила конформационную подвижность рибосомного туннеля, поддерживая целостность комплекса рибосома–хлорамфеникол и оставляя конформационную подвижность, связанную с движением гетероциклических оснований нуклеотидных остатков. На основании анализа взаимодействий рибосома – хлорамфеникол – CmlAL была отобрана поза № 6, так как для неё при сохранении положения хлорамфеникола в неканоническом сайте связывания наблюдались наиболее интенсивные взаимодействия между CmlAL с одной стороны и хлорамфениколом и рибосомным туннелем – с другой стороны. Центроид наиболее населённого кластера, выделенного из траектории позы № 6, использовался для дальнейших расчётов.
Чтобы уточнить решение, найденное докингом и эволюционировавшее в ходе равновесной молекулярной динамики, необходимо сгенерировать различные конформации моделируемой системы, определяемые главным образом взаимодействием аминокислотных остатков лидерного пептида CmlAL со стенками рибосомного туннеля. Удобным методом решения этой задачи является моделирование молекулярной динамики со взвешенным ансамблем. Взвешивающие потенциалы для лидерного пептида CmlAL готовились нами следующим образом. Поскольку лидерный пептид CmlAL содержит всего 8 остатков, мы смогли построить априорный взвешивающий потенциалы V apriori для торсионных углов φ и ψ CmlAL достаточно детально, выполнив моделирование уравновешенной метадинамики с обменом потенциалами для CmlAL, закреплённого на CCA-концевом тринуклеотиде, в растворе. Симуляция протяжённостью 400 нс была выполена на восьми репликах, каждая из которых подстраивала компенсирующий потенциал для углов φ и ψ соответствующего аминокислотного остатка. Ширина гауссиан составляла 10 градусов, взвешивающий коэффициент составлял 8, гауссовы функции высотой 1 кДж/моль добавлялись к взвешивающим потенциалам каждые 2500 шагов (5 пс). В результате этого расчёта мы оценили взвешивающие потенциалы V aPOr ( ф,- , V, ) для каждого аминокислотного остатка CmlAL, учитывающие их конформационные предпочтения, связанные, в том числе, и с взаимодействиями остатков CmlAL друг с другом. Взвешивающие потенциалы использовались в форме матриц 360 х 360 значений V p'Or ( ф i , v , ) , при расчётах интерполируемых сплайнами.
С этими a priori взвешивающими потенциалами было выполнено моделирование молекулярной динамики со взвешенным ансамблем протяжённостью в 200 нс, в ходе которой значения торсионных углов φ i и ψ i записывались каждые 20 фс, а на сахарофосфатный остов остатков РНК так же были наложены позиционные ограничения. Полученная выборка величин торсионных углов использовалась для расчёта поправленных взвешивающих потенциалов У аР0рпоп ( ф , , V , ) :
V bzro' ( ф , , v , ) =RT in P ( ф i , V i ) +V£T ( 9 , , V , ) . (2)
Поправленные взвешивающие потенциалы VpppSter'or ( ф, , ф ) , учитывающие также и взаимодействия CmlAL со стенками рибосомного туннеля и окружения, использовались при следующем моделировании молекулярной динамики со взвешенным ансамблем протяжённостью в 400 нс с теми же позиционными ограничениями и частотой записи значений торсионных углов. Полученные в обоих расчётах взвешенной молекулярной динамики состояния были объединены и кластеризованы методом GROMOS с уровнем отсечки 0,1 нм по координатам тяжелых атомов CmlAL. Центроиды 6 наиболее населённых кластеров были вовлечены в дальнейшие расчёты равновесной молекулярной динамики.
Для каждой из отобранных конформаций системы была проведена равновесная динамика. Сначала на протяжении 20 нс имевшиеся в ранее проводимых расчётах позиционные ограничения сахарофосфатного остова РНК линейно ослаблялись до полного исчезновения, затем следовало 200 нс равновесной молекулярной динамики. Полученные состояния были объединены и кластеризованы методом GROMOS с уровнем отсечки 0,12 нм по координатам тяжелых атомов CmlAL, хлорамфеникола и ближайших остатков 23S рРНК. При этом пять наиболее населённых кластеров включают 99 % состояний, причём наиболее населённый кластер – 44 %. Для каждого кластера были проанализированы встречаемости водородных связей и стэкинг-взаимодействий, а также рассчитаны энергии нековалентных взаимодействий между хлорамфениколом, лидерным пептидом и остатками рибосомного туннеля. Конформации CmlAL в центроидах первого и второго по населённости кластеров были выбраны для дальнейшего моделирования с полноразмерной рибосомой E. coli на том основании, что в них в наибольшей степени сохраняются взаимодействия, характерные для хлорамфеникола, находящегося в неканоническом сайте связывания, а пептид CmlAL образует множество стабильных взаимодействий с рибосомным туннелем.
Лидерный пептид CmlAL, находящийся в конформациях, соответствующих центроидам выделенных выше первого и второго кластера, был установлен в рибосомный туннель полноразмерной A/A,P/P-рибосомы E. coli , из которой выделялись использованные выше фрагменты рибосомы. Для каждой из построенных таким образом систем были рассчитаны траектории равновесной МД протяжённостью 400 нс. Траектория, рассчитанная для системы, построенной с конформацией CmlAL из первого кластера, будет далее называться I траекторией, а траектория, рассчитанная для системы с конформацией CmlAL из второго кластера, – II траекторией.
Состояния, содержащиеся в анализируемых участках I и II траекторий, были кластеризованы методом GROMOS, для каждой траектории в отдельности. Кластеризация производилась по конформации пептида SecM и нуклеотидных и аминокислотных остатков, слагающих рибосомный туннель, причём атомы водорода не учитывались. Дальнейший анализ проводился для состояний, принадлежащих к наиболее населённым кластерам, и центроидов этих кластеров. Наиболее населённый кластер и его центроид, извлечённый из I траектории, будет называться I кластером и I конформацией, аналогично для II траектории – II кластер и II конформация.
Во II траектории хлорамфеникол покидает неканонический сайт связывания, поэтому мы исключили её из дальнейшего рассмотрения. Таким образом, именно I конформация может рассматриваться как структура тройного комплекса хлорамфеникол – CmlAL – рибосома.
В этой структуре нитрофенильный остаток хлорамфеникола располагается между основаниями Ψ2504 и U2506, образуя с ними гидрофобные контакты. Его гидроксильные группы образуют водородные связи с 2’-гидроксильной группой остатка G2505 и основанием G2061 (табл. 2). При этом карбонильная группа хлорамфеникола образует водородную связь с боковой аминогруппой остатка лизина, этерифицирующей A-тРНК (рис. 2). Лидерный пептид CmlAL при этом образует множество стабильных водородных связей с остатками рибосомного туннеля, контактируя с основаниями G2061, m2A2503, U2609 и C2610 (рис. 3).
Таблица 2
Водородные связи в тройном комплексе хлорамфеникол – CmlAL – рибосома, % состояний
|
Донор |
Акцептор |
Встречаемость, % состояний |
|
Clm/O3–H |
50S/G2061/O6 |
10 |
|
Clm/O1–H |
50S/G2505/O 4’ |
99 |
|
A–tRNA/Lys/N ε –H |
Clm/O 4 |
63 |
|
A–tRNA/Lys/N–H |
CmlAL/Ala7/O |
69 |
|
50S/G2061/N2–H |
CmlAL/Ser4/O β |
83 |
|
50S/C2063/O 2′ –H |
CmlAL/Asp8/O δ |
46 |
|
50S/C2063/O 2′ –H |
CmlAL/Asp8/O δ′ |
30 |
|
50S/m2A2503/N6–H |
CcmlAL/Ser4/O β |
53 |
|
50S/U2584/N3–H |
CmlAL/Asn6/O δ |
94 |
|
CmlAL/Met1/N–H |
50S/U2609/O2′ |
20 |
|
CmlAL/Met1/N–H |
50S/C2610/O4′ |
53 |
|
CmlAL/Ser2/N–H |
50S/C2610/O2 |
52 |
|
CmlAL/Asp8/N–H |
50S/U2585/O 4′ |
87 |
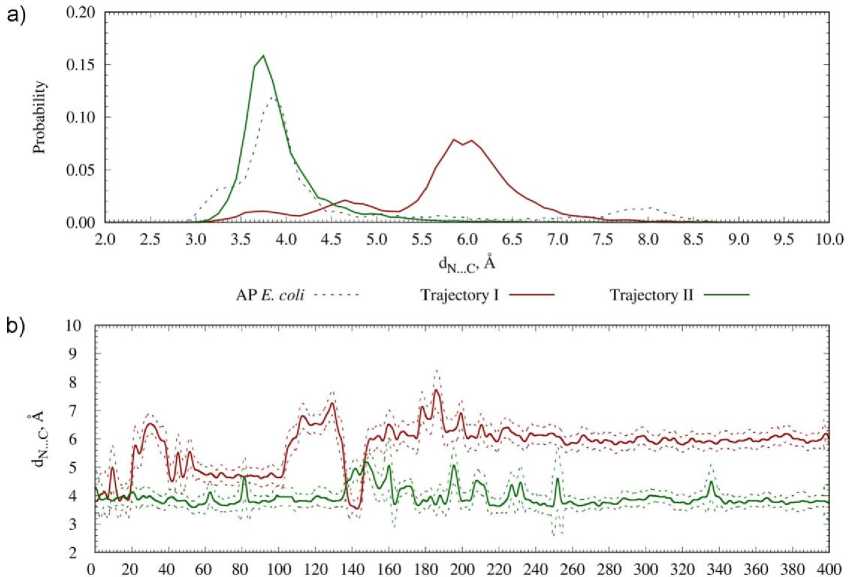
В I конформации расходятся субстраты пептидилтрансферазной реакции – аминогруппа остатка лизина, этерифицирующего 3’-конец A/A-тРНК, и карбонильный атом кислорода остатка аспарагиновой кислоты, находящегося на C-конце CmlAL и этерифицирующего 3’-конец P/P-тРНК. Хотя в начальных состояниях они сближены в обеих траекториях, в I траектории примерно через 20 нс субстраты начинают расходиться, и к 160 нс расстояние между их реакционными центрами стабилизируется на уровне 6Å, что хорошо видно по анализу распределения вероятности для этой величины (рис. 4). Это расхождение субстратов пептидилтрансфе-
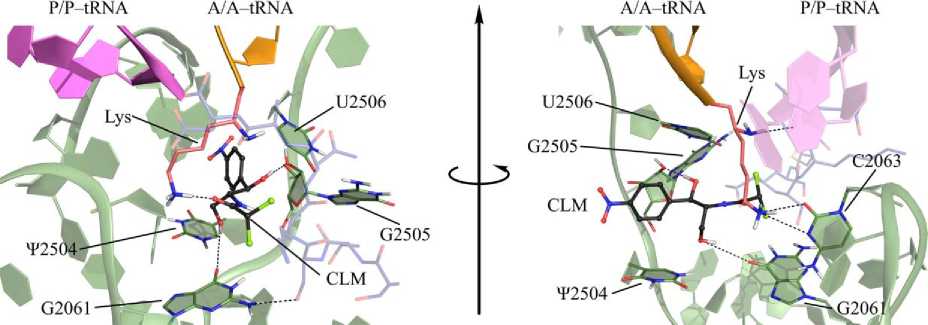
Рис. 2. Взаимодействие хлорамфеникола (черные стержни), связанного в неканоническом сайте, с A/A,P/P-рибосомой E. coli (зелёный), несущей лидерный пептил CmlAL, по данным молекулярно-динамического моделирования траектории I. Розовыми стержнями показан остаток лизина, ацилирующий A/A-тРНК.
Чёрным пунктиром показаны водородные связи
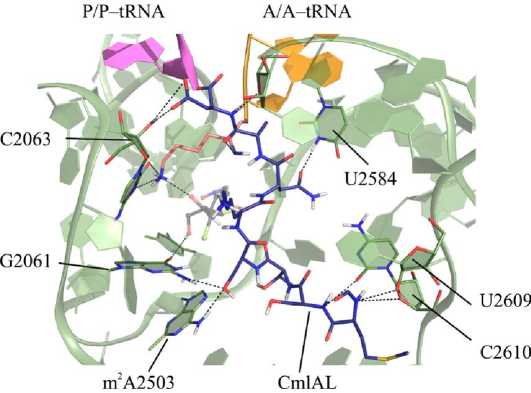
Рис. 3. Взаимодействия лидерного пептида CmlAL (синие стержни) с A/A,P/P-рибосомой E. coli (зелёный), по данным молекулярнодинамического моделирования траектории I. Розовыми стержнями показан остаток лизина, ацилирующий A/A-тРНК, чёрными стержнями – хлорамфеникол. Чёрным пунктиром показаны водородные связи разной реакции в тройном комплексе стабилизируется стабильными взаимодействиями между аминогруппой боковой цепи остатка лизина с одной стороны и основанием C2063 и хлорамфениколом – с другой. При этом α-аминогруппа остатка лизина образует водородную связь с карбонильной группой остатка Ala-7 CmlAL (рис. 5). Интересно заметить, что во II траектории, в которой хлорамфеникол покидает неканонический сайт связывания и выходит в рибосомный туннель, реакционные центры субстратов пептидилтрансферазной реакции остаются сближены на 4 Å, что соответствует ван-дер-ваальсову контакту между ними, наблюдающемуся также и в траекториях AP E. coli.
Заключение
Приступая к моделированию структуры тройного комплекса хлорамфеникол – CmlAL – рибосома, мы ожидали выявить специфические взаимодействия между хлорамфениколом и лидерным пептидом, но так и не нашли их при анализе результатов моделирования. Тем не менее, мы обнаружили стабильные взаимодействия между аминогруппой боковой цепи приходящего остатка лизина с одной стороны и основанием C2063 и хлорамфениколом – с другой. С их помощью связавшийся в неканоническом сайте хлорамфеникол понуждает приходящий остаток
Time, ns
Trajectory I ------- Trajectory II
Рис. 4. a - сравнение распределений плотности вероятности расстояния dC ... N между траекториями I и II, а также с AP E. Coli ; b - изменение расстояния dC ... N в траекториях I и II; сплошная линия изображает скользящее среднее, полученное свёрткой с гауссовой функцией с σ = 2 нс, а пунктир – скользящее стандартное отклонение
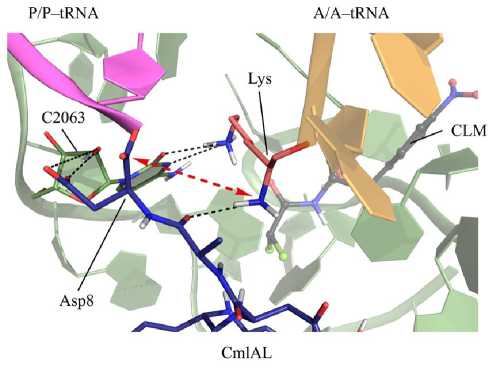
Рис. 5. Конформация субстратов пептидилтрансферазной реакции по данным молекулярно - динамического моделирования траектории I.
Чёрным пунктиром показаны водородные связи, красная стрелка показывает расстояние между атакующей аминогруппой остатка лизина и атакуемым карбонильным атомом остатка аспарагиновой кислоты лизина принять конформацию, препятствующую его нуклеофильной атаке на C-конец CmlAL, и таким образом останавливает его биосинтез. Специфическая же аминокислотная последовательность CmlAL требуется для прочного взаимодействия с рибосомным туннелем, делающего возможным уклонение приходящего остатка лизина, а не для прочного взаимодействия с хлорамфениколом.
Список литературы Взаимодействие лидерного пептида CMLAL с рибосомой E. coli, связавшей хлорамфеникол в неканоническом сайте
- Bissonnette L., Champetier S., Buisson J.P. et al. // Journal of Bacteriology. 1991. V. 173, No. 14. P. 4493. DOI: 10.1128/jb.173.14.4493-4502.1991.
- Edgar R., Bibi E. // Journal of Bacteriology. 1997. V. 179, No. 7. P. 2274. DOI: 10.1128/jb.179.7.2274-2280.1997.
- Bischoff K.M., White D.G., McDermott P.F. et al. // Journal of Clinical Microbiology. 2002. V. 40, No. 2. P. 389. DOI: 10.1128/jcm.40.2.389-394.2002.
- Arenz S., Meydan S., Starosta A. et al. // Mol. Cell. 2014. V. 56, P. 446. DOI: 10.1016/j.molcel.2014.09.014.
- Lovett P.S., Rogers E.J. // Microbiological Reviews. 1996. V. 60, No. 2. P. 366. DOI: 10.1128/mr.60.2.366-385.1996.
- Lovett P. S. // Gene. 1996. V. 179, No. 1. P. 157. DOI: 10.1016/s0378-1119(96)00420-9.
- Marks J., Kannan K., Roncase E.J. et al. // Proc. Natl. Acad. Sci. U.S.A. 2016. V. 113, No. 43. P. 12150. DOI: 10.1073/pnas.1613055113.
- Jack A. Dunkle J., Xiong L., Mankin A. et al. // Proc. Natl. Acad. Sci. U.S.A. 2010. V. 107, No. 40. P. 17152. DOI: 10.1073/pnas.1007988107.
- Bulkley D., Innis C.A., Blaha G. et al. // Proc. Natl. Acad. Sci. U.S.A. 2010. V. 107, No. 40. P. 17158. DOI: 10.1073/pnas.1008685107.
- Makarov G., Makarova T. // Biochim. Biophys. Acta. 2018. V. 1862, No. 12. P. 2940. DOI: 10.1016/j.bbagen.2018.09.012.
- Makarova T., Bogdanov A. // Biochimie. 2019. V. 167, P. 179. DOI: 10.1016/j.biochi.2019.09.019.
- Ruiz-Carmona S., Alvarez-Garcia D., Foloppe N. et al. // PLoS Comput Biol. 2014. V. 10, No. 4. P. e1003571. DOI: 10.1371/journal.pcbi.1003571.
- Horn H.W., Swope W.C., Pitera J.W. et al. // The Journal of Chemical Physics. 2004. V. 120, No. 20. P. 9665. DOI: 10.1063/1.1683075.
- Joung I.S., Cheatham T.E. // J. Phys. Chem. B. 2008. V. 112, No. 30. P. 9020. DOI: 10.1021/jp8001614.
- Athavale S., Petrov A., Hsiao C. et al. // Plos One. 2012. V. 7, P. 1. DOI: 10.1371/journal.pone.0038024.
- van der Spoel D., Lindahl E., Hess B. et al. // J. Comput. Chem. 2005. V. 26, P. 1701. DOI: 10.1002/jcc.20291.
- van der Spoel D., Lindahl E., Hess B. et al. // J. Chem. Theory Comput. 2008. V. 4, P. 435. DOI: 10.1021/ct700301q.
- Maier J.A., Martinez C., Kasavajhala K. et al. // Journal of Chemical Theory and Computation. 2015. V. 11, No. 8. P. 3696. DOI: 10.1021/acs.jctc.5b00255.
- Bayly C.I., Cieplak P., Cornell W. et al. // J. Phys. Chem. 1993. V. 97, No. 40. P. 10269. DOI: 10.1021/j100142a004.
- Bussi G., Donadio D., Parrinello M. // J. Chem. Phys. 2007. V. 126, P. 014107. DOI: 10.1063/1.2408420.
- Berendsen H., Postma J., van Gunsteren W. et al. // J. Chem. Phys. 1984. V. 81, P. 3684. DOI: 10.1063/1.448118.
- Darden T., York D., Pedersen L. // J. Chem. Phys. 1993. V. 98, P. 10089. DOI: 10.1063/1.464397.
- Hess B., Bekker H., Berendsen H.J.C. et al. // J. Comput. Chem. 1997. V. 18, No. 12. P. 1463. DOI: 10.1002/(SICI)1096-987X(199709)18:12<1463::AID-JCC4-3.0.CO;2-H.
- Makarov G.I., Sumbatyan N.V., Bogdanov A.A. // Biochemistry (Moscow). 2017. V. 82, No. 8. P. 925. DOI: 10.1134/S0006297917080077.
- Daura X., Gademann K., Jaun B. et al. // Angewandte Chemie International Edition. 1999. V. 38, No. 1-2. P. 236. DOI: 10.1002/(SICI)1521-3773(19990115)38:1/2<236::AID-ANIE236-3.0.CO;2-M.


