Взаимодействие нуклеиновых кислот с молекулами воды, белков и интеркаляторов (обзор)
Автор: Чесноков Ю.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Обзоры, проблемы
Статья в выпуске: 3 т.56, 2021 года.
Бесплатный доступ
Современные представления о межмолекулярных взаимодействиях в клетке неполны без понимания того, как формируются комплексы между нуклеиновыми кислотами и основными внутриклеточными компонентами - водой и белками и что определяет пространственную стабилизацию таких комплексов. То же справедливо в отношении интеркаляции - внутриклеточного межмолекулярного взаимодействия веществ планарной структуры, способных внедряться между соседними парами азотистых оснований в молекулы ДНК и РНК, которое играет особую роль в фармакологии и генетическом мутагенезе. Кроме того, интеркаляция, может оказывать сильное влияние на клеточный метаболизм, замедляя, а в некоторых случаях прекращая рост клеток, что в определенных условиях приводит как к апоптозу, так и к раковым заболеваниям либо, наоборот, к выздоровлению от такого рода заболеваний (M. Ashrafizadeh с соавт., 2020). Настоящий обзор посвящен рассмотрению молекулярных механизмов и биологической роли этих процессов. Известно, что двойная спираль ДНК может взаимодействовать с полипептидами при помощи образования специфических водородных связей между Уотсон-Криковскими парами оснований и боковыми цепями аминокислот (C.N. Pace с соавт., 2004), посредством интеркаляции боковых цепей ароматических аминокислот между парами оснований, при которой также проявляется некоторая специфичность (A. Bazzoli с соавт., 2017), и за счет непосредственного связывания белковых α-спиралей и β-слоев в желобках ДНК (E. Del Giudice с соавт., 2009). Предполагается, что последний тип взаимодействия имеет место, например, в комплексах ДНК с cro -репрессором экспрессии генной активности и с белком, активирующим катаболизм, для которого предложены две модели связывания α-спиралей с левосторонней и правосторонней двойной спиралью ДНК в В-форме. Указывается, что если известна структура молекулы нуклеиновой кислоты, то величину поверхности ДНК и РНК, доступной для молекул воды или иных растворителей, можно определить расчетным способом. При этом в случае сворачивания ДНК в растворе в двойную спираль ее молекула становится полярной. При такого рода гидратации вокруг молекулы ДНК образуются две гидратные оболочки. Первая из них, состоящая примерно из 20 молекул воды в расчете на один нуклеотид, непроницаема для катионов и по своей агрегатной структуре не похожа на лед, а вторая оболочка неотличима от обычной воды. Различия в структуре гидратных оболочек проливают свет на природу конформационного перехода между формами В - А, происходящего при уменьшении гидратации молекулы ДНК. Описано также взаимодействие нуклеиновых кислот с молекулами лекарственных и иных планарных веществ. При этом в обзоре рассмотрены только интеркаляционные комплексы с препаратами, молекулы которых имеют плоскую структуру или обладают плоскими функциональными группами. Продемонстрировано, что связывание таких веществ с двойной спиралью протекает в две стадии: на первой происходит их присоединение по периферии спирали, на второй осуществляется интеркаляция, то есть собственно встраивание интеркалятора в планарной плоскости между парами нуклеотидов. Такого рода интеркаляция сопровождается раскручиванием и удлинением спирали нуклеиновой кислоты, а также увеличением ее жесткости. В соответствии с принципом исключения ближайших мест связывания, согласно которому оно не происходит у каждого ближайшего соседа вдоль оси двойной спирали ДНК из-за пространственных ограничений, которые определяет стереометрия нуклеотидов, примыкающих к интеркаляторам, молекулы интеркаляторов заполняют лишь половину таких мест. В целом описанные в работе взаимодействия нуклеиновых кислот с молекулами воды, белков и интеркаляторов указывают на биологическую значимость такого рода взаимоотношений, поскольку, как известно, стабильность и регулярность процессов репликации и экспрессии генов играет важнейшую роль в практическом осуществлении взаимодействия генотип-среда, а также реализации генетической информации на молекулярном уровне.
Нуклеиновые кислоты, а-днк, в-днк, конформационные переходы, молекулы воды, гидратация днк, белки, лиганды, планарные интеркаляторы, межмолекулярные взаимодействия, репликация, генная экспрессия
Короткий адрес: https://sciup.org/142231361
IDR: 142231361 | УДК: 577.113.7:577.323.3:547.96:57.044 | DOI: 10.15389/agrobiology.2021.3.434rus
Текст обзорной статьи Взаимодействие нуклеиновых кислот с молекулами воды, белков и интеркаляторов (обзор)
Современные представления о межмолекулярных взаимодействиях в клетке неполны без понимания того, как формируются комплексы между нуклеиновыми кислотами и основными внутриклеточными компонентами — водой и белками и что определяет пространственную стабилизацию таких комплексов. В воде растворены все биоорганические компоненты и вещества внутри клетки. Это высокополярный растворитель, необходимый для всех живых организмов (1-3). Нуклеиновые кислоты хорошо растворяются в воде (4, 5). В ней происходят все основные биохимические реакции, поэтому роль воды в химической структуре и функционировании живых систем невозможно переоценить. В качестве реагента вода вовлечена во многие метаболические процессы. Так, гидролиз белков, жиров и углеводов происходит при непосредственном участии воды, а при гидролизе АТФ выделяется энергия, необходимая для осуществления энергетически невыгодных ферментативных реакций (6-9). В жидком состоянии вода практически не сжимается и, как следствие, служит своеобразным каркасом клетки. Ее молекулы характеризуются высокой когезионной прочностью (сцеплением между молекулами в объеме тела), что, в свою очередь, характеризует способность противостоять внешним воздействиям. Благодаря своим осмотическим свойствам вода создает избыточное давление внутри вакуолей растительных клеток (10). Это тургорное давление позволяет клеточной стенке сохранять эластичность и форму органов (например, листьев). Молекула воды — электрически нейтральный диполь, который имеет частичный положительный заряд в области атомов водорода, хотя внутри молекулы он распределен неравномерно. Атом кислорода (как более электроотрицательный, чем атомы водорода) оттягивает электронную плотность, образуя частично отрицательный заряд. Благодаря своей структуре молекулы воды могут взаимодействовать друг с другом и с другими биоорганическими молекулами внутри клетки посредством водородных связей (11, 12). Все это определяет важность воды как одного из основных элементов клетки в процессе ее функционирования, включая реализацию генетической информации на молекулярном и клеточном уровне.
Белки — еще один компонент, который играет важную роль в клетке (13, 14). Белки выполняют функцию не только строительного материала внутри клеток, но и позволяют реализовывать метаданные, закодированные в основном носителе генетической информации — нуклеиновых кислотах. Белки — это азотсодержащие высокомолекулярные органические соединения, мономерами которых служат аминокислоты. Белки характеризуются сложной пространственной структурой, которая имеет первичный, вторичный, третичный и четвертичный уровни организации. Для выполнения своих биологических функций белки принимают одну или несколько конкретных пространственных конфигураций благодаря нековалентным водородным, ионным, гидрофобным связям и другим межмолекулярным взаимодействиям (15-18). Сложность строения белковых молекул связана прежде всего с разнообразием их функций. В то же время структура молекул зависит от свойств окружающей среды и межмолекулярных взаимодействий. Взаимодействие белков с нуклеиновыми кислотами играет особую роль в жизненном цикле любого организма, поскольку именно эти взаимодействия обеспечивают постоянство и воспроизводимость генетической информации, закодированной в ДНК и РНК. Однако, как показывает практика, для белков и нуклеиновых кислот пространственные стереохимические механизмы взаимодействия до конца не выяснены, хотя их понимание особенно важно для возможности управления такого рода межмолекулярными взаимодействиями (19-21).
Еще один внутриклеточный межмолекулярный процесс, привлекающий внимание и играющий особую роль в фармакологии и генетическом мутагенезе, — это интеркаляция (22, 23). Некоторые молекулы (так назы- ваемые лиганды) могут связываться с ДНК несколькими способами. Лиганды обычно либо внедряются в молекулу ДНК в плоскость между азотистыми основаниями в двойной спирали, либо связываются с ней электростатически или ковалентно. Для того чтобы произошло интеркалирование, лиганд должен иметь соответствующий размер и химическую природу. Обычно интеркалирующие агенты — это ароматические полициклические молекулы, имеющие плоскую пространственную структуру. В химиотерапевтической фармакологии интеркалирующие лиганды используют в качестве агентов, которые ингибируют репликацию ДНК, чтобы остановить рост раковых клеток. Например, для лечения болезни Ходжкина применяют даунорубицин и адриамицин (24, 25), для лечения саркомы Юинга — дактиномицин (26). Антибиотик актиномицин D, внедряясь между соседними парами азотистых оснований в молекуле ДНК, ограничивает связывание РНК-полимеразы с матрицей ДНК и тем самым предотвращает взаимодействие фермента с цепью ДНК (27). В молекулярно-биологических исследованиях интеркалирующие лиганды применяют для флуоресцентного окрашивания молекул нуклеиновых кислот или для мутагенеза. Например, бромистый этидий обычно используется при электрофоретическом разделении нуклеиновых кислот в агарозном или полиакриламидном геле (28), а акридиновый оранжевый или акридиновый желтый — для индуцирования хромосомных мутаций, например делеции, приводящей к потере средней части хромосом, либо для окрашивания ядер клеток in vivo или исследования биологических мембран (29, 30).
В этом обзоре мы кратко рассмотрим межмолекулярные механизмы взаимодействия нуклеиновых кислот с водой, белками и интеркалирую-щими веществами, а также важность таких взаимодействий, играющих ключевую роль в стабильности и регуляции процессов репликации и экспрессии генов при практической реализации взаимодействия генотип—среда, реализации генетической информации на молекулярном уровне и в обеспечении постоянства и воспроизводимости генетической информации.
В ода и нуклеиновые кислоты. Излишне говорить, насколько важна роль водного окружения нуклеиновых кислот. Вода в клетке — это не просто среда, содержащая определенные вещества. Вода взаимодействует с растворенными молекулами, и именно она в основном стабилизирует вторичную и третичную структуру макромолекул (11, 31, 32). Это относится и к белкам, и к ДНК, причем к ДНК, возможно, даже в большей степени, поскольку высокая диэлектрическая проницаемость воды и гидратированных противоионов ослабляет электростатическое отталкивание фосфатов (33-35). Молекулы воды также принимают участие в процессе самосборки азотистых оснований в упорядоченные структуры, поскольку этот процесс во многом обусловлен гидрофобными взаимодействиями. Степень гидратации ДНК имеет решающее значение для ее конформации: при высокой относительной влажности среды ДНК находится в B-форме (основная часть нативной клеточной ДНК находится в этой форме) (рис. 1), уменьшение влажности (или увеличение ионной силы раствора) приводит к переходу ДНК из B- в C-, A- или (если позволяет первичная структура) в D- и Z-формы (32, 36).
Гидратация нуклеиновых кислот играет важную роль в формировании их пространственной структуры и отвечает за конформационные переходы A ↔ B (отметим, что в клетке А-форма ДНК образуется при транскрипции, обратной транскрипции и при отжиге РНК-праймеров) (37, 38).
Рис. 1. Сайты предпочтительного связывания молекул воды с B-формой ДНК (по 42). Цифры от 1 до 5 указывают порядок уменьшения прочности связи. Рядом с фосфатной группой находится примерно пять молекул воды.
Вокруг молекулы ДНК формируются две гидратные оболочки. Первая из них (примерно 20 молекул воды на нуклеотид) непроницаема для катионов. Структура этого слоя не похожа на кристаллическую решетку воды в составе льда — твердого агрегатного состояния воды. Вторая оболочка неотличима от обычной жидкой воды. Если структура нуклеиновых кислот известна, то доступную для молекул растворителя поверхность ДНК или РНК можно определить расчетным путем. Результаты таких расчетов объясняют некоторые особенности поведения ДНК в растворе и, в частности, свидетельствуют, что при сворачивании в двойную спираль молекула ДНК становится более полярной. Рентгеноструктурный анализ монокристаллов олигонуклеотидов показал, что в областях, состоящих из АТ-пар, в малой бороздке В-ДНК образуется гребень из молекул воды, связанных с азотистыми основаниями водородными связями (39). В A-ДНК образуются водные «нити», которые перекрестно соединяют фосфатные группы, лежащие на краях основной бороздки. Такие различия в строении гидратных оболочек проливают свет на природу перехода B → A, происходящего при уменьшении содержания воды в среде и снижении гидратированности молекулы ДНК. В кристаллах некоторых циклодекстринов и комплекса d(CpG) с профлавином обнаружены 4-, 5-и 6-членные циклические структуры, которые образуют соединенные водородными связями атомы кислорода или О-Н-группы (40, 41). Не исключено, что подобные структуры входят в гидратные оболочки макромолекул, в том числе нуклеиновых кислот.
При высокой активности воды, когда катионы не нарушают первичную гидратную оболочку, состоящую примерно из 20 молекул воды на нуклеотид, ДНК находится в B-форме. По мере уменьшения относительной влажности в волокне или пленке или увеличения концентрации соли в растворе степень гидратации также уменьшается. При определенном пороговом значении G (параметр, который определяет сольватацию или гидратацию макромолекулы, которая в случае ДНК равна количеству молей воды на моль нуклеотидов, что соответствует примерно 20 молекулам воды на нуклеотид) наблюдается структурный переход ДНК из B-формы в C- или A-форму в зависимости от природы присутствующего противоиона. Переход B → C происходит «непрерывно» (43, 44), как и должно быть в случае двух структурно близких форм. Что же касается переходов B → A и C → A, то из-за конформационного изменения углеводного остатка С2-эндо ^ Сз‘-эндо они должны происходить скачком, кооперативно.
В водных растворах спираль В-ДНК немного раскручена и угол ее поворота меньше, чем у В-ДНК в волокнах, получаемых для изучения обезвоженных молекул ДНК. Однако по мере увеличения концентрации соли происходит некооперативный внутрисемейственный структурный переход B → C. Когда концентрация соли достигает определенного значения, происходит резкий кооперативный межсемейственный переход C → A или
B → A. Такие кооперативные переходы (B → A или C → A) также возникают при изменении полярности среды, например при добавлении в систему этанола, изопропанола или диоксана до концентрации ∼ 80 % (45-47). В то же время, когда образуется двойная спираль, ДНК становится более полярной. Другими словами, если в раскрученной вытянутой молекуле ДНК фосфаты составляют ∼ 20 % площади поверхности, основания — ∼ 50 % и сахара — ∼ 30 %, то в двойной спирали ДНК на фосфаты приходится 45 % поверхности, на основания — 20 %, на сахара — 35 %. Таким образом, полярность молекулы ДНК увеличивается при образовании двойной спирали. То же наблюдается в белках при образовании глобулярных структур: полярные группы располагаются на поверхности, а неполярные гидрофобные — внутри. В целом можно сказать, что доступность поверхности ДНК для растворителя определяет ее свойства. Оценки доступной площади поверхности ДНК показывают, что основания B-ДНК открыты в основном со стороны большой бороздки, а основания A-ДНК — со стороны малой. Следовательно, когда белок взаимодействует с интактной двойной спиралью, специфичные взаимодействия между парами оснований и боковыми группами аминокислот в случае B-формы будут происходить в большой бороздке, а в случае A-формы — в малой. Это согласуется с данными о взаимодействии B-ДНК со специфическими белками — например, с РНК-полимеразы Escherichia coli с lac-промотором, lac-репрессором с lac-оператором, а также с данными о λ - и cro -репрессорах и взаимодействии ДНК с гистонами в нуклеосомах (48-50).
Взаимодействие белков и нуклеиновых кислот. Взаимодействия между белками и нуклеиновыми кислотами происходят на всех стадиях репликации и экспрессии ДНК и лежат в основе многочисленных регуляторных процессов, поэтому роль таких взаимодействий чрезвычайно важна. Тем не менее наши знания о молекулярных механизмах этих взаимодействий все еще ограничены. У нас нет четкого представления о том, как эндонуклеазы рестрикции связываются с ДНК и разрезают ее в определенных местах. Нет полной информации о геометрии распознавания операторного участка репрессором и о том, как аминоацил-тРНК-синтетазы распознают «свои» тРНК (51). Основная трудность в изучении этих сложных систем состоит в том, что необходимо одновременно наблюдать каждую из взаимодействующих макромолекул. В то же время спектроскопические методы, за редким исключением, дают неадекватные результаты, а кристаллизация комплексов белков с нуклеиновыми кислотами сопряжена со многими трудностями. Тем не менее в начале 1980-х годов прошлого века появились публикации, авторам которых удалось получить специфичный комплексы белков с ДНК (52, 53) и тРНК (54) в форме, пригодной для рентгеноструктурного анализа.
Чтобы упростить процесс изучения столь сложных систем, исследования модельных соединений проводятся с использованием как теоретических, так и экспериментальных подходов. Основная цель таких исследований — установить специфичность распознавания четырех типов оснований нуклеиновых кислот боковыми группами 20 аминокислот. В этих экспериментах используются как мономерные компоненты обоих партнеров взаимодействия, так и полимеры, в некоторых случаях синтетические. Также изучается связывание нуклеотидов (ингибиторов или коферментов) в активных центрах ферментов. Были получены кристаллы нескольких белков, распознающих определенные последовательности ДНК, и описана их пространственная структура. Это, пожалуй, максимум, что можно сделать, не 438
прибегая к анализу кристаллов специфичных ДНК-белковых комплексов. Такие структуры дают представление об общих принципах взаимодействий нуклеиновых кислот и белков, но последнее слово в конечном итоге остается за исследованиями самих комплексов (55, 56).
Если мы внимательно проанализируем структуру боковых групп аминокислот и полипептидного остова, мы увидим, что существует четыре потенциально возможных типа взаимодействий между белками и нуклеиновыми кислотами (57-59). Первый тип — это солевые мостики между фосфатами и положительно заряженными аминокислотными группами (Nξ-аминогруппа лизина, гуанидиновая группа аргинина и протонированный остаток His). Второй тип — водородные связи между фосфатами, сахарами, основаниями нуклеиновых кислот и пептидными группами или гидрофильными боковыми цепями аминокислотных остатков. Третий тип представляют стэкинг-взаимодействия между боковыми группами ароматических аминокислот (Trp, Tyr, Phe, His) и основаниями. Наконец, четвертый тип — это гидрофобные взаимодействия между основаниями нуклеиновых кислот и боковыми группами неполярных аминокислот. Энергия этих четырех типов взаимодействия обычно уменьшается в том порядке, в котором они перечислены. Поскольку основную роль играет притяжение противоположных зарядов, чрезвычайно важны данные о распределении зарядов в боковых цепях аминокислот и в пептидной группе. Их необходимо сравнить с аналогичными данными для компонентов нуклеиновых кислот, и тогда можно будет предсказать некоторые взаимодействия. Однако в действительности наблюдается более сложная ситуация, поскольку многочисленные слабые взаимодействия могут подавлять специфические взаимодействия типа заряд—заряд.
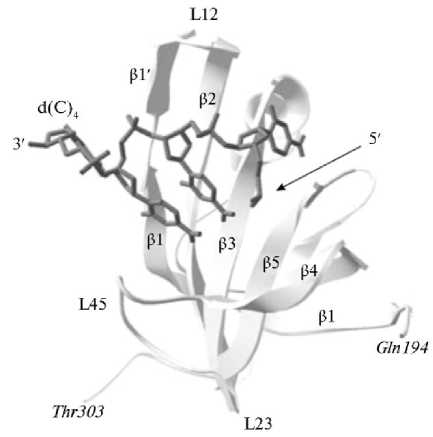
Рис. 2. Пример связывания нуклеиновых кислот «прямой полярности» в третичной структуре комплекса OB-домена p70A (a.b. 194-303) hsRPA с олигонуклеотидным остатком d(C)4 (выделено темно-серым) (по 63).
Двойная спираль ДНК может взаимодействовать с полипептидами несколькими способами: посредством образования специфических водородных связей между парами оснований Уотсона-Крика и боковыми цепями аминокислот; посредством интеркаляции боковых цепей ароматических аминокислот между парами оснований (модель «закладки»), которая также проявляет некоторую специфичность; за счет прямого связывания белковых α-спиралей и β-складок в бороздках ДНК. Предполагается, что последний тип взаимодействия (правильнее сказать — два типа взаимодействия) имеет место в комплексах ДНК с cro-репрессором (в этой модели α- спираль и β-складчатая структура белка попадают соответственно в большую и малую бороздки двойной спирали ДНК) и с белком, активирующим катаболизм, для чего были предложены две модели связывания α-спира-лей — с левосторонней и правосторонней двойной спиралью B-ДНК (60).
На основании данных о кристаллической структуре преальбумина был предложен гипотетический комплекс преальбумина с ДНК (61). Однако на самом деле такой комплекс, по-видимому, не образуется (62). И полилизин, и полиаргинин необратимо связываются с ДНК; в обоих случаях процесс присоединения кооперативный, но структура соответствующих комплексов различна.
Еще один пример — связывание нуклеиновых кислот «прямой полярности» в третичной структуре комплекса OB-домена, связывающего олигосахариды и олигонуклеотиды p70A hsRPA с олигонуклеотидным остатком d(C)4 (рис. 2). Полученные β -складки располагаются ортогонально друг другу и образуют β -цилиндр с топологией цепи β 1- β 2- β 3- β 5- β 4- β 1. Обычно каноническая поверхность взаимодействия OB-упаковки с лигандами находится в области цепей β 2 и β 3. Кроме того, взаимодействие может включать петли между β 1 и β 2 (петля L12), β 3 и α (петля L3a), α и β 4 (петля La4) и, наконец, между β 4 и β 5 (петля L45). Эти петли образуют желоб, который проходит по поверхности домена перпендикулярно оси топологического β -цилиндра (63).
Нуклеотиды и одноцепочечные РНК и ДНК при связывании с белками обычно принимают удлиненную форму, то есть торсионный угол γ для них лежит в области - ck или an . Такое изменение угла γ происходит, когда NAD+ связывается с дегидрогеназами, когда динуклеотидфосфаты связываются с рибонуклеазами A и S и когда РНК связывается с белком вируса табачной мозаики (64). Взаимодействия в комплексе белок-нуклеиновая кислота могут представлять собой контакты любого типа и влиять на любую часть молекулы каждого партнера. Например, в случае специфического комплекса рибонуклеазы Т1 с гуаниловой кислотой гуанин распознается за счет образования водородной связи с основной цепью пептида и укладки с боковой группой тирозина. Оказалось, что взаимодействия между нуклеотидами и атомами белкового остова, как правило, более распространены и более специфичны, чем взаимодействия с боковыми группами аминокислот (65).
Связывание ДНК с белком гена 5 фага fd способствует расхождению цепей двойной спирали. Если (одноцепочечная) ДНК и белок образуют комплекс, то есть если ДНК прикреплена к активному центру белка, который состоит исключительно из элементов β -структуры, начинается образование агрегатов белка. В этом случае белковые молекулы выстраиваются одна за другой, образуя спираль, на которую наматываются одноцепочечные участки ДНК (66).
Интеркаляция. ДНК как носитель генетической информации взаимодействует со многими лекарствами, канцерогенами и мутагенными веществами, а также с красителями, характерная особенность которых — наличие протяженных (гетеро)циклических ароматических хромофоров. К таким веществам относятся акридины (желтый и оранжевый), профлавин, бромистый этидий, эллиптицин, 3,5,6,8-тетраметил-N-метилфенантролин, 2-гидроксиэтантиолат-2,2′,2″- терпиридин-платина (III), дауномицин, актиномицин и ряд других (рис. 3). Поскольку ДНК играет ключевую роль в процессах репликации и биосинтеза белка, ее модификация при взаимодействии с этими соединениями оказывает сильное влияние на клеточный метаболизм, замедляя, а в некоторых случаях останавливая рост клеток. Все эти свойства упомянутых выше соединений вызвали к ним большой интерес, особенно возросший за последние десятилетия. Открыта возможность их использования в медицине; они также нашли широкое применение при изучении структуры и функций ДНК (67, 68). Одна из категорий таких соединений состоит из веществ, которые приводят к химической модификации ДНК (мишенью в первую очередь служит гуанин), другая — из веществ, которые связываются с ее двойной спиралью. Связывание происходит либо на периферии молекулы, либо (как в случае лекарственных препаратов да-уномицина и актиномицина) посредством интеркаляции между соседними парами оснований без нарушения спаривания Уотсона-Крика (рис. 4). Взаимодействие ДНК с лекарствами имеет большое значение для фармакологии (69). Если рассматривать только комплексы интеркаляции с лекарствами с плоскими группами, то связывание таких лекарств двойной спиралью происходит в два этапа: на первом они прикрепляются по периферии спирали, на втором происходит интеркаляция, т.е. сопровождается раскручиванием и удлинением спирали, а также увеличением ее жесткости. В соответствии с принципом исключения ближайших сайтов связывания, молекулы интеркалятора заполняют только половину таких сайтов.
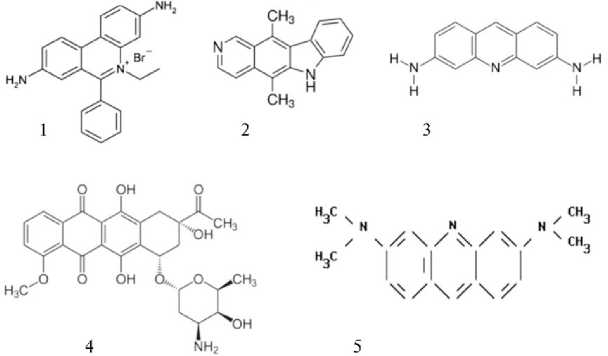
Рис. 3. Примеры некоторых лекарств и красителей, которые образуют комплексы с нуклеиновыми кислотами по типу интеркаляции: 1 — бромид этидия; 2 — эллиптицин; 3 — профлавин; 4 — дауномицин; 5 — акридин оранжевый.
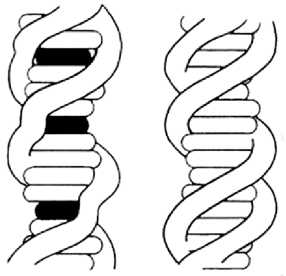
Рис. 4. Внедрение плоских молекул интеркалирующего агента (отмечен черным) в двойную спираль ДНК. Регулярная структура сахарофос-фатного остова (справа) нарушена в местах интеркаляции (слева).
Во всех известных кристаллических комплексах интеркаляторы расположены между двумя парами оснований в спирали Уотсона-Крика, образованными (в случае рибо-и дезоксирибозида) самокомплементарными динуклеозидмонофосфатами, в которых нуклеозид на 5′-конце всегда представлен пиримидином, а на 3′-конце — пурином. Если основания перевернуты, то либо кристаллы не образуются, либо комплекс будет иметь неспиральную структуру. Эта специфическая зависимость интеркаляции от последовательности была обнаружена и в водных растворах и получила теоретическое обоснование. Было показано, что как для ДНК, так и для РНК при интеркаляции в последовательность пи-римидин-3′,5′-пурина происходит гораздо бо- лее сильное перекрытие оснований с интеркалятором, чем при интеркаляции в последовательность пурин-3′,5′-пиримидин (70). В первом случае интеркаляция оказывается на 7-13 ккал/моль более энергетически выгодной, чем во втором (71). Это межмолекулярное взаимодействие стабилизируется внутримолекулярными электростатическими силами, которые также обеспечивают предпочтительную вставку в последовательность пи-римидин-3′,5′-пурин. Кроме того, в случае обратной последовательности могут возникать стерически неблагоприятные контакты, которые усложняют интеграцию.
При интеркаляции изменяются физические свойства двойных спиралей. Впервые предположение о возможном встраивании планарных ароматических молекул между соседними парами оснований было сделано на основании результатов гидродинамических и рентгеноструктурных исследований ДНК в присутствии акридиновых красителей (72). Если добавить акридиновый краситель в раствор ДНК, а затем подготовить обезвоженный препарат ДНК и облучить его рентгеновскими лучами, будет получена дифракционная решетка, на которой отражения регулярной спиральной структуры размываются и исчезают. Остаются только экваториальные рефлексы, свидетельствующие о регулярной упаковке молекул вдоль волокна, и сильный меридиональный рефлекс (0,34 нм), соответствующий межплоскостному расстоянию между парами (72, 73). Вообще говоря, при встраивании ароматических молекул толщиной 0,34 нм сама по себе схема укладки молекулы ДНК не нарушается. Однако для того, чтобы произошла интеркаляция, пары оснований должны раздвинуться, и это приведет к изменению геометрии сахаро-фосфатного остова и разрушению регулярной спиральной структуры (см. рис. 4). В соответствии с предлагаемой концепцией молекула ДНК должна удлиняться по мере добавления красителя в систему. Именно это подтверждают экспериментальные данные о повышении вязкости и уменьшении коэффициента седиментации обработанных препаратов ДНК (72). Оба эти эффекта также предполагают увеличение жесткости двойной спирали ДНК.
Интеркаляция приводит к раскручиванию спирали ДНК — пары оснований должны раздвинуться, чтобы освободить место для интеркалятора. Это осуществляется за счет одновременного растяжения двойной спирали В-ДНК вдоль оси ее раскручивания, что необходимо для того, чтобы саха-рофосфатный остов не разорвался при растяжении (73). О том, что раскручивание действительно происходит, свидетельствуют результаты экспериментов по интеркаляции различных соединений в плазмидную ДНК — кольцевую замкнутую двойную спираль, образующую правую суперспираль (67, 74). При интеркаляции наблюдается раскручивание двойной спирали (10-20 ° для каждой интеркалированной молекулы), при котором число правых суперспиралей уменьшается до тех пор, пока не будет достигнуто равновесие, то есть пока ДНК не станет кольцом без суперспиралей. При дальнейшем интеркалировании двойная спираль продолжает раскручиваться, и в результате образуется левая суперспираль. Этот процесс легко наблюдать по изменению коэффициента седиментации (75).
В наших экспериментах эффект интеркалирующих красителей и лазерного излучения регистрировали как по раскручиванию двойных спиралей нативного фага и высокомолекулярной растительной ДНК, так и по направленному образованию одно- и двухцепочечных разрывов для увеличения частоты и спектра рекомбинации для повышения генотипической изменчивости при селекции различных видов растений (76-79). В результате было обнаружено, что красители-интеркаляторы образуют устойчивый комплекс, который при облучении лазерным светом передает энергию от донора-интеркалятора к ДНК-акцептору, что приводит не только к раскручиванию двойных цепей ДНК, но и к образованию одно- и двухцепочечных разрывов. В то же время на участках, где не происходила интеркаляция, структура ДНК не изменяется и ДНК сохраняет способность к репликации, при этом интеркалирующие красители могут подавлять нуклеазную активность. Таким образом достигается не только мутагенный, но и рекомбиногенный эффект, который приводит к индукции генотипической изменчивости у растений и может быть использован в практике генетической селекции.
Примечательно, что при интеркаляции в двойные спирали А-типа все углеводные остатки могут оставаться в С з ‘ - эндо -конформации. Расхождение пар в основном связано с изменением конформационных углов каркаса Z и Y , значения которых смещены в область an (антипланарная); в этом случае процесс протекает без раскручивания и приемлемыми для образующихся структур, по-видимому, являются комплексы профлавина с CpG и с 5-йод-CpG (80, 81). Поскольку конформация и ориентация сахаров существенно не меняются, можно предположить, что при интеркаляции в A-ДНК и A-РНК принцип исключения ближайших сайтов связывания не выполняется, то есть физически невозможна интеркаляция в сайты, ближайшие к сайтам, уже имеющим интеркаляцию.
В случае B-ДНК, которая служит основной природной формой и в которой при интеркаляции в область an смещаются углы а и у , наблюдается иная картина. В лучшей с точки зрения интеркаляции модели (82) конформация сахара, расположенного на 5 ′ -области сайта интеркаляции, изменяется с С 2 ‘ - эндо на С 3 ‘ - эндо в соответствии с тем, что наблюдалось для комплексов динуклеозидмонофосфатов с интеркаляторами. Конформационные изменения не ограничиваются нарушением положения двух соседних с интеркалятором пар, а распространяются на другие, отдаленные пары. Раскручивание на 18 ° , связанное с включением каждой молекулы интеркалятора, затрагивает по крайней мере три пары до и после сайта интеркаляции.
В частности, это отдаленное действие хорошо прослеживается в комплексе дауномицина с гексадеоксинуклеотидом (83) и согласуется с принципом исключения ближайших сайтов связывания. Изучение кругового дихроизма показало, что плоскость интеркалятора не перпендикулярна оси спирали, а наклонена к ней под углом около 20 ° . Это влияет на положение как ближайшей пары, так и следующей за ней, то есть в этом случае возникает кооперативный эффект (84).
Таким образом, на основании представленных данных можно сделать вывод, что взаимодействия между белками и нуклеиновыми кислотами очень разнообразны: это могут быть солевые мостики, водородные связи, стэкинг- и гидрофобные взаимодействия. В них может быть задействована любая часть обеих молекул. Молекулы нуклеотидов, одноцепочечных ДНК и РНК обычно удлиняются при связывании с белками.
Вода — это не просто среда, в которой растворены те или иные молекулы. Вода стабилизирует вторичную и третичную структуру макромолекул. Это относится и к белкам, и к ДНК, причем к ДНК, пожалуй, даже в большей степени (85, 86). Гидратация нуклеиновых кислот, как упоминалось выше, играет важную роль в формировании их структуры и отвечает за конформационные переходы A ^ B в ДНК. Следует отметить, что B-форма — основная природная форма, в которой ДНК находится в клетке и взаимодействует с внутриклеточными белками, обеспечивая активность и репликацию генетической информации, закодированной в ДНК.
При сворачивании в двойную спираль молекула ДНК становится более полярной, и это может определять возможность интеркаляции плоских веществ в двойную цепь нуклеиновых кислот. Интеркаляция планарных лекарственных и других химических веществ или их планарных групп имеет большое значение не только для фармакологии (77, 78), но и влияет на репликацию, включая ингибирование нуклеазной активности, а также может использоваться для направленного мутагенеза и рекомбиногенеза в нуклеиновых кислотах (76, 79). Интеркаляция проявляет ключевое воздействие при модификации ДНК в процессах репликации и биосинтеза белка (87, 88), структурно изменяя ДНК при ее взаимодействии с этими соединениями и оказывая сильное влияние на клеточный метаболизм, замедляя, а в некоторых случаях прекращая рост клеток, что может приводить как к апоптозу, так и к раковым заболеваниям либо, наоборот, к выздоровлению от такого рода заболеваний (89, 90). Понимание механизмов взаимодействия позволяет установить роль каждого из интеркаляторов в решении практических задач генетики, физиологии и фармакологии. Например, препараты с планарной структурой, содержащие хелатный комплекс Pt с бипиридином, сильно изменяют структуру ДНК, которая принимает форму «веревочной лестницы» (91). Более того, в каждой паре Уотсона-Крика один нуклеотид находится в ñèí -форме, а другой — в àíòè -форме, как у Z-ДНК.
Итак, внутриклеточные взаимодействия нуклеиновых кислот с водой и молекулами белков чрезвычайно важны, поскольку это фиксирует и стабилизирует пространственную структуру ДНК и РНК на молекулярном уровне, что, в свою очередь, обеспечивает стабильность и регулярность процессов репликации и экспрессию генов, особенно при взаимодействии генотип—среда. Вода стабилизирует вторичную и третичную структуру растворенных в ней макромолекул. Для ДНК это крайне важно, поскольку от степени гидратации зависят конформационные изменения ее молекулы и в конечном счете функциональная активность ДНК как носителя генетической информации. Взаимодействие ДНК с лекарственными препаратами-интеркаляторами также имеет большое значение. Интеркаляция, влияя на процессы репликации и биосинтеза белка, воздействует на клеточный метаболизм, что при определенных условиях может оказывать лечебный эффект. Есть основания надеяться, что благодаря изучению взаимодействий нуклеиновых кислот с водой и с белками на внутриклеточном уровне в самом ближайшем времени удастся решить проблему нуклеиново-белкового узнавания и тем самым вплотную подойти к расшифровке механизмов этого процесса, положив начало управлению таким взаимодействием. Однако последнее слово в любом случае остается за исследованием самих комплексов, образуемых нуклеиновыми кислотами не только с водой, но и с белками, а в случае интеркаляции — и с интеркалирующими агентами.
Список литературы Взаимодействие нуклеиновых кислот с молекулами воды, белков и интеркаляторов (обзор)
- Pace C.N., Trevino S., Prabhakaran E., Scholtz J.M. Protein structure, stability and solubility in water and other solvents. Philosophical Transactions Royal Society London B, 2004, 359(1448): 1225-1235 (doi: 10.1098/rstb.2004.1500).
- Bazzoli A., Karanicolas J. “Solvent hydrogen-bond occlusion”: a new model of polar desolvation for biomolecular energetics. Journal Computational Chemistry, 2017, 38(15): 1321-1331 (doi: 10.1002/jcc.24740).
- Del Giudice E., Tedeschi A. Water and autocatalysis in living matter. Electromagnetic Biology and Medicine, 2009, 28(1): 46-52 (doi: 10.1080/15368370802708728).
- Barciszewski J., Jurczak J., Porowski S., Specht T., Erdmann V.A. The role of water structure in conformational changes of nucleic acids in ambient and high-pressure conditions. European Jour-nal of Biochemistry, 1999, 260(2): 293-307 (doi: 10.1046/j.1432-1327.1999.00184.x)
- Biedermannová L., Schneider B. Hydration of proteins and nucleic acids: advances in exper-iment and theory. A review. Biochimisry et Biophysics Acta, 2016, 1860(9): 1821-1835 (doi: 10.1016/j.bbagen.2016.05.036).
- Takahashi H., Umino S., Miki Y., Ishizuka R., Maeda S., Morita A., Suzuki M., Matubayasi N. Drastic compensation of electronic and solvation effects on ATP hydrolysis revealed through large-scale QM/MM simulations combined with a theory of solutions. Journal of Physical Chem-istry B, 2017, 121(10): 2279-2287 (doi: 10.1021/acs.jpcb.7b00637).
- Inada A., Oue T., Yamashita S., Yamasaki M., Oshima T., Matsuyama H. Development of highly water-dispersible complexes between coenzyme Q10 and protein hydrolysates. European Journal of Pharmaceutical Sciences, 2019, 136: 104936 (doi: 10.1016/j.ejps.2019.05.014).
- Chen J., Zhang C., Xia Q., Liu Dю, Tan X., Li Y., Cao Y. Treatment with subcritical water-hydrolyzed citrus pectin ameliorated cyclophosphamide-induced immunosuppression and modu-lated gut microbiota composition in ICR mice. Molecules, 2020, 25(6): 1302 (doi: 10.3390/mol-ecules25061302).
- Jamshidi A., Antequera T., Solomando J.C., Perez-Palacios T. Microencapsulation of oil and protein hydrolysate from fish within a high-pressure homogenized double emulsion. Journal of Food Science and Technology, 2020, 57(1): 60-69 (doi: 10.1007/s13197-019-04029-5).
- Bentrup F.W. Water ascent in trees and lianas: the cohesion-tension theory revisited in the wake of Otto Renner. Protoplasma, 2017, 254(2): 627-633 (doi: 10.1007/s00709-016-1009-4).
- Djikaev Y.S., Ruckenstein E. A probabilistic approach to the effect of water hydrogen bonds on the kinetics of protein folding and protein denaturation. Advances in Colloid and Interface Sciences, 2010, 154(1-2): 77-90 (doi: 10.1016/j.cis.2010.01.009).
- Andrić J.M., Stanković I.M., Zarić S.D. Binding of metal ions and water molecules to nucleic acid bases: the influence of water molecule coordination to a metal ion on water-nucleic acid base hydrogen bonds. Acta Crystallographica Section B: Structural Science, Crystal Engineering and Materials, 2019, 75(3): 301-309 (doi: 10.1107/S2052520619001999).
- Li C., Liu M. Protein dynamics in living cells studied by in-cell NMR spectroscopy. FEBS Letters, 2013, 587(8): 1008-1011 (doi: 10.1016/j.febslet.2012.12.023).
- Wang L., Xue Y., Xing J., Song K., Lin J. Exploring the spatiotemporal organization of membrane proteins in living plant cells. Annual Review of Plant Biology, 2018, 69: 525-551 (doi: 10.1146/an-nurev-arplant-042817-040233).
- Lee S., Wang C., Liu H., Xiong J., Jiji R., Hong X., Yan X., Chen Z., Hammel M., Wang Y., Dai S., Wang J., Jiang C., Zhang G. Hydrogen bonds are a primary driving force for de novo protein folding. Acta Crystallographica Section D: Structural Biology, 2017, 73: 955-969 (doi: 10.1107/S2059798317015303)
- Durell S.R., Ben-Naim A. Hydrophobic-hydrophilic forces in protein folding. Biopolymers, 2017, 107(8): e23020 (doi: 10.1002/bip.23020).
- Persson F., Soderhjelm P., Halle B. The spatial range of protein hydration. The Journal of Chem-ical Physics, 2018, 148(21): 215104 (doi: 10.1063/1.5031005).
- Meyer A.J., Riemer J., Rouhier N. Oxidative protein folding: state-of-the-art and current avenues of research in plants. New Phytologist, 2019, 221(3): 1230-1246 (doi: 10.1111/nph.15436).
- Zhang J., Ma Z., Kurgan L. Comprehensive review and empirical analysis of hallmarks of DNA-, RNA- and protein-binding residues in protein chains. Brief Bioinformatics, 2019, 20(4): 1250-1268 (doi: 10.1093/bib/bbx168).
- Sharma M., Ganeshpandian M., Majumder M., Tamilarasan A., Sharma M., Mukhopadhyay R., Islam N.S., Palaniandavar M. Octahedral copper(II)-diimine complexes of triethylenetetramine: effect of stereochemical fluxionality and ligand hydrophobicity on CuII/CuI redox, DNA binding and cleavage, cytotoxicity and apoptosis-inducing ability. Dalton Transactions, 2020, 24: 8282-8297 (doi: 10.1039/d0dt00928h).
- Bartas M., Červeň J., Guziurová S., Slychko K., Pečinka P. Amino acid composition in various types of nucleic acid-binding proteins. International Journal of Molecular Sciences, 2021, 22(2): 922 (doi: 10.3390/ijms22020922).
- Hendry L.B., Mahesh V.B., Bransome E.D. Jr., Ewing D.E. Small molecule intercalation with double stranded DNA: implications for normal gene regulation and for predicting the biological efficacy and genotoxicity of drugs and other chemicals. Mutation Research, 2007, 623(1-2): 53-71 (doi: 10.1016/j.mrfmmm.2007.03.009).
- Soni A., Khurana P., Singh T, Jayaram B. A DNA intercalation methodology for an efficient prediction of ligand binding pose and energetics. Bioinformatics, 2017, 33(10): 1488-1496 (doi: 10.1093/bioinformatics/btx006).
- Sieber M., Bredenfeld H., Josting A., Reineke T., Rueffer U., Koch T., Naumann R., Boiss-evain F., Koch P., Worst P., Soekler M., Eich H., Müller-Hermelink H.K., Franklin J., Pau-lus U., Wolf J., Engert A., Diehl V. German Hodgkin’s Lymphoma Study Group. 14-day variant of the bleomycin, etoposide, doxorubicin, cyclophosphamide, vincristine, procarbazine, and pred-nisone regimen in advanced-stage Hodgkin’s lymphoma: results of a pilot study of the German Hodgkin’s Lymphoma Study Group. Journal of Clinical Oncology, 2003, 21(9): 1734-1739 (doi: 10.1200/JCO.2003.06.028).
- Cabrera C.M.E., Puga L.B., Torres V., Salinas M. Evaluación del tratamiento de linfoma de Hodgkin con esquema ABVD en Chile [Treatment of Hodgkin lymphoma. Analysis of 915 pa-tients]. Revista medica de Chile, 2019, 147(4): 437-443 (doi: 10.4067/S0034-98872019000400437).
- Gaspar N., Hawkins D.S., Dirksen U., Lewis I.J., Ferrari S., Le Deley M.C., Kovar H., Grimer R., Whelan J., Claude L., Delattre O., Paulussen M., Picci P., Sundby Hall K., van den Berg H., Ladenstein R., Michon J., Hjorth L., Judson I., Luksch R., Bernstein M.L., Marec-Bérard P., Brennan B., Craft A.W., Womer R.B., Juergens H., Oberlin O. Ewing sarcoma: current management and future approaches through collaboration. Journal of Clinical Oncology, 2015, 33(27): 3036-3046 (doi: 10.1200/JCO.2014.59.5256).
- Paulino E., de Melo A.C. Actinomycin D shortage in the Brazilian market: new challenges for successful treatment of gestational trophoblastic neoplasia. Journal of Gynecologic Oncology, 2019, 30(4): e87 (doi: 10.3802/jgo.2019.30.e87).
- Haines A.M., Tobe S.S., Kobus H.J., Linacre A. Properties of nucleic acid staining dyes used in gel electrophoresis. Electrophoresis, 2015, 36(6): 941-944 (doi: 10.1002/elps.201400496).
- Teuber M., Rögner M., Berry S. Fluorescent probes for non-invasive bioenergetic studies of whole cyanobacterial cells. Biochimica et Biophysica Acta, 2001, 1506(1): 31-46 (doi: 10.1016/s0005-2728(01)00178-5).
- Bruno J.G. An acridine orange spore germination fluorescence microscopy versus spectral para-dox. Journal of Fluorescence, 2015, 25(1): 211-216 (doi: 10.1007/s10895-014-1498-5).
- Hopfinger A.J. Intermolecular interaction and biomolecular organization. Wiley, New York, 1977: 159-169.
- Hadži S., Lah J. Origin of heat capacity increment in DNA folding: the hydration effect. Biochimica et Biophysica Acta — General Subjects, 2021, 1865(1): 129774 (doi: 10.1016/j.bbagen.2020.129774).
- Kuntz I.D. Jr., Kauzmann W. Hydration of proteins and polypeptides. Advances in Protein Chem-istry, 1974, 28: 239-345 (doi: 10.1016/s0065-3233(08)60232-6).
- Edelhoch H., Osborne J.C. Jr. The thermodynamic basis of the stability of proteins, nucleic acids, and membranes. Advances in Protein Chemistry, 1976, 30: 183-250 (doi: 10.1016/s0065-3233(08)60480-5).
- Anandakrishnan R., Izadi S., Onufriev A.V. Why computed protein folding landscapes are sensi-tive to the water model. Journal of Chemical Theory and Computation, 2019, 15(1): 625-636 (doi: 10.1021/acs.jctc.8b00485).
- Чесноков Ю.В. Конформационная изменчивость двойных спиралей ДНК. Овощи России, 2020, 6: 51-57 (doi: 10.18619/2072-9146-2020-6-51-57).
- Khesbak H., Savchuk O., Tsushima S., Fahmy K. The role of water H-bond imbalances in B-DNA substate transitions and peptide recognition revealed by time-resolved FTIR spectroscopy. Journal of the American Chemical Society, 2011, 133(15): 5834-5842 (doi: 10.1021/ja108863v).
- Waters J.T., Lu X.J., Galindo-Murillo R., Gumbart J.C., Kim H.D., Cheatham T.E. 3rd, Har-vey S.C. Transitions of double-stranded DNA between the A- and B-forms. Journal of Physical Chemistry B, 2016, 120(33): 8449-8456 (doi: 10.1021/acs.jpcb.6b02155).
- Fuller W., Forsyth T., Mahendrasingam A. Water-DNA interactions as studied by X-ray and neutron fibre diffraction. Philosophical Transactions of the Royal Society B: Biological Sciences, 2004, 359(1448): 1237-1247 (doi: 10.1098/rstb.2004.1501).
- Aldawsari H., Altaf A., Banjar Z.M., Iohara D., Nakabayashi M., Anraku M., Uekama K., Hirayama F. Crystallization of a new polymorph of acetohexamide from 2-hydroxybutyl-β-cy-clodextrin solution: form VI with a high aqueous solubility. International Journal of Pharmaceutics, 2013, 453(2): 315-321 (doi: 10.1016/j.ijpharm.2013.06.026).
- Iohara D., Anraku M., Uekama K., Hirayama F. Modification of drug crystallization by cy-clodextrins in pre-formulation study. Chemical and Pharmaceutical Bulletin (Tokyo), 2019, 67(9): 915-920 (doi: 10.1248/cpb.c18-00752).
- Falk M., Hartman K.A., Lord R.C. Hydration of deoxyribonucleic acid. II. An infrared study. Journal of the American Chemical Society, 1963, 85(4): 387-391 (doi: 10.1021/ja00887a004).
- Wolf B., Hanlon S. Structural transitions of deoxyribonucleic acid in aqueous solutions. II. The role of hydration. Biochemistry, 1975, 14(8): 1661-1670 (doi: 10.1021/bi00679a018).
- Vikram K., Alapati P.R., Singh R.K. Temperature dependent Raman study of S(B)→S(C) tran-sition in liquid crystalline compound N-(4-n-pentyloxybenzylidene)-4'-heptylaniline (5O.7). Spectrochimica Acta Part A: Molecular and Biomolecular Spectroscopy, 2010, 75(5): 1480-1485 (doi: 10.1016/j.saa.2010.02.002).
- Ivanov V.I., Minchenkova L.E., Schyolkina A.K., Poletayev A.I. Different conformations of dou-ble stranded nucleic acids in solution as revealed by circular dichroism. Biopolymers, 1973, 12(1): 89-100 (doi: 10.1002/bip.1973.360120109).
- Noy A., Pérez A., Laughton C.A., Orozco M. Theoretical study of large conformational transitions in DNA: the B ↔ A conformational change in water and ethanol/water. Nucleic Acids Research, 2007, 35(10): 3330-3338 (doi: 10.1093/nar/gkl1135).
- Zhang H., Fu H., Shao X., Dehez F., Chipot C., Cai W. Changes in microenvironment modulate the B- to A-DNA transition. Journal of Chemical Information and Modeling, 2019, 59(5): 2324-2330 (doi: 10.1021/acs.jcim.8b00885).
- Alden C.J., Kim S.-H. Solvent-accessible surfaces of nucleic acids. Journal of Molecular Biology, 1979, 132(3): 411-434 (doi: 10.1016/0022-2836(79)90268-7).
- Borukhov S., Lee J. RNA polymerase structure and function at lac operon. Comptes Rendus Biologies, 2005, 328(6): 576-587 (doi: 10.1016/j.crvi.2005.03.007).
- Sendy B., Lee D.J., Busby S.J., Bryant J.A. RNA polymerase supply and flux through the lac operon in Escherichia coli. Philosophical Transactions of the Royal Society B: Biological Sciences, 2016, 371(1707): 20160080 (doi: 10.1098/rstb.2016.0080).
- Rajendran V., Kalita P., Shukla H., Kumar A., Tripathi T. Aminoacyl-tRNA synthetases: Struc-ture, function, and drug discovery. International Journal of Biological Macromolecules, 2018, 111: 400-414 (doi: 10.1016/j.ijbiomac.2017.12.157).
- McPherson A., Jurnak F., Wang A., Kolpak F., Rich A. The structure of a DNA unwinding protein and its complexes with oligodeoxynucleotides by X-ray diffraction. Biophysical Journal, 1980, 32(1): 155-173 (doi: 10.1016/S0006-3495(80)84931-9).
- Young T.-S., Kim S.-H., Modrich P., Beth A., Jay E. Preliminary X-ray diffraction studies of EcoRI restriction endonuclease-DNA complex. Journal of Molecular Biology, 1981, 145(3): 607-610 (doi: 10.1016/0022-2836(81)90548-9).
- Ruff M., Cavarelli J., Mikol V., Lorber B., Mitschler A., Giege R., Thierry J.C., Moras D. A high-resolution diffracting crystal form of the complex between yeast tRNAAsp and aspartyl-tRNA synthetase. Journal of Molecular Biology, 1988, 201(1): 235-236 (doi: 10.1016/0022-2836(88)90450-0).
- Havrylenko S., Mirande M. Aminoacyl-tRNA synthetase complexes in evolution. International Journal of Molecular Sciences, 2015, 16(3): 6571-6594 (doi: 10.3390/ijms16036571).
- Stasyuk O.A., Jakubec D., Vondrášek J., Hobza P. Noncovalent interactions in specific recogni-tion motifs of protein-DNA complexes. Journal of Chemical Theory and Computation, 2017, 13(2): 877-885 (doi: 10.1021/acs.jctc.6b00775).
- von Hippel P.H., McGhee J.D. DNA-protein interactions. Annual Review of Biochemistry, 1972, 41(10): 231-300 (doi: 10.1146/annurev.bi.41.070172.001311).
- Nucleic acid-protein recognition /H.J. Vogel (ed.). Academic Press, New York, 1977.
- Helene C., Maurizot J.-C. Interactions of oligopeptides with nucleic acids. CRC Critical Reviews in Biochemistry, 1981, 10(3): 213-258 (doi: 10.3109/10409238109113600).
- Pabo C.O., Sauer R.T. Protein-DNA recognition. Annual Review of Biochemistry, 1984, 53: 293-321 (doi: 10.1146/annurev.bi.53.070184.001453).
- Blace C.C.F., Oatley S.J. Protein-DNA and protein-hormone interactions in prealbumin: a model of the thyroid hormone nuclear receptor? Nature, 1977, 268(5616): 115-120 (doi: 10.1038/268115a0).
- Schulz G.E., Schirmer H. Principles of protein structure. Springer Verlag, New York, 1979.
- Bochkarev A., Bochkareva E. From RPA to BRCA2: lessons from single-stranded DNA binding by the OB-fold. Current Opinion in Structural Biology, 2004, 14(1): 36-42 (doi: 10.1016/j.sbi.2004.01.001).
- Ishibashi K., Ishikawa M. Template selection by replication protein of tobacco mosaic virus. Uirusu, 2014, 64(1): 3-10 (doi: 10.2222/jsv.64.3) (In Japanese).
- Ruszkowski M., Dauter Z. Structures of Medicago truncatula L-histidinol dehydrogenase show rearrangements required for NAD+ binding and the cofactor positioned to accept a hydride. Scientific Reports, 2017, 7: 10476 (doi: 10.1038/s41598-017-10859-0).
- Oliver A.W., Bogdarina I., Schroeder E., Taylor I.A., Kneale G.G. Preferential binding of fd gene 5 protein to tetraplex nucleic acid structures. Journal of Molecular Biology, 2000, 301(3): 575-584 (doi: 10.1006/jmbi.2000.3991).
- Persil O., Hud N.V. Harnessing DNA intercalation. Trends in Biotechnology, 2007, 25(10): 433-436 (doi: 10.1016/j.tibtech.2007.08.003).
- Rescifina A., Zagni C., Varrica M.G., Pistarà V., Corsaro A. Recent advances in small organic molecules as DNA intercalating agents: synthesis, activity, and modeling. European Journal of Medicinal Chemistry, 2014, 74: 95-115 (doi: 10.1016/j.ejmech.2013.11.029).
- Portugal J., Barceló F. Noncovalent binding to DNA: still a target in developing anticancer agents. Current Medicinal Chemistry, 2016, 23(36): 4108-4134 (doi: 10.2174/0929867323666160902153511).
- Pack G.R., Loew G. Origins of the specificity in the intercalation of ethidium into nucleic acids. A theoretical analysis. Biochimica et Biophysica Acta, 1978, 519(1): 163-172 (doi: 10.1016/0005-2787(78)90070-9).
- Tsai C.-C., Jain S.C., Sobell H.M. Visualization of drug-nucleic acid interactions at atomic resolution. I. Structure of an ethidium/dinucleoside monophosphate crystalline complex, ethidium: 5-iodocytidylyl(3′,5′)adenosine. Journal of Molecular Biology, 1977, 114(3): 301-305 (doi: 10.1016/0022-2836(77)90252-2).
- Lerman L.S. Structural considerations in the interaction of DNA and acridines. Journal of Mo-lecular Biology, 1961, 3: 18-30 (doi: 10.1016/S0022-2836(61)80004-1).
- Fuller W., Warning M. A molecular model for the interaction of ethidium bromide with deoxy-ribonucleic acid. Berichte der Bunsengesellschaft für physikalische Chemie, 1964, 68(8-9): 805-809 (doi: 10.1002/bbpc.19640680830).
- Watson J.D., Tooze J., Kurtz D.T. Recombinant DNA: a short course. Scientific American Books (W.H. Freeman), New York, 1983.
- Warning M. Variation of the supercoils in closed circular DNA by binding of antibiotics and drugs: evidence of molecular models involving intercalation. Journal of Molecular Biology, 1970, 54(2): 247-279 (doi: 10.1016/0022-2836(70)90429-8).
- Чесноков Ю.В., Стыранкевич Р.Г., Бурилков В.К. Эффекты лазерного излучения и бромистого этидия на препараты фаговой и растительной ДНК. Известия АН ССРМ. Серия биологических и химических наук, 1990, 6: 62-63.
- Чесноков Ю.В., Пащенко В.М., Бурилков В.К. Нуклеазная активность прорастающей пыльцы томата и ее ингибирование. Биополимеры и клетка, 1993, 9(5): 78-82 (doi: 10.7124/bc.000376).
- Пашенко В.М., Чесноков Ю.В., Бурилков В.К., Лысиков В.Н. Эффекты совместного действия ультрафиолетового излучения и красителей-сенсибилизаторов на прорастающую пыльцу. Известия АН РМ. Серия биологических и химических наук, 1992, 5: 19-23.
- Пащенко В.М., Бурилков В.К., Чесноков Ю.В. Возможный механизм взаимодействия лазерного излучения и комплекса ДНК-6-меркаптопурин. Известия АН РМ. Серия биологических и химических наук, 1993, 1: 33-36.
- Berman H.M., Stallings W., Carrell H.L., Glusker J.P., Neidle S., Taylor G., Achari A. Molecular and crystal structure of an intercalation complex: proflavine-cytidylyl-(3′,5′)-guanosine. Biopoly-mers, 1979, 18(10): 2405-2429 (doi: 10.1002/bip.1979.360181004).
- Reddy B.S., Seshadri T.P., Sakore T.D., Sobell H.M. Visualization of drug-nucleic acid inter-actions at atomic resolution. V. Structures of two aminoacridine-dinucleoside and acridine orange-5-iodocytidylyl(3′,5′)-guanisine. Journal of Molecular Biology, 1979, 135(4): 787-812 (doi: 10.1016/0022-2836(79)90513-8).
- Alden C.J., Arnott S. Visualization of planar drug intercalation in B-DNA. Nucleic Acids Research, 1975, 2(10): 1701-1717 (doi: 10.1093/nar/2.10.1701).
- Davies D.B., Eaton R.J., Baranovsky S.F., Veselkov A.N. NMR investigation of the com-plexation of daunomycin with deoxytetranucleotides of different base sequence in aqueous solution. Journal of Biomolecular Structure and Dynamics, 2000, 17(5): 887-901 (doi: 10.1080/07391102.2000.10506577).
- Hogan M., Dattagupta N., Crothers D.M. Transmission of allosteric effects in DNA. Nature, 1979, 278(5704): 521-524 (doi: 10.1038/278521a0).
- Mondal S., Bandyopadhyay S. Flexibility of the binding regions of a protein-DNA complex and the structure and ordering of interfacial water. Journal of Chemical Information and Modeling, 2019, 59(10): 4427-4437 (doi: 10.1021/acs.jcim.9b00685).
- Sinha S.K., Bandyopadhyay S. Conformational fluctuations of a protein-DNA complex and the structure and ordering of water around it. The Journal of Chemical Physics, 2011, 135(24): 245104 (doi: 10.1063/1.3670877).
- Hendry L.B., Mahesh V.B., Bransome E.D. Jr., Ewing D.E. Small molecule intercalation with double stranded DNA: implications for normal gene regulation and for predicting the biological efficacy and genotoxicity of drugs and other chemicals. Mutation Research, 2007, 623(1-2): 53-71 (doi: 10.1016/j.mrfmmm.2007.03.009).
- Tateishi-Karimata H., Sugimoto N. Biological and nanotechnological applications using interac-tions between ionic liquids and nucleic acids. Biophysical Reviews, 2018, 10(3): 931-940 (doi: 10.1007/s12551-018-0422-7).
- Khosravifar F., Dehghan G., Bidoki S.K., Mahdavi M. DNA-binding activity and cytotoxic and cell-cycle arrest properties of some new coumarin derivatives: a multispectral and computational investigation. Luminescence, 2020, 35(1): 98-106 (doi: 10.1002/bio.3702).
- Ashrafizadeh M., Mohammadinejad R., Samarghandian S., Yaribeygi H., Johnston T.P., Saheb-kar A. Anti-tumor effects of osthole on different malignant tissues: a review of molecular mech-anisms. Anti-Cancer Agents in Medicinal Chemistry, 2020, 20(8): 918-931 (doi: 10.2174/1871520620666200228110704).
- Arnott S., Bond P.J., Chandrasekaran R. Visualization of an unwound DNA duplex. Nature, 1980, 287(5782): 561-563 (doi: 10.1038/287561a0).


