Анализ полиморфизма гена дисферлина у генофондных пород кур
Автор: Баркова О.Ю., Крутикова А.А., Дементьева Н.В.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 4 т.56, 2021 года.
Бесплатный доступ
Дисферлин относится к белкам, участвующим в репарации мышечной мембраны. Предполагается, что некоторые мононуклеотидные замены в гене дисферлина ( DYSF ) связаны с формированием мышечной массы у домашней птицы. В настоящей работе впервые у кур породы русская белая выявлены четыре мононуклеотидные замены, находящиеся в 32-м интроне на 4-й хромосоме, - rs317801013 (G/A) в позиции 90672849, rs16455118 (С/A) в позиции 90672756, rs318045896 (A/G) в позиции 90672862, T/G в позиции 90672805. Мононуклеотидный полиморфизм T/G на 4-й хромосоме в позиции 90672805 представлен для регистрации в базу данных ENSEMBL, поскольку у вида Gallus gallus был выявлен впервые. Также впервые проведен анализ частоты встречаемости генотипов и отклонения наблюдаемого распределения генотипов от ожидаемого при равновесии Харди-Вайнберга у генофондных кур породы русская белая по всем вышеуказанным заменам в гене дисферлина. Целью работы было изучение мононуклеотидных полиморфизмов (SNPs) гена дисферлина у генофондных пород кур и выявление возможных ассоциаций полиморфизмов гена DYSF с хозяйственно ценными характеристиками. Материалом для исследования служили куры мясного (корниш), яичного направления (русская белая, род-айланд, аврора, австролорп черно-пестрый, ленинградская ситцевая) и декоративные породы (русская хохлатая, брама светлая, голошейная) из генофондной популяции Генетической коллекции редких и исчезающих пород кур (ВНИИГРЖ, г. Санкт-Петербург-Пушкин). ДНК выделяли фенольным методом из крови, взятой из подкрыльцовой вены. Для анализа полиморфизма rs16455118 пользовались базой данных, полученной в результате генотипирования с применением чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip («Illumina, Inc.», США). Проанализирована частота встречаемости генотипов АА , АС , СС и отклонения наблюдаемого распределения генотипов от ожидаемого при равновесии Харди-Вайнберга у генофондных кур по замене аденина на цитозин в гене дисферлина (rs16455118). Достоверность полученных данных оценивали с применением критерия χ2 Пирсона. Полиморфизм гена дисферлина анализировали методом секвенирования участка гена DYSF размером 237 п.н. на 4-й хромосоме у 76 кур породы русская белая. Провели анализ международных баз генетических данных NCBI и ENSEMBL для идентификации выявленных замен. По четырем обнаруженным заменам проведен анализ частоты генотипов и аллелей. В результате генотипирования 185 кур с использованием чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip был выявлен мононуклеотидный полиморфизм rs16455118. Наблюдали смещение частоты встречаемости аллелей в сторону увеличения гетерозиготных генотипов АС у кур декоративного направления, генотип АА присутствовал в меньшинстве. У кур яичного направления наибольшую частоту встречаемости имел гомозиготный генотип АА , генотип СС был малочисленным, а у популяции кур породы аврора полностью отсутствовал. Мясная порода корниш имела более равномерное распределение генотипов в сравнении с декоративными и яичными. Мы секвенировали участок гена дисферлина размером 237 п.н., расположенный на 4-й хромосоме у кур породы русская белая, и обнаружили четыре мононуклеотидные замены, находящиеся в 32-м интроне. Мононуклеодидные замены G/A (rs317801013), С/А (rs16455118), A/G (rs318045896) соответствовали заменам в геноме кур, зарегистрированным в международных генетических базах данных NCBI (https://www.ncbi.nlm.nih.gov/SNP) и ENSEMBL (https://www.ensembl.org/index.html). Мононуклеотидный полиморфизм T/G в позиции 90672805 выявлен впервые. Сдвиг генетического равновесия, который наблюдался у генофондных кур породы русская белая, свидетельствует об эффекте основателя либо селекционном давлении на район мононуклеотидной замены rs16455118. Практически полное отсутствие гетерозигот у яичных пород кур может указывать на инбридинг или сильное селекционное давление. Полученные результаты в дальнейшем могут быть использованы для поиска ассоциаций с показателями продуктивности у кур для создания системы молекулярно-генетических маркеров, что позволит ускорить селекционный прогресс.
Ген дисферлина, snp, однонуклеотидный полиморфизм, птицеводство, аллель, куры
Короткий адрес: https://sciup.org/142231374
IDR: 142231374 | УДК: 636.52/.58:575:575.116.4 | DOI: 10.15389/agrobiology.2021.4.641rus
Текст научной статьи Анализ полиморфизма гена дисферлина у генофондных пород кур
В настоящее время традиционная селекция птицы вышла на плато и прогресс в увеличении показателей продуктивности существенно снизился. Использование молекулярно-генетических маркеров становится наи- более эффективным методом ускорения селекционного процесса в животноводстве (1). В основе такого подхода лежит поиск мононуклеотидных полиморфизмов (single nucleotide polymorphisms, SNPs), ассоциированных с различными признаками домашней птицы, в том числе при помощи SNP-чиповых панелей. На основании нескольких миллионов SNP-локусов, выявленных в результате многолетних научных исследований, компанией «Illumina, Inc.» (США) созданы чипы средней и большой плотности для основных видов сельскохозяйственных животных, в том числе кур, что позволяет получить данные о локализации районов и генов, связанных с признаками. Секвенирование дает возможность более детально изучать участки генов-кандидатов для выявления вариантов генетического полиморфизма, ассоциированного с интересующими признаками. Так, с использованием чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip была найдена достоверная ассоциация области 32-го интрона на 4-й хромосоме в гене дисферлина (DYSF) с белым пухом у кур русской белой породы (1).
Наиболее актуальная задача в птицеводстве — повышение яичной и мясной продуктивности. Для работы в этом направлении нами был выбран ген DYSF , предположительно влияющий на формирование мышечной массы и яйценоскости у птицы (2-4).
Дисферлин (DYSF) — это трансмембранный белок II типа, который локализуется на периферии мышечных волокон и служит регулятором слияния везикул в сарколемме. Дисферлин играет важную роль в перемещении везикул, эндоцитозе, рециркуляции мембранных рецепторов, регенерации мышц и формировании Т-канальцев (2). Он может выполнять дополнительные функции в везикулярном переносе рецепторов факторов роста, способствующих росту и регенерации мышц. Предполагается, что дисферлин-за-висимый транспорт таких сигнальных молекул модулирует экспрессию генов и функцию взрослых мышечных стволовых (или сателлитных) клеток, ответственных за рост и регенерацию скелетных мышц у взрослых людей (5).
Мутации в гене DYSF вызывают ряд заболеваний мышц с различными клиническими проявлениями, известными как дисферлинопатии, включая мышечную дистрофию конечностей типа 2B (LGMD2B) и миопатию Ми¸си (6-8). Ферлины представляют собой белки, которые влияют на управляемую Ca2+ динамику мембран и принадлежат к суперсемейству белков с несколькими доменами C2 (MC2D), выполняющих общие функции при связывании мембраноассоциированных органелл и белков на клеточных мембранах. Часто эти белки описываются как сенсоры ионов кальция (Ca2+) для везикулярного переноса, способные формировать мембраны (3, 9, 10). У позвоночных существует шесть ферлиновых генов, у людей — гены дисферлина, отоферлина, миоферлина, Fer1L5 и Fer1L6 и длинный ген, не кодирующий РНК, — Fer1L4 (11).
Наиболее изучена роль дисферлина в восстановлении повреждений в поверхностной мембране поперечнополосатых мышечных волокон — сарколемме. Сокращение мышечных волокон механически воздействует на сарколемму, что приводит к ее микроразрушению. Процесс восстановления запускается притоком Ca2+ в саркоплазму, что зависит от ряда белков, включая дисферлин в качестве одного из ключевых участников (12-14). Вероятно, при восстановлении мембраны он способствует агрегации и слиянию мембран благодаря взаимодействию Ca2+ с отрицательно заряженными фосфолипидами (15-17).
В мировой научной литературе не встречаются работы, посвященные функциям дисферлина у кур, в том числе не изучена ассоциация дис- ферлина с продуктивностью птицы.
В настоящей работе у кур породы русская белая впервые выявлены четыре мононуклеотидные замены, находящиеся в 32-м интроне на 4-й хромосоме, — rs317801013 (G/A) в позиции 90672849, rs16455118 (С/A) в позиции 90672756, rs318045896 (A/G) в позиции 90672862 и мононуклеотидный полиморфизм (T/G) в позиции 90672805. Мононуклеотидный полиморфизм T/G на 4-й хромосоме в позиции 90672805 был представлен для регистрации в базу данных ENSEMBL. Также впервые проведен анализ частоты встречаемости генотипов и отклонения наблюдаемого распределения генотипов от ожидаемого при равновесии Харди-Вайнберга у генофондных кур породы русская белая по всем указанным выше заменам в гене дисферлина.
Целью работы было изучение мононуклеотидных полиморфизмов (SNPs) гена дисферлина у генофондных пород кур и выявление возможных ассоциаций полиморфизмов гена DYSF с хозяйственно ценными характеристиками.
Методика. Исследования проводили на курах ( Gallus gallus ) из гено-фондной популяции Генетической коллекции редких и исчезающих пород кур (Всероссийский НИИ генетики и разведения сельскохозяйственных животных, г. Санкт-Петербург—Пушкин).
ДНК выделяли фенольным методом из крови, взятой из подкрыльцовой вены в стандартные пробирки с антикоагулянтом (EDTA). Для анализа полиморфизма rs16455118 пользовались базой данных, полученной в результате генотипирования с применением чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip («Illumina, Inc.», США).
На первом этапе использовали в общей сложности 185 кур — мясного направления (корниш, n = 39), яичного направления (русская белая, n = 19; род-айланд, n = 18; аврора, n = 14; австролорп черно-пестрый, n = 20; ленинградская ситцевая, n = 20) и декоративных пород (русская хохлатая, n = 20; брама светлая, n = 18; голошейная, n = 17). Проанализировали частоту встречаемости генотипов АА , АС , СС и отклонения наблюдаемого распределения генотипов от ожидаемого при равновесии Харди-Вайнберга у генофондных кур по замене rs16455118 аденина на цитозин в гене дисферлина. Достоверность полученных данных оценивали с применением критерия х 2 Пирсона.
Дополнительно у 76 кур яичной породы русская белая более подробно анализировали полиморфизм гена дисферлина методом секвенирования участка гена DYSF размером 237 п.н. на 4-й хромосоме. Дизайн праймеров для амплификации проводили на основании базы данных NCBI при помощи компьютерной программы PRIMER_3 . Использовали последовательности праймеров: Fw — 5´-GGATGCCATAAGGACGTTGC-3´, Rv — 5´-TCCCCACAGCATCCCCTATAC-3´. ПЦР проводили в 10 мкл реакционной смеси, содержащей 67 мМ Tris-HCl (pH 8,6), 2,5 мM MgCl2, 16,6 мM NHOH, 0,125 мМ dNTP, 0,5 мкМ праймера, 50-100 нг геномной ДНК и 2,5 ед. Taq-полимеразы («Сибэнзим», Новосибирск) на амплификаторе C 1000 Touch («Bio-Rad», США). Режим амплификации: 5 мин при 95 °С (денатурация); 20 с при 95 °С, 20 с при 62 °С, 20 с при 72 °С (40 циклов); 4 мин при 72 °С (финальная элонгация). Анализ продуктов ПЦР проводили в 2 % агарозном геле.
ПЦР продукты очищали с использованием коммерческого набора ExoSAP-IT Express («Affimetrix», США) согласно протоколу производителя. Секвенирование по Сэнгеру проводили на генетическом анализаторе Applied
Biosystems 3500 («Thermo Fisher Scientific, Inc.», США) с применением коммерческого набора BigDye® Terminator v3.1 Cycle Sequencing Kit («Thermo Fisher Scientific, Inc.», США). Выравнивание и обработку сиквенсов выполняли с помощью программного обеспечения MEGA 6 .
Для идентификации выявленных замен провели анализ международных баз генетических данных NCBI и ENSEMBL . По четырем заменам осуществляли анализ частоты генотипов и аллелей. Отклонение наблюдаемой гетерозиготности от ожидаемой у кур породы русская белая были обрабо- таны статистически и рассчитаны достоверности полученных результатов с применением критерия χ2 Пирсона.
Результаты. По итогам генотипирования 185 особей кур с использованием чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip по замене rs16455118 (С/A) наблюдалось смещение в сторону увеличения частоты встречаемости аллеля С у птицы декоративного направления. У яичных кур наибольшую частоту имел гомозиготный генотип АА, генотип СС был самым малочисленным, а у популяции аврора полностью отсутствовал. Мясная порода корниш имела более равномерное распределение генотипов в сравнении с декоративными и яичными породами (рис.).
Анализ баз данных ;r=4:90672256-90673256;v=rs16455118;vdb=variation;vf=6811490) показал равное распределение аллелей А и С (C: 0,500; A: 0,500) у красной джунглевой курицы, белого плимутрока, белого леггорна и шелковой ку- рицы (местная китайская порода).
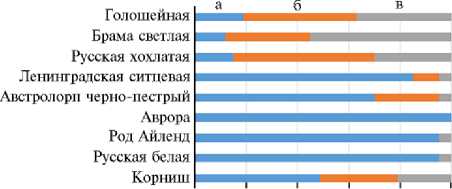
0 0,2 0,4 0,6 0,8 1,0
Частота встречаемости генотипов АА (а) , АС (б) , СС (в) по мо-нонуклеотидной замене A/C (rs16455118) в позиции 90672756 на 4-й хромосоме в гене дисферлина DYSF в популяциях гено-фондных пород из Генетической коллекции редких и исчезающих пород кур (Всероссийский НИИ генетики и разведения сельскохозяйственных животных, г. Санкт-Петер-бург—Пушкин).
При анализе распределения частот генотипов по замене rs16455118 среди генофондных пород было выявлено сильное смещение генотипов к гомозиготности. Отклонение наблюдаемой гетерозиготности (Ho) от ожидаемой (He) оказалось статистически недостоверным для пород голошейная (χ2 = 0,28, p = 0,9), брама светлая (χ2 = 0,28, p = 0,5), русская хохлатая (χ2 = 0,68, p = 0,71), австролорп чер- но-пестрый (χ2 = 0,009, p = 1) в соответствии с законом Харди-Вайнберга.
Иначе говоря, фактические частоты хорошо согласовывались с теоретически ожидаемыми. Отклонение наблюдаемого распределения генотипов от ожидаемого при равновесии Харди-Вайнберга было статистически значимо для популяций корниш (χ2 = 7,38 р = 0,025), род айленд (χ2 = 5,18, p = 0,05) ленинградская ситцевая (χ2 = 7,9, р = 0,019), русская белая (χ2 = 40, р = 0,00001). Полученное значение критерия χ2 были больше критического (3,84 при количестве степеней свободы 1), следовательно, сдвиг генетического равновесия в анализируемых популяциях свидетельствовал об эффекте основателя либо о селекционном давлении на район мононуклеотидной замены rs16455118, предположительно ассоциированной с яйценоскостью.
Практически полное отсутствие гетерозигот AC у яичных пород кур и небольшое количество гомозигот CC также свидетельствовали об ассоциации гомозиготного генотипа АА с яичной продуктивностью и сильном селекционном давлении на эти популяции, следствием чего стало повышение степени инбридинга. Видимо, отбор на повышение яйценоскости с использованием близкородственных скрещиваний приводил к эффекту основателя, в результате чего уменьшалась доля гетерозигот и особей с генотипом CC , что, в свою очередь, приводило к смещению распределения генотипов по закону Харди-Вайнберга (табл.).
Частота встречаемости генотипов и аллелей по четырем SNP в гене дисферлина DYSF у генофондных кур породы русская белая из Генетической коллекции редких и исчезающих пород кур (Всероссийский НИИ генетики и разведения сельскохозяйственных животных, г. Санкт-Петербург—Пушкин)
|
Идентификационный номер SNP |
Аллель, генотип |
Частота |
χ 2 |
p |
Н o |
Н е |
|
rs318045896 |
А |
0,593 |
6,004 |
0,014 |
13 |
20,75 |
|
G |
0,401 |
|||||
|
AA |
0,442 |
|||||
|
AG |
0,302 |
|||||
|
GG |
0,256 |
|||||
|
rs16455118 |
A |
0,623 |
45,1 |
1,026187e - 10 |
4 |
28.6557 |
|
С |
0,377 |
|||||
|
AA |
0,590 |
|||||
|
АС |
0,066 |
|||||
|
СС |
0,344 |
|||||
|
rs317801013 |
A |
0,280 |
4,31 |
0,037 |
18 |
24,52 |
|
G |
0,720 |
|||||
|
AA |
0,131 |
|||||
|
AG |
0,295 |
|||||
|
GG |
0,573 |
|||||
|
Отсутсвует в базе ENSEMBL |
Т |
0,180 |
2,68 |
0,1 |
15 |
18,8 |
G 0,820
ТТ 0,063
TG 0,234
___________________________ GG 0,703 Примечание. H o — наблюдаемая гетерозиготность, H e — ожидаемая гетерозиготность.
Секвенирование участка размером 237 п.н. гена дисферлина, расположенного на 4-й хромосоме у кур породы русская белая ( и = 76), выявило четыре мононуклеотидные замены, находящиеся в 31-м интроне в следующих положениях: G/A в позиции 90672849 (rs317801013), С/A в позиции 90672756, (rs16455118), A/G в позиции 90672862 (rs318045896), T/G в позиции 90672805. Мононуклеодидные замены rs317801013, rs16455118 и rs318045896 совпадали с известными заменами в геноме кур (базы данных NCBI и ENSEMBL). Мононуклеотидный полиморфизм T/G в позиции 90672805 был выявлен впервые и представлен нами для депонирования в базе ENSEMBL.
Несмотря на увеличение выборки яичных кур до 76 особей, при генотипировании с использованием чиповой технологии Illumina Chicken 60K SNP iSelect BeadChip по замене rs16455118 также наблюдалось отклонение фактического распределения генотипов от ожидаемого при равновесии Харди-Вайнберга ( χ 2 = 45,1, p = 1,026187e - 10), тенденция смещения к гомозиготности сохранялась, самым многочисленным оставался генотип АА , но увеличивалось число особей с генотипом СС .
В предыдущем исследовании (4) не было выявлено достоверной связи альтернативных генотипов АА, АС, СС по гену дисферлина с живой массой, возрастом первого снесения яйца и массой яйца у кур русской белой породы, однако была установлена связь с яйценоскостью. Куры с геноти- пом СС снесли за 180 сут в среднем на 10 яиц меньше (134,06±5,96), чем имеющие генотипы АА (145,00±2,35) и АС (143,94±2,31) (р < 0,05). Эта работа отчасти подтверждает гипотезу о влиянии генотипа АА на яйценоскость кур, поскольку у яичных пород преобладал генотип АА. Следовательно, замену A/С rs16455118 в гене дисферлина можно считать ассоциированной с яйценоскостью и использовать в качестве молекулярного маркера после более подробного изучения в составе панели маркерных полиморфизмов.
Поскольку мутация A/С находится в интроне и не приводит к замене аминокислоты, рассмотрим, как можно объяснять ее влияние на фенотип. У многих эукариот, включая млекопитающих, растения, насекомых и дрожжи, интроны могут повышать экспрессию генов, не функционируя в качестве сайта связывания для факторов транскрипции. Этот феномен получил название интрон-опосредованного усиления (18). Интроны могут увеличивать количество транскриптов, влияя на скорость транскрипции, ядерный экспорт и стабильность транскриптов, а также повышать эффективность трансляции мРНК (19-21). Проксимальные к промоторам интроны способны усиливать транскрипцию в клетках млекопитающих и растений (22-24). Анализ иммунопреципитации хроматина (ChIP) показал, что число сайтов связывания РНК-полимеразы II (Pol II) на репортерной конструкции, содержащей интрон, было в 4 раза выше по сравнению с конструкцией без интрона (25).
Интрон-опосредованное усиление транскрипции коррелирует с образованием петлевой конформации генов, которая объединяет их промо-торные и терминаторные области, возможно, облегчая рециклинг и повторную инициацию Pol II (26). По результатам полногеномного анализа стабильность мРНК положительно коррелировала с числом интронов у мыши и человека (27-30). Помимо увеличения содержания мРНК присутствие интронов повышает эффективность трансляции мРНК у дрожжей, растений, млекопитающих и других животных (31, 32).
Другая гипотеза о механизмах влияния мононуклеотидной замены в интроне заключается в изменении сплайсинга пре-мРНК, необходимого для соответствующей трансляции белка, которая зависит от наличия консенсусных цис-последовательностей, определяющих границы экзон-интрон и регуляторных последовательностей (33). Точечные мутации в этих консенсусных последовательностях могут вызвать неправильное распознавание экзонов и интронов и привести к образованию аберрантного транскрипта мутированного гена. Обычно такие мутации вызывают ошибки в процессе сплайсинга, приводят к неправильному удалению интрона и таким образом вызывают изменения открытой рамки считывания. Недавние исследования подчеркнули значительное количество и важность сплайсинговых мутаций в этиологии наследственных заболеваний.
В работе китайских ученых (34) была обнаружена достоверная ассоциация мононуклеотидных замен в интронах гена MAGI-1 (кодирует ассоциированную с мембраной гуанилаткиназу 1) и гена ACSF2 (кодирует аце-тил-коА-синтетазу — фермент митохондриального матрикса) с яйценоскостью у гусей. D.P. Bai с соавт. (35) также показали, что полиморфизм (A412G) в интроне 1 гена пролактина PRL достоверно ассоциирован с признаками яйценоскости у двух популяций китайских домашних уток. J. Arango с соавт. (36) показали наличие связи между полиморфизмом в интроне 3 гена гормона роста крупного рогатого скота BGH и массой тела в период первой течки и первого отела.
Следовательно, несмотря на то, что мутация rs16455118 (А/С) в гене дисферлина не меняет аминокислотную последовательность, она, возможно, влияет на экспрессию гена и стабильность его транскрипции, что приводит к изменению содержания белка DYSF (18-21). Мутация rs16455118 A/С в гене DYSF может быть связана с хозяйственно значимыми признаками посредством влияния на сплайсинг или стабильность мРНК, а также с неравновесием по сцеплению с неустановленной миссенс-мутацией, ассоциированной с признаком. Влияние гена дисферлина на яйценоскость предположительно связано со способностью суперсемейства ферлинов запускать приток важного для яйцекладки макроэлемента Ca2+ в половые пути курицы-несушки, поскольку эти белки описываются как сенсоры ионов кальция (Ca2+) и ключевые участники ряда физиологических процессов (11-16).
Мы планируем продолжить изучение возможных ассоциаций однонуклеотидных замен rs317801013, rs318045896 и представленной в базу данных ENSEMBL замены T/G в положении 4:90672805 с признаками продуктивности. Выявленные ассоциации в дальнейшем могут быть использованы в селекции высокопродуктивных линий отечественных пород кур.
Таким образом, исследование мононуклеотидного полиморфизма (SNPs) rs16455118 гена дисферлина у кур из генофондной популяции Генетической коллекции редких и исчезающих пород кур (Всероссийский НИИ генетики и разведения сельскохозяйственных животных) показало, что у птицы яичного направления наибольшую частоту встречаемости имел гомозиготный генотип АА , наименьшую — генотип СС , причем в популяции аврора последний полностью отсутствовал. Мясная порода корниш характеризовалась более равномерным распределением генотипов в сравнении с декоративными и яичными породами. В результате секвенирования участка гена дисферлина, расположенного на 4-й хромосоме, у кур породы русская белая выявлены четыре мононуклеотидные замены, находящиеся в 32-м интроне: rs317801013 (G/A) в позиции 90672849, rs16455118 (С/A) в позиции 90672756, rs318045896 (A/G) в позиции 90672862 и мононуклеотидный полиморфизм (T/G) в позиции 90672805. Анализ частоты встречаемости генотипов и отклонения их наблюдаемого распределения от ожидаемого по Харди-Вайнбергу показал сдвиг генетического равновесия по всем обнаруженным у генофондных кур заменам в гене дисферлина. Замена rs16455118 в гене DYSF ассоциирована с повышенной яичной продуктивностью и может быть следствием селекционного давления при усовершенствовании породы.
Список литературы Анализ полиморфизма гена дисферлина у генофондных пород кур
- Kudinov A.A., Dementieva N.V., Mitrofanova O.V., Stanishevskaya O.I., Fedorova E.S., Larkina T.A., Mishina A.I., Plemyashov K.V., Griffin D.K., Romanov M.N. Genome-wide association studies targeting the yield of extraembryonic fluid and production traits in Russian White chickens. BMC Genomics, 2019, 20(1): 270 (doi: 10.1186/s12864-019-5605-5).
- Bansal D., Miyake K., Vogel S.S., Groh S., Chen C.C., Williamson R., McNeil P.L., Campbell K.P. Defective membrane repair in dysferlin-deficient muscular dystrophy. Nature, 2003, 423: 168-172 (doi: 10.1038/nature01573).
- Johnson C.P. Emerging functional differences between the synaptotagmin and ferlin calcium sensor families. Biochemistry, 2017, 56(49): 6413-6417 (doi: 10.1021/acs.biochem.7b00928 ).
- Крутикова А.А., Яковлев А.Ф., Дементьева Н.В., Митрофанова О.В. Исследования SNP в гене дисферлина кур различного направления продуктивности. Птицеводство, 2019, 9-10: 28-34 (doi: 10.33845/0033-3239-2019-68-9-10-28-34).
- Bulankina A.V., Thoms S. Functions of vertebrate ferlins. Cells, 2020, 9(3): 534 (doi: 10.3390/cells9030534).
- Nguyen K., Bassez G., Krahn M., Bernard R., Laforêt P., Labelle V., Urtizberea J.A., Figarella-Branger D., Romero N., Attarian S., Leturcq F., Pouget J., Lévy N., Eymard B. Phenotypic study in 40 patients with dysferlin gene mutations: high frequency of atypical phenotypes. Archives of neurology, 2007, 64(8): 1176-1182 (doi: 10.1001/archneur.64.8.1176).
- Amato A.A., Brown Jr.R.H. Chapter 7 — Dysferlinopathies. In: Handbook of clinical neurology /R.C. Griggs, A.A. Amato (eds.). Elsevier, 2011, 101: 111-118 (doi: 10.1016/B978-0-08-045031-5.00007-4).
- Cárdenas A.M., González-Jamett A.M., Cea L.A., Bevilacqua J.A., Caviedes P. Dysferlin function in skeletal muscle: possible pathological mechanisms and therapeutical targets in dysferlinopathies. Experimental Neurology, 2016, 283(part A): 246-254 (doi: 10.1016/j.expneurol.2016.06.026).
- Pangrsic T., Vogl C. Balancing presynaptic release and endocytic membrane retrieval at hair cell ribbon synapses. FEBS Lett., 2018, 592(21): 3633-3650 (doi: 10.1002/1873-3468.13258).
- Kiselev A., Vaz R., Knyazeva A., Sergushichev A., Dmitrieva R., Khudiakov A., Jorholt J., Smolina N., Sukhareva K., Fomicheva Y., Mikhaylov E., Mitrofanova L., Predeus A., Sjoberg G., Rudenko D., Sejersen T., Lindstrand A., Kostareva A. Truncating variant in myof gene is associated with limb-girdle type muscular dystrophy and cardiomyopathy. Front. Genet., 2019, 10: 608 (doi: 10.3389/fgene.2019.00608).
- Peulen O., Rademaker G., Anania S., Turtoi A., Bellahcène A., Castronovo V. Ferlin. overview: from membrane to cancer biology. Cells, 2019, 8(9): 954 (doi: 10.3390/cells8090954).
- Azakir B.A., Di Fulvio S., Therrien C., Sinnreich M. Dysferlin interacts with tubulin and microtubules in mouse skeletal muscle. PLoS ONE, 2010, 5(4): e10122 (doi: 10.1371/journal.pone.0010122).
- Codding S.J., Marty N., Abdullah N., Johnson C.P. Dysferlin binds SNAREs (soluble N-ethylmaleimide-sensitive factor (NSF) attachment protein receptors) and stimulates membrane fusion in a calcium sensitive manner. Journal of Biological Chemistry, 2016, 291(28): 14575-14584 (doi: 10.1074/jbc.M116.727016).
- Barthélémy F., Defour A., Lévy N., Krahn M., Bartoli M. Muscle сells fix breaches by orchestrating a membrane repair ballet. Journal of Neuromuscular Diseases, 2018, 5(1): 21-28 (doi: 10.3233/JND-170251).
- Abdullah N., Padmanarayana M., Marty N.J., Johnson C.P. Quantitation of the calcium and membrane binding properties of the C2 domains of dysferlin. Biophysical Journal, 2014, 106(2): 382-389 (doi: 10.1016/j.bpj.2013.11.4492).
- Therrien C., Di Fulvio S., Pickles S., Sinnreich M. Characterization of lipid binding specificities of dysferlin C2 domains reveals novel interactions with phosphoinositides. Biochemistry, 2009, 48(11): 2377-2384 (doi: 10.1021/bi802242r ).
- Tjondrokoesoemo A., Park K. H., Ferrante C., Komazaki S., Lesniak S., Brotto M., Ko J.-K., Zhou J., Weisleder N., Ma J. Disrupted membrane structure and intracellular Ca2+ signaling in adult skeletal muscle with acute knockdown of Bin1. PLoS ONE, 2011, 6(9): e25740 (doi: 10.1371/journal.pone.0025740).
- Shaul O. How introns enhance gene expression. The International Journal of Biochemistry & Cell Biology, 2017, 91: 145-155 (doi: 10.1016/j.biocel.2017.06.016).
- Gallegos J.E., Rose A.B. Intron DNA sequences can be more important than the proximal promoter in determining the site of transcript initiation. The Plant Cell, 2017, 29(4): 843-853 (doi: 10.1105/tpc.17.00020).
- Laxa M. Intron-mediated enhancement: a tool for heterologous gene expression in plants? Frontiers in Plant Science, 2017, 7: 1977 (doi: 10.3389/fpls.2016.01977).
- Moore M.J., Proudfoot N.J. Pre-mRNA processing reaches back to transcription and ahead to translation. Cell, 2009, 136(4): 688-700 (doi: 10.1016/j.cell.2009.02.001).
- Brinster R.L., Allen J.M., Behringer R.R., Gelinas R.E., Palmiter R.D. Introns increase transcriptional efficiency in transgenic mice. Proceedings of the National Academy of Sciences, 1988, 85(3): 836-840 (doi: 10.1073/pnas.85.3.836).
- Furger A., O'Sullivan J.M., Binnie A., Lee B.A., Proudfoot N.J. Promoter proximal splice sites enhance transcription. Genes & Development, 2002, 16(21): 2792-2799 (doi: 10.1101/gad.983602).
- Samadder P., Sivamani E., Lu J.L., Li X.G., Qu R.D. Transcriptional and post-transcriptional enhancement of gene expression by the 5' UTR intron of rice rubi3 gene in transgenic rice cells. Molecular Genetics and Genomics, 2008, 279(4): 429-439 (doi: 10.1007/s00438-008-0323-8).
- Laxa M., Müller K., Lange N., Doering L., Pruscha J.T., Peterhänsel C. The 5' UTR intron of Arabidopsis GGT1 aminotransferase enhances promoter activity by recruiting RNA polymerase II. Plant Physiology, 2016, 172(1): 313-27 (doi: 10.1104/pp.16.00881).
- Moabbi A.M., Agarwal N., El Kaderi B., Ansari A. Role for gene looping in intron-mediated enhancement of transcription. Proceedings of the National Academy of Sciences,2012, 109(22): 8505-8510 (doi: 10.1073/pnas.1112400109).
- Duan J.B., Shi J.X., Ge X.J., Dolken L., Moy W., He D.L., Shi S., Sanders A.R., Ross J., Gejman P.V., Genome-wide survey of interindividual differences of RNA stability in human lymphoblastoid cell lines. Scientific Reports, 2013, 3(1): 1318 (doi: 10.1038/srep01318).
- Narsai R., Howell K.A., Millar A.H., O'Toole N., Small I., Whelan J. Genome-wide analysis of mRNA decay rates and their determinants in Arabidopsis thaliana. The Plant Cell, 2007,19(11): 3418-3436 (doi: 10.1105/tpc.107.055046).
- Sharova L.V., Sharov A.A., Nedorezov T., Piao Y., Shaik N., Ko M.S.H. Database for mRNA half-life of 19 977 genes obtained by DNA microarray analysis of pluripotent and differentiating mouse embryonic stem cells. DNA Research, 2009, 16(1): 45-58 (doi: 10.1093/dnares/dsn030).
- Wang H.-F., Feng L.A., Niu D.-K. Relationship between mRNA stability and intron presence. Biochemical and Biophysical Research Communications, 2007, 354(1): 203-208 (doi: 10.1016/j.bbrc.2006.12.184 ).
- Akua T., Shaul O. The Arabidopsis thaliana MHX gene includes an intronic element that boosts translation when localized in a 5' UTR intron. Journal of Experimental Botany, 2013, 64(14): 4255-4270 (doi: 10.1093/jxb/ert235).
- Hoshida H., Kondo M., Kobayashi T., Yarimizu T., Akada R. 5'-UTR introns enhance protein expression in the yeast Saccharomyces cerevisiae. Applied Microbiology and Biotechnology, 2017, 101(1): 241-25 (doi: 10.1007/s00253-016-7891-z).
- Abramowicz A., Gos M. Splicing mutations in human genetic disorders: examples, detection, and confirmation. Journal of Applied Genetics, 2018, 59(3): 253-268 (doi: 10.1007/s13353-018-044-7).
- Yang Y., An C., Yao Y., Cao Z., Gu T., Xu Q., Chen G. Intron polymorphisms of MAGI-1 and ACSF2 and effects on their expression in different goose breeds. Gene, 2019, 701: 82-88 (doi: 10.1016/j.gene.2019.02.102).
- Bai D.P., Hu Y.Q., Li Y.B., Huang Z.B., Li A. Polymorphisms of the prolactin gene and their association with egg production traits in two Chinese domestic ducks. British Poultry Science, 2019, 60(2): 125-129 (doi: 10.1080/00071668.2019.1567909).
- Arango J., Echeverri J.J., López A. Association between a polymorphism in intron 3 of the bovine growth hormone gene and growth traits in Holstein heifers in Antioquia. Genetics and Molecular Research, 2014, 13(3): 6191-6199 (doi: 10.4238/2014.August.15.1).


