Анализ полиморфизма микросателлитных локусов ДНК рыжика посевного Camelina sativa
Автор: Логинова Е.Д., Рамазанова С.А.
Рубрика: Селекция, семеноводство и биотехнология сельскохозяйственных растений
Статья в выпуске: 4 (200), 2024 года.
Бесплатный доступ
Рыжик (Camelina sativa) - это двудольное масличное растение семейства Brassicaceae, родом из Южной Европы и Юго-Западной Азии. Рыжик требует меньше воды и удобрений и устойчив к вредителям и патогенам в отличие от других культур. Масло рыжика содержит ненасыщенные жирные кислоты: линоленовую, линолевую, олеиновую и эйкозеновую, имеет низкий уровень эруковой кислоты. Оно применяется в кормопроизводстве, производстве биодизеля, полимеров, лаков, олифы и красок, косметологии и пищевой промышленности. Для эффективного создания новых сортов рыжика необходимо изучать его генетическое разнообразие с помощью молекулярно-генетических методов, включая SSR-маркеры. Микросателлиты широко используются в анализе генома растений. Целью данной работы было изучение генетического разнообразия озимой и яровой форм рыжика селекции ВНИИМК с использованием новых экспериментальных SSR-маркеров. В работе изучали 18 сортообразцов рыжика посевного озимой и яровой форм из коллекции ВНИИМК и два образца сорта Крепыш. Генетическое разнообразие исследовали с помощью девяти SSR-маркеров. Продукты ПЦР детектировали методом капиллярного электрофореза на генетическом анализаторе Нанофор-05. В результате было выявлено 42 аллеля, от 2 до 7 на локус. Среднее эффективное число аллелей - 3,00. Среднее значение индекса полиморфного информационного содержания (PIC) - 0,66. Из 20 генотипов восемь не удалось отличить по девяти SSR-маркерам. Для их идентификации необходимо увеличить количество SSR-локусов. Образцы сорта Крепыш отличаются по семи локусам. Исследование показало генетическую неоднородность четырех сортов рыжика. Изучение внутрисортового полиморфизма у сортов рыжика посевного селекции ВНИИМК будет продолжено.
Рыжик посевной (camelina sativa), ssr-маркеры, микросателлиты, генетическое разнообразие, аллели, дендрограмма
Короткий адрес: https://sciup.org/142243661
IDR: 142243661 | УДК: 631.522:633.854 | DOI: 10.25230/2412-608X-2024-4-200-63-69
Текст научной статьи Анализ полиморфизма микросателлитных локусов ДНК рыжика посевного Camelina sativa
Введение . Рыжик посевной ( Camelina sativa (L.) Crantz) – это двудольное масличное растение, принадлежащее к семейству Brassicaceae. Его родиной являются Южная Европа и Юго-Западная Азия. Археологические находки подтверждают существование рыжика в Европе и свидетельствуют о том, что его выращивание началось еще в 4000 году до нашей эры [1].
Рыжик отличается от других культур этого семейства тем, что способен переносить почвенную и воздушную засуху, а также для его возделывания требуется меньшее количество удобрений. Кроме того, он обладает высокой устойчивостью к распространенным вредителям и патогенам, которые обычно поражают родственные виды [2].
В основном рыжиковое масло содержит ненасыщенные жирные кислоты. Наиболее распространёнными из них являются линоленовая, линолевая, олеиновая и эй-козеновая [3]. По сравнению с другими представителями семейства Brassicaceae рыжиковое масло выделяется низким содержанием эруковой кислоты [4].
Благодаря своему уникальному составу, рыжиковое масло находит широкое применение в различных областях промышленности [5]. В кормопроизводстве оно может выступать как заменитель или дополнение к другим жирам, таким как рыбий и бройлерный, в рационах животных. В производстве биодизеля рыжиковое масло используется в качестве основы для реактивного топлива [6]. В полимерной промышленности оно применяется для производства экологически чистых полимеров, лаков, олифы и красок. Это связано с тем, что масло рыжика является одним из быстросохнущих масел из-за высокого содержания полиненасыщенных жирных кислот. В косметической отрасли рыжиковое масло применяется в качестве средства для ухода за кожей. Кроме того, из-за высокого содержания жирных кислот омега-3 и низкого уровня эруковой кислоты рыжиковое масло находит применение в пищевой промышленности. Оно используется в качестве заменителя молочного жира, что делает его ценным ингредиентом для разнообразных продуктов питания [7; 8].
Рыжик посевной – это культура, которая имеет большой потенциал для развития. Чтобы эффективно решать селекционные задачи по созданию новых сортов, необходимо проводить тщательный отбор исходного материала. Изучение генетического разнообразия культуры с использованием современных молекулярно-генетических методов позволит усовершенствовать этот процесс. Однако исследования по генотипированию сортов C. sativa с помощью молекулярных маркеров пока ещё не проводились в достаточном объёме.
Впервые образцы рыжика были изучены с применением метода RAPD (Random Amplified Polymorphic DNA) [9]. Затем были проведены работы по генотипированию сортов из разных географических регионов с использованием метода AFLP (Amplified Fragment Length Polymorphism) [10]. ДНК-маркеры уже давно зарекомендовали себя как надёжный инструмент для генотипирования многих видов растений, в том числе тех, которые имеют незначительные фенотипические отличия. Одним из наиболее эффективных типов ДНК-маркеров для идентификации генотипов являются микросателлиты, также известные как SSR-маркеры (Simple Sequence Repeats). Мик-росателлитные локусы характеризуются высокой степенью полиморфизма, кодоминантным наследованием, простотой де- текцией результатов [11]. Они представляют собой тандемные повторы 1–6 нуклеотидов и расположены как в кодирующей, так и в некодирующей областях генома всех организмов. Высокая степень аллельной изменчивости, выявляемая с помощью микросателлитных маркеров, обусловлена вариацией числа повторяющихся мотивов в локусе, вызванной проскальзыванием репликации и/или неравным кроссинговером во время мейоза [12]. Несмотря на недостаточное понимание функций мотивов SSR-локусов, микросателлиты широко используются в анализе генома растений [13].
-
А. Manca с соавторами разработали 16 микросателлитных маркеров и провели анализ генетической изменчивости коллекции из 40 образцов рыжика посевного [14]. В России аналогичное исследование было проведено на 18 сортах рыжика с использованием тех же маркеров SSR [1 5]. Авторам не удалось различить с использованием примененной системы маркеров некоторые сорта рыжика.
Целью данной работы было изучение генетического разнообразия озимой и яровой форм рыжика селекции ВНИИМК с использованием новых, разработанных нами экспериментальных SSR-маркеров.
Материалы и методы . В данной работе изучали 18 сортообразцов рыжика посевного коллекции ВНИИМК, из которых десять озимой формы: РО- 1/22 (1), РО 22-2 (2), РО 22-4 (4), РО 01-15/13 (13), РО 22-15 (15), РО 22-16 (16), РО 08-14/17 (17), РО 22-18 (18), РО 22-19 (19), РО 01-15/30 (30), и восемь – яровой: РЯ-1009/35 (35), РЯ-1012/38 (38), РЯ-1016/42 (42), РЯ-1017/43 (43), РЯ 45К-684/14 (45), РЯ-1026/2798 (51), РЯ-1030/54 (54), РЯ 49К-684/14 (49), а также два индивидуальных семени сорта Крепыш – К1 и К2 (в скобках указан порядковый номер образца).
Генетическое разнообразие генотипов рыжика исследовали с помощью девяти разработанных нами экспериментальных SSR-маркеров: Рсh13.2, Рсh14.1, Рсh14.2, Рсh12.2, Рсh18.2, Рсh14.3, Рсh3.2, Рсh1.2,
Рсh20.3, и синтезированных ООО «НПФ Синтол» (Россия).
Экстракцию ДНК проводили на автоматической станции Auto-Pure 96 (Allsheng, Китай) с помощью набора МагноПрайм ФИТО. ДНК выделяли из смеси трех семян каждого образца рыжика, за исключением сорта Крепыш, каждый образец данного сорта выделен из отдельного семени.
Реакционная смесь для полимеразной цепной реакции (ПЦР) объемом 25 мкл содержала: 10х ПЦР-буфер, 2,5 мМ MgCl 2 , 2,5 мМ dNTP; 1 е.а. ДНК-полимеразы; концентрация прямого (F) и обратного (R) праймеров – 10 пкМ/мкл; ДНК – 100 нг. Амплификацию проводили в термоциклере Applied Biosystems MiniAmp (Thermo Fisher Scientific, США) по следующим температурно-временным режимам: начальная денатурация 94 °С – 3 мин, далее 35 циклов: денатурация 94 °С – 25 сек, отжиг праймера 60 °С – 25 сек, элонгация 72 °С – 40 сек, и финальная элонгация 72 °С – 3 мин.
Детекция ПЦР-продуктов проводилась с помощью высокоразрешающего капиллярного электрофореза в денатурирующих условиях на генетическом анализаторе Нанофор-05 (Синтол, Россия). Для проведения фрагментного анализа прямые праймеры (F) были модифицированы одной из четырех флуоресцентных меток (табл. 1).
Анализ информативности микросател-литных локусов включал определение количества аллелей (n), эффективного числа аллелей (n e ) и индекса полиморфного информационного содержания (PIC – Polymorphic Index Content). Вычисления проводили по следующим формулам:
n
ne= 1/ Σ P ji² (1) j=1
n
PICi = 1- Σ Pji² , (2)
j=1
где Pi – частота j паттерна для локуса i и суммирование распространяется на n паттернов [16].
Для построения дендрограммы генетического подобия использовали программное обеспечение Past версии 4.7.0.0 [17].
Результаты и обсуждение . Для анализа генетического разнообразия образцов рыжика посевного были выбраны наиболее полиморфные микросателлитные локусы, отобранные нами в исследованиях, проведенных ранее [18]. Их характеристика представлена в таблице 1. В результате исследования полиморфизма девяти микросателлитных локусов ДНК рыжика посевного всего в данной группе образцов было выявлено 42 аллеля, от 2 до 7 на локус, что в среднем составило 4,6. Наибольшее количество аллелей обнаружено у локуса Рсh14.3 (291, 297, 307, 310, 313, 316, 319). У локуса Рсh20.3 выявлено всего два аллеля (388, 401).
Таблица 1
Характеристика изученных SSR-локусов
|
SSR-локус |
Флуоресцентная метка |
Диапазон выявленных аллелей |
Количество аллелей |
Эф-фек-тивное число аллелей |
PIC |
|
Рсh14.1 |
R6G |
151-169 |
6 |
3,48 |
0,71 |
|
Рсh14.2 |
R6G |
223-229 |
3 |
2,89 |
0,65 |
|
Рсh14.3 |
R6G |
291-319 |
7 |
3,71 |
0,73 |
|
Рсh13.2 |
FAM |
267-282 |
5 |
3,50 |
0,71 |
|
Рсh12.2 |
TAMRA |
277-301 |
6 |
4,02 |
0,75 |
|
Рсh18.2 |
FAM |
268-292 |
5 |
2,69 |
0,63 |
|
Рсh3.2 |
R6G |
264-270 |
4 |
3,16 |
0,68 |
|
Рсh1.2 |
TAMRA |
279-294 |
3 |
2,42 |
0,59 |
|
Рсh20.3 |
ROX |
388-401 |
2 |
2,00 |
0,50 |
|
Среднее |
- |
- |
4,6 |
3,00 |
0,66 |
Показатели уровня полиморфизма изученных микросателлитных локусов были достаточно высокими в данной выборке образцов рыжика. Значение эффективного числа аллелей варьировало от 2,00 до 4,02 и в среднем составило 3,00. Этот показатель характеризует выявленные аллели по частоте их встречаемости в изучаемой выборке генотипов. Количество выявленных аллелей будет равно количеству эффективных, если частоты их встречаемости одинаковы. Важным показателем, отражающим информативность микросателлит-ных локусов, является индекс полиморфного информационного содержания (PIC).
Этот индекс характеризует не только количество выявленных аллелей, но и относительные частоты их встречаемости. Редкие аллели оказывают меньшее влияние на значение PIC по сравнению с аллелями, которые встречаются чаще. Значения PIC могут варьироваться от 0 до 1. Для изученных нами SSR-локусов индекс PIC варьировался от 0,5 для локуса Рсh20.3 до 0,75 для локуса Рсh12.2. Среднее значение индекса полиморфного информационного содержания для изученной группы генотипов рыжика составило 0,66.
В таблице 2 представлены данные по аллельному составу и длинам полученных фрагментов изученных образцов. Поскольку в нашем исследовании анализ ДНК сортообразца проводили не у индивидуальных генотипов, а их смесей, в некоторых образцах было обнаружено по два аллеля разной длины, что свидетельствует о наличии внутрисортового полиморфизма. Например, у образца РО 22-2 внутрисортовой полиморфизм был обнаружен по четырем локусам Рсh14.1, Рсh12.2, Рсh14.3, Рсh3.2 (табл. 2).
Как видно из таблицы большинство изученных сортообразцов различаются по нескольким аллелям. Однако мы не смогли с использованием данного набора праймеров различить шесть образцов яровой формы рыжика (№ 35, 38, 42, 45, 51 и 54) и два озимой формы (№ 1 и 18). При этом образцы 35, 38 и 42 имеют одинаковое происхождение, но отличаются от образцов 45, 51 и 54, которые в свою очередь все имеют разное происхождение. Образцы озимой формы 1 и 18 также имеют одинаковое происхождение, поэтому, вероятно, для их идентификации необходимо увеличить количество SSR-локусов. В то же время семена из сорта Крепыш отличились друг от друга по семи локусам. Вторая семянка (К2), очевидно, является гибридным растением, так как по трем локусам у него идентифицировано два фрагмента разной длины.
Следует обратить внимание, что локус Рсh20, по-видимому, является специфичным для озимой и яровой форм рыжика, так как у всех образцов озимой формы выявлен фрагмент 401 п.н., а у яровой – 388 п.н. (табл. 2).
Аллельный состав образцов рыжика посевного
Таблица 2
Озимые формы рыжика посевного
|
№ образца |
Рсh13.2 |
Рсh14.1 |
Рсh14.2 |
Рсh12.2 |
Рсh18.2 |
Рсh14.3 |
Рсh3.2 |
Рсh1.2 |
Рсh20.3 |
|
1 |
270* |
154 |
226 |
280 |
268 |
291 |
264 |
279 |
401 |
|
2 |
267 |
151/154 |
223 |
280/301 |
280 |
291/313 |
261/264 |
291 |
401 |
|
4 |
276 |
154 |
223 |
301 |
292 |
297 |
267 |
279 |
401 |
|
13 |
267 |
151 |
223 |
280 |
280 |
313/319 |
261 |
279 |
401 |
|
15 |
267 |
160 |
223 |
277 |
268 |
313 |
267 |
279 |
401 |
|
16 |
267 |
160 |
223 |
286 |
268 |
313 |
261 |
291 |
401 |
|
17 |
270 |
157 |
226 |
301 |
268 |
291 |
264 |
279 |
401 |
|
18 |
270 |
154 |
226 |
280 |
268 |
291 |
264 |
279 |
401 |
|
19 |
270 |
154 |
223 |
280 |
268 |
316 |
267 |
279 |
401 |
|
30 |
267 |
154 |
223 |
286 |
268 |
297 |
261/267 |
279 |
401 |
|
Яровые формы рыжика посевного |
|||||||||
|
№ образца |
Рсh13.2 |
Рсh14.1 |
Рсh14.2 |
Рсh12.2 |
Рсh18.2 |
Рсh14.3 |
Рсh3.2 |
Рсh1.2 |
Рсh20.3 |
|
35 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
38 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
42 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
43 |
270/276 |
157 |
226 |
292 |
268 |
291 |
261 |
279 |
388 |
|
45 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
49 |
270 |
157 |
226 |
292 |
274 |
291 |
264 |
279 |
388 |
|
51 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
54 |
279 |
160 |
229 |
295 |
280 |
310 |
270 |
294 |
388 |
|
К 1 |
282 |
169 |
229 |
295 |
280 |
313 |
270 |
279 |
388 |
|
К 2 |
279 |
163/169 |
229 |
301 |
271 |
307/316 |
264/267 |
291 |
388 |
* – длина фрагмента, пар нуклеотидов
Чтобы наглядно продемонстрировать генетическое разнообразие и степень родства между исследуемыми генотипами, была построена дендрограмма иерархической кластеризации, основанная на методе UPGMA (Unweighted Pair Group Method with Arithmetic Mean) (рисунок).
Как видно из рисунка, образцы разделились на два основных кластера. В первый кластер вошли некоторые образцы яровой формы рыжика посевного и два образца сорта Крепыш. Второй кластер составляют преимущественно озимые сортооб-разцы за исключением двух генотипов (43, 49). В данном кластере практически все генотипы отличались друг от друга, что свидетельствует о большей генетической разнородности озимых форм рыжика.
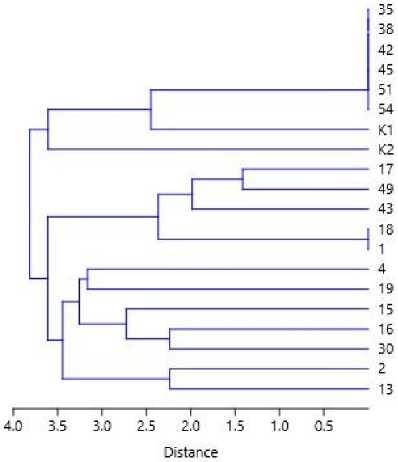
Рисунок – Дендрограмма генетического сходства 20 генотипов рыжика посевного на основе анализа полиморфизма девяти
SSR-локусов ДНК
Заключение . Таким образом из 20 генотипов рыжика посевного восемь не удалось отличить по изученным девяти SSR-маркерам. Это сортообразцы ярового рыжика РЯ-1009/35 (35), РЯ-1012/38 (38), РЯ-1016/42 (42) (имеют одинаковое происхождение), РЯ 45К-684/14 (45), РЯ-1026/2798 (51), РЯ-1030/54 (54), и два сортообразца озимого рыжика – РО-1/22 (1), РО 22-18 (18), также имеющих одинаковое происхождение. Поэтому в дальнейших исследованиях следует увеличить выборку SSR-маркеров для анализа.
Два образца сорта Крепыш отличились друг от друга по семи SSR-локусам. Это исследование показало генетическую неоднородность четырех сортов рыжика посевного. Изучение внутрисортового полиморфизма у сортов рыжика селекции ВНИИМК будет продолжено.
Список литературы Анализ полиморфизма микросателлитных локусов ДНК рыжика посевного Camelina sativa
- Popa A.L., Jurcoane Ş., Dumitriu B.Ş. Camelina sativa oil – a review // Scientific Bulle-tin, Series F. Biotechnologies. – 2017. – Vol. 21 – P. 233–238.
- Kim N., Li Y., Sun X.S. Epoxidation of Camelina sativa oil and peel adhesion properties // Industrial Crops and Products. – 2015. – Vol. 64. – Р. 1–8.
- Karvonen H.M. Aro A., Tapola N.S. [et al.]. Effect of [alpha]-linolenic acid [ndash] rich Camelina sativa oil on serum fatty acid composi-tion and serum lipids in hypercholesterolemic subjects // Metabolism Clinical and Experi-mental. – 2002. – Vol. 51. – No. 10. – P. 1253–1260.
- Chantsalnyam B., Otgonbayar C., Enkhtun-galag O. [et al.]. Physical and chemical character-istics and fatty acids composition of seeds oil iso-lated from Camelina sativa (L.) cultivated in Mongolia // Mongolian Journal of Chemistry. – 2013. – Vol. 14. – P. 80–83.
- Mondor M., Hernández‐Álvarez A.J. Came-lina sativa composition, attributes, and applica-tions: a review // European Journal of Lipid Sci-ence and Technology. – 2022. – Vol. 124. – No. 3. – Art. No. 2100035.
- Pecchia P., Russo R., Brambilla I. [et al.]. Biochemical seed traits of Camelina sativa – an emerging oilseed crop for biofuel: environmental and genetic influences // Journal of Crop Im-provement. – 2014. – Vol. 28. – No. 4. – P. 465–483.
- Zubr J. Dietary fatty acids and amino acids of Camelina sativa seed // Journal of Food Qual-ity. – 2003. – Vol. 26. – No. 6. – P. 451–462.
- Rokka T., Alеn K., Valaja J. [et al.]. The ef-fect of a Camelina sativa enriched diet on the composition and sensory quality of hen eggs // Food Research International. – 2002. – Vol. 35 (2). – P. 253–256.
- Vollmann J., Grausgruber H., Stift G. Ge-netic diversity in camelina germplasm as revealed by seed quality characteristics and RAPD poly-morphism // Plant Breeding. – 2005. – Vol. 124. – №. 5. – Р. 446–453.
- Ghamkhar K., Croser J., Aryamanesh N. [et al.]. Camelina (Camelina sativa (L.) Crantz) as an alternative oilseed: molecular and ecogeographic analyses // Genome. – 2010. – Vol. 53 (7). – P. 558–567.
- Kashi Y., Nave A., Darvasi A. [et al.]. How is it that microsatellites and random oligonucleo-tides uncover DNA fingerprint patterns // Mamm. Genome. – 1994. – Vol. 5. – Vol. 5. – P. 525–530.
- Ellegren H. Microsatellites: simple se-quences with complex evolution // Nature reviews genetics. – 2004. – Vol. 5. – №. 6. – Р. 435-445.
- Kalia R.K., Rai M.K., Kalia S. [et al.]. Mi-crosatellite markers: an overview of the recent progress in plants // Euphytica. – 2011. – Vol. 177 (3). – P. 309–334.
- Manca A., Galasso I. Development of sim-ple sequence repeat (SSR) markers in Camelina sativa (L.) Crantz // Minerva Biotec. – 2010. – Vol. 22. – P. 43–45.
- Базанов Т.А., Ущаповский И.В., Логи-нова Н.Н. [и др.]. Оценка генетического разнообразия сортов рыжика посевного (Camelina sativa L.) с использованием SSR-маркеров // Аграрная наука. – 2021. – №. 9. – С. 108–112.
- Nei M. Analysis of gene diversity in subdi-vided populations // Proc. of the National Acad-emy of Sciences USA. – 1973. – Vol. 70 (12). – Р. 3321–3323. DOI: 10.1073/pnas.70.12.3321.
- Hammer Ø., Harper D.A.T., Ryan P.D. Past: paleontological statistics software package for education and data analysis // Palaeontologia electronica. – 2001. – Vol. 4 (1). – Р. 1–9.
- Логинова Е.Д., Рамазанова С.А., Гучетль С.З. Проверка SSR-маркеров для геноти-пирования рыжика посевного // Актуальные проблемы науки и практики в исследованиях молодых ученых: сб. I-й междунар. науч.-практич. конф., Новосибирск, 21–22 мая 2024 г. – Новосибирск: ИЦ НГАУ «Золотой колос», 2024. – С. 11–15.


