Анализ сцепления гена устойчивости к расе G заразихи с микросателлитными локусами у линии-донора подсолнечника селекции ВНИИМК RGP1
Автор: Гучетль С.З., Савиченко Д.Л.
Рубрика: Селекция и семеноводство сельскохозяйственных растений
Статья в выпуске: 2 (186), 2021 года.
Бесплатный доступ
Одним из главных биотических факторов, лимитирующих получение высоких урожаев подсолнечника, является заразиха (Orobanche cumana Wallr.). Наиболее эффективным и экологически безопасным методом борьбы считается возделывание устойчивых сортов и гибридов подсолнечника. Создание резистентных генотипов включает в себя поиск и использование источников устойчивости в селекционном процессе, а также обеспечение точных и производительных процедур оценки материала. Целью исследования являлся анализ сцепления гена Or7 с микросателлитными локусами у линии-донора устойчивости к расе G заразихи коллекции ВНИИМК. Материал исследования - линия-донор устойчивости к расе G заразихи RGP1 и восприимчивая к этой расе линия ВК678 селекции ВНИИМК. Растения подсолнечника были скрещены в полевых условиях для получения поколения F1. Также проведено самоопыление растений F1 с получением потомства F2. Растения тестировали в теплице методом ранней диагностики на инфекционном фоне из семян заразихи расы G. ДНК подсолнечника экстрагировали из верхушечных листьев молодых побегов вегетирующих растений. Для ПЦР анализа использовали три SSR-праймера, показавшие полиморфизм у родительских линий: ORS 683, ORS 1040, ORS 1112. Был проведен тест на совместное наследование гена Or7 c этими локусами, а также SSR-локусов друг с другом. Показано независимое наследование гена Or7 c локусами ДНК ORS 683, ORS 1040, ORS 1112, а также SSR-локусов ORS 1040 с ORS 1112 и ORS 1040 с ORS 683. Локусы ORS683 - ORS 1112 сцеплены с частотой рекомбинации 0,27 ± 0,41 (27 сМ). В результате проведенных исследований было исключено расположение гена Or7 в области непосредственной близости к микросателлитным локусам ORS 683, ORS 1040, ORS 1112. На основании изученных литературных источников и репрезентативного генома подсолнечника HanXRQr2.0-SUNRISE составлена частичная физическая карта LG3 для определения области дальнейшего поиска локализации гена Or7 и ДНК-маркеров, косегрегирующих с ним.
Подсолнечник, заразиха, раса G, устойчивость, микросателлитные локусы, ПЦР, сцепление
Короткий адрес: https://sciup.org/142229254
IDR: 142229254 | УДК: 633.854.78:575:632.9 | DOI: 10.25230/2412-608X-2021-2-186-3-9
Текст научной статьи Анализ сцепления гена устойчивости к расе G заразихи с микросателлитными локусами у линии-донора подсолнечника селекции ВНИИМК RGP1
Введение. Одним из главных биотических факторов, лимитирующих получение высоких урожаев подсолнечника, является паразитическое растение заразиха ( Orobanche cumana Wallr.). При отсутствии резистентности у возделываемых гибридов ущерб урожаю, наносимый данным паразитом, может составить до 100 % [1]. На данный момент сообщается об обнаружении, по крайней мере, восьми рас, обозначенных буквами от А до Н в порядке возрастания вирулентности [2].
Наиболее эффективным и экологически безопасным методом борьбы с заразихой считается возделывание устойчивых сортов и гибридов подсолнечника. Создание резистентных генотипов включает в себя поиск и использование источников устойчивости в селекционном процессе, а также обеспечение точных и производительных процедур оценки материала. Учитывая, что устойчивость подсолнечника быстро преодолевается с появлением более вирулентных рас паразита, необходимо непрерывно вести процесс внедрения и комбинирования генов устойчивости [3; 4].
Устойчивость подсолнечника к расам заразихи контролируется различными способами: одним доминантным [5], од-4
ним или двумя рецессивными и двумя частично доминантными аллелями генов [4; 6]. Это говорит о наличии различных источников устойчивости, обусловленных происхождением генетического материала. Тем самым для обеспечения оценки селекционного материала с помощью ДНК-маркеров (маркер-ассо-циированной селекции) необходимо картировать ген устойчивости для каждого источника. Пять локусов количественных признаков (QTL), обеспечивающих устойчивость к расе E, и шесть QTL – к расе F, были обнаружены в семи разных хромосомах [5]. Расоспецифичный ген, контролирующий устойчивость к расе E – Or 5 картирован в теломерной области хромосомы 3, находится на 7,5 сМ выше маркера ORS1036 [7]. В той же хромосоме был обнаружен ген устойчивости к расе выше F, который предварительно обозначен как or ab-vl-8 и локализован на 1,5 сМ ниже маркера ORS683 и 19,6 сМ ниже маркера ORS1036 [4]. Несмотря на близкое расположение к гену Or 5 , доказано, что эти гены различны. Позже, I. Ime-rovski et al. [8] картировали от 2 до 23 значимых QTL в геноме подсолнечника. Два основных QTL располагались в хромосоме 3. Так же один ген устойчивости к расе F O. cumana был описан и картирован в хромосоме 7 [9].
Во ВНИИМК были созданы линии подсолнечника, устойчивые к расе G заразихи. Установлено, что устойчивость у этих линий контролируется одним геном Or 7 с неполным доминированием [10]. Эти линии представляют ценный источник для переноса генов с использованием ДНК-маркеров (МАС). Использование данных генов в селекции позволит создавать безопасные и качественные продукты питания с использованием современных генетических методов. В связи с этим целью исследования является анализ сцепления гена Or 7 с микросателлитными локусами у линии-донора устойчивости к расе G заразихи RGP1 коллекции ВНИИМК.
Материалы и методы. Материал исследования – линия-донор устойчивости к расе G заразихи RGP1 и восприимчивая к этой расе линия ВК678 селекции ВНИИМК. Растения подсолнечника были скрещены в полевых условиях для получения гетерозиготного поколения F 1. Затем проведено самоопыление растений F 1 с получением потомства F 2. Растения тестировали в теплице на устойчивость и восприимчивость на инфекционном фоне из семян заразихи расы G, применяя метод ранней диагностики [10].
ДНК подсолнечника экстрагировали из верхушечных листьев молодых побегов вегетирующих растений. Выделение ДНК производилось по методу Vroh et аl. с добавлением активированного угля [11]. Для проведения полимеразной цепной реакции использовали 25 мкл реакционной смеси следующего состава: 67 мМ трис-HCl, рН 8,8; 16,6 мM сульфата аммония; 1,5–3 мM MgCl 2 ; 0,01 % Tween 20; по 0,2 мM дезоксирибонуклеозидфосфатов; по 10 пМ праймеров; 10 нг матричной ДНК и 1 ед. рекомбинантной термостабильной ДНК полимеразы (Си-бэнзим, Россия). Для амплификации использовали термоциклер S1000тм (BioRad, США). Для увеличения специфичности гибридизации праймеров с матрицей ДНК использовали ступенчатую ПЦР (touchdown PCR). Условия амплификации: начальная денатурация 95 °C в течение 2 мин, 1 цикл при соблюдении температурно-временного режима: 94 °C – 30 сек, 64 °C – 30 сек, 65 °C – 45 сек, затем шесть циклов с уменьшением температуры отжига на 1 °C на цикл, следом 32 цикла при соблюдении температурновременного режима: 94 °C – 30 сек, 58 °C – 30 сек и 65 °C – 45 сек. Финальная элонгация 65 °C – 20 мин. Для ПЦР анализа использовали три SSR-праймера, показавшие полиморфизм у родительских линий в предыдущем исследовании: ORS 683, ORS 1040, ORS 1112 [12].
Электрофорез продуктов амплификации проводили в полиакриламидном геле
(8 %, 1хТБЕ) с использованием камеры VE-20 для вертикального электрофореза (Хеликон, Россия) в течение 2,5–3 часов при силе тока 40–50 mA и напряжении 200–230 V. Результаты электрофореза документировали при помощи гель-доку-ментирующей видеосистемы BIO-PRINT (Vilber Lourmat, Франция). Размер фрагментов ДНК определяли с использованием программного обеспечения BioCapture (Vilber Lourmat, Франция) относительно маркера длины фрагментов ДНК GeneRuler 100 bp DNA Ladder Thermo Scientific (Сибэнзим, Россия). Математическую обработку результатов расщепления проводили с использованием χ2-критерия соответствия фактических расщеплений теоретически ожидаемым в моно- и дигибридных скрещиваниях. Расчет частоты рекомбинации r и ошибку рекомбинации s p по результатам расщепления в F 2 рассчитывали по методу максимального правдоподобия [13].
Результаты и обсуждение. Для установления локализации гена, контролирующего устойчивость к расе G заразихи, I. Imerovski et al. была составлена частичная SSR карта LG3 подсолнечника, содержащая ген or ab-vl-8 . Согласно карте, данный ген локализуется в верхней части хромосомы [4]. С использованием BSA-seq I. Imerovski et al . также были обнаружены предполагаемые гены-кандидаты устойчивости – HanXRQChr03g0076321 и HanXRQChr03g0065841 [8]. Основываясь на полученных этими авторами результатах, мы проводили подбор генетических маркеров к гену Or 7 [12]. В рамках настоящего исследования используемые в работе SSR-локусы были сопоставлены со сборкой репрезентативного генома подсолнечника HanXRQr2.0-SUNRISE. По этим данным с помощью инструмента Basic Local Alignment Search Tool (BLAST®) составлена физическая карта положения локусов в LG3 (рисунок).
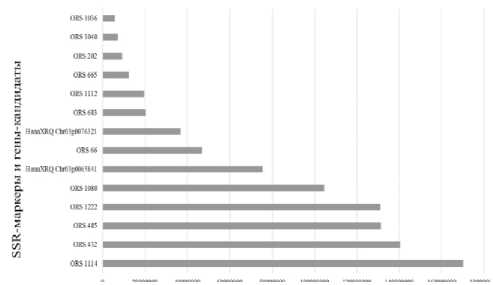
Таблица 1
Аллельные состояния SSR-локусов ДНК подсолнечника у гибрида F 1 (ВК678 × RGP1) и его родительских линий, восприимчивой и устойчивой к расе G заразихи
Рисунок – Положение SSR-локусов, предположительно сцепленных с геном Or 7 , и генов-кандидатов в LG3 H. annuus
Согласно этой карте, в верхней части LG3 располагается группа локусов (ORS 683, ORS 1112, ORS 1040, ORS 665, ORS 202, ORS 1036), находящихся выше предполагаемого нахождения гена устойчивости к расе G заразихи (рисунок).
Для Or 7 у были RGP1,
установления локализации гена линий селекции ВНИИМК нами выбраны линии подсолнечника устойчивая к расе G заразихи, и
ВК 678, восприимчивая к этой расе. У этих линий обнаружено три маркерных микросателлитных локуса с контрастными аллельными вариантами [12]. Это локусы ORS 1040, ORS 1112, ORS 683. Полученные после скрещивания родительских линий семена подсолнечника F 1 (ВК678 × RGP1) были проверены на гиб-ридность по ДНК локусам. По результатам анализа было установлено, что два локуса – ORS 683, ORS 1112 – имели кодоминантное наследование аллелей. По этим локусам у гибрида выявили две родительские фракции ДНК. Представляет интерес тот факт, что I. Imerovski et al. [4] определили эти локусы, как локусы с доминантным наследованием аллелей. У локуса ORS 1040 выявлена одна фракция, следовательно, имеет место доминирование одного аллеля над другим (табл. 1).
После получения потомства F 2 анализ расщепления по устойчивости подсолнечника к расе G заразихи для данной комбинации скрещивания показал, что фактически наблюдаемое расщепление соответствовало теоретически ожидаемой модели 3 : 1 при моногенном наследовании признака (табл. 2).
Таблица 2
Наследование признака устойчивости подсолнечника к расе G заразихи в F 2 при скрещивании линий ВК678 и RGP1
|
Ген |
Всего растений, шт. |
Теоретически ожидаемое соотношение |
χ2 |
df |
Р |
|
Or 7 |
107 |
3 : 1 |
0,24 |
1 |
0,7–0,8 |
Ранее было установлено, что у линии подсолнечника RGP1 устойчивость обеспечивается действием одного гена Or 7 с доминантным типом наследования [10]. Наши исследования также подтвердили этот тип наследования у данного генотипа.
Также был проведен гибридологический анализ результатов расщепления в F 2 для трех ДНК-локусов. Локусы ORS 683 и ORS 1112 являются кодоминантными, и во втором поколении расщепление соответствует модели 1 : 2 : 1, а у доминантного локуса ORS 1040 – модели 3 : 1 (табл. 3).
Таблица 3
Наследование SSR-локусов ДНК ORS 683, ORS 1040 и ORS 1112 при скрещивании линий ВК678 и RGP1
|
Локус |
Всего растений, шт. |
Теоретически ожидаемое соотношение |
χ2 |
df |
Р |
|
ORS 683 |
98 |
1:2:1 |
1,16 |
2 |
0,56 |
|
ORS 1112 |
94 |
1:2:1 |
2,37 |
2 |
0,67 |
|
ORS 1040 |
102 |
3:1 |
3,84 |
1 |
0,05 |
Для данных локусов χ2-критерий достоверно подтверждал соответствие фактических расщеплений теоретически ожидаемым. Следовательно, комбинация скрещивания пригодна для дальнейшего анализа. Проведен тест на совместное наследование гена Or 7 c этими локусами, а также SSR-локусов друг с другом (табл. 4).
Таблица 4
Значения χ2 между геном Or 7 и SSR-локусами ДНК в потомстве F 2 (ВК 678 × RGP1)
|
Расщепляющиеся локусы |
Всего растений, шт. |
Теоретически ожидаемое соотношение |
χ2 |
df |
Р |
|
Or 7 –ORS 683 |
98 |
3:6:3:1:2:1 |
4,96 |
5 |
0,08 |
|
Or 7 – ORS 1112 |
89 |
3:6:3:1:2:1 |
7,42 |
5 |
0,19 |
|
Or 7 – ORS 1040 |
102 |
9:3:3:1 |
7,27 |
3 |
0,06 |
|
ORS 683 – ORS 1112 |
94 |
1:2:1:2:4:2:1:2:1 |
19,2 |
8 |
< 0,05 |
|
ORS 1040 – ORS 683 |
97 |
3:6:3:1:2:1 |
8,33 |
5 |
0,15 |
|
ORS 1040 – ORS 1112 |
88 |
3:6:3:1:2:1 |
2,06 |
5 |
0,30 |
В таблице 4 представлены данные по оценке совместного наследования гена Or 7 c ДНК локусами, где отмечено значение χ2, которое характеризует отклонение от независимого расщепления приведенных локусов. Показаны также вероятность нулевой гипотезы о независимом наследовании (P) между анализируемыми генами. Тест на совместное наследование показал независимое наследование гена Or 7 c локусами ДНК ORS 683, ORS 1040, ORS 1112, а также SSR-локусов ORS 1040 с ORS 1112 и ORS 1040 с ORS 683. Независимое наследование пары локусов ORS683 – ORS 1112 не подтвердилось (χ2 = 19,2; Р <0,05) (табл. 4).
Была выполнена оценка частоты рекомбинации между парами локусов Or 7 – ORS 683, ORS 1040 – ORS 1112, ORS 683 – ORS 1112 (табл. 5).
Таблица 5
Значения частот рекомбинации между геном Or 7 и SSR-локусами ДНК в потомстве F 2 (ВК678 х RGP1)
|
Расщепляющиеся локусы |
r |
s p |
|
Or 7 – ORS 683 |
0,39 |
0,06 |
|
ORS 683 – ORS 1112 |
0,27 |
0,41 |
|
ORS 1040 – ORS 1112 |
0,56 |
0,60 |
Тест подтвердил сцепление локусов ORS 683 – ORS 1112 с частотой рекомбинации 0,27 ± 0,41 (27 сМ). Частота рекомбинации локусов Or 7 – ORS 683 составила 0,39 ± 0,06 (39 сМ). Вероятно, сцепление между генами отсутствует или очень слабое. Коэффициент рекомбинации между локусами ORS 1040 – ORS 1112 составил более 50 %, что говорит об отсутствии сцепления между этими локусами. Мы предполагаем следующий порядок расположения генов в данном сегменте хромосомы подсолнечника LG3: Or 7 – ORS 683 – ORS 1112 – ORS 1040 с расстояниями между ними 39, 27 и 56 сМ соответственно. Такой порядок расположения локусов согласуется с их расположением на SSR [4] и физической карте, сконструированной нами. Однако полученные данные частот рекомбинации между исследуемыми генами не совпадают с результатами исследований других авторов. Так, согласно I. Imerovski et al. [4], SSR-локус ORS 683 находится на расстоянии 1,5 сМ выше гена or ab-vl-8 . У этих же авторов дистанция между ORS 683 и ORS 1112 составляет 9,8 сМ. Tang et al. [7] определили дистанцию между ORS 683 и ORS 1112 в 0,9 сМ, а между ORS 683 и ORS 1040 – 7 сМ. Этими авторами было отмечено, что на оценку расстояния между локусами могут оказать влияние фенотипические ошибки, потому что идентификация восприимчивого фенотипа как устойчивого при классификации приводит к увеличению класса рекомбинантных генотипов и искажает дистанции на генетической карте [7]. Кроме того, оценка частоты рекомбинации зависит от генотипов, которые используются для анализа [14].
Вероятно, ген, который контролирует устойчивость к расе G заразихи у линии RGP1, не является идентичным гену orab-vl-8, описанному сербскими учеными. Исходя из литературных данных, мы предполагаем, что он локализуется в этой же хромосоме ниже области, в которой производился поиск. Предложенные гены-кандидаты располагаются также в области ниже маркера ORS 683 [8]. В связи с этим будет продолжена работа по поиску местонахождения гена Or7 и локусов ДНК, сцепленных с ним, в нижних областях LG3.
Выводы. В результате проведенных исследований было исключено расположение гена Or 7 в области непосредственной близости к микросателлитным локусам ORS 683, ORS 1040, ORS 1112. Локусы ORS 683 – ORS 1112 показали сцепление с частотой рекомбинации 0,27 ± 0,41 (27 сМ). На основании изученных литературных источников и репрезентативного генома подсолнечника HanXRQr2.0-SUNRISE составлена частичная физическая карта LG3 для определения области дальнейшего поиска локализации гена Or 7 и ДНК-маркеров, косегрегирующих с ним.
Список литературы Анализ сцепления гена устойчивости к расе G заразихи с микросателлитными локусами у линии-донора подсолнечника селекции ВНИИМК RGP1
- Molinero-Ruiz L., Delavault P., Pérez-Vich B., Pacureanu-Joita M., Bulos M., Altieri E., Domínguez J. History of the race structure of Orobanche cumana and the breeding of sunflower for resistance to this parasitic weed // A review. J. Agric. Res. - 2015. - V. 13.- e10R01 DOI: 10.5424/sjar/2015134-8080.
- Martín-Sanz A., Malek J., Fernández-Martínez J., Pérez-Vich B., Velasco L. Increased virulence in sunflower broomrape (Orobanche cumana Wallr.) populations from Southern Spain is associated with greater genetic diversity // Front. Plant Sci. - 2016. - V. 7. - P. 589. DOI: 10.3389/fpls.2016.00589.
- Fernández-Martínez J., Pérez-Vich B., Ve-lasco L. Sunflower broomrape (Orobanche cu-mana Wallr.) // Sunflower (Urbana: AOCS Press) 2015. - P. 129-155.
- Imerovsk, I., Dimitrijevic A., Miladinovic D., Dediv B., Jocic S., Kocis Tubic N., Cvejic S. Mapping of a new gene for resistance to broom-rape races higher than F // Euphytica. - 2016. -V. 209. - P. 281-289.
- Pérez-Vich B., Akhtouch B., Knapp S., Leon A., Velasco L., Fernández-Martínez J., Berry S.T. Quantitative trait loci for broomrape (Oro-banche cumana Wallr.) resistance in sunflower // Theor. Appl. Genet. - 2004. - V. 109. - P. 92102.
- Akhtouch B., del Moral L., Leon A., Velasco L., Fernández-Martínez J., Pérez-Vich B. Genetic study of recessive broomrape resistance in sunflower // Euphytica. - 2016. - V. 209. - P. 419-428.
- Tang S., Heesacker A., Kishore V., Fernandez A., Sadik E., Cole G., Knapp S. Genetic mapping of the Or5 gene for resistance to Orobanche race E in sunflower // Crop Sci. - 2003. -V. 43. - P. 1021-1028.
- Imerovsk I., Dedic B., Cvejic S., Miladinovic D., Jocic S., Owens G., Tubic N., Rieseberg L. BSA-seq mapping reveals major QTL for broomrape resistance in four sunflower lines // Mol. Breeding. - 2019. - V. 39. - P. 3941. DOI.org/10.1007/s11032-019-0948-9.
- Duriez P., Vautrin S., Auriac M., Bazerque J., Boniface M., Callot C., Carrére S., Cauet S., Chabaud M., Gentou F. A receptor-like kinase enhances sunflower resistance to Orobanche cumana // Nat. Plants. - 2019. - V. 5. - P. 12111215.
- Гучетль С.З., Арасланова Н.М., Антонова Т.С., Челюстникова Т.А., Питинова Ю.В. Генетический анализ устойчивости к расе G Orobanche cumana Wallr. в F2 и ВС1 линий подсолнечника RGP1, RGP2, RGB, RGL1, RGL2 // Масличные культуры. - 2019. -Вып. 4 (180). - С. 23-28. DOI: 10.25230/2412-608Х-2019-4-180-23-28.
- Vroh Bi I., Harvengt L., Chandelier A., Mergeai P. Improved RAPD amplification of recalcitrant plant DNA by the use of activated charcoal during DNA extraction // Plant breeding. - 1996. - Vol. 115. - Is. 3 - P. 205-206.
- Савиченко Д.Л., Гучетль С.З., Голо-ватская А.В. Поиск и анализ нуклеотидных последовательностей в геноме подсолнечника, ассоциированных с генами устойчивости к расам заразихи Orobanche cumana Wallr. // Масличные культуры. - 2020. - Вып. 3 (183). -С. 11-19. DOI: 10.25230/2412-608X-2020-3-183-11-19.
- Гершензон С.М. Основы современной генетики. - Киев: Наукова думка, 1979. - 508 с.
- Карабицина Ю.И., Гаврилова В.А., Ал-патьева Н.В., Кузнецова Е.Б., Анисимова И.Н. Особенности наследования признака восстановления фертильности пыльцы подсолнечника при цитоплазматической мужской стерильности // Генетика. - 2019. - Т. 55. - № 11. - C.1327-1334.


