Бактерии рода Bacillus, выделенные из почв района солеразработок
Автор: Ястребова О.В., Ананьина Л.Н., Плотникова Е.Г.
Журнал: Вестник Пермского университета. Серия: Биология @vestnik-psu-bio
Рубрика: Микробиология
Статья в выпуске: 9, 2008 года.
Бесплатный доступ
Из почв района солеразработок (г. Березники, Пермский край) методом накопительных культур выделено семь штаммов бактерий рода Bacillus. По результатам анализа REP- и BOX-ПЦР большинство выделенных штаммов входят в одну геномогруппу (I), штаммы SN501, И10b и И12b различаются между собой по REP- и BOX-ДНК профилям и выделены в три геномогруппы (II, III и IV, соответственно). Принадлежность штаммов SN501, И10b и И12b к разным геномогруппам подтверждена результатами рестрикционного анализа амплифицированных рибосомальных ДНК (ARDRA). Филогенетический анализ нуклеотидных последовательностей гена 16S рРНК показал, что штаммы И12b, И23 и И27 являются представителями рода Bacillus, штамм И10b принадлежит к роду Paenibacillus. Большинство штаммов растет при 9-11% NaCl в среде культивирования и в щелочных условиях (pH 7.0-9.0). Штамм Bacillus sp. SN501 растет на нафталине как единственном источнике углерода и энергии при концентрации NaCl 5%.
Короткий адрес: https://sciup.org/147204437
IDR: 147204437 | УДК: 579.222:579.851.11
Текст научной статьи Бактерии рода Bacillus, выделенные из почв района солеразработок
Бактерии рода Bacillus представлены грампо-ложительными, аэробными, каталазоположительными, палочковидными клетками, образующими овальные эндоспоры, высокоустойчивые ко многим неблагоприятным воздействиям. Бактерии данного рода широко распространены в почвенных, водных экосистемах и варьируют по физиологическим свойствам. Ряд галофильных и алка-лофильных штаммов рода Bacillus выделен из соленых и содовых озер (Nielsen et al., 1995; Vargas et al., 2005; Lim et al., 2006); термофильные штаммы Bacillus thermophleovorans (Annweiler et al., 2000) и Bacillus sp. JF8 (Shimura et al., 1999) изолированы при 60°C с использованием нафталина в качестве субстрата. Из нефтезагрязненных почв и донных отложений выделен ряд штаммов Bacillus spp., использующих фенантрен (Doddamani, Nin-nekar, 2000) и дизельное топливо в качестве единственного источника углерода (Stapleton et al., 2000). Данная группа бактерий представляет интерес в связи с высоким биотехнологическим потенциалом для получения осмопротектерных соединений, термостабильных и гидролитических ферментов; создания препаратов для очистки почв и стоков со сложным, полихимическим характером загрязнения (Margesin, Schinner, 2001; Zhuang et al., 2002).
Цель работы – характеристика бактерий рода Bacillus, выделенных из засоленных почв.
Методы исследования
Бактериальные культуры. В работе использованы бактериальные штаммы, выделенные из почв и донных отложений, загрязненных отходами соледобывающего предприятия БКРУ-1 г. Березники (Пермский край), обозначенные SN501, И10b, И12b, И23, И26, И27, И28.
Среды и условия культивирования. Бактерии выращивались в минеральной среде Раймонда (Розанова, Назина, 1982) с разным содержанием хлорида натрия (от 3 до 20%). В качестве полноценной среды использовали среду Раймонда при добавлении триптона (5 г/л) и дрожжевого экстракта (2.5 г/л). При культивировании в жидкой минеральной среде нафталин, фенантрен, гентизат и орто -фталат использовали как субстраты в концентрации 1 г/л, салицилат – в концентрации 0.5 г/л. При выращивании бактерий на агаризованной среде нафталин, фенол, жидкие алифатические углеводороды (октан, декан, пентадекан) и дизельное топливо добавляли на крышку перевернутой чашки Петри. Культивирование проводили при температуре 28˚С.
Изучение физиологических свойств бактерий. Устойчивость к NaCl (3, 5, 7, 9, 10, 11, 13, 17, 20%) определяли на полноценной агаризованной минеральной среде Раймонда. Рост учитывали на 7-й день.
Рост при разных значениях рН определяли при концентрации Na+ 0.8–0.85 М в буферных системах (для рН 5 – ацетатный буфер, для рН 7–8 –
фосфатный буфер, для рН 9–10 – трис-HCl буфер) (Методы общей бактериологии, 1983), приготовленных на основе минеральной среды Раймонда, содержащей глюкозу в качестве источника углерода и энергии. Штаммы культивировались на агаризованной среде при рН 5, 7, 8, 9, 10. Рост учитывали на 7-й день.
Идентификация бактерий. Культуральные, морфологические и биохимические свойства изучали согласно руководств «Определитель бактерий Берджи» (1997) и «Методы общей бактериологии» (1983). Морфологию и жизненный цикл бактерий изучали у выращенных на агаризованной среде 12–72-часовых культур с использованием световой микроскопии (фазововый контраст).
Амплификация гена 16S рРНК. Для амплификации использовали бактериальные праймеры 27F и 1492R. Амплификацию проводили так, как описано у Е.Ю. Гавриш с соавт. (2004).
Определение нуклеотидной последовательности гена 16S рРНК. Секвенирование гена 16S рРНК, амплифицированного с использованием универсальных бактериальных праймеров (Гавриш и др., 2004), проводили с применением набора реактивов CEQ Dye Terminator Cycle Sequencing Kit на автоматическом секвенаторе MegaBASE 1000 (JSC GE Healthcare, США).
Филогенетический анализ. При предварительном анализе нуклеотидной последовательности гена 16S рРНК использовали программы BLAST . Далее нуклеотидную последовательность 16S рДНК изучаемого изолята сравнивали с нуклеотидными последовательностями типовых штаммов близкородственных видов с помощью программы CLUSTAL W (Thompson et al., 1994) и корректировали вручную. Эволюционное расстояние, выраженное как число замен на 100 нуклеотидов, рассчитывали согласно Jukes, Cantor (1969). Построение филогенетического древа производили с помощью пакета программ TREECON (van de Peer, DeWachter, 1994) c использованием метода «neighbor-joining» (NEIGHBOR). Статистическую достоверность ветвления («bootstrap-анализ») оценивали с использованием соответствующей функции программы TREECON на основе 1000 альтернативных деревьев.
REP-ПЦР (полимеразная цепная реакция повторяющихся экстрагенетических палиндромных последовательностей ДНК) и BOX-ПЦР (полимеразная цепная реакция повторяющихся Box-элементов) проводили по методу J. Versalovic et al. (1994). Продукты реакции разделяли электрофорезом в 1.5% агарозном геле, приготовленном на 1 х ТВЕ буфере (Маниатис и др., 1984).
Расщепление ДНК эндонуклеазами рестрикции. 5 мкл ПЦР продукта 16S рДНК были обрабо- таны 1 ед. рестрикционных ферментов HhaI, MseI, HaeIII (Fermentas, Литва) с использованием для каждой эндонуклеазы рекомендованного буфера и при соответствующем температурном режиме (Scortichini et al., 2002). Фрагменты рестрикции разделяли электрофорезом в 1.2% агарозном геле.
Результаты и их обсуждение
При использовании метода накопительных культур из почв, загрязненных отходами химических и соледобывающего производств (г. Березники, Пермский край), выделен ряд штаммов грамположительных бактерий, способных к деструкции различных алифатических и ароматических углеводородов (Плотникова и др., 2001; Плотникова и др., 2007).
Семь штаммов были идентифицированы как представители рода Bacillus . Данные штаммы имели круглые, гладкие, непрозрачные колонии бледно-желтого цвета. Клетки штаммов – грампо-ложительные, аэробные палочки, каталазоположительные, оксидазоотрицательные. Образуют округлые, расположенные на концах клеток споры.
Проведена сравнительная характеристика исследуемых штаммов с использованием методов REP-ПЦР и BOX-ПЦР (рис. 1, 2). Анализ полученных профилей фрагментов ДНК выявил, что штаммы И23, И26 и И27 входят в одну геномо-группу (I группа). Штаммы SN501, И10b и И12b не входят в данную геномогруппу, различаются между собой по REP- и BOX-ДНК профилям, они выделены в три геномогруппы (II, III и IV группы соответственно) (рис. 1, 2).
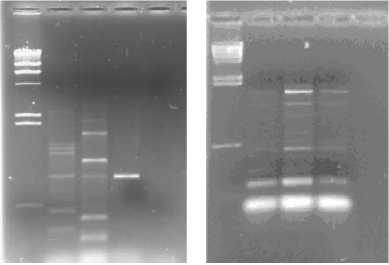
1 2 3 4 5 1 2 3 4 5
I
II
Рис. 1. Электрофореграмма продуктов REP-ПЦР штаммов Bacillus spp.:
I : 1 - маркер λ/ Hind III; 2 – SN501; 3 – И10b;
4 – И12b; 5 – отрицательный контроль;
II : 1 - маркер λ/ Hind III; 2 – И23 ; 3 – И26;
4 – И27; 5 – отрицательный контроль
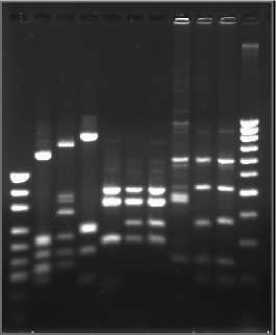
Результаты рестрикционного анализа ампли-фицированных рибосомальных ДНК (ARDRA) с использованием рестриктаз Mbo I, Hha I и Mse I подтвердили принадлежность штаммов SN501, И10b и И12b к разным геномогруппам (рис. 3).
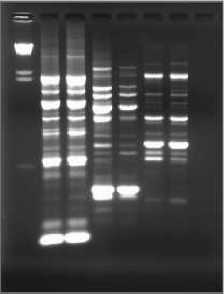
1 2 3 4 5 6 7 8
1 2 3 4 5 6 7 8 9 10 11
Рис. 2. Электрофореграмма продуктов BOX-ПЦР штаммов Bacillus spp.:
1 – маркер λ/ Hind III; 2, 3 – SN501; 4, 5 – И10b; 6, 7 – И12b; 8 – отрицательный контроль
У штаммов И10b, И12b, И23 и И27 определены нуклеотидные последовательности 16S рДНК. Филогенетический анализ нуклеотидных последовательностей гена 16S рРНК показал, что штаммы И12b, И23 и И27 являются представителями рода Bacillus. На филогенетическом древе изоляты по-
Рис. 3. Электрофореграмма гидролиза ПЦР-продуктов амплификации 16S рДНК, обработанных эндонуклеазами рестрикции Mbo I ( 2 – 4 ), Hha I ( 5 – 7 ), Mse I ( 8 - 10 ):
1 - маркер длин фрагментов pUC19 DNA/ Msp I; 2 , 5 , 8 – штамм SN501; 3, 6, 9 – штамм И10b; 4, 7, 10 – штамм И12b; 11 - маркер длин фрагментов 1kb DNA Ladder (Life Technologies, США)
падают в разные 16S рДНК-группы бацилл (Ash et al., 1991): штаммы И23 и И27 группируются с представителями 1-й группы, а штамм И12b образует кластер с видами 6-й группы (рис. 4).
0.02
Bacillus mojavensis IFO15718T (AB021191)
Bacillus subtilis DSM10T (AJ276351)
Bacillus tequilensis 10bT (AY197613)
Bacillus subtilis subsp. spizizenii NRRL B-23049T (AF074970)
58 Bacillus vallismortis DSM11031T (AB021198)
г Bacillus amyloliquefaciens ATCC 23350T (X60605)
100 Bacillus atrophaeus JCM9070T (AB021181)
100 Bacillus sonorensis NRRL B-23154T (EU138473)
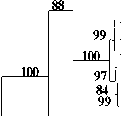
Bacillus licheniformis DSM 13T (X68416)
Bacillus aerophilus 28KT (AJ831844)
Bacillus stratosphericus 41KF2aT (AJ831841)
Bacillus altitudinis 41KF2bT (AJ831842)
Bacillus safensis FO-036bT (AF234854)
Bacillus pumilus DSMZ27T (AY456263)
И23
И27
Bacillus idriensis SMC 4352-2T (AY904033)
0 Bacillus indicus Sd/3T (AJ583158)
Bacillus cibi JG-30T (AY550276)
Bacillus decolorationis LMG 19507T (AJ315075)
Bacillus taeanensis BH030017T (AY603978)
Bacillus algicola KMM 3737T (AY228462)
И12b
Bacillus hwajinpoensis SW-72T (AF541966)
100 г Paenibacillus macquariensis DSM 2T (AB073193)
Paenibacillus antarcticus LMG 22078T (AJ605292)
Paenibacillus xylanilyticus XIL14T (AY427832)
Paenibacillus illinoisensis JCM 9907T (AB073192)
Paenibacillus barcinonensis BP-23T (AJ716019)
Paenibacillus pabuli JCM 9074T (AB073191)
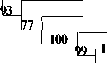
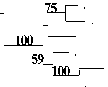
И10b
Paenibacillus amylolyticus NBRC 15957T (AB363743)
Alicyclobacillus acidocaldarius NBRC 15652T (AB271754)
Рис. 4. Филогенетическое древо, построенное с использованием метода «neighbor-joining», основанное на сравнении нуклеотидных последовательностей 16S рДНК видов родов Bacillus и Paenibacillus , близкородственных исследуемым штаммам. Масштаб соответствует 2 нуклеотидным заменам на каждые 100 нуклеотидов. Цифрами показана статистическая достоверность порядка ветвления, определенная с помощью «bootstrap»-анализа 1000 альтернативных деревьев (приведены значения выше 50%)
Максимальный уровень 16S рДНК сходства, составляющий 99.5%, штаммы И23 и И27 имеют с видами Bacillus pumilus и Bacillus safensis, а изолят И12b – с видом Bacillus hwajinpoensis. На данном этапе работы штаммы идентифицированы как Bacillus spp.
Филогенетический анализ нуклеотидной последовательности гена 16S рРНК штамма И10b выявил принадлежность его к роду Paenibacillus , который был предложен для объединения видов, относящихся к 3-й 16S рДНК-группе рода Bacillus (Ash et al., 1994). Наибольший уровень сходства (98%) изолят имеет с Paenibacillus amylolyticus . Штамм идентифицирован как Paenibacillus sp.
Исследована способность выделенных штаммов к росту на различных ароматических и алифатических углеводородах. Установлено, что штаммы И10b и И12b используют дизельное топливо, нафталин и декан в качестве субстрата. Штаммы И23, И26, И27 и И28 растут на дизельном топливе и пентадекане. Штамм SN501 способен к росту на нафталине, фенантрене, салицилате, орто -фталате, а также на феноле и октане.
Установлено, что оптимальными для роста выделенных штаммов являются температуры в диапазоне 24–28°С. Большинство штаммов хорошо растет при щелочных значениях рН (до 9.0) среды культивирования, а штамм И23 – при pH 7.0–10.0. Изоляты способны к росту в полноценной среде в условиях повышенных концентраций хлорида натрия: до 9% – штаммы SN501, И10b, И12b, И23, И27 и до 11% – штаммы И26, И28. Штамм SN501 растет на нафталине при концентрации NaCl в среде культивирования – до 5%.
Работа поддержана грантами РФФИ-Урал №07-04-96078_а и РФФИ-Урал №07-04-97625_офи.
Список литературы Бактерии рода Bacillus, выделенные из почв района солеразработок
- Гавриш, Е.Ю. Три новых вида бревибактерий -Brevibacterium antiguum sp. nov., Brevibacterium aurantiacum sp. nov. и Brevibacterium permense sp. nov./Е.Ю. Гавриш, В.И. Краузова, Н.В. Потехина, С.Г. Карасев, Е.Г. Плотникова, О.В. Алтынцева, Л.А. Коростелева, Л.И. Евтушенко//Микробиология. 2004. Т. 73, ¢ 2. С. 218-225.
- Маниатис, Т. Методы генетической инженерии/Т. Маниатис, Э. Фрич, Дж.Сэмбрук//Молекулярное клонирование. М.: Мир, 1984. 390с.
- Методы общей бактериологии/пер. с англ.; под ред. Ф. Герхардт и др. Т. 1-3. М.: Мир, 1983.
- Определитель бактерий Берджи/пер. с англ.; под ред. Дж. Хоулта и др. Т. 1, 2. М.: Мир, 1997.
- Плотникова, Е.Г. Бактерии-деструкторы полициклических ароматических углеводородов, выделенные из почв и донных отложений района солеразработок/Е.Г. Плотникова, О.В. Алтынцева, И.А. Кошелева, И.Ф. Пунтус, А.Е. Филонов, Е.Ю. Гавриш, В.А. Демаков, А.М. Боронин//Микробиология. 2001. Т. 70, ¢ 1. С. 61-69.
- Плотникова, Е.Г. Разнообразие и функционирование микроорганизмов из техногенных почв района солеразработок Верхнекамья/Е.Г. Плотникова, Л.Н. Ананьина, О.В. Ястребова, Д.О. Рыбкина//Биотехнология: состояние и перспективы развития: материалы конгресса. Ч. 2. М., 2007. С. 135.
- Розанова, Е.П. Углеводородокисляющие бактерии и их активность в нефтяных пластах/Е.П. Розанова, Т.Н. Назина//Микробиология. 1982. Т. 51. С. 324-348.
- Annweiler, E. Naphthalene degradation and incorporation of naphthalene-derived carbon into biomass by the thermophile Bacillus thermoleovorans/E. Annweiler, H.H. Richnow, G. Antranikian, S. Hebenbrok, C. Garms, S. Franke, W. Francke, W. Michaelis//Appl. Environ. Microbiol. 2000. Vol. 66. P. 518-523.
- Ash, C. Phylogenetic heterogeneity of the genus Bacillus revealed by comparative analysis of small subunit -ribosomal RNA sequences/C. Ash, J.A.E. Farrow, S. Wallbanks, M.D. Collins//Lett. Appl. Microbiol. Vol. 13. P. 202-206.
- Ash, C. Molecular identification of rRNA group 3 bacilli (Ash, Farrow, Wallbanks and Collins) using a PCR probe test/C. Ash, F.G. Priest, M.D. Collins//Antonie van Leeuwenhoek. 1993. Vol. 64. P. 253-260.
- Doddamani, H.P. Biodegradation of phenanthrene by a Bacillus species/H.P. Doddamani, H.Z. Ninnekar//Curr. Microbiol. 2000. Vol. 41. P. 11-14.
- Jukes, T.H. Evolution of protein molecules/T.H. Jukes, C.R. Cantor//Mamallian protein metabolism/Ed. H.N. Munro New York: Academic press. 1969. P. 21-132.
- Lim, J.-M. Bacillus salarius sp. nov., a halophilic, spore-forming bacterium, isolated from a salt lake in China/J.-M. Lim, C.O. Jeon, S.-M. Lee, J.-C. Lee, L.-H. Xu, C.-L. Jiang, C.-J. Kim//Int. J. Syst. Evol. Microbiol. 2006. Vol. 56. P. 373-377.
- Margesin, R. Potential of halotolerant and halophilic microorganisms for biotechnology/R. Margesin, F. Schinner//Extremophiles. 2001. Vol. 5. P. 73-83.
- Nielsen, P. Phenetic diversity of alkaliphilic Bacillus strains; proposal for nine new species/P. Nielsen, D. Fritze, F.G. Priest//Microbiology. 1995. Vol. 141. P. 1745-1761.
- Scortichini, М. Bacteria associated with hazelnut (Corylus avellana L.) decline are of two groups: Pseudomonas avellanae and strains resembling P. syringae pv. syringae/М. Scortichini, U. Marchesi, M.-P. Rossi, P.D. Prospero//Appl. Environ. Microbiol. 2002. Vol. 68. P. 476-484.
- Shimura, M. Isolation and characterization of a thermophilic Bacillus sp. JF8 capable of degrading polychlorinated biphenyls and naphthalene/M. Shimura, G. Mukerjee-Dhar, K. Kimbara, H. Nagato, H. Kiohara, T. Hatta//FEMS Microbiol. Lett. 1999. Vol. 178. P. 87-93.
- Stapleton, R.D. Catabolic and genetic diversity of degradative bacteria from fuel-hydrocarbon contaminated aquifers/R.D. Stapleton, N.G. Bright, G.S. Sayler//Microb. Ecol. 2000. Vol. 39. P. 211-221.
- Thompson, J.D. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position specific gap penalties and weight matrix choice/J.D. Thompson, D.G. Higgins, T.J. Gibson//Nucleic Acids Res. 1994. Vol. 22. P. 4673-4680.
- van de Peer, Y. TREECON for Windows a software package for the construction and drawing of evolutionary trees for the Microsoft Windows environment/Y. van de Peer, R. DeWachter//Comput. Appl. Biosci. 1994. Vol. 10. P. 569-570.
- Vargas, A.V. Bacillus bogoriensis sp. nov., a novel alkalophilic, halotolerant bacterium isolated from Kenyan soda lake/A.V. Vargas, O.D. Delgado, R. Hatti-Kaul, B. Mattiasson//Int. J. Syst. Evol. Microbiol. 2005. Vol. 55. P. 899-902.
- Versalovic, J. Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction/J. Versalovic, M. Schneider, F.J. de Bruijn, J.R. Lupski//Meth. Mol. Cell. Biol. 1994. Vol. 5. P. 25-40.
- Zhuang, W.Q. Bacillus naphthalinovorans sp. nov. from oil-contaminated tropical marine sediments and its role in naphthalene biodegradation/W.Q. Zhuang, J.-H. Tay, A.M. Maszenan, S.T.L. Tay//Appl. Microbiol. Biotechnol 2002. Vol.58. P. 547-553.


