Биоразнообразие и метаболические функции микробиома рубца у молочных коров в разные физиологические периоды
Автор: Лаптев Г.Ю., Йылдырым Е.А., Дуняшев Т.П., Ильина Л.А., Тюрина Д.Г., Филиппова В.А., Бражник Е.А., Тарлавин Н.В., Дубровин А.В., Новикова Н.И., Большаков В.Н., Пономарева Е.С.
Журнал: Сельскохозяйственная биология @agrobiology
Рубрика: Генетика и геномика
Статья в выпуске: 4 т.56, 2021 года.
Бесплатный доступ
В условиях интенсификации скотоводства негативное влияние на микробиоту рубца и, как следствие, на физиологию крупного рогатого скота оказывает совокупность стресс-факторов, в частности экстремально высокая молочная продуктивность, несогласованность нейрогуморальной и гормональной регуляции потребления корма и синтеза молока, отрицательный энергетический баланс, использование кормов, содержащих избыточное количество крахмала. В представленной работе впервые показано, что физиологические периоды у молочного скота - это важный фактор, определяющий относительное изобилие неатрибутируемых бактерий из кандидатных семейств vadinBE97 и WCHB1-41, функции которых практически не изучены. Наиболее выраженные изменения метаболического потенциала микробиоты, а именно угнетение различных типов обмена веществ в химусе рубца, в частности энергетического (цикл трикарбоновых кислот), белкового, углеводного, липидного, включая синтез летучих жирных кислот (ЛЖК), наблюдаются у коров в период стабилизации и спада лактации по сравнению с сухостойными, новотельными и раздойными животными. Цель работы - изучение состава и метаболического потенциала микробиома рубца у коров молочного направления в различные физиологические периоды. Эксперимент проводили летом 2020 года в АО «Агрофирма Дмитрова Гора» (Тверская обл.) на 15 коровах (Bos taurus) молочного направления черно-пестрой голштинизированной породы 2-3-й лактации. Животные были разделены на пять групп (по n = 3): I группа - сухостойные (в среднем за 30 сут до отела), II - новотельные (среднее число дней доения - 20), III - в период раздоя (среднее число дней доения - 90), IV - в период стабилизации лактации (208-е сут лактации), V - в период спада лактации (310-е сут лактации). Рационы коров рассчитывали в программе AMTS.Cattle.Professional в соответствии с общепринятыми требованиями. Тотальную ДНК из образцов химуса рубца выделяли с использованием набора Genomic DNA Purification Kit («Fermentas, Inc.», Литва). Оценку бактериального сообщества рубца проводили методом NGS-секвенирования на платформе MiSeq («Illumina, Inc.», США) с применением праймеров для V3-V4 региона 16S рРНК. Секвенирование осуществляли с использованием реагентов для подготовки библиотек Nextera® XT IndexKit («Illumina, Inc.», США), для очистки ПЦР продуктов Agencourt AMPure XP («Beckman Coulter, Inc.», США) и для проведения секвенирования MiSeq® ReagentKit v2 (500 cycle) («Illumina, Inc.», США). Биоинформатический анализ данных выполняли с помощью программного обеспечения Qiime2 ver. 2020.8. Фильтрацию шумовых последовательностей проводили методом Deblur. Для построения филогении de novo применяли программный пакет MAFFT. Для анализа таксономии использовали справочную базу данных Silva 138 (https://www.arb-silva.de/documentation/release-138/). Реконструкцию и прогнозирование функционального содержания метагенома осуществляли при помощи программного комплекса PICRUSt2 (v.2.3.0). Для анализа метаболических путей и ферментов пользовались базой данных MetaCyc. Тотальную РНК из образцов рубцового содержимого выделяли с помощью набора Aurum Total RNA («Bio-Rad», США). На матрице РНК получали кДНК (набор iScript RT Supermix, «Bio-Rad», США). Относительную экспрессию генов Ldh-L и Ldb 0813 бактерий определяли в количественной ПЦР. Реакцию амплификации с праймерами гена Ldb 0813 , связанного с синтезом D-лактатдегидрогеназы, и L-лактатдегидрогеназы (ген Ldh-L ) проводили при помощи набора SsoAdvanced Universal SYBR Green Supermix («Bio-Rad», США). В результате 16S метагеномного секвенирования содержимого рубца показано снижение α-разнообразия бактериальной микробиоты у коров из IV и V групп (р ≤ 0,05). Обнаружены 12 суперфилумов и филумов микроорганизмов. Суперфилум Bacteroidota и филум Firmicutes были определены как «доминантные» бактерии рубца (соответственно до 59,94±1,86 и 46,82±14,40 % сообщества). Бактерии суперфилума Actinobacteriota полностью элиминировались из рубца лактирующих коров, присутствуя только в рубце сухостойных животных. Бактерии суперфилума Armatimonadota исчезали из рубца новотельных коров и животных в период стабилизации лактации, филума Chloroflexi - в периоды раздоя и стабилизации лактации. Выявлены существенные различия между животными по 8 бактериальным семействам, в том числе Muribaculaceae , Prevotellaceae , Erysipelatoclostridiaceae , Oscil-lospiraceae , Ruminococcaceae , Saccharimonadaceae , кандидантым семействам WCHB1-41, vadinBE97. Обнаружено появление в составе микробиома коров в новотельный период и персистирование в последующие периоды лактации родов Asteroleplasma , Sharpea , Moryella , Oribacterium , Shuttleworthia . Эти бактерии отсутствовали в составе микробиома рубца в период сухостоя. Предсказанный функциональный потенциал 17 метаболических путей микробиома различался (р ≤ 0,01) у коров из разных опытных групп.
Микробиом рубца, жвачные, молочные коровы, рационы, крахмал, клетчатка, ngs-секвенирование, picrust2, metacyс, метаболические пути
Короткий адрес: https://sciup.org/142231398
IDR: 142231398 | УДК: 636.2:591.1:579.62:575:577.2 | DOI: 10.15389/agrobiology.2021.4.619rus
Текст научной статьи Биоразнообразие и метаболические функции микробиома рубца у молочных коров в разные физиологические периоды
В условиях интенсификации скотоводства негативное влияние на микробиоту рубца и, как следствие, на физиологию крупного рогатого скота оказывает совокупность стресс-факторов, в частности экстремально высокая молочная продуктивность, несогласованность нейрогуморальной и гормональной регуляции потребления корма и синтеза молока, отрицательный энергетический баланс, использование кормов, содержащих избыточное количество крахмала. В представленной работе впервые показано, что физиологические периоды у молочного скота — это важный фактор, определяющий относительное изобилие неатрибутируемых бактерий из кандидатных семейств vadinBE97 и WCHB1-41, функции которых практически не изучены. Наиболее выраженные изменения метаболического потенциала микробиоты, а именно угнетение различных типов обмена веществ в химусе рубца, в частности энергетического (цикл трикарбоновых кислот), белкового, углеводного, липидного, включая синтез летучих жирных кислот (ЛЖК), наблюдаются у коров в период стабилизации и спада лактации по сравнению с сухостойными, новотельными и раздойными животными. Цель работы — изучение состава и метаболического потенциала микробиома рубца у коров молочного направления в различные физиологические периоды. Эксперимент проводили летом 2020 года в АО «Агрофирма Дмитрова Гора» (Тверская обл.) на 15 коровах (Bos taurus) молочного направления черно-пестрой голштинизированной породы 2-3-й лактации. Животные были разделены на пять групп (по n = 3): I группа — сухостойные (в среднем за 30 сут до отела), II — новотельные (среднее число дней доения — 20), III — в период раздоя (среднее число дней доения — 90), IV — в период стабилизации лактации (208-е сут лактации), V — в период спада лактации (310-е сут лактации). Рационы коров рассчитывали в программе в соответствии с общепринятыми требованиями. Тотальную ДНК из образцов химуса рубца выделяли с использованием набора Genomic DNA Purification Kit («Fermentas, Inc.», Литва). Оценку бактериального сообщества рубца проводили методом NGS-секвенирования на платформе MiSeq («Illumina, Inc.», США) с применением праймеров для V3-V4 региона 16S рРНК. Секвенирование осуществляли с использованием реагентов для подготовки библиотек Nextera® XT IndexKit («Illumina, Inc.», США), для очистки ПЦР продуктов Agencourt AMPure XP («Beckman Coulter, Inc.», США) и для проведения секвенирования MiSeq® ReagentKit v2 (500 cycle) («Illumina, Inc.», США). Биоинфор-матический анализ данных выполняли с помощью программного обеспечения Qiime2 ver. 2020.8. Фильтрацию шумовых последовательностей проводили методом Deblur. Для построения филогении de novo применяли программный пакет MAFFT. Для анализа таксономии использовали справочную базу данных Silva 138 . Реконструкцию и прогнозирование функционального содержания метагенома осуществляли при помощи программного комплекса PICRUSt2 (v.2.3.0). Для анализа метаболических путей и ферментов пользовались базой данных MetaCyc. Тотальную РНК из образцов рубцового содержимого выделяли с помощью набора Aurum Total RNA («Bio-Rad», США). На матрице РНК получали кДНК (набор iScript RT Supermix, «Bio-Rad», США). Относительную экспрессию генов Ldh-L и Ldb 0813 бактерий определяли в количественной ПЦР. Реакцию амплификации с праймерами гена Ldb 0813, связанного с синтезом D-лактатдегидрогеназы, и L-лактатдегидрогеназы (ген Ldh-L) проводили при помощи набора SsoAdvanced Universal SYBR Green Supermix («Bio-Rad», США). В результате 16S мета-геномного секвенирования содержимого рубца показано снижение α-разнообразия бактериальной микробиоты у коров из IV и V групп (р ≤ 0,05). Обнаружены 12 суперфилумов и филумов микроорганизмов. Суперфилум Bacteroidota и филум Firmicutes были определены как «доминантные» бактерии рубца (соответственно до 59,94±1,86 и 46,82±14,40 % сообщества). Бактерии суперфи-лума Actinobacteriota полностью элиминировались из рубца лактирующих коров, присутствуя только в рубце сухостойных животных. Бактерии суперфилума Armatimonadota исчезали из рубца новотельных коров и животных в период стабилизации лактации, филума Chloroflexi — в периоды раздоя и стабилизации лактации. Выявлены существенные различия между животными по 8 бактериальным семействам, в том числе Muribaculaceae, Prevotellaceae, Erysipelatoclostridiaceae, Oscil-lospiraceae, Ruminococcaceae, Saccharimonadaceae, кандидантым семействам WCHB1-41, vadinBE97.
Основная проблема современной стратегии молочного животноводства состоит в необходимости обеспечить максимальное использование питательных веществ рационов при минимизации риска дисбиоза экосистемы рубца, расстройства пищеварения и возникновения метаболических нарушений. Известно, что потребности молочных коров в энергии удовлетворяются на 70 % за счет микробной ферментации. Это указывает на важнейшую роль микробиоты рубца в метаболизме животных и необходимости изучения состава и функций микроорганизмов (1).
Уникальная особенность метаболизма жвачных — их способность переваривать растительную клетчатку, содержащую целлюлозу, гемицеллюлозу и ксиланы, благодаря сложившемуся в процессе эволюции симбиозу с микробиотой рубца (2-4). Представители микробиома пищеварительной системы, различающиеся по филогенезу, неразрывно связаны между собой, их симбиотические отношения и метаболические сети играют центральную роль в функционировании рубца, особенно в переваривании клетчатки (5). Так, в исследованиях D. Akin с соавт. (6) и P.H. Janssen с соавт. (7) продемонстрирована междоменная взаимозависимость на примере бактерий и архей. Бактерии, разлагающие лигноцеллюлозный материал, выделяют водород (6), который, в свою очередь, используют метаногенные археи (7). Аналогичным образом большая часть лактата, произведенного одними микроорганизмами, далее метаболизируется бактериями, утилизирующими это вещество (8). В результате образуются летучие жирные кислоты (ЛЖК), которые всасываются через стратифицированный плоский эпителий рубца. С одной стороны, ЛЖК обеспечивают прямое пополнение энергетических субстратов (прежде всего, для глюконеогенеза) (9), внося важный вклад в формирование метаболического пула животного (10). С другой стороны, преобразование лактата в ЛЖК способствует буферизации содержимого рубца, что служит важным механизмом противодействия развитию ацидоза (11).
В условиях интенсивного скотоводства негативное влияние на микробиоту рубца и, как следствие, физиологию крупного рогатого скота оказывает совокупность стресс-факторов, в частности экстремально высокая молочная продуктивность, несогласованность нейрогуморальной и гормональной регуляции потребления корма и синтеза молока, отрицательный энергетический баланс, использование кормов с избыточным количеством крахмала. Известно, что кормление сухостойных коров должно быть направлено на снижение риска возникновения послеотельных осложнений, что предопределяет использование кормов высокого качества, содержащих повышенное количество клетчатки и умеренную долю концентратов. Это стимулирует жевательную активность, моторику рубца, повышает рН, восстанавливает микробиоту рубца и, следовательно, общую метаболическую активность животных (12-14). Ряд исследователей (4, 15, 16) отмечают высокое разнообразие рубцовой микробиоты, состоящей в основном из целлюлолитических бактерий, у коров в сухостойный период.
Как считает G. Henderson с соавт. (17), в отличие от моногастричных животных, у жвачных роль состава рациона в формировании микробного сообщества рубца значительно выше, чем генотипа и индивидуальных физиологических характеристик хозяина. Новотельный период — критический в отношении становления физиологического статуса коров и молочной продуктивности (18). В результате стремительного нарастания потребностей в глюкозе у новотельных коров выход энергии для производства молока становится больше, чем потребление энергии из корма, что приводит к отрицательному энергетическому балансу (14). Как результат низкого содержания глюкозы и инсулина в плазме крови включается физиологический механизм преодоления дефицита энергии: организм активно мобилизуют триглицериды из жировой ткани в попытке удовлетворить высокие энергетические потребности (19). Все это индуцирует дисбаланс в соотношении глюкогенных и липогенных соединений плазмы крови, что имеет тяжелые последствия для метаболизма (20).
Известно, что на физиологию новотельных животных отрицательно влияют корма, содержащие крахмал и моносахара, которые служат основными энергетическими компонентами, используемыми в рационе кормления лактирующих коров (21, 22). В то же время крахмал и моносахара — это основной источник гликогенных предшественников (например, пропионата) и ферментируемой энергии для микроорганизмов рубца. Тем не менее избыточное образование короткоцепочечных жирных кислот снижает рН рубца (23), поскольку на фоне возрастания продукции ЛЖК увеличивается численность кислотоустойчивых бактерий филума Bacteroidetes и снижается численность Firmicutes (24). Осуществляемый бактериями филума Bacteroidetes активный синтез лактата как промежуточного продукта ферментации крахмала и неспособность микробиоты поддерживать соответствующий кислотно-щелочной баланс рубца приводят к внутрипросветному накоплению протонов и последующему падению рН ниже физиологического диапазона (14, 25, 26). Это состояние широко известно как ацидоз рубца. На фоне ацидоза в разы увеличивается риск ожирения печени и кетоза, метрита, смещения сычуга. Подобные нарушения имеют далеко идущие последствия для здоровья и продуктивности крупного рогатого скота, приводя к хромоте (27, 28), ухудшению репродуктивных функций, снижению молочной продуктивности и сокращению продуктивного долголетия (29, 30). По мнению Q. Zebeli с соавт. (14), частота и тяжесть метаболических расстройств зависят от количества крахмала в рационе и потребления корма. Показано, что постепенное увеличение концентратов на 0,25 кг сухого вещества (СВ) в сутки в послеотельный период обеспечивает лучшую адаптацию микробиоты рубца, чем увеличение суточной нормы концентрата на 1 кг сухого вещества СВ в сутки (18). Совокупность таких факторов, как резкий переход к высокоэнергетическому рациону, стресс от отела и лактации, отрицательный энергетический баланс связаны с высоким риском развития метаболических нарушений у коров в новотельный период (31, 32).
Коровы в периоды раздоя, стабилизации и спада лактации также подвержены высокому риску нарушений микробиома рубца и возникновения метаболических расстройств (14). По наблюдению нескольких исследователей (26, 33, 34), высокие концентрации растворимого крахмала в рубце коров в период раздоя были связаны с увеличением численности Lactobacillus sp. и амилолитического микроорганизма Streptococcus bovis, про- дуцирующих лактат (34). Это индуцировало снижение рН и угнетение целлюлозолитических и ЛЖК-продуцирующих бактерий, а также нарушения в процессах синтеза ЛЖК.
Изучение микробиома рубца — один из фундаментальных подходов к разработке эффективных мер профилактики метаболических нарушений на молочных фермах. Представляет интерес комплексный анализ изменений состава и метаболического потенциала микробиома рубца в различные физиологические периоды у молочных коров. Влияние физиологических периодов и рационов на таксономический состав микробиома рубца достаточно подробно изучено отечественными учеными с применением классических методов микробиологии (35-37) и зарубежными исследователями (13, 14, 23). Однако конкретные биологические и метаболические функции микробиома рубца для разных физиологических периодов молочных коров полностью не раскрыты. Кроме того, микробиомы животных, выращенных в соответствии с практикой молочного скотоводства Российской Федерации, мало исследовались с использованием молекулярнобиологических методов.
В представленной работе мы впервые показали, что период физиологического состояния молочного скота — это важный фактор, определяющий относительное изобилие неатрибутируемых бактерий из кандидатных семейств vadinBE97 и WCHB1-41, функции которых практически не изучены. Наиболее выраженные изменения метаболического потенциала микробиоты, а именно угнетение различных типов обмена веществ в химусе рубца, в частности энергетического (цикл трикарбоновых кислот), белкового, углеводного, липидного, включая синтез ЛЖК, наблюдаются у коров в период стабилизации и спада лактации по сравнению с сухостойными, новотельными и раздойными животными.
Цель работы — изучение состава и метаболического потенциала микробиома рубца у коров молочного направления в различные физиологические периоды.
Методика. Работу проводили летом 2020 года в АО «Агрофирма Дмитрова Гора» (Тверская обл.) на 15 коровах ( Bos taurus ) молочного направления черно-пестрой голштинизированной породы 2-3-й лактации. Выбор коммерческого животноводческого хозяйства в качестве опытной площадки был основан на экспериментах, описанных в ранее опубликованных исследованиях микробиома дойных коров (38, 39). Животные находились в одинаковых условиях на привязном содержании.
Коров разделили на пять групп (по n = 3): I группа — сухостойные (в среднем за 30 сут до отела), II — новотельные (среднее число дней доения — 20), III — в период раздоя (среднее число дней доения — 90), IV — в период стабилизации лактации (208-е сут лактации), V — в период спада лактации (310-е сут лактации). Сухостойных коров отбирали по ожидаемой дате отела. Число животных в группах выбирали в соответствии с методиками, представленными в ранее опубликованных исследованиях (40, 41). Средняя живая масса животных в I группе — 703 кг. Средний суточный надой коров из II группы составлял 27,0 л/гол., содержание жира в молоке — 3,7 %, масса животных — 637 кг. Средний суточный надой коров в период раздоя — 38,5 л/гол., содержание жира в молоке — 3,7 %, масса животных — 650 кг. Живая масса коров из IV и V групп — соответственно 667 и 681 кг.
Пробы химуса (30-50 г от каждой коровы) отбирали из верхней части вентрального мешка рубца вручную стерильным зондом с максимально возможным соблюдением условий асептики.
Тотальную ДНК из образцов выделяли с использованием набора Genomic DNA Purification Kit («Fermentas, Inc.», Литва) согласно прилагаемой инструкции (3). Анализ был основан на селективном осаждении ДНК из субстрата с применением растворов для лизиса клеточных стенок, осаждения ДНК, 1,2 М хлорида натрия, хлороформа.
Бактериальное сообщество рубца оценивали методом NGS-секвенирования на платформе MiSeq («Illumina, Inc.», США) с праймерами для V3-V4 региона 16S рРНК. Прямой праймер: 5´-TCGTCGGCAGCGTCAG-ATGTGTATAAGAGACAGCCTACGGGNGGCWGCAG - 3´, обратный праймер: 5´- GTCTCGTGGGCTCGGAGATGTGTATAAGAGACAGGACTACH-VGGGTATCTAATCC-3´. Секвенирование проводили с использованием реагентов для подготовки библиотек Nextera® XT IndexKit («Illumina, Inc.», США), для очистки ПЦР-продуктов Agencourt AMPure XP («Beckman Coulter, Inc.», США) и для секвенирования MiSeq® ReagentKit v2 (500 cycle) («Illumina, Inc.», США). Максимальная длина полученных последовательностей составила 2½250 п.н.
Биоинформатический анализ данных выполняли с помощью программного обеспечения Qiime2 ver. 2020.8 . После первоначального импорта последовательностей в формат Qiime2 парные строки прочтений выравнивали. Далее последовательности фильтровали по качеству с использованием настроек по умолчанию. Шумовые последовательности фильтровали методом Deblur, при этом использовали максимальную длину последовательности обрезки, равную 250 п.н. . Для построения филогении de novo применяли программный пакет MAFFT, далее проводили маскированное выравнивание последовательностей. Для анализа таксономии использовали справочную базу данных Silva 138 .
На основании полученной таблицы оперативно-таксономических единиц (ОТЕ) с помощью плагинов программного пакета Qiime2 рассчитывали индексы α -разнообразия и строили графики зависимости числа ОТЕ от числа прочтений. При статистическом анализе индексов разнообразия их дополнительного преобразование не проводили.
Реконструкцию и прогнозирование функционального содержания метагенома, семейств генов, ферментов выполняли при помощи программного комплекса PICRUSt2 (v.2.3.0) . С программой работали согласно рекомендованному сценарию, все настройки использовали по умолчанию. Для анализа метаболических путей и ферментов пользовались базой данных MetaCyc . Прогнозируемые профили метаболических путей MetaCyc оценивали по обилию ASV (Amplicon Sequence Variants) (42).
Тотальную РНК из образцов рубцового содержимого выделяли с помощью набора Aurum Total RNA («Bio-Rad», США) согласно инструкции производителя. На матрице РНК получали кДНК (набор iScript RT Supermix, «Bio-Rad», США) (43). Относительную экспрессию генов Ldh-L и Ldb 0813 бактерий, способных осуществлять молочнокислое брожение, анализировали при помощи количественной ПЦР. Реакцию амплификации с праймерами гена Ldb 0813 , связанного с синтезом D-лактатдегидрогеназы (F: 5´-CTGGGATCCGTTGAGGGAGATGCTTAAG - 3´, R: 5´-TCCGAAGCTTTT-AGTTGACCCGGTTGAC - 3´) и L-лактатдегидрогеназы (ген Ldh-L ) (F: 5´-CA-TCAAAAAGTTGTGTTAGTCGGCG - 3´, R: 5´-TCAGCTAAACCGTCGTT-AAGCACTT - 3´) проводили на детектирующем амплификаторе ДТ Lite-4
(ООО «НПО ДНК-Технология», Россия). Условия амплификации: 1 мин при 95 ° С (1 цикл); 15 с при 95 ° С, 1 мин при 50 ° С (45 циклов). Использовали реакционную смесь для амплификации, которая входила в состав набора SsoAdvanced Universal SYBR Green Supermix («Bio-Rad», США), приготовленную согласно протоколу производителя.
Относительную экспрессию рассчитывали методом 2 -AA CT (44). Универсальный ген, кодирующего 16S-рибосомальную субъединицу прокариот (соответствующая пара праймеров: F — 5'-AGGCCTTCGGGTTGTAAAGT - 3', R — 5'-CGGGGATTTCACATCTCACT - 3'), служил референсным.
Индексы биоразнообразия микробиома рубца Сhao1, Шеннона (Н), Симпсона (D) вычисляли согласно описанию (45).
Математическую и статистическую обработку результатов осуществляли методом однофакторного дисперсионного анализа (ANOVA) в программах Microsoft Excel XP/2003, R-Studio (Version 1.1.453) . Для коррекции ошибки 1-го типа рассчитывали критерий достоверно значимой разности Тьюки (Tukey’s HSD test) . Результаты представлены как средние значения (M) и стандартные ошибки средних (±SEM). Уровень статистической значимости — р < 0,05. Для оценки достоверности использовали t-критерий Стьюдента.
Результаты. Рационы коров в каждой группе рассчитывали с помощью программы в соответствии с общепринятыми требованиями (46-48) (табл.). Рецепты премиксов для каждой группы рассчитывали индивидуально. Состав премиксов полностью обеспечивал потребности в витаминно-минеральном питании под заданную продуктивность.
Показатели питательности рационов для черно-пестрых голштинизированных коров ( Bos taurus ) молочного направления в разные физиологические периоды
|
Показатель |
Группа |
||||
|
I |
II |
III |
IV |
V |
|
|
Содержание сухого вещества (СВ), % |
39,02 |
45,10 |
42,31 |
43,50 |
42,50 |
|
Потребление СВ, кг |
11,82 |
18,94 |
25,00 |
25,60 |
27,60 |
|
Обеспеченность обменной энергией из рациона |
|||||
|
к норме, % |
106,86 |
103,82 |
104,87 |
105,52 |
100,20 |
|
Обеспеченность обменным протеином из рациона |
|||||
|
к норме, % |
103,47 |
97,08 |
104,57 |
105,33 |
105,43 |
|
Сырой протеин (СП), % |
14,86 |
16,44 |
17,48 |
17,50 |
17,60 |
|
Чистая энергия лактации, МДж/кг |
6,29 |
6,47 |
6,86 |
6,60 |
6,30 |
|
Нейтрально-детергентная клетчатка, % СВ |
41,63 |
25,75 |
28,28 |
29,28 |
30,20 |
|
Кислотно-детергентная клетчатка, % СВ |
26,64 |
16,25 |
17,75 |
18,50 |
18,80 |
|
Расщепляемый в рубце протеин, % от СП |
56,0 |
57,0 |
52,0 |
55,0 |
56,0 |
|
Не расщепляемый в рубце протеин, % от СП |
44,0 |
43,0 |
48,0 |
45,0 |
44,0 |
|
Крахмал, % СВ |
16,20 |
23,54 |
26,64 |
26,45 |
24,50 |
|
Са, % СВ |
0,49 |
0,82 |
0,74 |
0,71 |
0,68 |
|
Р, % СВ |
0,51 |
0,39 |
0,40 |
0,40 |
0,41 |
|
Mg, % СВ |
0,47 |
0,39 |
0,37 |
0,39 |
0,42 |
|
Примечание. Описание групп см. в разделе «Методика». |
|||||
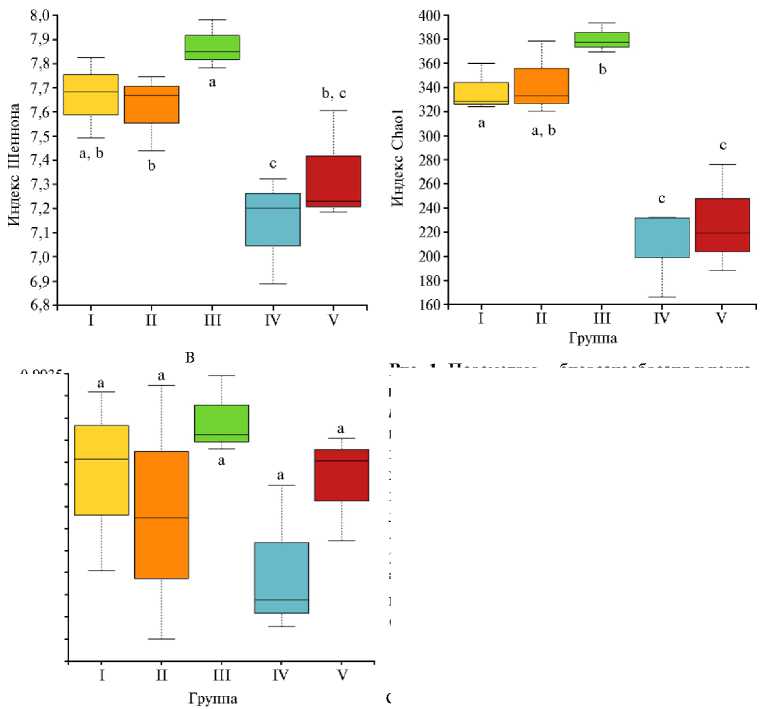
Мы изучили состав и структуру бактериального сообщества рубца коров методом NGS-секвенирования. При вычислении индексов Шеннона и Chao1 оказалось, что их значения у животных из II группы (новотельные) и I группы (сухостойные) не различались (р > 0,05) (рис. 1). Это интересный результат, поскольку обе метрики оценивают степень видового разнообразия (49). Ожидаемым было более выраженное в том или ином направлении изменение разнообразия в новотельный период по сравнению с сухостойным в связи с действием ряда негативных факторов на организм животного в совокупности с резкими изменениями в структуре рациона, стрессами от отела и начала лактации. Так, A. Bach с соавт. (50) наблюдали снижение значений индекса Н у микробного сообщества пищеварительной системы коров после отела относительно сухостойного периода. В нашем исследовании индекс Н немного увеличивался (р ≤ 0,05) в период раздоя по сравнению с новотельным. Резкое снижение (р ≤ 0,05) значений индексов Н и Chao1 при сопоставлении с предыдущими физиологическими периодами отмечали в период стабилизации (IV группа) и спада лактации (V группа). В целом известно, что уменьшение разнообразия микробиоты пищеварительной системы происходит при дисбиотических нарушениях, в частности при лечении антимикробными препаратами (51), и гастроэнтеритах (52). Тем не менее разнообразие микробных сообществ рубца — это достаточно стабильный показатель, поскольку в нашем исследовании у коров в новотельный период и период раздоя не отмечалось снижения значений индексов Шеннона и Chao1. При расчете индекса Симпсона для каждой исследованной группы оказалось, что полученные значения не имели достоверных различий (р ≥ 0,05) (см. рис. 1, В).
А
Б
A
|
=
=
Чтобы выяснить, были ли связаны вариации в α -биоразнооб-разии с составом и функциями мик-
0,9935"
0,9930
0,9925
0,9920
0,9915
0,9910
0,9905
0,9900
0,9895
0,9890
0,9885
0,9880
0,9875
0,9870-
Рис. 1. Параметры α -биоразнообразия у чернопестрых голштинизированных коров ( Bos taurus ) молочного направления в разные физиологические периоды: А — индекс Шеннона, Б — индекс Chao1, В — индекс Симпсона; I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации (АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год).
a-c При отсутствии в обозначении одинаковых букв различия статистически значимы (р ≤ 0,05).
роорганизмов, мы оценили изменения в таксономической структуре микробиоты рубца на основе данных NGS-секвенирования.
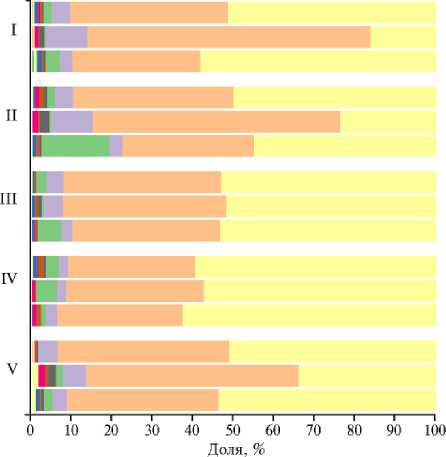
В составе микробиома рубца коров были обнаружены 12 суперфи-лумов и филумов микроорганизмов (рис. 2), из которых суперфилум Bacteroidota и филум Firmicutes можно считать доминантными бактериями рубца (соответственно до 59,94±1,86 и 46,82±14,40 % микробиоты). Две эти таксономические группы допустимо рассматривать как ядро бактериального микробиома, поскольку они присутствуют в значительном количестве практически у всех Bos taurus (12, 53, 54). Доминирующие бактерии, обнаруженные в нашем исследовании, вероятно, ответственны за большую часть трансформации веществ в рубце, преимущественно целлюлозы, гемицеллюлозы, крахмала, органических кислот и белка, так как эти вещества служат компонентами рациона и промежуточными энергетическими субстратами (1).
Bacteroidota Firmicutes Patescibacteria Proteobacteria
-
■ Planctomycetota
-
■ Spirochaetota
-
■ Cyanobacteria
-
■ Fibrobacterota
Verrucomicrobiota
Armatimonadota Chloro/lext Actinobacteriota
Рис. 2. Таксономический состав микробиома рубца у черно-пестрых голштинизированных коров ( Bos taurus ) молочного направления в разные физиологические периоды: I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации (анализ методом NGS-секвенирования; АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год).
Оказалось, что снижение α -разнообразия рубцового микробиома имело связь со уменьшением относительной численности некоторых таксонов, что было зафиксировано уже на уровне филумов. Например, бактерии суперфилума Actinobacteriota полностью элиминировались из рубца лакти-рующих коров, присутствуя только в рубце у сухостойных животных; бактерии суперфилума Armatimonadota исчезали из рубца новотельных коров и коров в период стабилизации лактации, филума Chloroflexi — в период раздоя и стабилизации лактации. Исчезновение этих микроорганизмов могло внести отрицательный вклад в процессы метаболизма в период лактации коров. Так, бактерии суперфилума Actinobacteriota — это широко распространенные симбионты эукариот (55), их целлюлолитические ферменты способствуют более эффективному расщеплению клетчатки (56).
Обилие бактерий суперфилума Verrucomicrobiota уменьшалось в рубце коров в период раздоя и стабилизации лактации по сравнению с сухостойными животными (р ≤ 0,05). Несмотря на повсеместное распространение указанных микроорганизмов, в чистой культуре были получены только немногочисленные изоляты (57), поэтому их экологическая значимость до сих пор точно не установлена. Известно, что Verrucomicrobia в значительном количестве присутствуют в кишечнике термитов, где участвуют в превращении растительных полисахаридов в ацетат (57). Недавно открыта способность некоторых представителей суперфилума Verrucomic-robia окислять метан (свойство, описанное ранее только для протеобакте- рий) в кислых условиях среды (рН 0,8-2,0) (58). Поэтому снижение содержания бактерий суперфилума Verrucomicrobiota в нашем эксперименте в периоды раздоя и стабилизации лактации может быть связано с угнетением процессов переваривания клетчатки, а также метаногенеза при высококонцен- тратном кормлении.
Особый интерес представляет снижение количества микроорганизмов суперфилума Patescibacteria в период стабилизации лактации по сравнению с сухостойным (р ≤ 0,05). Дело в том, что представители суперфи-лума Patescibacteria имеют значительно редуцированные по сравнению с большинством других микроорганизмов геномы и для получения энергии используют простые промежуточные метаболиты пищеварительной системы хозяина, например глюкозу и пировиноградную кислоту, а функции переваривания сложных полисахаридов у них утрачены (59). Возможно, снижение доли бактерий суперфилума Patescibacteria в период стабилизации лактации связано с нарушением синтеза этих питательных субстратов в рубце коров.
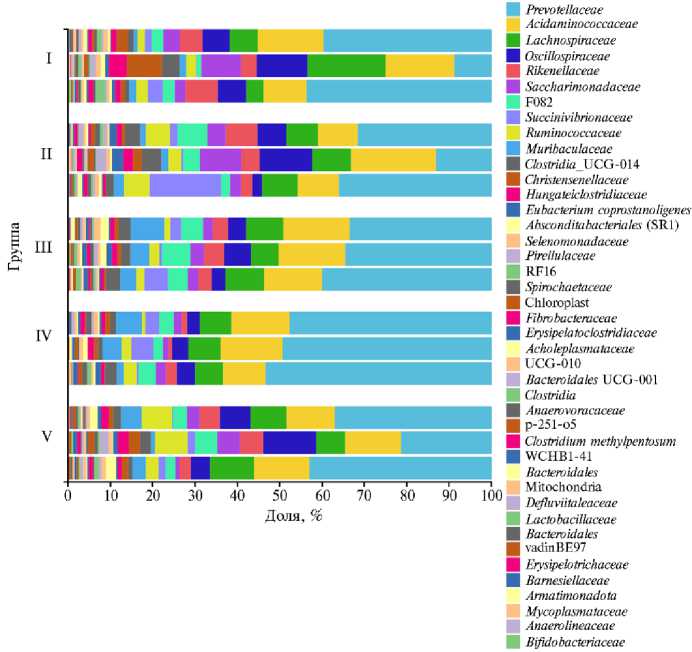
Рис. 3. Таксономический состав микробиома рубца на уровне семейств у черно-пестрых голшти-низированных коров ( Bos taurus ) молочного направления в разные физиологические периоды: I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации (анализ методом NGS-секвенирования; АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год).
Детальный анализ состава микробиома (рис. 3) показал, что некоторые выявленные в составе микробиоты семейства были относительно равномерно распределены между животными из разных экспериментальных групп. В то же время анализ позволил выявить существенные различия по 8 бактериальным семействам: Muribaculaceae , Prevotellaceae , Erysipelatoclost-ridiaceae , Oscillospiraceae , Ruminococcaceae , Saccharimonadaceae , кандидантым семействам WCHB1-41, vadinBE97 (см. рис. 3).
Мы наблюдали увеличение численности бактерий семейства Prevo-tellaceae суперфилума Bacteroidota , представленных преимущественно родом Prevotella , у коров из IV группы по сравнению с I группой (р ≤ 0,05). Присутствие в рубце бактерий семейства Prevotellaceae на фоне рационов, богатых крахмалом и монасахарами, закономерно. Известно (60, 61), что эти микроорганизмы используют крахмал для синтеза ЛЖК, однако избыточная продукция короткоцепочечных жирных кислот связана со снижением рН рубца и может привести к лактатному ацидозу, сопровождающемуся дисбактериозом (62). Также у коров из IV группы по сравнению с I группой наблюдалось снижение численности бактерий семейств Oscillospiraceae фи-лума Firmicutes и Saccharimonadaceae суперфилума Patescibacteria (р ≤ 0,05). Это могло свидетельствовать о дисбиотических нарушениях микробиома при высококонцентратном кормлении, поскольку представители семейства Oscillospiraceae обладают значительным набором гликозидгидролаз. Последние выполняя разнообразные функции, в частности связанные с деградацией целлюлозы и гемицеллюлозы кормов, образуют основу биологического аппарата разрушения гликозидных связей (63).
А
Б
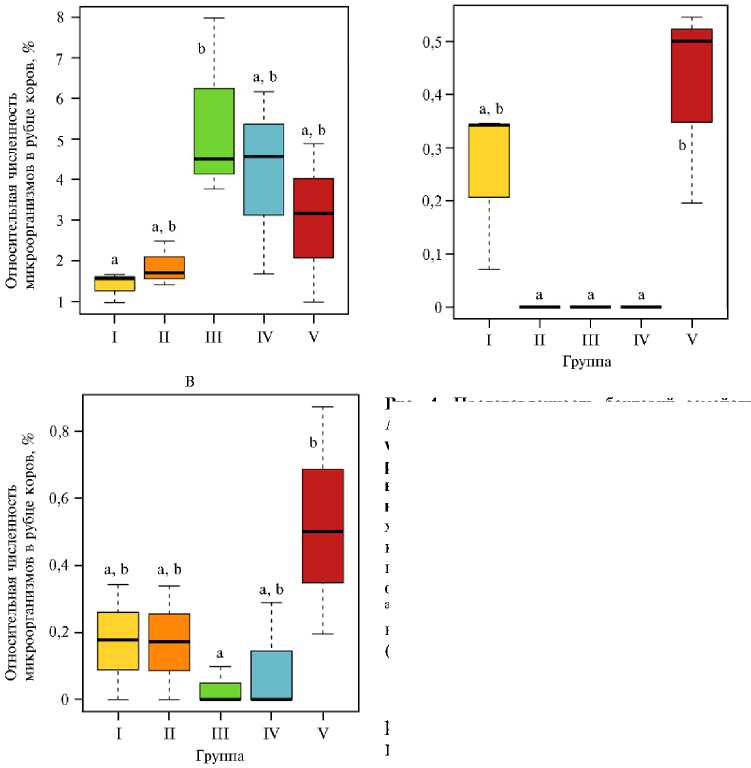
Рис. 4. Представленность бактерий семейства Muribaculaceae (А) и кандидатных семейств vadinBE97 (Б) и WCHB1-41 (В) в составе микробиома рубца у черно-пестрых голштинизиро-ванных коров ( Bos taurus ) молочного направления в разные физиологические периоды: I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации (АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год). a-b При отсутствии в обозначении одинаковых букв различия статистически значимы (р ≤ 0,05).
Бактериальное разнообразие рубца жвачных огромно, и в настоящее время описаны далеко не все его представители. Очень важно сосре доточить внимание на ранее не культивируемых формах микроорганизмов, чтобы иметь возможность пра- вильно анализировать такие важные процессы, как циклы превращений питательных веществ и модуляции функций организма, ассоциированных со здоровьем хозяина. В этой связи интересен факт выявления бактерий семейства Muribaculaceae суперфилума Bacteroidota в микробиоме рубца коров (рис. 4). До 2019 года (64) таксон классифицировался как некультивируемые бактерии семейства S24-7, несмотря на широкую представленность в микробиоте кишечника многих позвоночных животных. Поскольку таксономическая принадлежность была неоднозначной, сложно было оценить их взаимодействие с хозяином.
Численность бактерий семейства Muribaculaceae в нашем опыте увеличивалось (р ≤ 0,05) у коров в период лактации по сравнению с сухостойными животными (см. рис. 4). Ранее K.L. Ormerod с соавт. (65) продемонстрировали наличие значительного и универсального набора ферментов, связанных с расщеплением углеводов, в анализируемых геномах видов S24-7. Приспособленность к деградации углеводов, возможно, объясняет присутствие в рубце коров этих микроорганизмов в условиях высококонцентратного кормления. Наличие у бактерий семейства Muribaculaceae защитных механизмов в отношении действия некоторых органических кислот (64) может также объяснять особенности их колонизационного поведения.
Вытеснение из состава микробиоты рубца неатрибутируемых бактерий из кандидатного семейства vadinBE97 у лактирующих коров из II, III, IV групп и снижение доли бактерий кандидатного семейства WCHB1-41 у коров из III, IV групп (см. рис. 4) указывало на то, что кормление — важный фактор, определяющий относительное изобилие этих микроорганизмов в рубце. Такой вывод представляет фундаментальный интерес, поскольку в настоящее время функции представителей этих семейств, принадлежащих к суперфилуму Verrucomicrobiota , практически не изучены из-за отсутствия возможности их лабораторного культивирования.
Обращает на себя внимание появление в составе рубцовой микробиоты в новотельный период и персистирование в последующие периоды лактации микроорганизмов родов Asteroleplasma , Sharpea (сем. Erysipelatoclos-tridiaceae ) и Moryella , Oribacterium , Shuttleworthia (сем. Lachnospiraceae ), отсутствующих в микробиоме в период сухостоя. Эти роды можно обозначить как маркерные таксоны, которые появлялись в ответ на стресс-факторы, связанные с отелом, отрицательным энергетическим балансом, изменениями в составе рациона.
Интересно, что Asteroleplasma anaerobium (единственный представитель рода Asteroleplasma в составе микробиома исследованных коров) обладает ферментами лактатдегидрогеназами, которые активируются фруктозо-1,6-бисфосфатом (66). Лактатдегидрогеназы — ключевые ферменты молочнокислого брожения, приводящего к образованию лактата (67, 68). Лактат в качестве одного из основных конечных продуктов метаболизма также образуют представители родов Moryella (69), Oribacterium (70) и Shuttleworthia (71). Появление бактерий этих таксонов в составе микробиома в период лактации можно объяснить метаболическими нарушениями, нередко наблюдаемыми при высококонцентратном кормлении (34), поскольку лактат индуцирует снижение рН и гибель целлюлозолитических и ЛЖК-синтезиру-ющих микроорганизмов (34).
Отдельно остановимся на виде Sharpea azabuensis — единственном представителе рода Sharpea, зафиксированном в составе микробиома коров в новотельный период, а также периоды раздоя и стабилизации лактации. Эта бактерия способна синтезировать в рубце промежуточные транс-11-изомеры линолевой и линоленовой кислот (72). В содержащих концен- траты рационах жвачных обнаруживаются в основном С18 ненасыщенные жирные кислоты (α-линоленовая, линолевая, олеиновая и др.) (73). Известно, что при увеличении содержания крахмала и снижении количества клетчатки в рационе в рубце может наблюдаться сдвиг метаболизма в сторону пути формирования транс-10 изомеров жирных кислот через транс-11 промежуточные продукты (73). Присутствие в рубце транс-10 промежуточных продуктов часто ассоциируется со снижением содержания молочного жира (74).
Тем не менее в составе микробиома коров мы не наблюдали типичных изменений (34, 75), свидетельствующих о возможных метаболических расстройствах. В частности, не происходило увеличения численности бактерий семейства Lactobacillaceae , которые в большинстве случаев (34) индуцируют снижение рН и провоцируют метаболические нарушения на фоне высококонцентратных рационов. Высоковирулентный патоген жвачных Fusobacterium necrophorum, способный продуцировать гемагглютинин, эндотоксин и лейкотоксин (76) и нередко получающий конкурентное преимущество при высококонцентратном кормлении (76), полностью отсутствовал в микробиоме.
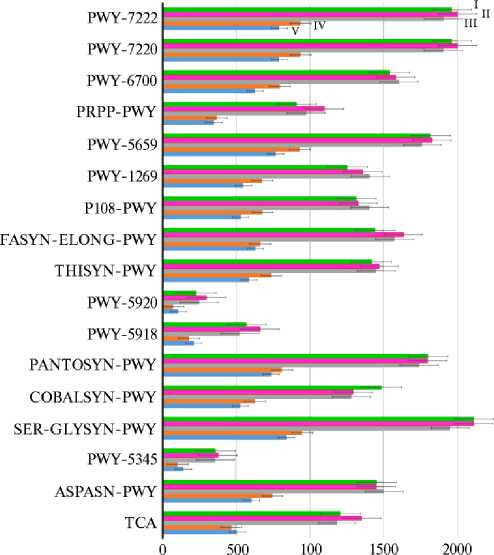
Нуклеотиды, нуклеозиды
Углеводный
Липидный
Витамины, кофакторы, коферменты
Белковый
| Энергетический 2500
Нормализованные условные значения log, для каждого пути
Рис. 5. Данные (р ≤ 0,01) функциональной аннотации метаболических путей метагеномного сообщества рубца у черно-пестрых голштинизированных коров ( Bos taurus ) молочного направления в разные физиологические периоды: I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации (АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год). ТСА — цикл Кребса, ASPASN-PWY — биосинтез L-аспартата и L-аспарагина, PWY-5345 — биосинтез L-метионина (через сульфгидрилирова-ние), SER-GLYSYN-PWY — биосинтез L-серина и глицина, COBALSYN-PWY — биосинтез аденозилкобаламина из кобинамида I, PANTOSYN-PWY — биосинтез пантотената и кофермента А I, PWY-5918 — биосинтез гема из глутамата, PWY-5920 — биосинтез гема из глицина, THISYN-PWY — биосинтез тиаминдифосфата I, FASYN-ELONG-PWY — удлинение цепи насыщенных жирных кислот, P108-PWY — ферментация пирувата до пропаноата I, PWY-
1269 — биосинтез CMP-3-дезокси-D-маннооктулозоната I, PWY-5659 — биосинтез GDP-маннозы, PRPP-PWY — биосинтез гистидина, пурина и пиримидина, PWY-6700 — биосинтез квевозина, PWY-7220 — аденозин-дезоксирибонуклеотидный биосинтез, PWY-7222 – гуано-зин-дезоксирибонуклеотидный синтез.
Чтобы установить, связаны ли сдвиги в составе рубцового микробиома, сопутствующие физиологическим периодам, с изменениями в его функциональности, мы провели реконструкцию и прогнозирование функционального содержания метагеномного сообщества рубца с использованием программного комплекса PICRUSt2 и базы данных Kyoto Encyclopedia of Genes and Genomes (KEGG: Kyoto Encyclopedia of Genes and Genomes) . В результате были аннотированы 282 различных метаболических пути. По 17 путям предсказанный функциональный потенциал изменялся (р ≤ 0,01) у коров разных групп (рис. 5). Наиболее выраженные (р ≤ 0,01) изменения метаболического потенциала микробиоты, а именно угнетение различных типов обмена веществ в химусе рубца, в частности энергетического (цикл трикарбоновых кислот), белкового, углеводного, липидного, включая, синтез ЛЖК, наблюдали у коров из IV и V групп по сравнению с I, II и III группами (см. рис. 5).
Снижение (р ≤ 0,01) связанных с осуществлением цикла Кребса прогнозируемых метаболических возможностей микробиома у коров из IV и V группы по сравнению с I, II и III группами могло привести к нарушению энергетического обмена веществ в рубце, поскольку цикл Кребса играет центральную регуляторную роль у макро- и микроорганизмов. Он включает в себя сложную, многоступенчатую последовательность реакций, поставляющих энергетические и пластические субстраты, восстановленные и фосфорилированные кофакторы для основных биосинтетических путей (77), которые во многом определяют последующую интенсивность и направленность главных метаболических потоков (78).
Следовательно, представляется закономерным снижение потенциала микробиома в отношении синтеза пантотената и кофермента А (КоА) у коров из IV и V групп по сравнению с I, II и III группами (р ≤ 0,01). Ацетилированная форма кофермента А (ацетил-КоА) — одно из ключевых звеньев регуляторного механизма активности пируват- и альфа-кетоглутаратдегидрогеназ в цикле Кребса (79). Предшественник биосинтеза КоА — 3-метил-2-оксобутаноат, который служит промежуточным звеном в синтезе L-валина. На первом этапе это соединение преобразуется в (R)-пантоат, затем в (R)-4´-фосфопантотенат. Далее взаимодействие L-цистеина и (R)-4´-фосфопантотената приводит к образованию (R)-4´-фосфопантотеноил-L-цистеина с его последующим декарбоксилированием до 4´-фосфопанте-теина. Конечная реакция катализируется ферментом дефосфо-КоА-киназой (ЕС 2.7.1.24), которая преобразует 4´-фосфопантетеин в КоА (80, 81).
Снижение потенциала баланса энергии в рубце у коров из IV и V групп по сравнению с I, II и III группами (р ≤ 0,01) могло иметь связь с нарушением синтеза микробиальных аминокислот (L-аспартата, L-аспарагина, L-метионина, L-серина и глицина) (см. рис. 5). Действительно, энергия и азот служат основными детерминантами количества микробиального белка, синтезируемого в рубце (82). Микробиальный белок представляет собой основной источник аминокислот для жвачных животных (83). Возможна связь описанных изменений потенциала с кормлением. Ранее (84) было продемонстрировано снижение скорости синтеза микробиального белка у коров на фоне высококонцентратного рациона.
На состоянии здоровья коров из IV и V групп могло негативно сказаться снижение числа генов, вовлеченных в преобразование пирувата до пропаноата, поскольку пропаноат служит важнейшим предшественником в глюконеогенезе у жвачных и образуются исключительно в результате деятельности рубцовой микробиоты (9). На первом этапе сукцинил-КоА превращается в (R)-метилмалонил-КоА, который расщепляется, перенося единицу С1 на пируват. Эта реакция, катализируемая ферментом метилмало-нил-КоА-карбоксилтрансферазой, приводит к образованию пропаноил-КоА и оксалоацетата. Образующийся пропаноил-КоА действует как донор кофермента А для другой стадии цикла, на которой он переносится на сукцинат. В результате последующей реакции, катализируемой сукцинатной КоА-трансферазой, образуется конечный продукт — пропаноат (85). Снижение рН в рубце на фоне высококонцентратного кормления подавляет размножение чувствительных к подкислению среды продуцентов ЛЖК, в частности пропаноата (86). Вследствие этого скорость образования пропаноата у коров из IV и V групп могла быть замедлена, что угнетало глюконеогенез.
Кроме того, в рубце коров из IV и V групп был снижен (р ≤ 0,01) метаболический потенциал синтеза аденозинкобаламина из кобинамида. Аденозинкобаламин — это витамин В 12, который продуцируют представители рубцового микробиома (87). Многие бактерии не обладают полным набором генов, необходимым для биосинтеза аденозилкобаламина de novo, однако способны преобразовывать кобинамид в кобаламин (88). Это происходит посредством присоединения верхнего лиганда 5´-дезоксиаденозина к аденозилкобинамиду с последующим фосфорилированием до получения аденозилкобинамид-фосфата — промежуточного звена биосинтетического пути de novo.
Описанные модификации функционального потенциала у коров из IV и V групп могли быть связаны с метаболическими нарушениями (89). Ранее J. Lima с соавт. (90) показали, что изменение числа генов рубцовой микробиоты, связанных с биосинтезом витамина В 12, было обусловлено количеством потребленного корма. I. Ogunade с соавт. (91), изучая функциональную аннотацию микробиома рубца голштинских бычков с помощью киотской энциклопедии генов и геномов, установили, что потенциал 10 метаболических путей, включая метаболизм углеводов, аминокислот, энергии, витаминов, кофакторов, а также формирования бактериальных биопленок, был изменен у скота с симптомами подострого ацидоза. В нашем исследовании изменения метаболического потенциала корреспондировались со снижением α -биоразнообразия бактерий в рубце, а также с исчезновением одних таксонов микроорганизмов (суперфилума Armatimonadota , филума Chloroflexi ) и снижением численности других (суперфилума Verrucomicro-biota , семейства Oscillospiraceae филума Firmicutes , семейства Saccharimona-daceae суперфилума Patescibacteria и др.) в рубце коров IV и V групп.
Таким образом, в основном наиболее выраженными оказались следующие различия: по составу рубцового микробиома — при раздое и стабилизации лактации по сравнению с сухостоем, по метаболическому потенциалу — при стабилизации и спаде лактации по сравнению с сухостоем. Эти данные свидетельствует о зависимости биоразнообразия и функций микроорганизмов от состояния и кормления коров в разные физиологические периоды. Кроме того, полученные результаты указывают на то, что рубцовый микробиом и его функциональный потенциал в периоды раздоя, стабилизации и спада лактации подвержены более сильным негативным изменениям, нежели в новотельный период, что предполагает переоценку об- щепринятых представлений (13, 14).
Как уже отмечалось, образование молочной кислоты микробиотой рубца служит ключевым механизмом провокации метаболических нарушений в организме жвачных (92). Мы сопоставили данные таксономических исследований и оценки метаболического потенциала рубцовых микроорганизмов с анализом экспрессии бактериальных генов, связанных с синтезом лактатдегидрогеназ. Для анализа выбрали количественную ПЦР с обратной транскрипцией как метод оценки экспрессии генов бактерий, обладающий общепризнанной воспроизводимостью, высокой чувствительностью и специфичностью (93). Лактат образуется в результате молочнокислого брожения из предшественников под действием двух различных форм НАДсвязанных лактатдегидрогеназ: одна из них (ЕС 1.1.1.27) производит изомер L(+)-лактат и кодируется преимущественно геном Ldh-L (51), другая (ЕС 1.1.1.28) — изомер D( - )-лактат и кодируется геном Ldb 0813 (94). По мнению исследователей (95), наблюдается ощутимая разница в воздействии двух энантиомеров на жвачных, хотя стереоизомеры обладают сходными физическими и химическими свойствами. Важное различие между изомерами — их почечная экскреционная способность, более низкая для D-лактата, что определяет его первостепенную роль в провокации метаболического ацидоза (95). Поэтому гены Ldh-L и Ldb 0813 следует рассматривать, на наш взгляд, как важные кандидатные биомаркеры, способные дать представление об активности синтеза молочной кислоты в рубце коров.
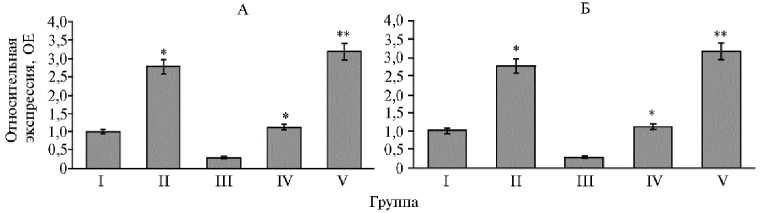
Рис. 6. Относительная экспрессия генов Ldh-L (А) и Ldb 0813 (Б) в микробном сообществе рубца у черно-пестрых голштинизированных коров ( Bos taurus ) молочного направления в разные физиологические периоды: I — сухостойные, II — новотельные, III — раздойные коровы, IV — животные в период стабилизации, V — в период спада лактации; ОЕ — относительные единицы экспрессии по сравнению с экспрессией гена у коров из I группы, принятой за единицу (АО «Агрофирма Дмитрова Гора», Тверская обл., 2020 год).
*, ** Различия при сравнении II, III, IV и V групп с I группой статистически значимы соответственно при p ≤ 0,01 и p ≤ 0,001.
В период раздоя (III группа) экспрессия гена Ldh-L снижалась в 11,3 раза, Ldb 0813 — в 9,9 раза по сравнению с таковой у новотельных коров (II группа) (p ≤ 0,001). То есть у животных на раздое организм лучше противодействовал негативному эффекту от введения в рацион большого количества крахмала, чем у новотельных коров. Вероятно, это связано с формированием адаптивных механизмов у микробного сообщества рубца.
Значительное повышение экспрессии генов Ldh-L (в 10,6 раза при p ≤ 0,001) и Ldb 0813 (в 2,8 раза при p ≤ 0,05) происходило на спаде лактации (V группа по сравнению с IV). Возможно, это было связано с увеличением численности представителей родов Asteroleplasma , Sharpea , Moryella , Oribac-terium , Shuttleworthia , образующих лактат в качестве одного из основных продуктов брожения.
Подобные вариации в экспрессии генов корреспондировались со снижением α-разнообразия микробного сообщества рубца и угнетением ме- таболического потенциала микробиоты, связанного с углеводным, белковым, энергетическим, липидным обменом. Основываясь на полученных нами данных, можно сделать вывод, что животные в период спада лактации также составляют группу риска возникновения метаболических расстройств, что важно учитывать при разработке стратегии снижения количества метаболических заболеваний у дойных коров. Вероятно, ряд изменений в составе и функциях микроорганизмов в рубце коров взаимосвязаны с особенностями обмена веществ, кормления, стресса (например, вследствие отела и начала лактации), иммунного и гормонального статуса животных, обусловленных спецификой различных физиологических периодов.
Итак, у молочных коров с использованием молекулярно-биологических и биоинформатических методов подробно изучена структура микробиома рубца и предсказан ее функциональный потенциал в разные физиологические периоды. Проведенное 16S метагеномное секвенирование показано снижение α -разнообразия рубцовой бактериальной микробиоты в периоды стабилизации и спада лактации. Обнаружены 12 суперфилумов и фи-лумов микроорганизмов, из которых суперфилум Bacteroidota и филум Firmicutes определены как доминантные бактерии рубца (соответственно до 59,94±1,86 и 46,82±14,40 % сообщества). Установлена связь между присутствием определенных таксонов рубцовых микроорганизмов и физиологическими периодами у молочных коров. Бактерии суперфилума Actinobacteriota были обнаружены только в рубце сухостойных животных и полностью элиминировались из рубца лактирующих. Бактерии суперфилума Armatimonadota отсутствовали в рубце новотельных коров и коров в период стабилизации лактации, филума Chloroflexi — в период раздоя и стабилизации лактации. Детальный анализ состава микробиома выявил существенные различия между животными по восьми бактериальным семействам ( Muribaculaceae , Prevotellaceae , Erysipelatoclostridiaceae , Oscillospiraceae , Ruminococcaceae , Sac-charimonadaceae , кандидантым семействам WCHB1-41, vadinBE97). В составе микробиома в новотельный период появлялись и в последующие периоды лактации персистировали роды Asteroleplasma , Sharpea , Moryella , Ori-bacterium , Shuttleworthia , отсутствующие в период сухостоя. Появление этих бактерий в микробиоме во время лактации может быть связано с метаболическим дисбалансом, поскольку лактат — один из их основных продуктов. Предсказанный функциональный потенциал рубцовой микробиоты по 17 метаболическим путям различался. Наиболее выраженные (р ≤ 0,01) изменения, а именно угнетение разных типов обмена веществ в химусе рубца, наблюдались у коров в периоды стабилизации и спада лактации. Выявлено увеличение экспрессии генов Ldh-L (p < 0,01) и Ldb 0813 (p < 0,05), связанных с синтезом лактатдегидрогеназ, у новотельных животных по сравнению с сухостойными. В бактериальном сообществе рубца отмечено значительное повышение экспрессии генов Ldh-L (в 10,6 раза при p < 0,001) и Ldb 0813 (в 2,8 раза при p ≤ 0,05) в период спада лактации по сравнению с периодом стабилизации.
Список литературы Биоразнообразие и метаболические функции микробиома рубца у молочных коров в разные физиологические периоды
- Bergman E. Energy contributions of volatile fatty acids from the gastrointestinal tract in various species. Physiological Reviews, 1990, 70(2): 567-590 (doi: 10.1152/physrev.1990.70.2.567).
- Koike S., Kobayashi Y. Fibrolytic rumen bacteria: their ecology and functions. Asian-Australasian Journal of Animal Sciences, 2009, 22(1): 131-138 (doi: 10.5713/ajas.2009.r.01).
- Ильина Л.А. Изучение микрофлоры рубца крупного рогатого скота на основе молекулярно-биологического метода T-RFLP с целью разработки способов ее оптимизации. Канд. диc. Дубровицы, 2012.
- McCann J.C., Wickersham T.A., Loor J.J. Rumen microbiome and its relationship with nutrition and metabolism. Bioinformatics and Biology Insights, 2014, 8(8): 109-125 (doi: 10.4137/BBI.S15389).
- Creevey C.J., Kelly W.J., Henderson G., Leahy S.C. Determining the culturability of the rumen bacterial microbiome. Microbial Biotechnology, 2014, 7(5): 467-479 (doi: 10.1111/1751-7915.12141).
- Akin D., Borneman W., Windham W. Rumen fungi: morphological types from Georgia cattle and the attack on forage cell walls. Biosystems, 1988, 21(3-4): 385-391 (doi: 10.1016/0303-2647(88)90037-8).
- Janssen P.H., Kirs M. Structure of the archaeal community of the rumen. Applied and Environmental Microbiology, 2008, 74(12): 3619-3625 (doi: 10.1128/AEM.02812-07).
- Qumar M., Khiaosa-ard R., Pourazad F., Wetzels S., Klevenhusen F., Kandler W., Aschenbach J., Zebeli Q. Evidence of in vivo absorption of lactate and modulation of short chain fatty acid absorption from the reticulorumen of non-lactating cattle fed high concentrate diets. PLoS ONE 11(10): e0164192 (doi: 10.1371/journal.pone.0164192).
- Reynolds C.K., Huntington G.B., Tyrrell H.F., Reynolds P.J. Net metabolism of volatile fatty acids, d-β-hydroxybutyrate, nonesterified fatty acids, and blood gases by portal-drained viscera and liver of lactating Holstein cows. Journal of Dairy Science, 1988, 71(9): 2395-2405 (doi: 10.3168/jds.S0022-0302(88)79824-0).
- Aschenbach J.R., Penner G.B., Stumpff F., Gäbel G. Ruminant nutrition symposium: role of fermentation acid absorption in the regulation of ruminal pH. Journal of Animal Science, 2011, 89(4): 1092-1107 (doi: 10.2527/jas.2010-3301).
- Penner G.B., Aschenbach J.R., Gäbel G., Rackwitz R., Oba M. Epithelial capacity for apical uptake of short chain fatty acids is a key determinant for intraruminal pH and the susceptibility to sub-acute ruminal acidosis in sheep. Journal of Nutrition, 2009, 139(9): 1714-1720 (doi: 10.3945/jn.109.108506).
- Beauchemin K.A., Yang W.Z. Effects of physically effective fiber on intake, chewing activity, and ruminal acidosis for dairy cows fed diets based on corn silage. Journal of Dairy Science, 2005, 88(6): 2117-2129 (doi: 10.3168/jds.S0022-0302(05)72888-5).
- Jouany J.-P. Optimizing rumen functions in the close-up transition period and early lactation to drive dry matter intake and energy balance in cows. Animal Reproduction Science, 2006, 96(3-4): 250-264 (doi: 10.1016/j.anireprosci.2006.08.005).
- Zebeli Q., Aschenbach J.R., Tafaj M., Boghun J., Ametaj B.N., Drochner W. Invited review: Role of physically effective fiber and estimation of dietary fiber adequacy in high-producing dairy cattle. Journal of Dairy Science, 2012, 95(3): 1041-1056 (doi: 10.3168/jds.2011-4421).
- Steele M.A., Schiestel C., AlZahal O., Dionissopoulos L., Laarman A.H., Matthews J.C., McBride B.W. The periparturient period is associated with structural and transcriptomic adaptions of rumen papillae in dairy cattle. Journal of Dairy Science, 2015, 98(4): 2583-2595 (doi: 10.3168/jds.2014-8640).
- Dieho K., Dijkstra J., Klop G., Schonewille J.T., Bannink A. Changes in rumen microbiota composition and in situ degradation kinetics during the dry period and early lactation as affected by rate of increase of concentrate allowance. Journal of Dairy Science, 2017, 100(4): 2695-2710 (doi: 10.3168/jds.2016-11982 ).
- Henderson G., Cox F., Ganesh S. Jonker A., Young W., Janssen P.H. Rumen microbial community composition varies with diet and host, but a core microbiome is found across a wide geographical range. Scientific Reports, 2015, 5(1): 14567 (doi: 10.1038/srep14567).
- Kumar S., Indugu N., Vecchiarelli B., Pitta D.W. Associative patterns among anaerobic fungi, methanogenic archaea, and bacterial communities in response to changes in diet and age in the rumen of dairy cows. Frontiers in Microbiology, 2015, 6(6): 781 (doi: 10.3389/fmicb.2015.00781).
- Rukkwamsuk T., Kruip T.A. Meijer G.A., Wensing T. Hepatic fatty acid composition in periparturient dairy cows with fatty liver induced by intake of a high energy diet in the dry period. Journal of Dairy Science, 1999, 82(2): 280-287 (doi: 10.3168/jds.S0022-0302(99)75234-3).
- Schulz K., Frahm J., Meyer U., Kersten S., Reiche D., Rehage J., Dänicke S. Effects of prepartal body condition score and peripartal energy supply of dairy cows on postpartal lipolysis, energy balance and ketogenesis: An animal model to investigate subclinical ketosis. Journal of Dairy Research, 2014, 81(3): 257-266 (doi: 10.1017/S0022029914000107).
- Солун А.С. Петухова Е.А., Емелина Н.Т. Эффективность балансирования сахаро-протеинового отношения в летних рационах коров. Кормление сельскохозяйственных животных, 1971, 9: 201-209.
- Топорова Л.В. Теория и практика кормления высокопродуктивных молочных коров. Ветеринария сельскохозяйственных животных, 2005, 7: 67-74.
- Koenig M., Beauchemin K.A., Rode L.M. Effect of grain processing and silage on microbial protein synthesis and nutrient digestibility in beef cattle fed barley-based diets. Journal of Animal Science, 2003, 81(4): 1057-1067 (doi: 10.2527/2003.8141057x).
- Pitta D., Kumar S., Vecchiarelli B., Shirley D., Bittinger K., Baker L., Ferguson J.D., Thomsen N. Temporal dynamics in the ruminal microbiome of dairy cows during the transition period. Journal of Animal Science, 2014, 92(9): 4014-4022 (doi: 10.2527/jas.2014-7621).
- Krause K., Oetzel G. Understanding and preventing subacute ruminal acidosis in dairy herds: a review. Animal Feed Science and Technology, 2006, 126(3-4): 215-236 (doi: 10.1016/j.anifeedsci.2005.08.004).
- Plaizier J.C., Khafi E., LiS., Gozho G.N., Krause D.O. Subacute ruminal acidosis (SARA), endotoxins and health consequences. Animal Feed Science and Technology, 2012, 172(1-2): 9-21 (doi: 10.1016/j.anifeedsci.2011.12.004).
- Ospina P.A., Nydam D.V., Stokol T., Overton T.R. Evaluation of non-esterified fatty acids and b-hydroxybutyrate in transition dairy cattle in the north eastern United States: critical thresholds for prediction of clinical diseases. Journal of Dairy Science, 2010, 93(2): 546-554 (doi: 10.3168/jds.2009-2277).
- McArt J.A., Nydam D.V., Oetzel R. A field trial on the effect of propylene glycol on displaced abomasum, removal from herd and reproduction in fresh cows diagnosed with subclinical ketosis. Journal of Dairy Science, 2012, 95(5): 2505-2512 (doi: 10.3168/jds.2011-4908).
- Duffield T.F., Lissemore K.D., McBride B.W., Leslie K.E. Impact of hyperketonemia in early lactation dairy cows on health and production. Journal of Dairy Science, 2009, 92(2): 571-580 (doi: 10.3168/jds.2008-1507).
- Ospina P.A., Nydam, D.V., Stokol T., Overton T.R. Associations of elevated non-esterified fatty acids and b-hydroxybutyrate concentrations with early lactation reproductive performance and milk production in transition dairy cattle in the north eastern United States. Journal of Dairy Science, 2010, 93(2): 1596-1603 (doi: 10.3168/jds.2009-2852).
- Bell A. Regulation of organic nutrient metabolism during transition from late pregnancy to early lactation. Journal of Animal Science, 1995, 73(9): 2804-2819 (doi: 10.2527/1995.7392804x).
- Roche J.R., Bell A.W., Overton T.R., Loor J.L. Nutritional management of the transition cow in the 21st century — a paradigm shift in thinking. Animal Production Science, 2013, 53(9): 1000-1023 (doi: 10.1071/AN12293).
- Slyter L.L. Influence of acidosis on rumen function. Journal of Animal Science, 1976, 43(4): 910-929 (doi: 10.2527/jas1976.434910x).
- Russell J.B., Hino T. Regulation of lactate production in Streptococcus bovis: a spiraling effect that contributes to rumen acidosis. Journal of Dairy Science, 1985, 68(7): 1712-1721 (doi: 10.3168/jds.S0022-0302(85)81017-1).
- Шишов В.П. Липолитические и глицеринферментирующие бактерии рубца овец, содержащихся на разных рационах. Канд. дис. Боровск, 1969.
- Пивняк И.Г., Тараканов Б.В. Микробиология пищеварения жвачных. М., 1982.
- Тараканов Б.В. Нормальная микрофлора преджелудков жвачных. Сельскохозяйственные животные: физиологические и биохимические параметры организма. Боровск, 2002: 259-334.
- Lima F.S., Oikonomou G., Lima S.F. Bicalho M.L.S., Ganda E.K., de Oliveira Filho J.C., Lorenzo G., Trojacanec P., Bicalho R.C. Prepartum and postpartum rumen fluid microbiomes: characterization and correlation with production traits in dairy cows. Applied and Environmental Microbiology, 2015, 81(4): 1327-1337 (doi: 10.1128/AEM.03138-14).
- Zhong Y., Xue M., Liu J. Composition of rumen bacterial community in dairy cows with different levels of somatic cell counts. Frontiers in Microbiology, 2018, 9: 3217 (doi: 10.3389/fmicb.2018.03217).
- Sundset M.A., Edwards J.E., Cheng Y.F., Senosiain R.S., Fraile M.N., Northwood K.S., Praesteng K.E., Glad T., Mathiesen S.D., Wright A.D. Molecular diversity of the rumen microbiome of Norwe-gian reindeer on natural summer pasture. Microbial Ecology, 2009, 57(2): 335-348 (doi: 10.1007/s00248-008-9414-7).
- Zeng H., Guo C., Sun D., Seddik H.E., Mao S. The ruminal microbiome and metabolome alterations associated with diet-induced milk fat depression in dairy cows. Metabolites, 2019, 9(7): 154 (doi: 10.3390/metabo9070154).
- Callahan B., McMurdie P., Holmes S. Exact sequence variants should replace operational taxonomic units in marker-gene data analysis. The ISME Journal, 2017, 11(12): 2639-2643 (doi: 10.1038/ismej.2017.119).
- Zeka F., Vanderheyden K., De Smet E., Cuvelier C.A., Mestdagh P., Vandesompele J. Straightforward and sensitive RT-qPCR based gene expression analysis of FFPE samples. Scientific Reports, 2016, 6: 21418 (doi: 10.1038/srep21418).
- Livak K.J., Schmittgen T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCT method. Methods, 2001, 25(4): 402-408 (doi: 10.1006/meth.2001.1262).
- Kim E.J., Huws S.A., Lee M.R.F., Scollan N.D. Dietary transformation of lipid in the rumen microbial ecosystem. Asian-Australasian Journal of Animal Sciences, 2009, 22(9): 1341-1350 (doi: 10.5713/ajas.2009.r.11).
- Калашников А.П., Фисинин В.И., Щеглов В.В., Первое Н.Г., Клейменов Н.И., Стрекозов Н.И., Калышцкий Б.Д., Егоров И.А., Махаев Е.А., Двалишвили В.Г, Калашников В.В., Владимиров В.Л., Груздев Н.В., Мысик А. Т., Балакирев Н.А., Фицев А.И., Кирилов М.П., Крохина В. А., Наумепко П. А., Воробьева СВ., Трухачев В.И. Злыднев Н.Э., Свиридова Т.М., Левахин В.И., Галиев Б.Х., Арилов А.Н., Бугдаев И.Э. Нормы и рационы кормления сельскохозяйственных животных. М., 2003.
- Макарцев Н.Г. Кормление сельскохозяйственных животных. Калуга, 2012.
- Некрасов Р.В., Головин А.В., Махаев Е.А., Аникин А.С., Первов Н.Г., Стрекозов Н.И., Мысик А.Т., Дуборезов В.М., Чабаев М.Г., Фомичев Ю.П., Гусев И.В. Нормы потребностей молочного скота и свиней в питательных веществах. М., 2018.
- Reese A.T., Dunn R.R. Drivers of microbiome biodiversity: a review of general rules, feces, and ignorance. mBio, 2018, 9(4): e01294-18 (doi: 10.1128/mBio.01294-18).
- Bach A., López-García A., González-Recio O., Elcoso G., Fàbregas F., Chaucheyras-Durand F., Castex M. Changes in the rumen and colon microbiota and effects of live yeast dietary supplementation during the transition from the dry period to lactation of dairy cows. Journal of Dairy Science, 2019, 102(7): 6180-6198 (doi: 10.3168/jds.2018-16105).
- Robinson C.J., Young V.B. Antibiotic administration alters the community structure of the gastrointestinal microbiota. Gut Microbes, 2010, 1(4): 279-284 (doi: 10.4161/gmic.1.4.12614).
- Duvallet C., Gibbons S.M., Gurry T., Irizarry R.A., Alm E.J. Meta-analysis of gut microbiome studies identifies disease-specific and shared responses. Nature Communications, 2017, 8(1): 1784 (doi: 10.1038/s41467-017-01973-8).
- Fernando S.C., Purvis H.T., Najar F.Z., Sukharnikov L.O., Krehbiel C.R., Nagaraja T.G., Roe B.A., DeSilva U. Rumen microbial population dynamics during adaptation to a high-grain diet. Applied and Environmental Microbiology, 2010, 76(22): 7482-7490 (doi: 10.1128/AEM.00388-10).
- Qiu Q., Gao C., ur Rahman M.A., Cao B., Su H. Digestive ability, physiological characteristics, and rumen bacterial community of holstein finishing steers in response to three nutrient density diets as fattening phases advanced. Microorganisms, 2020, 8(3): 335 (doi: 10.3390/microorganisms8030335).
- Lewin G.R., Carlos C., Chevrette M.G., Horn H.A., McDonald B.R., Stankey R.J., Fox B.G., Currie C.R. Evolution and ecology of Actinobacteria and their bioenergy applications. Annual Review of Microbiology, 2016, 70(1): 235-254 (doi: 10.1146/annurev-micro-102215-095748).
- Berlemont R., Martiny A.C. Phylogenetic distribution of potential cellulases in bacteria. Applied and Environmental Microbiology, 2013, 79(5): 1545-1554 (doi: 10.1128/AEM.03305-12).
- Wertz J.T., Kim E., Breznak J.A., Schmidt T.M., Rodrigues J.L. Genomic and physiological characterization of the Verrucomicrobia isolate Geminisphaera colitermitum gen. nov., sp. nov., reveals microaerophily and nitrogen fixation genes. Applied and Environmental Microbiology, 2012, 78(5): 1544-1555 (doi: 10.1128/AEM.06466-11).
- Dunfield P.F., Yuryev A., Senin P., Smirnova A.V., Stott M.B., Hou S., Ly B., Saw J.H., Zhou Z., Ren Y., Wang J., Mountain B.W., Crowe M.A., Weatherby T.M., Bodelier P.L.E., Liesack W., Feng L., Wang L., Alam M. Methane oxidation by an extremely acidophilic bacterium of the phylum Verrucomicrobia. Nature, 2007, 450(7171): 879-882 (doi: 10.1038/nature06411).
- Tian R., Ning D., He Z., Zhang P., Spencer S.J., Gao S., Shi W., Wu L., Zhang Y., Yang Y., Adams B.G., Rocha A.M., Detienne B.L., Lowe K.A., Joyner D.C., Klingeman D.M., Arkin A.P., Fields M.W., Hazen T.C., Stahl D.A., Alm E.J., Zhou J. Small and mighty: adaptation of superphylum Patescibacteria to groundwater environment drives their genome simplicity. Microbiome, 2020, 8(1): 51 (doi: 10.1186/s40168-020-00825-w).
- Flint H.J., Duncan S.H., Scott K.P., Louis P. Links between diet, gut microbiota composition and gut metabolism. Proceedings of the Nutrition Society, 2015, 74(1): 13-22 (doi: 10.1017/S0029665114001463).
- Franke T., Deppenmeier U. Physiology and central carbon metabolism of the gut bacterium Prevotella copri. Molecular Microbiology, 2018, 109(4): 528-540 (doi: 10.1111/mmi.14058).
- Meissner S., Hagen F., Deiner C., Günzel D., Greco G., Shen Z., Aschenbach J.R. Key role of short-chain fatty acids in epithelial barrier failure during ruminal acidosis. Journal of Dairy Science, 2017, 100(8): 6662-6675 (doi: 10.3168/jds.2016-12262).
- Pascual J., Hahnke S., Abendroth C., Langer T., Ramm P., Klocke M., Luschnig O., Porcar M. Draft genome sequence of a new Oscillospiraceae bacterium isolated from anaerobic digestion of biomass. Microbiology Resource Announcements, 2020, 9(27): e00507-20 (doi: 10.1128/MRA.00507-20).
- Lagkouvardos I., Lesker T.R., Hitch T.C.A., Gálvez E.J.C., Smit N., Neuhaus K., Wang J., Baines J.F., Abt B., Stecher B., Overmann J., Strowig T., Clavel T. Sequence and cultivation study of Muribaculaceae reveals novel species, host preference, and functional potential of this yet undescribed family. Microbiome, 2019, 7(1): 28 (doi: 10.1186/s40168-019-0637-2).
- Ormerod K.L., Wood D.L., Lachner N., Gellatly S.L., Daly J.N., Parsons J.D., Dal’Molin C.G.O., Palfreyman R.W., Nielsen L.K., Cooper M.A., Morrison M., Hansbro P.M., Hugenholtz P. Genomic characterization of the uncultured Bacteroidales family S24-7 inhabiting the guts of homeothermic animals. Microbiome, 2016, 4(1): 36 (doi: 10.1186/s40168-016-0181-2).
- Petzel J.P., McElwain M.C., DeSantis D.J., Manolukas M.V., Williams M.V., Hartman P.A., Allison M.J., Pollack J.D. Enzymic activities of carbohydrate, purine, and pyrimidine metabolism in the Anaeroplasmataceae (class Mollicutes). Archives of Microbiology, 1989, 152(4): 309-316 (doi: 10.1007/BF00425166).
- Zhang X., Zhang S., Shi Y., Shen F., Wang H. A new high phenyl lactic acid-yielding Lactobacillus plantarum IMAU10124 and a comparative analysis of lactate dehydrogenase gene. FEMS Microbiology Letters, 2014, 356(1): 89-96 (doi: 10.1111/1574-6968.12483).
- Yeswanth S., Kumar Y.N., Prasad U.V., Swarupa V., Koteswara rao V., Sarma P.V.G.K. Cloning and characterization of l-lactate dehydrogenase gene of Staphylococcus aureus. Anaerobe, 2013, 24: 43-48 (doi: 10.1016/j.anaerobe.2013.09.003).
- Carlier J.P., K'ouas G., Han X.Y. Moryella indoligenes gen. nov., sp. nov., an anaerobic bacterium isolated from clinical specimens. International Journal of Systematic and Evolutionary Microbiology, 2007, 57(4): 725-729 (doi: 10.1099/ijs.0.64705-0).
- Sizova M.V., Muller P.A., Stancyk D., Panikov N.S., Mandalakis M., Hazen A., Hohmann T., Doerfert S.N., Fowle W., Earl A.M., Nelson K.E., Epstein S.S. Oribacterium parvum sp. nov. and Oribacterium asaccharolyticum sp. nov., obligately anaerobic bacteria from the human oral cavity, and emended description of the genus Oribacterium. International Journal of Systematic and Evolutionary Microbiology, 2014, 64(8): 2642-2649 (doi: 10.1099/ijs.0.060988-0).
- Downes J., Munson M.A., Radford D.R., Spratt D.A., Wade W.G. Shuttleworthia satelles gen. nov., sp. nov., isolated from the human oral cavity. International Journal of Systematic and Evolutionary Microbiology, 2002, 52(5): 1469-1475 (doi: 10.1099/00207713-52-5-1469).
- Dewanckele L., Jeyanathan J., Vlaeminck B., Fievez V. Identifying and exploring biohydrogenating rumen bacteria with emphasis on pathways including trans-10 intermediates. BMC Microbiology, 2020, 20(1): 198 (doi: 10.1186/s12866-020-01876-7).
- Ferlay A., Bernard L., Meynadier A., Malpuech-Brugère C., Ferlay A., Bernard L., Meynadier A., Malpuech-Brugère C. Production of trans and conjugated fatty acids in dairy ruminants and their putative effects on human health: a review. Biochimie, 2017, 141: 107-120 (doi: 10.1016/j.biochi.2017.08.006).
- Bauman D.E., Griinari J.M. Nutritional regulation of milk fat synthesis. Annual Review of Nutrition, 2003, 23(1): 203-227 (doi: 10.1146/annurev.nutr.23.011702.073408).
- Neubauer V., Petri R.M., Humer E., Kröger I., Reisinger N., Baumgartner W., Wagner M., Zebeli Q. Starch-rich diet induced rumen acidosis and hindgut dysbiosis in dairy cows of different lactations. Animals, 2020, 10(10): 1727 (doi: 10.3390/ani10101727).
- Tadepalli S., Narayanan S.K., Stewart G.C., Chengappa M.M., Nagaraja T.G. Fusobacterium necrophorum: a ruminal bacterium that invades liver to cause abscesses in cattle. Anaerobe, 2009, 15(1-2): 36-43 (doi: 10.1016/j.anaerobe.2008.05.005).
- Krebs H.A., Eggleston L.V. Metabolism of acetoacetate in animal tissues. Biochemical Journal, 1945, 39(5): 408-419.
- Галочкина В.П. Взаимосвязь ферментов цикла Кребса и метаболизма пирувата с продуктивностью выращиваемых на мясо бычков и птицы. Канд. дис. Боровск, 2007.
- Филиппович Ю.Б. Основы биохимии. М., 1999.
- Begley T.P., Kinsland C., Strauss E. The biosynthesis of coenzyme A in bacteria. Vitamins and Hormones, 2001, 61: 157-171 (doi: 10.1016/S0083-6729(01)61005-7).
- Leonardi R., Zhang Y.M., Rock C.O., Jackowski S. Coenzyme A: back in action. Progress in Lipid Research, 2005, 44(2-3): 125-153 (doi: 10.1016/j.plipres.2005.04.001).
- Clark J.H., Davis C.L. Some aspects of feeding high producing dairy cows. Journal of Dairy Science, 1980, 63(6): 873-885 (doi: 10.3168/jds.S0022-0302(80)83021-9).
- Abdo K.M., King K.W., Engel R.W. Protein quality of rumen microorganisms. Journal of Animal Science, 1964, 23(3): 734-736 (doi: 10.2527/jas1964.233734x).
- Hilton W.M. Nutrient requirements of beef cattle (7th edn.). National Academy Press, Washington, DC, 1996 (doi: 10.17226/9791).
- Swick R.W., Wood H.G. The role of transcarboxylation in propionic acid fermentation. Proceedings of the National Academy of Sciences USA, 46(1): 28-41 (doi: 10.1073/pnas.46.1.28).
- Wang X., Li X., Zhao C., Hu P., Chen H., Liu Z., Liu G., Wang Z. Correlation between composition of the bacterial community and concentration of volatile fatty acids in the rumen during the transition period and ketosis in dairy cows. Applied and Environmental Microbiology, 2012, 78(7): 2386‐2392 (doi: 10.1128/AEM.07545-11).
- Smith S.E., Loosli J.K. Cobalt and vitamin 12 in ruminant nutrition: a review. Journal of Dairy Science, 1957, 40(10): 1215-1227 (doi: 10.3168/JDS.S0022-0302(57)94618-0).
- Lawrence J.G., Roth J.R. Evolution of coenzyme B(12) synthesis among enteric bacteria: evidence for loss and reacquisition of a multigene complex. Genetics, 1996, 142(1): 11-24.
- Gebreyesus G., Difford G.F., Buitenhuis B., Lassen J., Noel S.J., Højberg O., Plichta D.R., Zhu Z., Poulsen N.A., Sundekilde U.K., Løvendahl P., Sahana G. Predictive ability of host genetics and rumen microbiome for subclinical ketosis. Journal of Dairy Science, 2020, 103(5): 4557-4569 (doi: 10.3168/jds.2019-17824).
- Lima J., Auffret M.D., Stewart R.D., Dewhurst R.J., Duthie C.A., Snelling T.J., Walker A.W., Freeman T.C., Watson M., Roehe R. Identification of rumen microbial genes involved in pathways linked to appetite, growth, and feed conversion efficiency in cattle. Frontiers in Genetics, 2019, 10: 701 (doi: 10.3389/fgene.2019.00701).
- Ogunade I., Pech-Cervantes A., Schweickart H. Metatranscriptomic analysis of sub-acute ruminal acidosis in beef cattle. Animals, 2019, 9(5): 232 (doi: 10.3390/ani9050232).
- Nocek J.E. Bovine acidosis: implications on laminitis. Journal of Dairy Science, 1997, 80(5): 1005-1028 (doi: 10.3168/jds.S0022-0302(97)76026-0).
- Bustin S.A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems. Journal of Molecular Endocrinology, 2002, 29(1): 23-39 (doi: 10.1677/jme.0.0290023).
- Huang Y., You C., Liu Z. Cloning of D-lactate dehydrogenase genes of Lactobacillus delbrueckii subsp. bulgaricus and their roles in D-lactic acid production. Biotechnology, 2017, 7(3): 194 (doi: 10.1007/s13205-017-0822-6).
- Hernández J., Benedito J.L., Abuelo A., Castillo C. Ruminal acidosis in feedlot: from aetiology to prevention. The Scientific World Journal, 2014: 702572 (doi: 10.1155/2014/702572).


